牦牛、犏牛、藏黄牛和普通牛HIOMT基因CDS序列的比较
2014-01-07泽让东科文勇立
张 云,泽让东科,艾 鹥,王 永,文勇立*
(1.西南民族大学 生命科学与技术学院,四川 成都610041;2.西南民族大学 青藏高原研究院,四川 成都610041)
生物节律是生命活动的基本特征,具有内源性、遗传性、环境适应性等性质。褪黑激素(Melatonin,MLT)主要由松果体组织分泌,是调控昼夜节律、生殖节律等多种生物节律,且具有复杂生理功能的一种吲哚类激素[1]。外界光节律信息被编码到MLT信号调控系统,在MLT靶组织中获得解码,从而调控如季节性生殖等节律性生命活动[2]。MLT的合成分别由4种酶按先后步骤进行的,其中,后两步酶分别为五羟色胺-氮-乙酰转移酶(arylalkylamine-N-acetyltransferase,AANAT)和羟基吲哚-氧-甲基转移 酶 (hydroxyindole-O-methyltransferase,HIOMT)是MLT合成的两种主要酶。研究表明,这两种酶的活性及基因转录、表达的节律性共同决定了MLT分泌的波动模式,从而完成对复杂生物节律的调控[3-10]。据文献,HIOMT基因的功能和结构比AANAT更为复杂,同时报道相对较少,对小鼠和鸡松果体的研究表明,松果体是机体唯一有HIOMT酶存在的器官,且其活性在夜间比白天高10%~20%,与褪黑激素合成波动性呈正相关[11-14]。此外,过去的研究绝大多数以小鼠等小型动物为对象,而鲜见对大型哺乳类动物的相关研究。自然条件下,牦牛的生殖对策表现出很强的环境适应性,除繁殖活动具有明显的季节性(7~9月发情,次年4~6月产犊)外,成年母牦牛还呈现1年或2年的产犊节律性。据调查,在青藏高原许多地区约30%的成年雌体可每年产1胎,而70%左右的成年雌体表现为隔1年产1胎。牦牛血浆MLT分泌同其他季节性繁殖动物一样具有近日节律[15],提示开展牦牛MLT合成酶基因相关研究是深入了解MLT生殖调控机制的重要线索。本研究采用RT-PCR方法克隆并分析了牦牛、藏黄牛和犏牛褪黑激素合成酶HIOMT基因CDS序列,研究结果将为进一步研究牦牛乃至哺乳动物繁殖周期等生物节律调控机制积累资料。
1 材料与方法
1.1 试验动物与样品采集
于四川省阿坝藏族羌族自治州红原县国中食品有限公司,随机选取健康、发育良好的成年雄性牦牛、藏黄牛和犏牛各1头,宰杀后立即取出松果体,液氮保存。
1.2 引物设计
1.2.1 CDS序列引物 根据普通牛HIOMT基因mRNA 序 列 (Genbank Accession Number M81862)设 计 同 源 性 引 物:HIOMT-F1(5'-TAGACTGGTGAAGCCCAAAGC-3')和 HIOMTR1(5'-GACCATCCGTGG GCATAAAC-3'),由上海Invitrogen生物技术有限公司合成。
1.2.2 DNA序列引物 为了获得基因组中HIOMT基因的DNA片断(用于验证下述牦牛HIOMT基因CDS缺失序列是否存在于基因组),通过http://genome.ucsc.edu/数据库检索得知普通牛HIOMT基因位于chr Un_JH125805,在其中35541-34838位点处设计同源性引物HIOMT-F2(5'-ATTGAGGCAGGCTGACAAGG-3')和 HIOMT-R2(5'-CCCACGACGAAGATGGAAGA-3'),由上海Invitrogen生物技术有限公司合成。
1.3 RNA、DNA 提取与 RT-PCR TRIzol(Invitrogen)提取总RNA,采用Ta KaRa反转录试剂盒合成cDNA;DNA提取采用经典酚/氯仿抽提法。PCR总体系25μL,其中2×Master Mix Taq 12.5 μL,cDNA 2μL,上下游引物各1μL,dd H2O 8.5 μL。cDNA扩增反应条件为94℃预热3 min;94℃变性45 s,56.8℃退火1 min,72℃延伸1.5 min,持续30个循环;72℃延伸5 min后4℃保存。DNA扩增反应条件为94℃预热3 min;94℃变性45 s,52.4℃退火1 min,72℃延伸1 min,持续30个循环;72℃延伸5 min后4℃保存。PCR产物经1%琼脂糖凝胶电泳检测后送大连宝生物工程有限公司(TaKaRa)测序。
1.4 外显子分析
将克隆所得牦牛、藏黄牛、犏牛和下载的普通牛HIOMT 基因 CDS序列于 http://genome.ucsc.edu/数据库中进行Blast比对,获得该基因外显子相关信息。
中国教师:全民阅读是一项系统的大工程,您主持的新教育实验一直把阅读作为最根本、最基础的工程来推进,并取得了显著的成效,积累了丰富的经验。在推广阅读方面,新教育实验有哪些好的经验可以和我们分享?
1.5 生物信息学分析
测序结果由Clustal X软件进行排序[16],并手工调校。采用MEGA 4.0分析核苷酸和氨基酸变异位点[17],ExPASy的 ProtParam 工具分析 HIOMT编码氨基酸基本性质 (http://web.expasy.org/protparam/),Compute p I/Mw分析其理论等电点和相对分子量(http://web.expasy.org/compute_pi/),ProtScale计算其疏水性图谱(http://web.expasy.org/protscale/)。采用 TMPRED 软件分析跨膜结构 (http://www.ch.embnet.org/software/TMPRED_form.html),通过 HNN 在线预测蛋白二级结构(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_hnn.html),SWISS-MODEL 预 测 蛋 白 三 级 结构[18]。基于Kimura双参数法使用 MEGA 4.0构建其NJ系统进化树。
2 结果与分析
2.1 PCR扩增与测序
牦牛、藏黄牛和犏牛的HIOMT基因CDS序列PCR扩增条带明显,均位于1 000-1 500 bp之间,与预测1 286 bp基本吻合。藏黄牛条带略大于牦牛,犏牛有2条带,其中1条长带与藏黄牛相似,另1条短带与牦牛相似(图1)。将牦牛和藏黄牛PCR产物纯化,同时将犏牛的长、短带分别切胶回收纯化后测序。对牦牛、藏黄牛HIOMT基因DNA序列的部分序列扩增,所得条带明显,位于700 bp左右,与预测704 bp基本一致(图2),产物经回收后测序。
2.2 序列比对
双向测序分析表明,牦牛HIOMT基因和犏牛的短带CDS序列均为915 bp,长带均为1 038 bp。与普通牛比较,藏黄牛和犏牛长带CDS序列长度与其一致,而牦牛和犏牛短带CDS序列连续缺失123 bp,并为3的倍数。藏黄牛与普通牛共存在10个变异位点,其中2处位于密码子第1位(643:G→T,862:A→G),另2处位于密码子第2位(320:T→A,965:G→C);犏牛长带与普通牛比对共存在17个变异位点,其中2处位于密码子第1位(28:A→C,862:A→G),另5处位于密码子第2位(155:A→G,287:G→A,716:A→C,788:C→G,965:G→C);犏牛短带与普通牛比较,除连续缺失123 bp外,还存在13个变异位点,其中2处位于密码子第1位(28:A→C,862:A→G),另4处位于密码子第2位(155:A→G,287:G→A,503:G→A,965:G→C);牦牛与普通牛比较,除连续缺失123bp外,还存在11个变异位点,其中2处位于密码子第1位(28:A→C,862:A→G),另2处位于密码子第2位(155:A→G,965:G→C)(见表1)。

图1 牦牛、藏黄牛和犏牛HIOMT基因PCR扩增结果M.DNA marker 100bp ladder;1.藏黄牛;2.牦牛;3.犏牛Fig.1 PCR amplification of HIOMT in yak,Tibetan-cattle and cattle-yakM.DNA marker 100bp ladder;1.Tibetan cattle;2.Yak;3.Cattle-yak
2.3 HIOMT的外显子分析
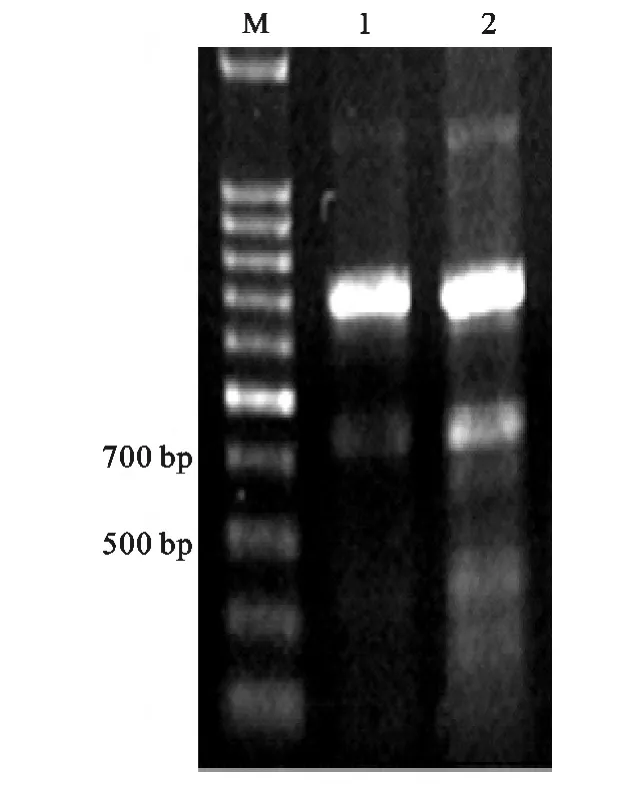
图2 基因组HIOMT基因DNA片断扩增结果M.DNA Marker 100 bp ladder;1.牦牛;2.藏黄牛Fig.2 PCR amplification segment of the partly HIOMT gene M.DNA Marker 100bp ladder;1.Yak;2.Tibetan cattle
藏黄牛、犏牛长序列和普通牛共有7处与chr Un_JH125805序列一致,因此推测该基因共有7个外显子,第1-7外显子分别依次位于1~69、70~244、371~443、444~562、563~703、704~826和827~1038位点处;而牦牛和犏牛该基因的短序列共有6处与chr Un_JH125805序列一致,因此推测存在6个外显子,分别依次位于1~69、70~244、371~443、444~562、563~703和704~915位点处,且最后1个外显子(704~915)与藏黄牛、犏牛长序列和普通牛的第7外显子位置(827-1038)相符,而所缺失的123 bp与藏黄牛及犏牛的长序列和普通牛序列的第6外显子(704-826)相符,因此推测,牦牛、犏牛的短序列缺失了第6外显子。
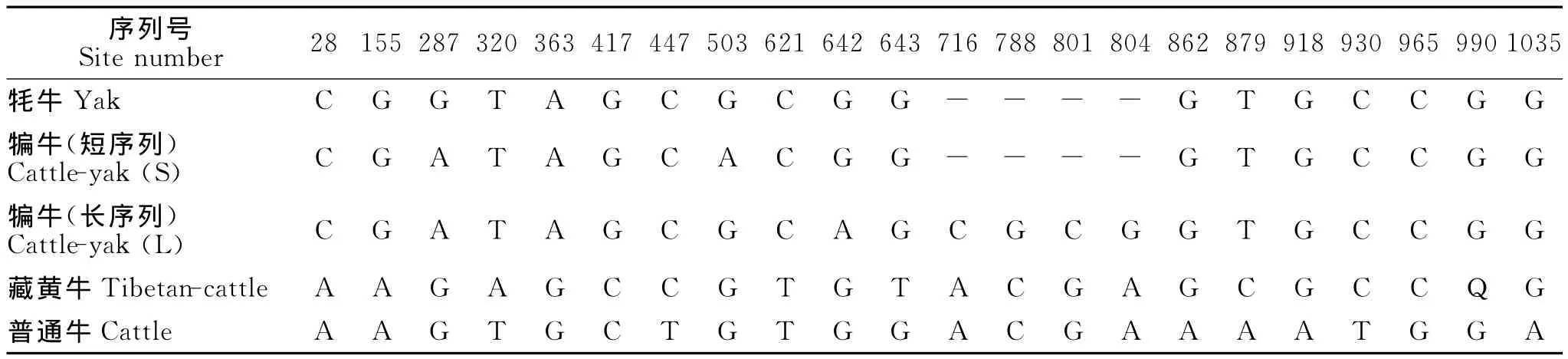
表1 牦牛、犏牛、藏黄牛和普通牛HIOMT基因CDS序列变异位点分析Table 1 Variation analysis of CDS of HIOMT between yak,cattle-yak,Tibetan-cattle and cattle

表2 牦牛、犏牛、藏黄牛和普通牛HIOMT基因编码氨基酸变异位点分析Table 2 Amino acid sequences substitute between yak,cattle-yak,Tibetan-cattle and cattle

图3 普通牛基因组chr Un_JH125805序列(35541-34838)与牦牛、藏黄牛比对划线部分为牦牛和犏牛的缺失序列Fig.3 Alignment of cattle genome chr Un_JH125805(35541-34838)with yak and Tibetan-cattleThe sequences with underline indicate the deletion of yak and cattle-yak
2.4 基因组中HIOMT基因对应缺失CDS片断的克隆
为了验证牦牛、犏牛短序列缺失的123 bp是否存在于基因组,将克隆测序的牦牛(699 bp)、藏黄牛(702 bp)DNA 序列与普通牛基因组chr Un_JH125805序列35541-34838区段(704 bp)比对,显示除少数核苷酸缺失和变异外,序列基本一致,表明牦牛、犏牛短序列HIOMT基因CDS序列的缺失并非来源于基因组缺失(图3)。
2.5 HIOMT基因的氨基酸编码、相对分子量、理论等电点和疏水性图谱
HIOMT基因的氨基酸编码、相对分子量、理论等电点pI、GRAVE(Grand average of hydropathicity)值均见表3,可见由突变引起的氨基酸变异造成了这些基本属性的一定变化。结合疏水性示意图(图略)和GRAVE值预测该蛋白均为亲水性蛋白。

表3 HIOMT基因氨基酸编码Table 3 Amino acid composition coding by HIOMT
2.6 HIOMT蛋白跨膜结构和空间结构预测
跨膜区分析发现跨膜方向由内向外和由外向内两组值,均存在2个具有较高可能性的由内向外的跨膜区,除犏牛短序列仅2个由外向内的跨膜区外,其余均存在3个由外向内的跨膜区(图略)。

图4 HIOMT蛋白二级结构预测a.牦牛;b.犏牛(短序列);c.犏牛(长序列);d.藏黄牛;e.普通牛;蓝色表示α螺旋;红色表示β折叠;粉色表示无规则卷曲Fig.4 Secondary structure prediction of HIOMTa.Yak;b.Cattle-yak-S;c.Cattle-yak-L;d.Tibetan cattle;e.Cattle;Blue region stands forα-helix;Red region is forβ-sheet,pink region random coils
牦牛、犏牛短序列HIOMT蛋白的二级结构由304个氨基酸组成,而犏牛长序列、藏黄牛和普通牛由365个氨基酸组成,其结构存在一定的差异(图4)。其中牦牛HIOMT存在15处α螺旋区域(50.33%),11处β折叠区域(12.50%)和20处无规则卷曲区域(37.17%);而普通牛则存在16处α螺旋 富 集 区 域 (53.62%),10 处 β 折 叠 区 域(11.30%)和20处无规则卷曲区域(35.07%),且牦牛氨基酸缺失区域位于α螺旋区域。
由SWISS-MODEL预测的牦牛、犏牛短带、犏牛长带、藏黄牛和普通牛HIOMT蛋白空间结构(图5),表明牦牛和犏牛短带间,犏牛长带、藏黄牛和普通牛间该基因核苷酸序列的变异所引起的编码氨基酸序列的差异,对该蛋白空间结构的影响不大,但是牦牛和犏牛短大面积的碱基缺失使该蛋白的预测空间结构产生了一定的改变,这些改变可能会改变其活性甚至生物学功能。

图5 HIOMT蛋白空间结构预测(红色区域表明结构不一致部分)A.牦牛;B.犏牛(短序列);C.犏牛(长序列);D.藏黄牛;E.普通牛Fig.5 Spatial structure prediction of HIOMT (Red region indicates the different structure)A.Yak;B.Cattle-yak-S;C.Cattle-yak-L;D.Tibetan cattle;E.Cattle
2.7 系统进化树
由牦牛、犏牛、藏黄牛、普通牛、山羊、绵羊、双峰驼、羊驼、白犀牛、小耳大婴猴和普通狨HIOMT基因CDS序列构建的系统进化树见图6。各节点自举值均大于80,且总共分为4大分支。首先犏牛与牦牛聚为一类,再与同属牛亚科的藏黄牛和普通牛聚为一类,最后于同属牛科的羊亚科山羊、绵羊聚为一类;骆驼属的双峰驼和羊驼聚为一类;犀科的白犀牛单独聚为一类;灵长类的小耳大婴猴和普通狨聚为一类。4大分支亲缘关系由近及远分别为牛科、骆驼属、犀科、灵长类。
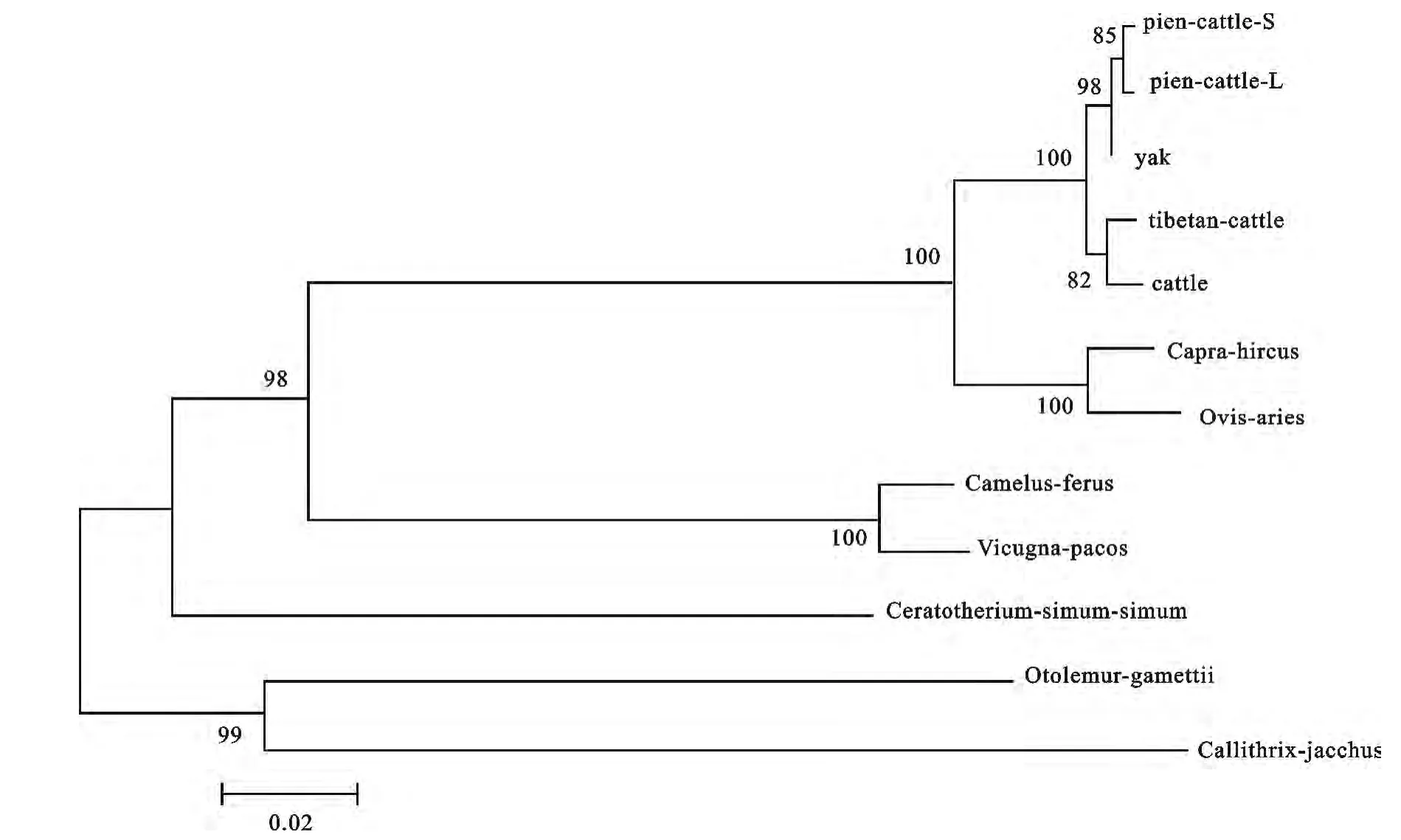
图6 牦牛HIOMT基因与其他哺乳动物构建NJ系统进化树Fig.6 NJ tree between yak and other mammals based on HIOMT
3 讨 论
本研究克隆了牦牛、藏黄牛和犏牛HIOMT基因CDS序列,对比发现藏黄牛HIOMT序列全长1 038 bp,牦牛915 bp,后者连续缺失123 bp,而犏牛兼有藏黄牛1 038 bp和牦牛915 bp两条序列。进一步分析表明,牦牛缺失的123 bp序列并非来源于基因组,此外,基于普通牛HIOMT基因的外显子分析表明,该缺失片段属第7外显子区段,由此判断,牦牛915 bp的序列缺失第6外显子,可能是HIOMT基因的一个可变剪接体。此结果暗示,牦牛极有可能还存在其他可变剪接体。据报道,80%的可变剪接发生在5′UTR区,19%在3′UTR区,仅1%发生在CDS区域[19],而本研究所发现的缺失片断位于CDS区域,因此,属于一种低概率变异现象,有必要在后续研究中,对这些变异体进行不同载体表达和酶活检测,研究其与生物周期调控是否存在相关性,对于阐明生物节律机制具有重要意义。
藏黄牛与普通牛虽为同一物种,但其HIOMT基因核苷酸序列仍然存在10个变异位点,且4处为非同义突变,这将导致编码氨基酸序列的改变,该现象是否与藏黄牛长期生存于高海拔、寒冷、强紫外线照射等恶劣环境有关[20],尚待进一步研究。
本研究发现犏牛HIOMT基因存在1 038 bp和915 bp两条CDS序列,虽然分别与牦牛和藏黄牛(包括普通牛)序列一致,并且,犏牛染色体50%来自公黄牛、50%来自母牦牛,但还是不能确定两条序列的来源。如果长、短变异体现象均存在于黄牛和牦牛,且可能性相同,则犏牛的两条序列中的任何1条都可能来自父本或来自母本;如果短变异体现象仅存在于牦牛,或存在于牦牛的可能性高于黄牛,则判断犏牛的长序列来自父本黄牛,短序列来自母本牦牛的正确性就较大,对此尚需进一步研究。
本研究结果显示,无论是牦牛、藏黄牛、普通牛还是犏牛,其HIOMT基因的核苷酸序列突变位点均较少,且突变主要集中在密码子第3位,基本为同义突变,表明该基因较为保守。但是,也存在较少突变位点密码子位于第1、2位的现象,并发生了编码氨基酸序列的改变,从而可能改变蛋白质构象,影响生物学功能。因此,有必要对这些非同义突变与牦牛生殖节律是否存在相关性开展研究,以便建立其分子标记,有助于牦牛繁殖性状的辅助选择[21]。
牦牛与普通牛氨基酸序列比较,除缺失部分外,还共存在4个变异位点,分别发生在第10、52、288和322位,这些变异导致预测的蛋白二级结构产生一定改变,对此需要进一步验证,并研究这种改变对蛋白活性的影响[22],将有助于阐明牦牛繁殖节律的分子调控机制。
本试验所获得的牦牛HIOMT基因编码氨基酸序列、相对分子量、理论等电点、疏水性、信号肽、跨膜结构和糖基化位点等结果,将有助于对牦牛HIOMT蛋白开展深入研究,为阐释该蛋白对牦牛繁殖及环境适应性影响的生理机制提供基础数据。
由牦牛和其他哺乳动物HIOMT基因CDS序列构建的NJ进化树能够较准确地反映物种之间的亲缘关系,表明将HIOMT基因用于哺乳动物科、属和种间的分子进化研究,具有一定意义。
[1] Voisin P,Guerlotte J,Bernard M,et al.Regulation of hydroxyindole-O-methyltransferase gene expression in the pineal gland and retina[J].Acta Neurobiol Exp(Wars),1994,54:41-46.
[2] Gall C,Lewy A,Schomerus C,et al.Transcription factor dynamics and neuroendocrine signalling in the mouse pineal gland:a comparative analysis of melatonin-deficient C57BL mice and melatonin-proficient C3H mice[J].Eur J Neurosci,2000,12:964-970.
[3] Kasahara T,Abe K,Mekada K,et al.Genetic variation of melatonin productivity in laboratory mice under domestication[J].Proceedings of the National Academy of Sciences,2010,107(14):6 412-6 417.
[4] Konturek S J,Konturek P C,Brzozowski T,et al.Role of melatonin in upper gastrointestinal tract[J].J Physiol Pharmacol,2007,58(6):23-52.
[5] Nisembaum L G,Tinoco A B,Moure A L,et al.The arylalkylamine-N-acetyltransferase(AANAT)acetylates dopamine in the digestive tract of goldfish:a role in intestinal motility[J].Neurochem Int,2013,62(6):873-880.
[6] Sakaguchi K,Itoh M T,Takahashi N,et al.The rat oocyte synthesises melatonin[J].Reprod Fertil Dev,2013,25(4):674-682.
[7] Fukuda T,Akiyama N,Ikegami M,et al.Expression of hydroxyindole-O-methyltransferase enzyme in the human central nervous system and in pineal parenchymal cell tumors[J].J Neuropathol Exp Neurol,2010,69(5):498-510.
[8] Velarde E,Cerdá-Reverter J M,Alonso-Gómez A L,et al.Melatonin-synthesizing enzymes in pineal,retina,liver,and gut of the goldfish(Carassius):mRNA expression pattern and regulation of daily rhythms by lighting conditions[J].Chronobiol Int,2010,27(6):1 178-1 201.
[9] Park S,Byeon Y,Back K.Functional analyses of three ASMT gene family members in rice plants[J].J Pineal Res,2013,55(4):409-415.
[10] Vuilleumier R,Boeuf G,Fuentes M,et al.Cloning and early expression pattern of two melatonin biosynthesis enzymes in the turbot (Scophthalmus maximus)[J].Eur J Neurosci,2007,25(10):3 047-3 057.
[11] Axelrod J,Wurtman R.Melatonin synthesis in the hen pineal gland and its control by light[J].Nature,1964,201:1 134.
[12] Axelrod J,Wurtman R,Snyder S H.Control of hydroxyin-dole-0-methyltransferase activity in the rat pineal gland by environmental lighting[J].J Biol Chem,1965,240:949-954.
[13] Voisin P,Guerlotte J,Bernard M,et al.Regulation of gene expression in the pineal gland and retina[J].Acta neurobiol exp,1994,54:41-46.
[14] Klein D C.Arylalkylamine N-acetyltransferase:“the Timezyme”[J].Journal of biological chemistry,2007,282(7):4 233-4 237.
[15] Sarkar M,Prakash B S.Circadian variations in plasma concentrations of melatonin and prolactin during breeding and non-breeding seasons in yak[J].Animal Reproduction Science,2005,90:149-162.
[16] Thompson J D,Higgins D G,Gibson T J.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic acids research,1994,22:4 673-4 680.
[17] Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular biology and evolution,2007,24(8):1 596-1 599.
[18] Arnold K,Bordoli L,Kopp J,et al.The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J].Bioinformatics,2006,22(2):195-201.
[19] Mironov A A,Fickett J W,Gelfand M S.Frequent alternative splicing of human genes[J].Genome research,1999,9(12):1 288-1 293.
[20] Qiu Q,Zhang G,Ma T,et al.The yak genome and adaptation to life at high altitude[J].Nature genetics,2012,946-951.
[21] El-Raey M,Geshi M,Somfai T,et al.Evidence of melatonin synthesis in the cumulus oocyte complexes and its role in enhancing oocyte maturation in vitro in cattle[J].Molecular reproduction and development,2011,78(4):250-262.
[22] Kripke D F,Nievergelt C M,Tranah G J,et al.Polymorphisms in melatonin synthesis pathways:possible influences on depression[J].J Circadian Rhythms,2011,9(8):1-7.
