基于ITS2和16S rRNA的西施舌群体遗传差异分析
2013-12-16孟学平赵娜娜陈建安董志国程汉良阎斌伦
孟学平,申 欣,赵娜娜,2,田 美,曾 云,陈建安,董志国,程汉良,阎斌伦
(1.淮海工学院海洋学院,江苏省海洋生物技术重点实验室,连云港 222005;2.南京农业大学资源与环境科学学院,江苏省海洋生物重点实验室,南京 210095)
ITS2(The internal transcribed spacer 2)是真核生物5.8S和28S核糖体RNA(rRNA)基因间的DNA序列,此区转录产物可为rRNA前体正确有效加工提供结构元件和二级结构。通常ITS2二级结构5'端的2个结构域非常保守,而3'端的结构域变异较大。由于ITS2上述结构特点,常被用于鉴别相关的种或推断种内和科间甚至更高分类水平的系统关系[1-3]。线粒体核糖体16S rRNA也适合解决双壳类近缘种或类群的系统关系[4-6]。来自细胞核的ITS2和线粒体DNA的16S一级结构和二级结构相结合也被用来研究双壳类同科不同种类的遗传关系[1]或同属不同种的系统发生分析[7]。西施舌(Coelomactra antiquata)是双壳纲蛤蜊科(Mactridae)的一种海产名贵经济贝类,其肉质细腻,营养丰富[8],具有极高经济价值[9]。我国专家对西施舌群体形态[10-12]、生化[13-14]、分子遗传差异及系统发生关系进行了卓有成效的研究工作,但目前西施舌群体遗传背景尚不清晰,研究结果存在分歧:多数资料认为福建(长乐、漳州)西施舌发生了明显遗传分化,可能是一个亚种或隐种[15-20];少数资料认为长乐西施舌与其它群体未达到种间差异的水平[14];此外,一些研究认为广西北海西施舌与江苏、山东、辽宁西施舌群体亲缘关系近,与福建群体亲缘关系远[15,17-18],另一些研究结果认为广西西施舌与福建西施舌遗传关系近[12];要澄清上述分歧、确定福建西施舌是否是一个新种,需要更多的形态、生物和分子生物学资料佐证。本研究拟从核基因组DNA和线粒体基因组DNA两个方面研究我国不同地理群体西施舌遗传差异,从ITS2和16S一、二级结构2个层次分析群体间差异,为我国西施舌群体遗传关系和福建西施舌分类地位的确定提供更多分子生物学证据。对西施舌的增养殖学和野生资源保护和科学利用具有重要意义。
1 材料和方法
1.1 样本采集
本研究西施舌样本分别来自辽宁大连(DL)(样本数为4)、山东胶南(JN、JD)(2)、江苏连云港(LYG)(32)、启东(QD、XM)(34),福建的长乐(CL)(35),广西北海(GX)(16)、越南(YN)(1)等海区,共124个;另一部分序列来源于GenBank中其他作者提交的序列,分别隶属QD(3)、GX(1)、CL(7,包括X1)、山东的日照(RZ)(2)、即墨(JM)(4)和JN(4)、平潭(X2),LYG(X3)等海区,共计23个序列。样本信息见表1、表2,因提交的序列为单倍型或基因型,GenBank中未反映出每个单倍型或基因型的样本数,因此,本实验把每个单倍型或基因型作为1个个体对待。本实验所用实验样本在2005年12月到2011年12月采集。

表1 西施舌ITS2核苷酸序列信息Table 1 The information of ITS2 nucleotide sequences of Coelomactra antiquata
1.2 DNA提取、扩增及序列测定
样本总DNA按2010年Oliverio[21]的方法从闭壳肌或斧足肌肉中提取。纯化的DNA定量测定后作为ITS2和16S序列PCR扩增的模板。ITS2 PCR引物(primer,P)为ITS2-P1(5'GTTTCTTTTCCTCCGCTTACT3')和ITS2-P2(5'CACACTGAACATCGACAGCTT3'),扩增产物450bp左右,包括5.8S和28S rRNA基因部分序列以及 ITS2全序列。16S部分序列 PCR引物为 16S3L(5'TGAGCGTGCTAAGGTAG3')和 16S4H(5'AGCCAACATCGAGGTCGC3')。扩增产物为 350 bp。在 25 μL 反应体系中,ddH2O 17.3 μL,dNTP(10 mmol/L)0.5 μL,10×buffer 2.5 μL,Mg2+(10 mmol/L)2.5 μL,引物(25 μmol/L)各 0.5 μL,模板(20—50 ng)1 μL,Taq 酶(5 U/μL)0.2 μL。反应程序为 94 ℃ 预变性 3 min,然后 35 个循环:94 ℃ 变性 30',52 ℃ 退火30',72℃延伸50',最后72℃延伸5 min。PCR反应在Bio-Rad C1000 PCR仪上进行。PCR产物用ABIPRISM3730测序仪双向测序。
1.3 序列比对分析
DNA序列用DNAStar软件组装,结合测序峰图进行序列人工校对,用 ClustalX[22]软件排序取齐,通过DnaSP 5.0软件统计各群体的单倍型或基因型,利用MEGA 4[23]分析序列特征、计算遗传差异和遗传距离,构建NJ(Neighbor-joining)和 MP(Maximum parsimony)系统进化树;用The mfold Web Server(http://mfold.rna.albany.edu/?q=mfold)进行DNA和RNA二级结构预测。
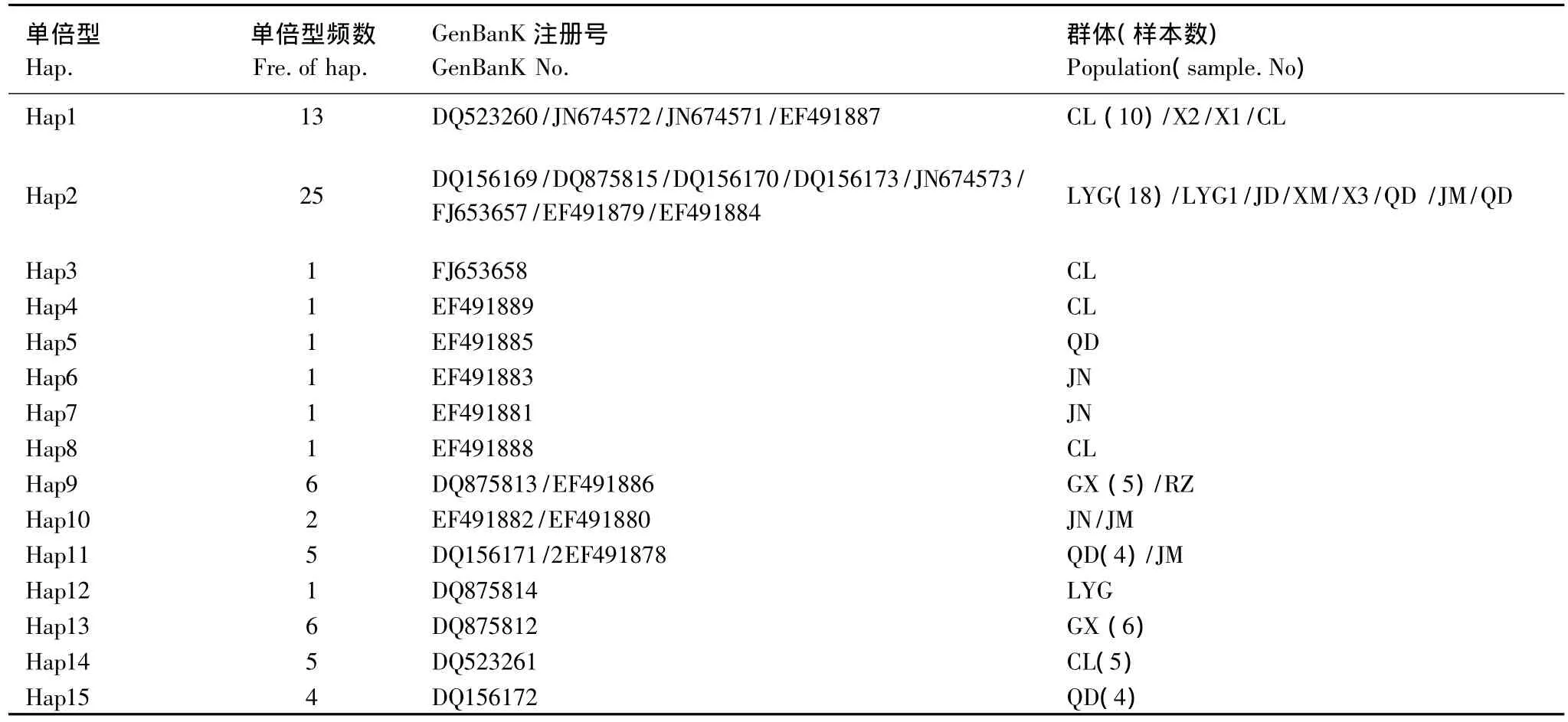
表2 西施舌16S rRNA基因片段信息Table 2 The information of 16S rRNA gene fragments of Coelomactra antiquata
2 结果
2.1 ITS2和16SrRNA基因序列信息
本研究西施舌样本来源于我国南北沿海的9个海区及邻近广西北海的越南海域,共获得147条DNA序列,其中ITS2序列74条,16S序列73条。本研究扩增获得西施舌ITS2序列68条,16S序列56条(表2);从GenBank下载的ITS2序列6条,16S序列17条。
用DnaSPv5分析ITS2获得17种基因型(genotype,Gen)(Gen1-17)(表1),每种基因型的出现频率不同,其中Gen3出现频率最高,为32.4%(24/74),Gen2出现频率次之,为23.0%,频率为5%—7%的基因型有4种,其余11种基因型出现频率分别为1.4%和4.1%。CL(包括YN群体)群体特有基因型有9种(Gen6—14),不与其它群体共享。此外,GX、JM、JN和RZ群体各有1—2个群体特有基因型。Gen1—Gen3为非长乐群体交叉共享基因型,其中Gen3为GX、QD、LYG、DL 4个群体共享;Gen1为GX、QD、LYG 3个群体共享。广西北海群体的ITS2基因型与北方群体基因型有交叉共享现象,与南方的长乐群体无共享。
16S序列共有15个单倍型(haplotype,Hap)(表2),其中 Hap1、3、4、8、14 为 CL 群体特有单倍型,有4 种单倍型为2个以上群体交叉共享。如Hap2为胶南、即墨、连云港、启东和X群体共享;Hap9为北海(GX)和日照群体共享。QD、JM 2个群体共享Hap11,JN、JM 2个群体共享Hap10。胶南、连云港、启东、北海群体除具体有共享单倍型外,尚有1—2个群体特有的单倍型。Hap2和Hap1出现频率较高,分别为34.2%和17.8%,其余单倍型出现频率均低于8%。
2.2 ITS2和16S rRNA序列比对分析
本研究测得的西施舌ITS2全序列长度在389—401 bp之间,有核苷酸的插入/缺失现象。
对ITS2序列两端取齐后获得370—380 bp片段用于序列比对。ITS2序列分析显示,17种基因型有23个变异位点(图1A),占比对位点的5.7%(23/401),其中简约信息位点16个,占4.0%(16/401)。CL群体9种基因型有6个共享变异位点,分别是第226位的碱基T,273—275位的TCT,317位的T和381位的G。而其它群体无共享变异位点。
根据ITS2 17种基因型核苷酸序列的变异,计算了不同群体间遗传距离(考虑文章篇幅太大,不同基因型间的遗传距离表格略),结果发现,CL群体与nCL群体间的遗传距离在0.019—0.042之间,平均为0.029,群体内的遗传距离为0.005—0.022,平均为0.012,CL和 nCL群体间与群体内遗传距离之比为2.42(0.029/0.012),而其余的DL、JM、JN、RZ、LYG、QD和GX群体间有交叉共享基因型,因此,将其列为一组,组内遗传距离为0.003—0.014,平均为0.007。由此可见,西施舌CL群体与nCL群体遗传差异极为明显。
本研究所测定的16S片段多数为306bp,GenBank下载的其他作者提交的序列有的多于360 bp,序列比对前将两端多余序列去除取齐,获得的比对序列长度均为306 bp。对15种16S单倍型序列比对发现,有35个变异位点(图1B),占比对位点的11.40%(35/306),其中简约信息位点24个,占7.84%。CL群体5个单倍型变异位点28个(9.15%),其中有23(7.5%)个变异位点为5种单倍型共享。

图1 西施舌ITS2(基因型)(A)和16S rRNA基因片段(单倍型)(B)变异位点Fig.1 The ITS2(genotype,Gen)(A)and 16S rRNA gene fragments(haplotype,Hap)(B)diversity sites of Coelomactra antiquata
基于16S序列所得各单倍型间的遗传距离如表3,灰色背景里的数字为CL群体与nCL群体间的遗传距离,在7.7%—9.5%之间,平均为8.1%。长乐群体内个体间的遗传距离在0.3%—1.0% 之间,平均为0.7%,群体间遗传距离与群体内遗传距离之比为11.6。长乐以外的JM、JN、LYG、QD和GX 5个群体9种单倍型间的遗传距离在0.3%—1.7%之间,平均为0.9%,远远小于长乐群体与非长乐群体之间的遗传距离。

表3 西施舌16S rRNA基因片段的遗传距离Table 3 The genetic distances based on 16S rRNA gene fragments of Coelomactra antiquata
2.3 基于ITS2和16S核苷酸序列的系统发育分析
基于ITS2和16S核苷酸序列,以与西施舌共属一个科的中国蛤蜊(Mactra chinensis)和四角蛤蜊(Mactra veneriformis)作为外群,用邻接法(NJ)和最大吝啬法(MP)分析了西施舌9个群体亲缘关系(图2),两套DNA资料构建的系统进化树均显示CL群体(包括YN群体)均聚为支持率很高的单系支,在ITS2的NJ和MP树中支持率分别为96和95;在16S的NJ(图2,B)和MP(图略)树中,支持率分别为100和99,而其余的8个群体无一个群体聚为单系支,只是交叉聚为支持率很高的一支[16S:支持率为98/96(NJ/MP)]。因ITS2和16S资料之间不是对应关系,因此,未将两套资料排列在一起构建进化树,尽管如此,但分别聚类结果也高度一致。

图2 基于ITS2基因型(A)和16S rRNA基因单倍型(B)的西施舌不同群体N-J树Fig.2 The N-J tree based on ITS2(genotypic,Gen)(A)and 16S rRNA gene fragments(haplotype,Hap)(B)of C.antiquata different stocks
2.4 ITS2和16S二级结构分析
根据ITS2和16S的序列进行二级结构预测。对基因型Gen3(DL、LYG、QD、GX群体共享)、Gen6(CL群体的一种基因型,命名为CL12-)和Gen10(CL群体的另一种基因型,命名为CL12+)3个有代表性的ITS2二级结构进行详细分析(图3)。结果显示nCL群体ITS2二级结构高度相似,几乎无差异。CL群体与nCL群体ITS2二级结构的a、b 2个结构域相同,但位于a、b区之间的c、d、e三个区域有明显的差异,c、d区是CL群体特有区域,而e区是nCL区域特有。根据c区的差异又可把CL群体的ITS2二级结构分为CL12-(图3B-c)和CL12+(图3C-c)2种类型。CL12-型和CL12+型二级结构极为相似,但结构域c有差异,结构域c的茎环结构中茎部结构完全相同,但是环状结构差异很大,B-c中的环由ACTCGTT共7个碱基组成,而C-c比B-c环大,由19个碱基组成,在B-c中环的G与T之间插入一个12个核苷酸(CTTTACCGCTTG)序列;之外,图3B-c和C-c茎环结构的3'侧翼碱基组成也不同,B-c中3'侧翼只有一个碱基T,而C-c中3'侧翼为TCGC,比B-c多3个核苷酸(CGC)。上述结果证实ITS2基因型Gen3与Gen6、Gen10二级结构形态明显不同,极易区分。而CL群体与非长乐群体ITS2二级结构a、b区相同,难以区别。长乐群体的CL12-和CL12+2种类型与生理或其它形态性状有什么关系,深入研究。

图3 西施舌ITS2二级结构比较Fig.3 Comparation of the ITS2 secondary structure of Coelomactra antiquata
根据二级结构形状,可将16S片段15个单倍型分为2大类,第一类包括5种单倍型(Hap1,3,4,8,14)(图4A),这一类16S二级结构的共同特点是主干茎环区较短,5种二级结构中的DI和DII区均相同(图4ADI,A-DII),DIV 区在其中的2 种(Hap1,14)二级结构中相同;第二类包括 10 种单倍型(Hap2,5-7,9-13,15),其共同的二级结构特点是主干茎环区较长(图4B),有2个区域(B-DI,B-DII)在10种单倍型中相同,且这两个区域碱基所占比例较大。有趣的是第一类16S二级结构中包含的单倍型均为CL群体所有,第二类二级结构中全部为nCL群体所有。A-DI、A-DII为第一类二级结构特有结构域,B-DI、B-DII(多数)是第二类二级结构所特有,通过这四个结构域极易将西施舌CL群体和nCL群体区别开来。
3 讨论
3.1 ITS2和16S资料支持福建西施舌是腔蛤蜊属的一个新种
一直以来,中国南北沿海西施舌被认为是一个种,无人置疑过。近年来,一部分分子生物学和形态学的资料认为福建群体与其它群体间的差异达到了种间差异水平,福建漳州或长乐西施舌是西施舌的一个亚种或隐种。但是要确定长乐西施舌的分类地位,仅现有的资料还不够充分,还需更多的生物学和分子生物学资料的支持。本研究用核DNA(ITS2)和线粒体DNA(16S)序列资料对我国南北沿海9个群体西施舌遗传差异分析结果显示,基于16S核苷酸序列的CL群体与nCL群体间遗传距离平均为8.1%,而nCL群体间的遗传距离平均为0.7%。与西施舌同属帘蛤目(Veneroida)的文蛤属的文蛤Meretrix meretrix(GQ463598)、丽文蛤Meretrix lusoria(GQ903339)和中华文蛤Meretrix petechialis(EU145977)3种贝类间(基于16S)遗传距离为0.5%—6.7%;贻贝目贻贝属(Mytilus)的紫贻贝Mytilus galloprovincialis(AY363687),贻贝Mytilus edulis(AY823623)和油黑壳菜蛤Mytilus trossulus(DQ198225)3种贝类(基于16S)的遗传距离为0.2%—0.7%。均小于西施舌CL群体与nCL群体间的遗传距离,这些资料从侧面证实CL群体与nCL群体的差异达到了种间差异水平。ITS2、16S二级结果显示CL群体内不同基因(单倍)型结构相似,nCL群体内各群体间二级结构相似(图3,图4),但CL、nCL群体间的二级结构明显不同。ITS2和16S二级结构比较结果提示中国西施舌存在新种,即福建西施舌可能是腔蛤蜊属的一个新种。这个结果与 ITS1[15]、COX1[18]、cytb[17]、AFLP[19]、形态学分析[10,11]结果一致。
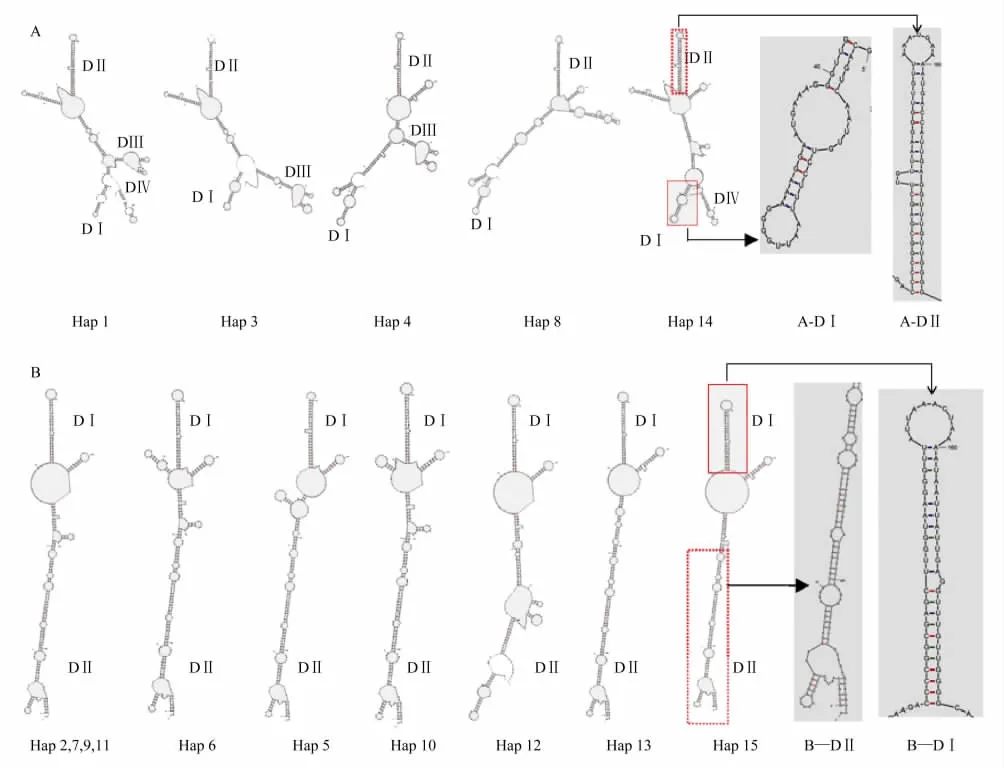
图4 西施舌16S rRNA基因单倍型二级结构比较Fig.4 Comparation of the16S rRNA gene hyplotype secondary structure of Coelomactra antiquata
Hebert等[24]基于细胞色素C氧化酶亚基3(cox3)核苷酸序列,计算了软体动物的1155个同属内物种对的遗传距离,平均为11.1%。此外,Hebert等[25]提出群体间差异若为群体内个体间差异的10倍,则认为这种差异达到种的水平,即“10×”规则。日照西施舌与漳州(同长乐西施舌的差异属种内差异)[18]西施舌cox1全序列核苷酸差异(divergence)为14.5%,大于软体动物同属不同种间的差异水平,也达到“10×”规则的水平。Ni等[26]对蛤蜊科11种贝类cox1和16S序列差异分析显示,cox1的种内遗传距离低于2.5%,而属内种间遗传距离为8.2%—28.4%,是种内遗传距离的3.2—11.4倍,最小种间(M.coralline和 M.lignaria)遗传距离为8.5%—9.3%。由此可见,CL和nCL群体西施舌遗传差异也达到蛤蜊科同属不同种间差异水平;11种贝类16S的种内和种间遗传距离分别为0—0.9%,1.4%—27.1%,种间遗传距离是种内的1.6—30.1倍。本研究结果显示西施舌16S的CL与nCL群体间遗传距离为8.1%,是CL群体内(0.7%)的11.6倍,此差异水平在蛤蜊科11种贝类种间差异水平范围内。孟学平比较漳州与日照西施舌线粒体全基因组显示,两者的主非编码区(MNR)所在位置虽然相同,但大小和序列存在明显差异,前者无串联重复序列,而后者有11个拷贝的99个核苷酸的串联重复序列,且两者的MNRs无可比性[27]。Xu等[28]对巴非蛤属(Paphia)4种不同贝类线粒体全基因组比较研究显示,Paphia undulata和P.textile的MNR所在位置相同,均有串联重复区,只是串联重复单位大小和的拷贝数不同。同种不同个体mtDNA MNR若存在串联重复区,其拷贝数可能有变化(异质性),但同种不同个体MNR的有和无的现象目前在双壳类中尚未发现。因此,mtDNA MNR的差异也强有力地支持漳州西施舌与日照西施舌的差异达到种的水平,这与本研究的结果一致。
3.2 西施舌广西北海群体与辽宁、山东和江苏群体差异用ITS2、16S标记不易区分
广西北海大陆海岸线离北方沿海(如江苏启东)最近也有7000多公里,本研究ITS2和16S资料证实西施舌北海群体(GX)与北方群体(JM、JN、RZ、LYG、QD和DL)亲缘关系近,基于16S和ITS2的遗传距离分别为0.005—0.010 和0.005—0.016,与福建群体遗传关系远(遗传距离 16S/ITS2:0.080—0.088/0.022—0.036)。此外,广西群体16S和ITS2均有与北方群体共享单倍型或基因型,与CL群体无共享单倍型或基因型。这充分说明广西群体与北方群体的遗传关系近,与福建群体遗传关系远。cox1[18]、cob[17]、ITS1[15]资料也与本研究结果一致。这种现象与地理距离与遗传关系远近的一般规律不相符。此种现象在巴非蛤[29]和青蛤[30]不同群体中也存在。造成这种现象的原因之一可能是人为因素造成。尤仲杰对山东胶南、江苏南通、浙江台州、福建福州、广西北海5个群体的RAPD分析证实广西北海西施舌分化最大,可能形成了一个地理种群,而福州西施舌未形成地理种群[12];等位酶的研究也认为北海西施舌形成了地理种群[14]。本研究结果与RAPD[12]、等位酶的研究结果有分歧。因此,关于广西西施舌与其它群体的遗传关系需要用更有说服力的生物及分子生物学资料作深入研究。
致谢:感谢东华大学孟清教授、意大利博洛尼亚大学Plazzi博士和美国北卡罗来纳州立大学Raley博士对本文英文摘要的修改润色。
[1] Salvi D,Bellavia G,Cervelli M,Mariottini P.The analysis of rRNA sequence-structure in phylogenetics:An application to the family Pectinidae(Mollusca:Bivalvia).Molecular Phylogenetics and Evolution,2010,56(3):1059-1067.
[2] Salvi D,Mariottini P.Molecular phylogenetics in 2D:ITS2 rRNA evolution and sequence-structure barcode from Veneridae to Bivalvia.Molecular Phylogenetics and Evolution,2012,65(2):792-798.
[3] Yu D H,Chu K H.Species identity and phylogenetic relationship of the pearl oysters in Pinctada Röding,1798 based on ITS sequence analysis.Biochemical Systematics and Ecology,2006,34(3):240-250.
[4] Barucca M,Olmo E,Schiaparelli S,Canapa A.Molecular phylogeny of the family Pectinidae(Mollusca:Bivalvia)based on mitochondrial 16S and 12S rRNA genes.Molecular Phylogenetics and Evolution,2004,31(1):89-95.
[5] Tao Y,He M X,Huang L M.Intraspecific genetic variation in mitochondrial 16S rRNA and COI genes in domestic and wild populations of Huaguizhikong scallop Chlamys nobilis Reeve.Aquaculture,2009,289(1/2):19-25.
[6] Puslednik L,Serb J M.Molecular phylogenetics of the Pectinidae(Mollusca:Bivalvia)and effect of increased taxon sampling and outgroup selection on tree topology.Molecular Phylogenetics and Evolution,2008,48(3):1178-1188.
[7] Wood A R,Apte S,MacAvoy E S,Gardner J P A.A molecular phylogeny of the marine mussel genus Perna(Bivalvia:Mytilidae)based on nuclear(ITS1&2)and mitochondrial(COI)DNA sequences.Molecular Phylogenetics and Evolution,2007,44(2):685-698.
[8] Meng X P,Gao R C,Dong Z G,Cheng H L,Yan B L.Analysis and evaluation of nutritive composition in edible part of Coelomactra antiquata.Marine Sciences,2007,31(1):17-22.
[9] Wu J F,Chen L X,Chen S W.Current status,and prospects for culture and proliferation of Coelomactra antiquata in China.Journal of Dailian Fisheries University,2005,20(2):137-141.
[10] Liu D J,Zhu S Y.Study of morphological difference of Coelomactra antiquata in Fujian and Jiangsu coasts.South China Fisheries Science,2010,6(2):29-34.
[11] Kong L F,Li Q,Qiu Z X.Genetic and morphological differentiation in the clam Coelomactra antiquata(Bivalvia:Veneroida)along the coast of China.Journal of Experimental Marine Biology and Ecology,2007,343(1):110-117.
[12] You Z J,Bao Y B,Zhang A J.Morphological and RAPD variation among five polulations of Coelomactra antiquata.Acta Oceanologica Sinica,2007,29(3):98-104.
[13] Li Z B,Wang Z L,Zhang G L,Chen J,Zhao B L,Wu N,Lin X Y.Genetic diversity and differentiation of Coelomactra antiquata(spengler)population.Oceanologia et Limnologia Sinica,2009,40(30):302-306.
[14] Bao Y B,Yu D D,You Z J,Lin Z H.Allozyme analysis of genetic diversity in five natural populations of Coelomactra antiquata from coast of China.Journal of Hydroecology,2010,3(3):76-79.
[15] Meng X P,Gao R C,Shen X,Wang S,Cheng H L,Dong Z G,Yan B L.ITS1 sequences variation and phylogenetic analysis on five geographical stocks of Coelomactra antiquata.Acta Ecologica Sinica,2010,30(20):5555-5561.
[16] Meng X P,Wang S,Gao R C,Shen X,Cheng H L,Tian M.RAPD analysis of genetic structure and differentiation of Coelomactra antiquata.Marine Sciences,2010,35(2):6-10,43-43.
[17] Meng X P,Shen X,Zhang B,Cheng H L,Tian M,Zheng W W.Divergences in cyt b gene nucleotides in clam Coelomactra antiquata.Fisheries Science,2010,29(9):537-542.
[18] Meng X P,Gao R C,Shen X,Zheng W W,Zhao N N,Cheng H L,Tian M.DNA barcodes of clam Coelomactata antiquata Bivalvia:Veneroida)in China based on COI gene.Fisheries Science,2011,30(10):626-630.
[19] Kong L F,Li Q.Genetic comparison of cultured and wild populations of the clam Coelomactra antiquata(Spengler)in China using AFLP markers.Aquaculture,2007,271(1/4):152-161.
[20] Kong L F,Li Q.Genetic evidence for the existence of cryptic species in an endangered clam Coelomactra antiquata.Marine Biology,2009,156(7):1507-1515.
[21] Oliverio M,Mariottini P.A molecular framework for the phylogeny of Coralliophila and related muricoids.Journal of Molluscan Studies,2001,67(2):215-224.
[22] Larkin M A,Blackshields G,Brown N P,Chenna R,McGettigan P A,McWilliam H,Valentin F,Wallace I M,Wilm A,Lopez R,Thompson J D,Gibson T J,Higgins D G.Clustal W and Clustal X version 2.0.Bioinformatics,2007,23(21):2947-2948.
[23] Tamura K,Dudley J,Nei M,Kumar S.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0.Molecular Biology and Evolution,2007,24(8):1596-1599.
[24] Hebert P D N,Ratnasingham S,de Waard J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species.Proceedings of the Royal Society B:Biological Sciences,2003,270(Suppl.1):S96-S99.
[25] Hebert P D N,Stockle M Y,Zemlak T S,Francis C M.Identification of birds through DNA barcodes.PLoS Biology,2004,2(10):e312.
[26] Ni L H,Li Q,Kong L F,Huang S Q,Li L J.DNA barcoding and phylogeny in the family Mactridae(Bivalvia:Heterodonta):Evidence for cryptic species.Biochemical Systematics and Ecology,2012,44:164-172.
[27] Meng X P,Shen X,Zhao N N,Tian M,Liang M,Hao J,Cheng H L,Yan B L,Dong Z G,Zhu X L.Mitogenomics reveals two subspecies in Coelomactra antiquata(Mollusca:Bivalvia).Mitochondrial DNA,2012;Early Online:1-3.
[28] Xu X D,Wu X Y,Yu Z N.Comparative studies of the complete mitochondrial genomes of four Paphia clams and reconsideration of subgenus Neotapes(Bivalvia:Veneridae).Gene,2012,494(1):17-23.
[29] Liu J Y,Wu J X,Shong C B.Morphological variations of different geographic populations of Paphia undulata off southeast China.Oceanologia et Limnologia Sinica,2010,41(1):114-120.
[30] Gu H G,Liu J Y,Yang G H.Analysis on morphological variations of four wild populations of Cyclina sinensis from the South China Sea.Journal of Guangdong Ocean University,2010,33(3):1-5.
参考文献:
[8] 孟学平,高如承,董志国,程汉良,阎斌伦.西施舌营养成分分析与评价.海洋科学,2007,37(1):17-22.
[9] 吴进锋,陈利雄,陈素文.西施舌增养殖的研究现状与展望.大连水产学院学报,2005,20(2):137-141.
[10] 刘德经,朱善央.福建与江苏西施舌群体形态差异研究.南方水产,2010,6(2):29-34.
[12] 尤仲杰,包永波,张爱菊.中国沿海西施舌5个自然群体形态差异和RAPD分析.海洋学报,2007,29(3):98-104.
[13] 黎中宝,王展林,张桂玲,陈锦,赵斌丽,吴宁,林小云.西施舌(Coelomactra antiquata)群体遗传多样性与分化的研究.海洋与湖沼,2009,40(30):302-306.
[14] 包永波,余冬冬,尤仲杰,林志华.中国沿海5个自然西施舌群体等位酶的遗传变异研究.水生态学杂志,2010,3(3):76-79.
[15] 孟学平,高如承,申欣,王帅,程汉良,董志国,阎斌伦.西施舌5个地理群体ITS1序列变异及系统发生分析.生态学报,2010,30(20):5555-5561.
[16] 孟学平,王帅,高如承,申欣,程汉良,田美.西施舌群体遗传结构及分化的RAPD分析.海洋科学,2011,35(2):6-10,43-43.
[17] 孟学平,申欣,张波,程汉良,田美,郑雯雯.西施舌Cyt b基因核苷酸差异分析.水产科学,2010,29(9):537-542.
[18] 孟学平,高如承,申欣,郑雯雯,赵娜娜,程汉良,田美.基于COI的中国西施舌DNA条形码.水产科学,2011,30(10):626-630.
[29] 刘建勇吴继兴 孙成波.我国东南沿海5个波纹巴非蛤(Paphia undulata)地理群体的形态差异分析.海洋与湖沼,2010,41(1):114-120.
[30] 古恒光,刘建勇,杨国宏.我国南海4个青蛤野生群体的形态差异分析.广东海洋大学学报,2010,33(3):1-5.
