我国特有植物青檀遗传结构的ISSR分析
2013-12-08李晓红王德元邵剑文张小平
李晓红,张 慧,王德元,张 莉,邵剑文,2,*,张小平,3
(1. 安徽师范大学生命科学学院, 芜湖 241000;2. 生物环境与生态安全安徽省高校重点实验室, 芜湖 241000;3. 安徽省重要生物资源保护与利用研究重点实验室, 芜湖 241000)
我国特有植物青檀遗传结构的ISSR分析
李晓红1,张 慧1,王德元1,张 莉1,邵剑文1,2,*,张小平1,3
(1. 安徽师范大学生命科学学院, 芜湖 241000;2. 生物环境与生态安全安徽省高校重点实验室, 芜湖 241000;3. 安徽省重要生物资源保护与利用研究重点实验室, 芜湖 241000)
青檀是我国特有的第三纪残遗植物,是宣纸纤维来源的重要原材料,现已被列入国家Ⅲ级保护植物。采用简单序列重复区间扩增多态性(ISSR)标记技术,对采自27个地理种群的628个青檀个体的遗传多样性和遗传结构进行了检测。8对引物共得到66个清晰的扩增位点,其中多态性位点63个,多态性位点百分率为95.45%。分析结果表明,青檀在物种水平上具有很高的遗传多样性(PPB=95.45%、Ao=1.9545、Ae=1.5729、He=0.3335、I=0.4980、Hb=0.3437)。在种群水平上的遗传多样性(PPB=69.98%、Ao=1.6998、Ae=1.4449、He=0.2561、I=0.3793、Hb=0.2656)和种群间遗传分化水平(Gst=0.23,ФST=0.25)均处于中等水平。华北地区(30°—42°N)和华南地区(22°—30° N)青檀的遗传多样性和遗传结构的差异并不显著。古老的起源历史、较广的分布范围、异交的繁育系统、风媒种子、长命的生活史以及华南和华北地形和植被的差异可能是导致青檀目前遗传格局的主要原因。结合叶绿体序列(cpDNA)序列的遗传结构特征对青檀的保护策略进行了探讨。
青檀;ISSR;遗传多样性;遗传结构;保护策略
青檀(PteroceltistatarinowiiMaxim.)为我国特有第三纪残遗温带落叶树种,隶属于榆科(Ulmaceae)唯一的单种属——青檀属 (Pteroceltis)。该树种对气候的适应幅度较宽,抗逆性高,萌生能力强,是钙质土壤石灰岩植物群落中的先锋树种,在我国的分布范围十分广泛,在华北、华东、华中和华南均有分布,其分布中心在华东,其中以安徽宣城、宁国、泾县分布最为集中[1- 2]。作为我国特有的经济纤维树种,青檀是“文房四宝”之首——宣纸的重要原材料(韧皮纤维)来源,已被列入国家Ⅲ级珍稀保护植物名录[3- 4]。目前许多研究人员从不同角度对该物种进行了相关研究,包括青檀的群落种群特征[5- 8]、种子休眠机制[9]、檀皮质量的影响因素、化学成分、人工林的苗木培育技术[1,10]、内生真菌[11]等方面。
了解一个物种的遗传多样性及其结构不仅可以加深人们对该植物进化历史和适应机制的认识[12- 14],还可为该植物的保护、品种选育和栽培利用提供指导[15- 17]。在青檀种内遗传多样性的研究方面,李晓红等2012年对代表青檀分布区的28个种群(共284个个体)的叶绿体序列 (trnS-trnG 和psbA-trnH) 进行了分析,从母系遗传的角度揭示了中国南部(北纬30°以南)青檀遗传多样性较高,种群间分化显著,而中国北部(北纬30°以北)遗传多样性相对较低,种群间分化相对较小,并推测青檀在第四纪冰期可能具有多个避难所[18]。然而一个物种双亲遗传的核基因多样性才是该物种遗传多样性的主体部分,更能体现该物种适应和进化的潜力[19]。至今,有关青檀核基因的遗传多样性和遗传结构的研究尚不全面。Chai等利用ISSR分子标记对安徽、山东、江苏和河南四省5个青檀野生种群的样品进行了研究[20];李建华等对湖北大贵寺国家森林公园不同海拔高度的野生青檀的种群遗传多样性进行了分析[21],这些研究使我们对青檀的核基因遗传多样性有了初步的了解,然而由于两者的取样点相对于青檀的整体分布区相对较少,样点分布也偏于集中,因而均未能全面揭示广布种青檀物种水平上的遗传多样性和分布格局。
简单序列重复区间多态性(ISSR)是Zietkeiwitcz等于1994年发展起来的一种基于微卫星基础上的分子标记[22]。它虽然是一种显性遗传标记,不能区分纯合体与杂合体,然而由于ISSR比等位酶、限制性片段长度多态性(RFLP)、微卫星(SSR)等技术简单易行、多态性高,因而近年来应用ISSR技术分析植物遗传多样性的研究已有大量报道[23- 24]。本研究采用ISSR分子标记,在整个分布区取样的基础上,拟从核基因的角度研究:1)青檀的遗传多样性和遗传结构;2)中国南部和北部青檀的遗传多样性及其分化程度是否存在明显的差异? 3)结合青檀的生物学特性和种群历史,进一步探讨现存青檀遗传格局的形成及维持机制,为青檀种质资源的有效保护和栽培利用提供参考。
1 材料和方法
1.1 供试材料
根据文献资料记载,2008年至2011年对青檀分布区的27个自然种群进行了取样,每个种群采集21—24个植株,植株间至少相隔30—50m。选取每个植株健康幼嫩的叶片3—4片,硅胶迅速干燥后保存。根据Harrison(2001)对东亚植被演替的归纳总结[25],其中15个种群位于华北(30° — 42° N),主要为温带落叶林;12个种群位于华南(22° — 30° N),主要为温带常绿林。种群编号、地理位置、经纬度、海拔高度及样本大小详见表1,采样种群的地理位置分布见图1。
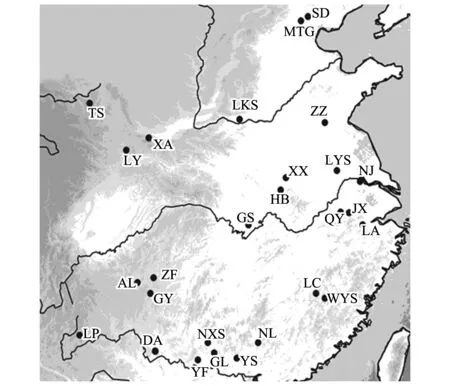
图1 青檀样品采集分布图 Fig.1 The sampled populations distribution of P. tatarinowii
1.2 基因组DNA的提取与聚合酶链式反应(PCR)扩增
采用改良十六烷基三甲基溴化铵(CTAB)法[26],从硅胶干燥后的青檀叶片中提取总基因组DNA。ISSR扩增反应条件经过比较和优化确定为20 μL的反应体系,内含:10 × PCR缓冲液 2.0 μL,2.00 mmol/L Mg2+,0.20 mmol/L dNTP,0.40 μ mol/L引物,1.25 UTaqDNA聚合酶,模板DNA 100 ng。扩增程序:94℃ 预变性 5 min,接着进行35个循环:94℃ 变性 30 s,55℃ 退火 45 s,72℃ 延伸 90s,循环结束后,72℃延伸 7 min,最后 4℃ 保存。PCR扩增结束后,用含有溴化乙锭(EB)的1.5%琼脂糖凝胶电泳,电泳缓冲液为 1 × TAE,以 DL 2000 Marker为分子量标记,电泳后用凝胶成像系统拍照。位点的命名由所用引物和条带的大小来确定。
1.3 引物筛选
在上海生物工程公司合成加拿大哥伦比亚大学(UBC)公布的第9套ISSR引物序列(100条),随机从10个种群中各选取1个个体,在20μL反应体系中进行扩增筛选。最终从100 条引物中筛选出8 条扩增条带清楚、重复性好的引物(表2)用于所有个体的扩增。
1.4 统计分析
由于ISSR为显性标记,电泳图谱的每条带都视为一个分子标记,代表引物的一个特异结合位点[27]。根据各分子标记的迁移率统计得到所有位点的二元数据,有带(包括弱带)记为“1”,无带记为“0”。
采用POPGENE 1.32 计算出多态位点百分率(PPB)、观测等位基因数(Ao)、有效等位基因数(Ae)、Nei′s基因多样性(He)、Shannon′s信息指数(I)、群体总基因多样性(Ht)、各群体间的遗传分化指数(GST)和Nei′s遗传距离[28]。利用不基于遗传平衡原理的 HICKORY version 1.0[29]在f-free模型下计算种群贝叶斯遗传多样性 (Hb)[30],参数为软件默认值。
根据Nei′s遗传距离,利用NTSYS pc2.1软件 的非加权配对算术平均法(UPGMA)对青檀群体间的遗传关系进行聚类分析[31]。采用MVSP(version 3.1)中的主成分分析(PCA)对群体的遗传关系进行聚类分析[32]。同时使用STRUCTURE 2.2中贝叶斯聚类法对所有个体进行聚类分析[33],K值设为10,5个重复,使用混合模型、burn-in为100000和run-length为1000000,根据每次运行不同的K值对应得lnP(D)值,计算ΔK值分析青檀可能的遗传结构[34]。采用Arlequin 3.0 软件对物种水平、华南、华北地区的青檀群体内、群体间和地区间的分子变异进行分析[35],进一步检测种群间的分化。遗传距离和地理距离之间的相关性进行中性检测(Mantel test)[36]。
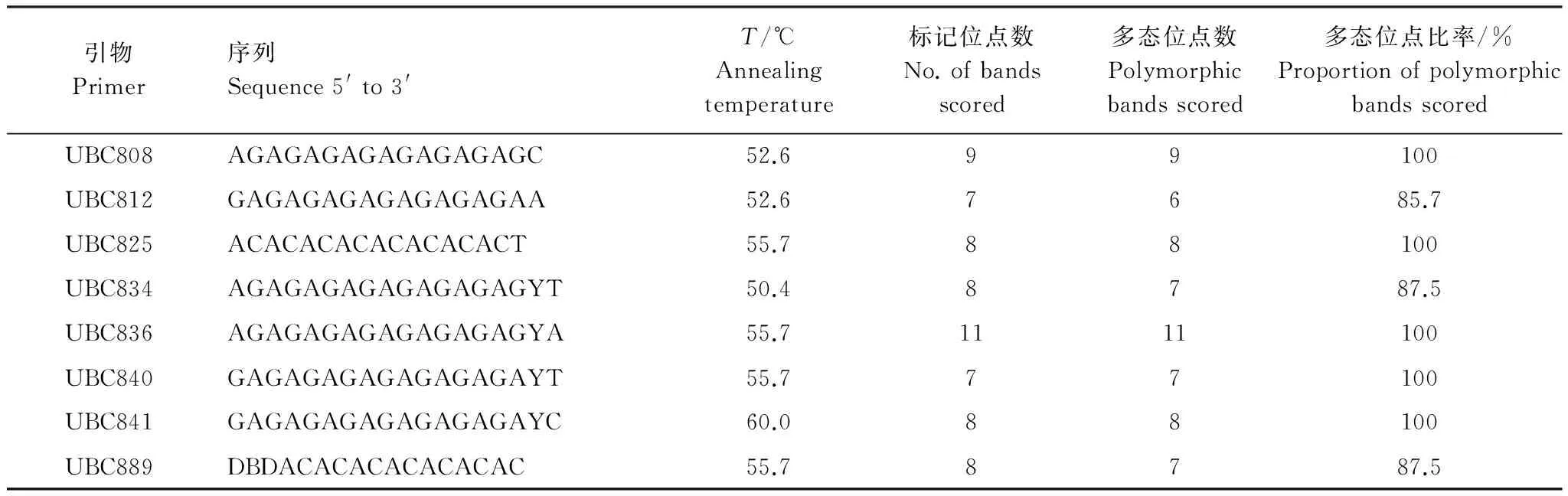
表2 筛选出的ISSR引物序列(5′—3′)及标记位点
R=A/T, Y=C/G, D=A
图2 UBC834引物的ISSR部分扩增结果Fig.2 Part results of UBC834 amplification of P.tatarinowii
2 结果
2.1 遗传多样性
8对引物共扩增出66条清晰、稳定的条带,每对引物条带数从7—11条不等(平均为8.25),片段大小分布在250—2500 bp之间(表2和图2)。在物种水平上,青檀多态性位点百分率(PPB)高达95.45%、观测等位基因数(Ao)为1.9545、有效等位基因数(Ae)为1.5729 、Nei′s基因多样性(He)为0.3336、Shannon′s信息指数(I)为0.4980、贝叶斯遗传多样性(Hb)为0.3437(表3)。种群水平的遗传多样性差异较大,平均值PPB=69.98%、Ao=1.6998、Ae=1.4449、He=0.2561、I=0.3793、Hb=0.2656,最高的出现在甘肃天水(TS)种群(PPB=84.85%、Ao=1.8485、Ae=1.5217、He=0.3033、I=0.4516、Hb=0.3060),最低的出现在广东南岭(NL)种群(PPB=54.55%、Ao=1.5455、Ae=1.3135、He=0.1841、I=0.2756、Hb=0.2130)。
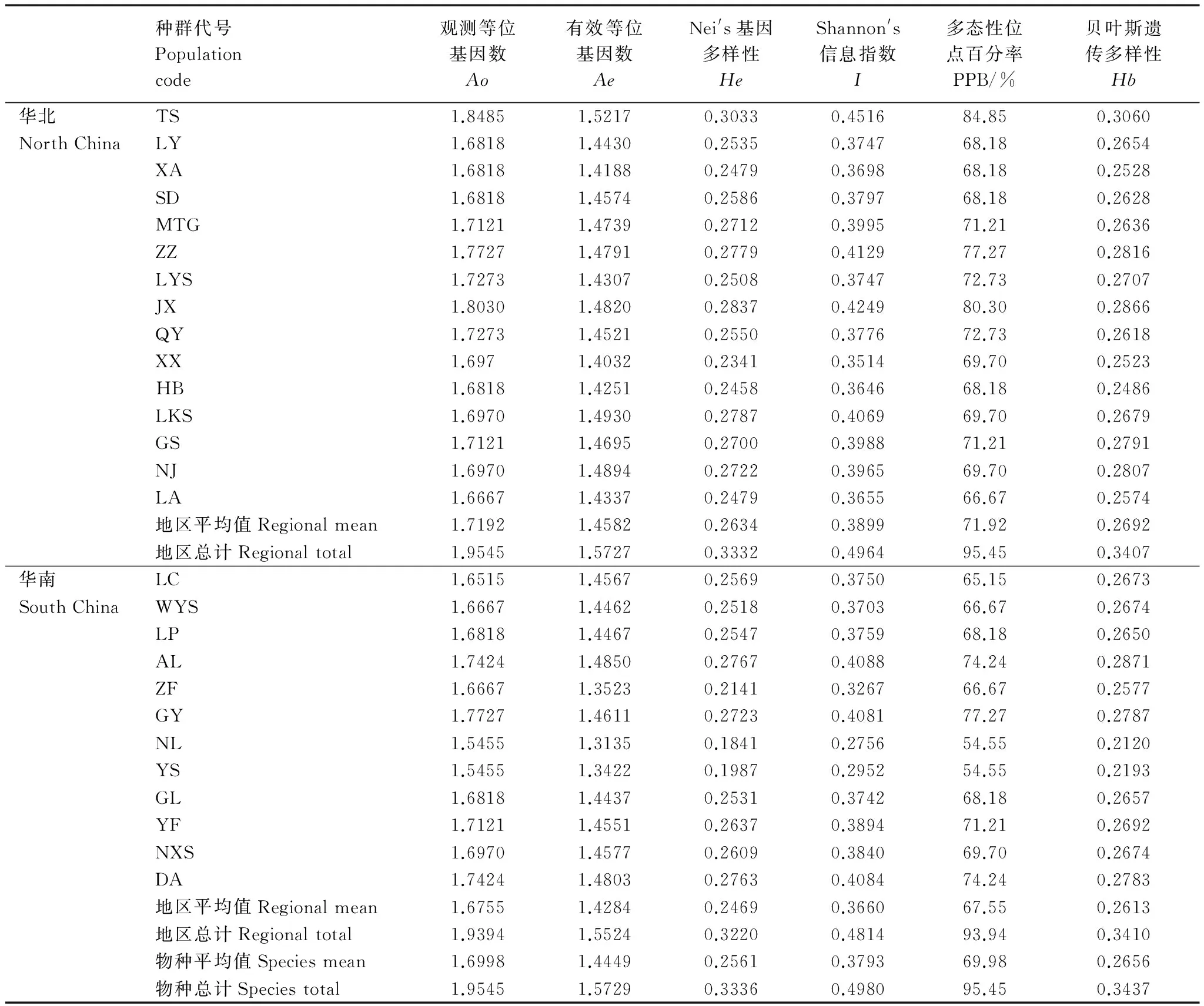
表3 基于ISSR检测的青檀遗传多样性水平
Ao: observed number of alleles per locus;Ae: effective number of alleles per locus;He: Nei′s gene diversity;I: Shannon′s information index; PPB: percentage of polymorphic loci
在地区水平上,华北地区总的多态性位点百分率(PPB=95.45%)、Nei′s基因多样性(He=0.3332)、Shannon′s信息指数(I=0.4964)均略高于华南地区 (PPB=93.94%、He=0.3220、I=0.4814),但种群的平均水平两者却没有显著差异(Pgt; 0.05)。两地区的贝叶斯遗传多样性(Hb)比较接近(表3)。
2.2 遗传结构
在物种水平上,青檀27个种群的总基因多样性(Ht)为0.3336,其中种群内平均遗传变异(Hpop)为0.2561,Nei′s基因分化系数(Gst)为0.2323,表明总的遗传变异中有23.23%来自于种群间,76.77%来自种群内,遗传变异主要存在于种群内。种群间基因流(Nm)为1.6524。在地区水平上,华南地区的基因分化系数(Gst=0.2326)略高于华北地区的基因分化系数(Gst=0.2100);华南地区的平均基因流(Nm=1.6497)略低于物种基因流平均水平(Nm=1.6524), 而华北地区的基因流(1.8814)略高于物种整体的基因流。
分子变异分析(AMOVA)分析的结果也同样显示(表5),青檀的主要遗传变异存在于种群内(占75.26%),种群间具中度的遗传分化水平(ФST=0.25)。华南和华北的遗传多样性也是主要存在于种群内,但华南地区(ФST=0.26)种群间的遗传分化水平略高于华北地区(ФST=0.23),华南和华北之间分化很小(ФST=0.02)。
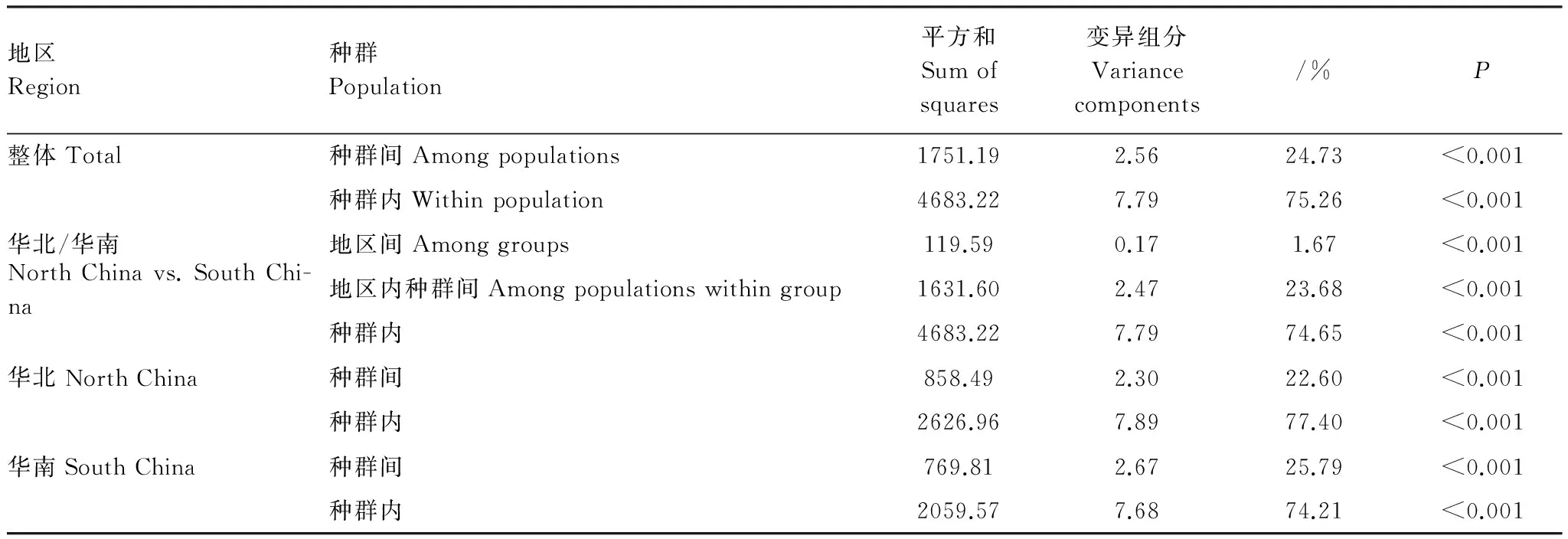
表5 青檀种群的AMOVA分析
P: the probability of null hypothesis; 1000次模拟的显著性检测
STRUCTURE软件分析结果的ΔK峰值出现在K=2,暗示着27个青檀种群的遗传结构可以大致分为两组。从图4可以看出,当K=2时华南和华北种群并没有明显分开,相比较而言LKS、NJ、ZZ、YF、LP、DA、AL、ZF、GY、NXS、LC 11个种群较相似(编号为蓝色),与NL、YS、GS、LYS种群(编号为红色)区别较大,其它种群(编号为黒色)混杂比较明显。
2.3 聚类分析及遗传距离与地理距离的关系
PCA聚类分析结果(图3)与UPGMA聚类结果(图5)相似,青檀各种群的遗传相似度较高,并没有形成明显的遗传结构,华南与华北地区并没有单独各聚在一起,而是互相掺杂。在物种水平上,遗传距离与地理距离没有明显的相关性(r=0.029,Pgt; 0.05);在地区水平上,华南地区遗传分化与地理距离呈中度相关(r=0.307,Plt; 0.05),而华北地区的遗传分化和地理距离不具相关性(r=-0.066,Pgt; 0.05)。
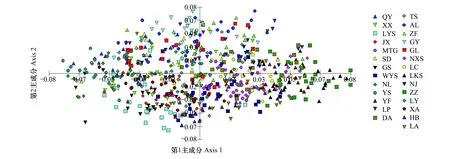
图3 青檀27个种群的主成分分析(PCA)分析Fig.3 PCA analysis of 27 populations of P. tatarinowii
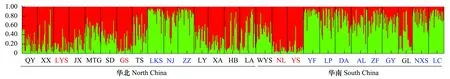
图4 K=2时贝叶斯聚类法的分析结果Fig.4 Histogram of the bayesian analysis with K=2 using STRUCTURE
3 讨论
3.1 青檀的遗传多样性和遗传结构
本文对代表青檀整个分布区的27个自然种群共628个个体遗传多样性分析显示,青檀在物种水平上具有很高的遗传多样性,其多态位点百分率(PPB)达到95.45%,明显高于Nybom 和Bartish对107个物种总结的平均遗传多样性(PPB=71.02%)[37],也明显高于一般木本植物的遗传多样性,如银杏PPB=70.45%[38]、长果秤锤树PPB=72.99%[39]、思茅木姜子PPB=87.01%[40]、板栗PPB=87.07%[41]等。Chai X Y和李建华对局部区域青檀的遗传多样分析的结果也揭示了类似的结果(Chai X Y: PPB=100%; 李建华:PPB=92.14% )。青檀种群内的遗传多样性(PPB=69.98%、Hpop=0.260、I=0.380)和种群间遗传分化程度(Gst=0.23,ФST=0.25)均处于中等水平,与异花传粉植物(Hpop=0.260,Gst=0.23,ФST=0.28)、种子风媒植物(Hpop=0.261,Gst=0.23,ФST=0.25)和长命植物(Hpop=0.242,Gst=0.23,ФST=0.25)的平均水平十分接近[38]。
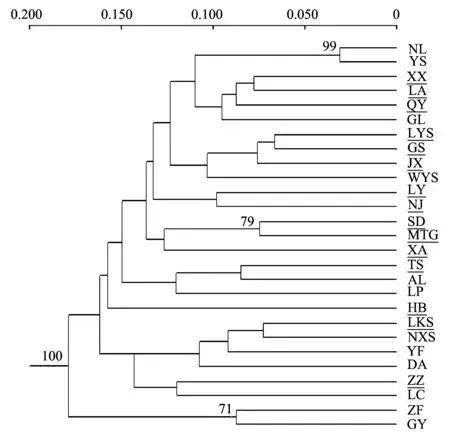
图5 青檀种群间Nei′s遗传距离建立的非加权配对算术平均法(UPGMA)聚类图Fig.5 Dendrogram of populations of P. tatarinowii generated from UPGMA cluster analysis according to Nei′s genetic distance有下划线的为北方种群,余为南方种群; 节点处数字代表gt;50%支持率
一个物种现有的遗传多样性和遗传结构是由其自身因素和环境因素共同作用的结果,其中包括物种进化历史、交配系统、基因流、基因漂变、地理分布等[37,42]。相对于青檀而言,古老的起源历史、较广的分布、异交的繁育系统、风媒种子及长命的生活史可能是其目前仍维持着较高遗传多样和中度遗传分化水平的主要原因。青檀是第三纪的孑遗植物[18],漫长的进化历史在时间尺度上为其基因突变、重组和变异积累提供了可能。此外,青檀主要分布于石灰岩地带,范围较为广泛,涵盖我国19个省市,大尺度的地理跨度、生境和气候的异质性为青檀遗传多样性提供了孕育场所。在繁殖方式上,青檀既可依靠有性繁殖也可依靠无性繁殖进行种群的自然更新。青檀雌雄异花同株、无花瓣,花丝直立将花药撑起、先花后叶等特征都有利于花粉借助风力散布;而且其种子两侧具薄宽翅,能借助自然风力进行较远距离传播[2]。因而这些特征十分有助于青檀各种群间的基因交流,维持了较大有效种群,可减小遗传漂变的影响[43]。另外,青檀是一种典型的长命木本植物,种群中具有多个世代、世代更替慢也可能是它维持现有遗传结构的原因之一[37,44]。
3.2 华南与华北地区青檀遗传多样性与遗传结构的差异
青檀是典型的落叶阔叶树种,现主要分布于我国华北(30°—42°N)的温带落叶林中和华南 (22°—30° N) 亚热带常绿阔叶林中[2, 25]。李晓红等采用母系遗传的cpDNA序列研究发现两种植被下青檀的遗传结构呈现明显差异,华北地区细胞质基因的遗传多样性(hT=0.32,πT=0.00064)明显低于华南地区的(hT=0.85,πT=0.00453),前者种群间的遗传分化水平(ФST=0.69)也明显低于后者(ФST=0.97)[18]。本文基于ISSR遗传标记研究显示,在核基因上,虽然华北地区的青檀遗传分化水平(ФST=0.22)略低于华南地区(ФST=0.25),前者总遗传多样性水平(PPB=95.45%,h=0.3332,I=0.4964)也略高于后者(PPB=93.94%,He=0.3220,I=0.48140),但种群水平的平均遗传多样性两者差异并不显著。青檀这种华南和华北地区间遗传多样性和遗传结构差异形成的主要原因可能是:华南地区(22°—30°N)地形复杂,存在多条明显的山脉,而华北地区(30°—42°N)青檀分布区地形平坦、没有山脉的阻碍,这种地形的差异已对种子的风媒传播产生了一定的影响,导致南部种子传播受到限制,种群间母系遗传的隔离明显,由于遗传漂变的原因,突变的单倍型易被保留下来(单倍型多样性较高,华南hT=0.85明显高于华北hT=0.32),种群间分化更明显(华南ФST=0.97高于华北ФST=0.69),然而这种地形差异目前对花粉流的影响还较低,还没有导致核基因遗传多样性与遗传结构上产生明显的差异[45]。这种地形的差异对种子流和花粉流的影响不同也可能是导致华南地区遗传分化与地理距离呈中度相关(r=0.307,Plt; 0.05)而华北地区的遗传分化和地理距离不具相关性(r=-0.066,Pgt;0.05)的主要原因。
3.3 保护建议
青檀是我国特有的经济纤维树种,它是宣纸的重要原材料(韧皮纤维)来源,现已被列入国家Ⅲ级珍稀保护植物名录[4]。物种保护的主要内容是保护其遗传多样性及进化潜力,种内遗传多样性愈丰富,物种对环境变化的适应能力愈强,其进化潜力愈大[46]。鉴于青檀在物种水平上仍维持着较高的遗传多样性,而且遗传多样性主要存在于种群内,因此我们认为青檀野生种群仍具有较高的进化潜力和适应能力,目前保护仍要以就地保护为主。考虑到青檀种群间已存在一定的遗传分化,要尽可能保护现所有的野生种群,尤其要注意对遗传多样性较高的种群(TS、JX、QY)和具有独特单倍型的种群(XA、SD、ML、NXS、LP、YF、WYS、GL、AL)优先保护详见Li,2012,必要时可以设置专门的保护区或保护点。此外,华南地区青檀种群由于与常绿阔叶林混生,建议可以选择性间伐部分常绿阔叶树种,开辟林窗以增强林下透光率,以利于青檀幼苗的生长和花粉及种子的散布,进而增加种群间的基因流。在人工林建立和种质资源库建立时,可以优先考虑上述重点保护的种群作为采样点,同时考虑到华南地区遗传多样性低于华北地区,而遗传分化高于华北地区,华南地区要在尽可能多的种群内收集种子或幼苗,华北地区则可以选择部分代表性的种群进行取样。
[1] Fang S Z, Li G Y, Fu X X. Biomass production and bark yield in the plantations ofPteroceltistatarinowii. Biomass and Bioenergy, 2004, 26(4): 319- 328.
[2] Fang S Z, Fu X X.PteroceltistatarinowiiMaxim in China. Beijing: Chinese Science and Culture Press, 2007.
[3] Cao T S. Xuan Paper of China. Beijing: China Light Industry Press, 1993: 20- 34.
[4] Fu L G. China Plant Red Data Book——Rare and Endangered Plants (Volume 1). Beijing: Science Press, 1992: 686- 686.
[5] Zhang L, Zhang X P, Lu C, Li X H. Spatial pattern ofPteroceltistatarinowiipopulations in Langya Moutain of Anhui province. Scientia Silvae Sinicae, 2012, 48(2): 9- 15.
[6] Zhang L, Lu C, Li X H, Wang L, Zhang X P. Age structure and inter-and intra- species competition ofPteroceltistatarinowiiin Huangcangyu Natural Reserve. Journal of Shanghai Jiaotong University: Agricultural Science, 2012, 30(1): 34- 40.
[7] Zhang X W, Zhang X P, Guo C Y, Ding J H. Quantitative dynamics ofPteroceltistatarinowiipopulations in Huangcangyu Nature Reserve of Anhui Province, East China. Chinese Journal of Ecology, 2012, 31(2): 282- 291.
[8] Zhang X W, Zhang X P, Hao C Y, Guo C Y. Population structure and spatial distribution pattern ofPteroceltistatarinowiiin Langyashan Mountain of Anhui Province. Guihaia, 2011, 31(6): 782- 788.
[9] Zhang X W, Cao J J, Gong Y X, Zhang X P. Seed dormancy and germination in rare plantPteroceltistatarinowiiMaxim. Journal of Biology, 2007, 24(1): 28- 31.
[10] Fang S Z. Cultivation ofPteroceltistatarinowiiand collecting and processing techniques for its barks. China Forestry Science and Technology, 1996, (4): 40- 42.
[11] Chai X Y, Chen S L. Diversity of endophytic fungi fromPteroceltistatarinowii. Mycosystema, 2011, 30(1): 18- 26.
[12] Hong DY, Ge S, Zhang D M, Wang X Q, Cheng S Z. Principles and approaches to studying the mechanisms of plant rarity and endangerment // Qian Y Q, Zheng R D. Advances in Biodiversity Research. Beijing: Science and Technology Press of China, 1995: 125- 133.
[13] Hamrick J L, Godt M J W, Sherman-Broyles S L. Factors influencing levels of genetic diversity in woody plant species. New Forests, 1992, 6(1/4): 95- 124.
[14] Hamrick J L, Godt M J W. Effects of life history traits on genetic diversity in plant species. Philosophical Transactions of the Royal Society B: Biological Sciences, 1996, 351(1345): 1291- 1298.
[15] Song Z Q, Wang J H, Wang H G, Wang M M, Xie Y L. Genetic diversity of different eco-geographical populations inSalviamiltiorrhizarevealed by ISSR Markers in Shandong, China. Acta Ecologica Sinica, 2008, 28(11): 5370- 5376.
[16] Zhang J, Wang J, Li H Y, Zhang H R, Wang Y C. Genetic diversity of different eco-geographical populations in endangered plantPrunusmongolicaby ISSR Markers. Acta Ecologica Sinica, 2012, 32(14): 4443- 4452.
[17] Tian B, Yang H Q, Wong K M, Liu A Z, Ruan Z Y. ISSR analysis shows low genetic diversity versus high genetic differentiation for giant bamboo,Dendrocalamusgiganteus(Poaceae: Bambusoideae), in China populations. Genetic Resources and Crop Evolution, 2012, 59(5): 901- 908.
[18] Li X H, Shao J W, Lu C, Zhang X P, Qiu Y X. Chloroplast phylogeography of a temperate treePteroceltistatarinowii(Ulmaceae) in China. Journal of Systematics and Evolution, 2012, 50(4): 325- 333.
[19] Sang T. Utility of low-copy nuclear gene sequences in plant phylogenetics. Critical Reviews in Biochemistry and Molecular Biology, 2002, 37(3): 121- 147.
[20] Chai X Y, Chen S L, Xu W. Using inter-simple sequence repeat markers to analyze the genetic structure of naturalPteroceltistatarinowiipopulations and implications for species conservation. Plant Systematics and Evolution, 2010, 285(1/2): 65- 73.
[21] Li J H, Li X S, Tian C Y, Zou L P, Wang D P. Genetic diversity of wild populations in DaGuiSi National Forest Park. Ecology and Environmental Sciences, 2011, 20(12): 1799- 1804.
[22] Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification. Genomics, 1994, 20(2): 176- 183.
[23] Esselman E J, Jianqiang L, Crawford D J, Windus J L, Wolfe A D. Clonal diversity in the rareCalamagrostisporterissp.insperata(Poaceae): comparative results for allozymes and random amplified polymorphic DNA (RAPD) and inter simple sequence repeat (ISSR) markers. Molecular Ecology, 1999, 8(3): 443- 451.
[24] Qian W, Ge S, Hong D Y. Genetic variation within and among populations of a wild riceOryzagranulatafrom China detected by RAPD and ISSR markers. Theoretical and Applied Genetics, 2001, 102(2/3): 440- 449.
[25] Harrison S P, Yu G, Takahara H, Prentice I C. Palaeovegetation (Communications arising): diversity of temperate plants in East Asia. Nature, 2001, 413(6852): 129- 130.
[26] Doyle J J. DNA protocols for plants: CTAB total DNA isolation // Hewitt G M, Johnston A. Molecular Techniques in Taxonomy. Berlin: Springer-Verlag, 1991: 283- 293.
[27] Williams J G K, Kubelik A R, Livak K J, Rafalski J A, Tingey S V. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research, 1990, 18(22): 6531- 6535.
[28] Yeh F C, Yang R C. POPGENE Version 1. 32, Microsoft window-based freeware for population genetic analysis. University of Alberta And Tim Boyle, Centre for International Forestry Research. 1999. http://ftp.microsoft.com/softlib/slfiles/hpgl.exe.
[29] Holsinger K E, Lewis P O. Hickory: a Package for the Analysis of Population Genetic Data, Version 1. 1. Department of Ecology and Evolutionary Biology, University of Connecticut, Storrs, Connecticut, USA. 2003.
[30] Zhivotovsky L A. Estimating population structure in diploids with multilocus dominant DNA markers. Molecular Ecology, 1999, 8(6): 907- 913.
[31] Rohlf F J. NTSYS-pc: Numerical Taxonomy and Multivariate Analysis System, version 2. 1. Exeter Software, Setauket, New York, USA. 2000.
[32] Kovach W L. MVSP---A multivariate statistical package for Windows, version 3. 1. Kovach Computing Services, Pentraeth, Wales, UK. 1999.
[33] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155(2): 945- 959.
[34] Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software structure: a simulation study. Molecular Ecology, 2005, 14(8): 2611- 620.
[35] Excoffier L, Laval G, Schneider S. Arlequin (version 3. 0): An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online, 2005, 1: 47- 50.
[36] Mantel N. The detection of disease clustering and a generalized regression approach. Cancer Research, 1967, 27: 209- 220.
[37] Nybom H, Bartich I V. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants. Perspectives in Plant Ecology, Evolution and Systematics, 2000, 3(2): 93- 114.
[38] Ge Y Q, Qiu Y X, Ding B Y, Fu C X. An ISSR analysis on population genetic diversity of the relict plantGinkgobiloba. Biodiversity Science, 2003, 11(4): 276- 287.
[39] Cao P J, Yao Q F, Ding B Y, Zeng H Y, Zhong Y X, Fu C X, Jin X F. Genetic diversity ofSinojackiadolichocarpa(Styracaceae), a species endangered and endemic to China, detected by inter-simple sequence repeat (ISSR). Biochemical Systematics and Ecology, 2006, 34(3): 231- 239.
[40] Chen J Q, Ci X Q, Li Q M, Li J. Genetic diversity ofLitseaszemaois, an endangered species endemic to China, detected by inter-simple sequence repeat (ISSR). Biodiversity Science, 2006, 14(5): 410- 420.
[41] Ai C X, Zhang L S, Wei H R, Jin S N, Yuan K J, Liu Q Z. Study on the genetic diversity of natural chestnut of Shandong by ISSR. Chinese Journal of Biotechnology, 2007, 23(4): 628- 633.
[42] Schaal B A, Hayworth D A, Olsen K M, Rauscher J T, Smith W A. Phylogeographic studies in plants: problems and prospects. Molecular Ecology, 1998, 7(4): 465- 474.
[43] Glemin S, Bazin E, Charlesworth D. Impact of mating systems on patterns of sequence polymorphism in flowering plants. Proceedings of the Royal Society B: Biological Sciences, 2006, 273(1604): 3011- 3019.
[44] Hamrick J L, Godt M J W. Allozyme diversity in plant species // Brown A H D, Clegg M T, Kahler A L, Weir B S. Plant Population Genetics, Breeding, and Genetic Resources. Sunderland: Sinauer Associates Inc., 1990: 43- 63.
[45] Ellstrand N C, Elam D R. Population genetic consequences of small population size: implications for plant conservation. Annual Review of Ecology and Systematics, 1993, 24(1): 217- 242.
[46] Zou Y P, Ge S, Wang X D. The Molecular Markers in Systematic and Evolutionary of Botany. Beijing: The Science Press, 2001: 140- 149.
参考文献:
[2] 方升佐, 洑香香. 中国青檀. 北京: 中国科学文化出版社, 2007.
[3] 曹天生. 中国宣纸. 北京: 中国轻工业出版社, 1993: 20- 34.
[4] 傅立国. 中国植物红皮书——稀有濒危植物(第一分册). 北京: 科学出版社, 1992: 686- 686.
[5] 张莉, 张小平, 陆畅, 李晓红. 安徽琅琊山青檀种群空间格局. 林业科学, 2012, 48(2): 9- 15.
[6] 张莉, 陆畅, 李晓红, 王磊, 张小平. 皇藏峪国家自然保护区青檀种群年龄结构及种内、种间竞争. 上海交通大学学报: 农业科学版, 2012, 30(1): 34- 40.
[7] 张兴旺, 张小平, 郭传友, 丁建华. 安徽皇藏峪自然保护区青檀种群数量动态. 生态学杂志, 2012, 31(2): 282- 291.
[8] 张兴旺, 张小平, 郝朝运, 郭传友. 安徽琅琊山青檀种群结构与空间分布格局研究. 广西植物, 2011, 31(6): 782- 788.
[9] 张兴旺, 操景景, 龚玉霞, 张小平. 珍稀植物青檀种子休眠与萌发的研究. 生物学杂志, 2007, 24(1): 28- 31.
[10] 方升佐. 青檀的栽培及檀皮采集加工技术. 林业科技开发, 1996, (4): 40- 42.
[11] 柴新义, 陈双林. 青檀内生真菌菌群多样性的研究. 菌物学报, 2011, 30(1): 18- 26.
[12] 洪德元, 葛颂, 张大明, 汪小全, 程树志. 植物濒危机制研究的原理和方法 // 钱迎倩, 甄仁德. 生物多样性研究进展. 北京: 科学与技术出版社, 1995: 125- 133.
[15] 宋振巧, 王建华, 王洪刚, 王明明, 解玉丽. 山东丹参(Salviamiltiorrhiza)不同地理居群的遗传多样性. 生态学报, 2008, 28(11): 5370- 5376.
[16] 张杰, 王佳, 李浩宇, 张慧荣, 王迎春. 濒危植物蒙古扁桃不同地理种群遗传多样性的ISSR分析. 生态学报, 2012, 32(14): 4443- 4452.
[21] 李建华, 李雪松, 田春元, 邹礼平, 汪殿蓓. 大贵寺国家森林公园野生青檀居群的遗传多样性. 生态环境学报, 2011, 20(12): 1799- 1804.
[38] 葛永奇, 邱英雄, 丁炳扬, 傅承新. 孑遗植物银杏群体遗传多样性的ISSR分析. 生物多样性, 2003, 11(4): 276- 287.
[40] 陈俊秋, 慈秀芹, 李巧明, 李捷. 樟科濒危植物思茅木姜子遗传多样性的ISSR分析. 生物多样性, 2006, 14(5): 410- 420.
[41] 艾呈祥, 张力思, 魏海蓉, 金松南, 苑克俊, 刘庆忠. 山东实生板栗居群遗传多样性ISSR分析. 生物工程学报, 2007, 23(4): 628- 633.
[46] 邹喻苹, 葛颂, 王晓东. 系统与进化植物学中的分子标记. 北京: 科学出版社, 2001: 140- 149.
ThegeneticstructureofendemicplantPteroceltistatarinowiibyISSRmarkers
LI Xiaohong1,ZHANG Hui1,WANG Deyuan1, ZHANG Li, SHAO Jianwen1,2,*, ZHANG Xiaoping1,3
1CollegeofLifeScience,AnhuiNormalUniversity,Wuhu241000 2TheKeyLaboratoryofBioticEnvironmentandEcologicalSafetyinAnhuiProvince,AnhuiNormalUniversity,Wuhu241000 3TheKeyLaboratoryofConservationandEmploymentofBiologicalResourcesofAnhui,Wuhu241000
PteroceltistatarinowiiMaxim. (Ulmaceae), a tertiary relict plant of a temperate deciduous tree species endemic to China, is widely distributed in bare limestone mountains across the mainland in China. The bark (phloem fiber) of this plant has long since been used as the sole raw material for manufacturing Chinese traditional Xuan Paper. However, the species are subject to many threats due to its distribution pattern characterized by small patches; its decreasing population size resulted from overexploitation, and reduction of the original forest ecosystem. Thus, it has now been listed as a rare and endangered plant (National Grand III) in China.
Using inter-simple sequence repeat markers, the genetic diversity and structure of 628 individuals from 27 populations ofP.tatarinowiiwere detected. A total of 66 bands, of which 63 were polymorphic, were presented from the 8 selected primers screening across all samples, with the percentage of polymorphic bands up to 95.45%. The result of POPGENE revealed quite high level genetic diversity for the plant at the species level (PPB=95.45%,Ao=1.9545,Ae=1.5729,He=0.3335,I=0.4980). At the population level, TS population from Gansu harbored the highest genetic diversity (PPB=84.85%,Ao=1.8485,Ae=1.5217,He=0.3033,I=0.4516), whereas NL population from Guangdong with the lowest genetic variation (PPB=54.55%,Ao=1.5455,Ae=1.3135,He=0.1841,I=0.2756). The mean population genetic level (PPB=69.98%,Ao=1.6998,Ae=1.4449,He=0.2561,I=0.3793) and population genetic differentiation ofP.tatarinowii(Gst=0.23,ФST=0.25) were both at the middle level compared to other species. Gene flow (Nm) was estimated to be 1.65. In addition, different genetic variation and patterns were found between North China and South China. Populations of North China presented higher genetic diversity (PPB=95.45%,He=0.3332,I=0.4964) and lower genetic differentiation (ФST=0.22) than those of South China (PPB=93.94%,He=0.3220,I=0.4814 and ФST=0.25). It would seem the extant genetic pattern ofP.tatarinowiiwas might mainly attributed to its long evolutionary history, wide-ranging distribution, outcross mating system, long life cycle and complex differences of terrain and vegetation between North China and South China.
According to our aforementioned results and the evidence from our cpDNA data,insituconservation was the preferred way to maintain the species′ high level genetic diversity. Especially, much more attention should be paid to populations with higher genetic diversity (TS, JX and QY) and populations harboring peculiar cpDNA haplotypes (XA,SD,ML,NXS,LP,YF,WYS,GL,AL). In the condition of establishment of artificial plantation and germplasm bank, the above peculiar populations should been given prior consideration. Regarding the genetic pattern difference between South China and North China, more populations in South China and fewer representative populations with more individuals in North China should been sampled to obtain the utmost genetic diversity ofP.tatarinowii.
Pteroceltistatarinowii; inter-simple sequence repeat(ISSR); genetic diversity; genetic structure; conservation strategies
国家自然科学基金项目(30970292, 30840020)
2012- 12- 12;
2013- 06- 03
*通讯作者Corresponding author.E-mail: shaojw@mail.ahnu.edu.cn
10.5846/stxb201212191825
李晓红,张慧,王德元,张莉,邵剑文,张小平.我国特有植物青檀遗传结构的ISSR分析.生态学报,2013,33(16):4892- 4901.
Li X H,Zhang H,Wang D Y, Zhang L, Shao J W, Zhang X P.The genetic structure of endemic plantPteroceltistatarinowiiby ISSR markers.Acta Ecologica Sinica,2013,33(16):4892- 4901.
