福建省不产毒O1群霍乱弧菌分子特征分析
2013-11-20陈爱平李曲文倪二茹徐海滨杨劲松郑金凤严延生
陈爱平,李曲文,倪二茹,徐海滨,杨劲松,郑金凤,严延生
3.福建医科大学公共卫生学院教学基地,福州 350001
霍乱弧菌是导致人类霍乱疾病的病原菌,包括O1和O139血清群。霍乱肠毒素是主要的毒力基因,能够产生霍乱肠毒素的霍乱弧菌(产毒株)可以导致严重的腹泻疾病和霍乱流行。同时,在外环境中还大量存在不产生霍乱肠毒素的O1群和O139群菌株(非产毒株)[1]。福建省2009年、2011和2012年均监测到由非产毒的O1群霍乱弧菌引起的腹泻病例,同时陆续从相关的外环境中分离到不产毒的O1群菌株。为了解福建省近年从病人和外环境中分离到O1群不产毒的霍乱弧菌的内在关系,揭示相关毒力基因在不同来源菌株中的分布情况以及基因组多态性等分子特征,为今后霍乱防控工作提供分子流行病学依据,我们对这些不产毒O1群菌株的遗传背景进行了分析,现将结果报告如下。
1 材料与方法
1.1 菌株 不产毒的O1群霍乱弧菌来自2009-2012年福建省霍乱监测中分离到的病人和相关的外环境菌株。菌株信息见表1。
1.2 引物 毒力相关基因检测引物序列及相应的扩增条件和参考文献见表2,寡核苷酸由上海生工生物工程技术服务有限公司合成。
1.3 PCR反应条件 PCR反应均采用20μL的反应体系,各组成成分的终浓度分别为:Ex Taq DNA聚合酶0.5U,dNTPs 200μmol/L,上游引物0.4μmol/L,下游引物0.4 μmol/L,DNA模板2μL。
1.4 模板的制备 分纯的细菌菌落,直接刮取适量于300 μL的纯水制备成菌悬液,100。C煮沸裂解10min,10 000r/min离心5min,取上清作为PCR检测的DNA模板。
1.5 PFGE实验方法 根据PulseNet USA(美国CDC)网络实验室推荐的标准方法经国家CDC实验优化条件的基础上进行PFGE实验[6]。采用NotI限制性内切酶对实验菌株进行酶切,标准菌株H9812采用XbaI限制性内切酶消化,主要的电泳参数为电压梯度6V/cm,电泳夹角120°,电泳温度14。C;电泳参数:2s~10s、13h,20s-25s、6h。
1.6 数据分析 PFGE图像导入BioNumerics(version 6.0,Applied Maths,Inc)软件包进行处理,选择Dice相关系数和UPGMA方法,tolerance设置为1.5%。
1.7 试剂 Taq DNA聚合酶、dNTP购自大连宝生物工程有限公司,琼脂糖为BioAsia分装品,Seakem Gold Agarose PFGE级琼脂糖购自Lonza公司,XbaⅠ和NotⅠ限制性内切酶以及蛋白酶K购自美国NEB公司,BIO-RAD公司的Basic电泳仪、Gel Doc XR+读胶仪、CHEF MAPPER脉冲场凝胶电泳仪,PCR扩增仪为G-Storm(英国GeneTech公司),BioMerieux Vitek公司的DENSIMAT细菌浊度仪。
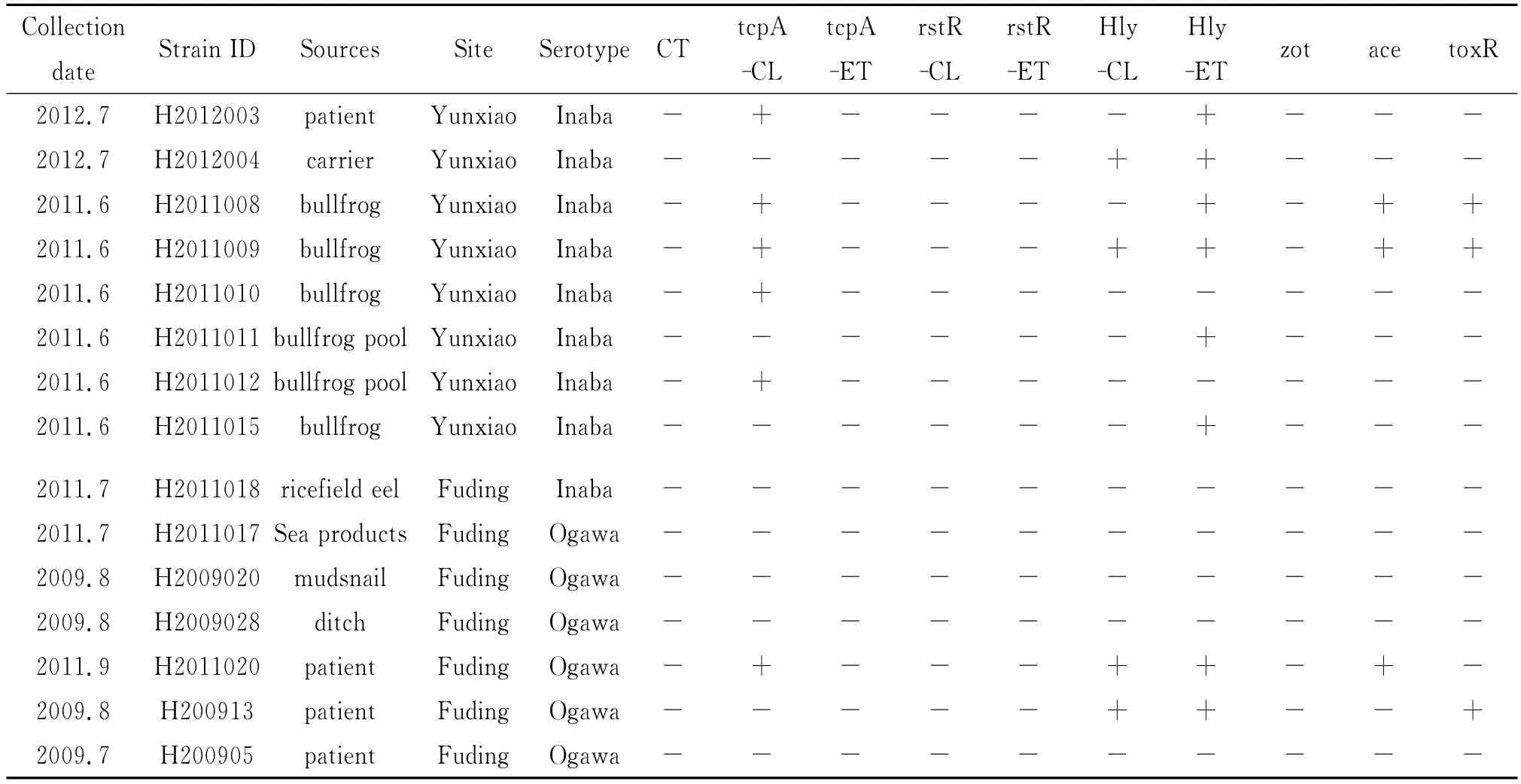
表1 15株O1群霍乱弧菌相关毒力基因检测结果Tab.1 Detection of virulence genes in 15O1-group V.cholerae strains
2 结 果
2.1 15株不产毒的霍乱弧菌毒力基因检测结果2009-2012年从病人和带菌者中分离到5株非产毒的O1群霍乱弧菌,同时从相关的外环境分离到10株菌,共15株非产毒株O1群霍乱弧菌,首先采用PCR扩增检测这些菌株是否携带以下7种毒力相关基 因,包 括 tcpA、rstR、hlyA、zot、ace、toxR、ctxA;其中tcpA、rstR、hlyA具有古典型和El Tor型两种基因,标记为tcpA-CL、tcpA-ET、rstR-CL、rstR-ET、hlyA-CL、hlyA-ET 均进行检测。检测发现各基因的阳检数分别为hly-ET(8)、tcpA-CL(6)、hly-Cl(4)、ace(3)、toxR(3),其余毒力基因均为阴性。详见表1。
2.2 PFGE分子分型结果 15株霍乱弧菌基因组DNA用NotⅠ酶切并进行脉冲场电泳后,DNA片段得到较好地分离,可见大小不一的电泳条带,分子量从20~400kb,条带数目在21~26之间。利用Bionumerics软件的Cluster analysis模块进行聚类分析,菌株间条带相似度在71.3%~100%之间;按照100%的相似度可将菌株酶切图谱分为12个PFGE型,其中P7、P10型和P12各有2株,其余各型均只有1株。根据TENOVER原则[7],只有1~3条带有差异的菌株可能有紧密相关性,按照90%的相似度将部分菌株酶切图谱划为同一个群,可得到2个主要的基因群G1-G2。见图1。

表2 O1群霍乱弧菌毒力相关基因检测引物序列Tab.2 Primer sequences for detection of virulence-associated genes in O1-group V.cholerae
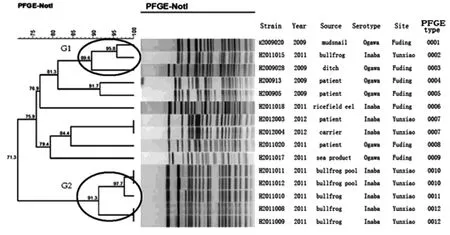
图1 15株不产毒霍乱弧菌NotI酶切聚类分析结果Fig.1 PFGE profile of Not I-digested genomes of the 15non-toxigenic O1-group V.cholerae strains
3 讨 论
全序列测定证实了霍乱弧菌的毒力基因有50多个,其中霍乱肠毒素是霍乱弧菌的主要毒力因子,由丝状噬菌体CTXФ编码,相邻的毒力基因还有zot、ace等毒力因子[8]。毒素协同调节菌毛 A(tcpA)是霍乱主要的定居因子,位于大染色体的VPI毒力岛上,菌毛的表达与ctxAB表达同受toxR基因的调控[1]。hlyA是溶血素基因,位于小染色体上。rstR基因是霍乱溶原性的丝状噬菌体CTXФ的调控基因,位于噬菌体核心区域之外。各种毒力因子广泛分散在大量的外环境菌株中,这些菌株的核糖体基因分型差异很大,分属于不同血清群,并具有不同的产毒能力。霍乱毒力基因的分布特性为毒力因子产生水平转移提供了环境毒力贮存库,成为霍乱持续流行的重要因素[9]。我们对近年来共15株非产毒的O1群霍乱弧菌进行上述毒力基因检测,结果发现这些毒力基因在病人株和环境株、不同血清型以及不同地区间的分布没有明显的差异。近几年福建省监测到5例散发的霍乱病例,患者分离株为霍乱肠毒素基因(ctxA)阴性,引起的症状都较轻,毒力相关基因检测表明这些不产毒的O1群霍乱病人菌株不同程度地携带有ctxA以外的其它毒力因子,说明其它致病因子也能够使不产毒菌株引起腹泻病。这些散发病例的影响范围有限,更没有导致大规模人群流行,表明这类菌株的致病力、复制和扩散传播能力较产毒株弱。
有研究表明近年来非典型埃尔托菌株(atypical El Tor strains)在东非、亚洲以及海地等地出现,主要特征表现为在埃尔托型基因组中杂合有部分古典型的相关基因,陆续发现了 Matlab变种(1991-1994年)、Mozambique变种(2004年)、突变的埃尔托型(2002年)以及杂交的埃尔托型(1991-2004年)等引起的局部流行,此类菌株的出现增加了霍乱流行的复杂性[10]。我们发现2009-2012年非产毒株O1群霍乱弧菌中,8株毒力相关基因阳性菌株里有6株菌同时携带有古典型和埃尔托型基因片段,说明非典型埃尔托型霍乱弧菌目前在福建省不产毒的O1群霍乱弧菌中较为常见。同时我们对福建省1962年以来从病人分离的产毒株进行研究,也发现此类杂合型的菌株较为广泛存在(未发表资料)。这些结果提示我们,应该密切关注这类菌株的出现对今后霍乱流行的影响。
随着PFGE技术的标准化以及PulseNet监测网络的建立,越来越多的公共实验室逐渐利用该技术平台进行各种细菌性病原体监测。我们对这些非产毒株O1群霍乱弧菌PFGE分子分型,结果发现按照100%的相似度,15个菌株存在有12个不同的PFGE型别,说明不产毒的O1群霍乱弧菌没有集中优势的PFGE型别,呈现基因组的多态性;同时病人株和外环境株并不存在100%相似的基因型别,说明所研究菌株间没有直接的传播关系。而H2012003和H2012004两个菌株PFGE分型100%一致,流行病学调查表明这两株菌分离自同一起疫情的病例和带菌者,说明PFGE分子分型技术能够在霍乱暴发或者流行时对共同暴露者进行确认,同时对疫情相关菌株进行溯源,因此具有实际的应用价值。
虽然这15株非产毒O1群霍乱弧菌的PFGE带型显示了这类菌株的基因组多样性,根据TENOVER原则[7]聚类仍然可以确定两个相对优势的基因群G1和G2。其中G2群的菌株分别来自同一年份和同一地区的牛蛙和牛蛙养殖池水,说明牛蛙及其养殖环境中可以存在由同一个克隆群组成的不产毒的霍乱菌株。由于丝状噬菌体CTXФ具有水平转移和溶原的性质,可以使不产毒的菌株转化为产毒株,应该加强对这类优势克隆群的监测,及时发现毒力基因的水平转移而引起的菌株变异,预防霍乱暴发流行。
[1]Reidl J,Klose KE.Vibriocholeraeand cholera:out of the water and into the host[J].FEMS Microbiol Rev,2002,26(2):125-139.
[2]Keasler SP,Hall RH.Detecting and biotypingVibriocholeraeO1with multiplex polymerase chain reaction[J].Lancet,1993,341(8861):1661.
[3]Singh DV,Isac SR,Colwell RR.Development of a hexaplex PCR assay for rapid detection of virulence and regulatory genes inVibriocholeraeandVibriomimicus[J].J Clin Microbiol,2002,40(11):4321-4324.DOI:10.1128/JCM.40.11.4321-4324.2002
[4]Chatterjee S,Ghosh SK,Raychoudhuri A,et al.Incidence,virulence factors,and clonality among clinical strains of non-O1,non-O139Vibriocholeraeisolates from hospitalized diarrheal patients in Kolkata,India[J].J Clin Microbiol,2009,47(4):1087-1095.DOI:10.1128/JCM.02026-08
[5]Bhowmick TS,Das M,Ruppitsch W,et al.Detection of virulence-associated and regulatory protein genes in association with phage typing of humanVibriocholeraefrom several geographical regions of the world[J].J Med Microbiol,2009,58(Pt 9):1160-1167.DOI:10.1099/jmm.0.008466-0
[6]Cooper KLF,Luey CKY,Bird M,et al.Development and validation of a PulseNet standardized pulsed-field gel electrophoresis protocol for subtyping ofVibriocholerae[J].Foodborne Pathog Dis,2006,3(1):51-58.DOI:10.1089/fpd.2006.3.51
[7]Tenover FC,Arbeit RD,Goering RV,et al.Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis:criteria for bacterial strain typing[J].J Clin Microbiol,1995,33(9):2233-2239.
[8]Waldor Mk,Mekalanos JJ.Lysogenic conversion by a filamentous phages encoding cholera toxin[J].Science,1996,272(5270):1910-1914.
[9]Clowell RR.Infectious disease and environment:cholera as a paradigm for waterborne disease[J].Int Microbiol,2004,7(4):285-289.
[10]Safa A,Nair GB,Kong RY.Evolution of new variants ofVibriocholeraeO1[J].Trends Microbiol,2010,18(1):46-54.DOI:10.1016/j.tim.2009.10.003
