野生大豆GsRNF12 基因的克隆及表达载体的构建
2013-10-16张红梅白云魏贤斌邓馨肖莉杰费志宏方淑梅韩毅强王北艳
张红梅 ,白云 ,2,魏贤斌 ,邓馨 ,肖莉杰 ,费志宏 ,方淑梅 ,韩毅强 ,王北艳
(1.黑龙江八一农垦大学生命科学技术学院,大庆 163319;2.大庆市质量技术监督局标准化所;3.黑龙江省八五三农场;4.中国科学院植物研究所;5.黑龙江八一农垦大学农学院)
中国野生大豆遗传资源数量多、类型丰富,受到世界大豆主产国高度重视。野生大豆具有极强的抗逆性和丰富的遗传多样性,为栽培大豆的亲缘较近的野生种,它是大豆育种极为重要的种质资源[1-2]。野生大豆资源研究开发利用,对拓宽栽培大豆的遗传基础,提高大豆育种水平具有重要价值。
大豆GmRNF12 基因属于锌指蛋白家族基因,对高盐、低温、金属胁迫具有较强的抗性[3]。但研究者对野生大豆的锌指蛋白家族基因研究较少。利用RT-PCR 技术扩增野生大豆GsRNF12 基因,并对基因编码的氨基酸进行序列比对分析,并构建了该基因的植物表达载体,为进一步研究该基因功能奠定基础。
1 材料与方法
1.1 材料
野生大豆材料由实验室收集。pGM-T vector 购自天根生化科技(北京)有限公司,大肠杆菌DH5α由实验室保存。
限制性内切酶、LA Taq 酶、T4-DNA 连接酶、RNA 提取试剂(RNAiso Reagent)、M-MLV 反转录酶(RNase H-)、引物 Oligo(dT)、2.5 mMdNTP、RNA 酶抑制剂、Protein Molecular Weight Marker(Low)、胶回收试剂盒均购自宝生物工程(大连)有限公司;引物由北京全式金生物公司合成,测序由上海生物工程公司完成。
1.2 方法
1.2.1 RNA 提取及cDNA 合成
利用TRIzol 试剂盒提取野生大豆叶片的总RNA,利用反转录试剂盒合成cDNA 第一链。
1.2.2 PCR 扩增
野生大豆与栽培大豆亲缘关系较近,因此根据GenBank 登录的栽培大豆的RNF12 基因全长设计特异引物,并在上下游引物的5′、3′端分别加AgeⅠ和ECORⅠ酶切位点序列,引物序列为:
5′-ACCGGTatgacgagcgcttcggagc-3′;
5′-C CGGAATTCttagttaattacaactatac-3′
在 25 μL 的反应体系中,dNTP 1 μL,上、下游引物各 1 μL ,模板 cDNA 1 μL,LA Taq 酶 0.5 μL,10 倍 buffer 2.5 μL(含 Mg2+),以 ddH2O 补至 25 μL。PCR 扩增条件为:94℃预变性3 min,94℃变性50 s,55 ℃ 50 s,72 ℃延伸 1 min,36 cycles,72 ℃延伸10 min。
1.2.3 目的片段克隆、测序分析
将 PCR 产物回收后,按 3∶1 比例与克体pMD18-T 连接。连接产物转化大肠杆菌DH5α感受态细胞,进行蓝白斑筛选,经PCR 及酶切鉴定含有阳性重组质粒的菌株送上海生物工程公司测序。将测序结果经 BLAST 比对,采用NCBI 的ORFfinder 确定该基因的开放阅读框,氨基酸多序列比对用DNAman 软件完成。
1.2.4 野大豆GsRNF12 基因的植物表达载体的构建
将重组质粒 pMDGsRNF12 和pEGAD 分别用AgeⅠ和ECORⅠ双酶切,回收目的片段和载体大片段,经T4 DNA 连接酶16℃过夜连接,连接产物转化DH5A 感受态细胞,在LB 培养基(含有10 μg ·mL-1的卡那霉素)上筛选重组子。挑取阳性克隆,经PCR检测和酶切鉴定验证重组质粒,命名为pEGADGsRNF12。
2 结果与分析
2.1 GsRNF12 基因 cDNA 序列的扩增及pMD GsRNF12 重组质粒PCR、酶切鉴定结果
根据大豆GmRNF12 基因ORF 序列设计特异引物,以野生大豆cDNA 为模板进行PCR 扩增,PCR扩增产物经1%琼脂糖凝胶电泳检测,大约在720 bp处出现特异性条带(图1),与克隆载体连接后,经AgeⅠ和ECORⅠ双酶切、PCR 鉴定后,与预期结果大小基本一致。测序结果表明,所得序列长度为723 bp,命名为 GsRNF12。

图1 质粒pMD-GsRNF12 的PCR 及酶切鉴定Fig.1 PCR amplication and restriction analysis of pMD-GsRNF12 plasmid
2.2 GsRNF12 基因生物信息学分析

图2 GsRNF12 ORF 序列及其所编码的氨基酸序列Fig.2 ORF nucleotide sequence and putative amino acid sequence of GsRNF12
野大豆基因GsRNF12 的cDNA 序列包含1 个完整的开放阅读框(1~723 bp),对GsRNF12 基因编码蛋白的结构进行分析,编码239 个氨基酸中(图2),预测其等电点为6.23,该蛋白理论分子量为26.807 kDa。利用NCBI 对氨基酸进行保守区域分析,GsRNF12 在178-220 氨基酸处有典型的C4HC3 结构域,属于C3HC4 锌指蛋白家族,利用PSORT 软件分析GsRNF12 蛋白存在细胞核中。利用DNAman 软件对野大豆、栽培大豆、苜蓿的RNF12 氨基酸序列进行分析,发现该基因的氨基酸序列与大豆、苜蓿同源性>80%。
2.3 GsRNF12 基因植物表达载体的构建
目的片段与表达载体连接、转化后,对筛选出的阳性克隆进行PCR 和AgeⅠ、ECORⅠ双酶切鉴定,1%琼脂糖凝胶电泳检测到约730 的目的带(图3),结果表明该片段大小正确,说明目的片段插入的位置和方向正确,成功构建出了植物表达载体pEGAD-GsRNF12。(图 4)。
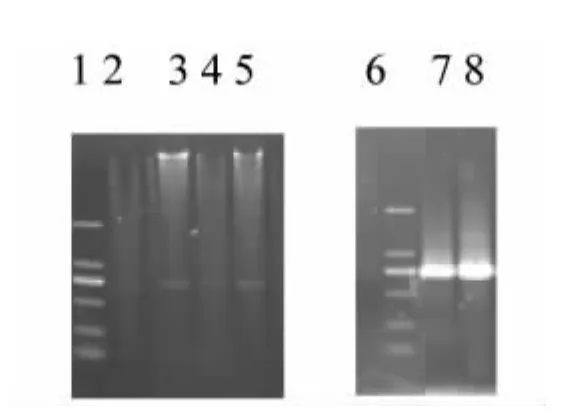
图3 植物表达载体pEGAD-GsRNF12 酶切、PCR 切鉴定Fig.3 Identification of pEGAD-GsRNF12 plant express ion vector by PCR amplification and enzyme digestion

图4 植物表达载体pEGAD-GsRNF12 的鉴定Fig.4 Identification of plant expression vector pEGAD-GsRNF12
3 讨论
锌指蛋白在真核生物基因组中十分丰富,根据半胱氨酸(C)和组氨酸(H)残基的数目和位置,将锌指结构分为 C2C2、C2H2、C3 H 2C3、C3 HC4(RI NG finger)4 个亚类[5]。针对非生物逆境的胁迫,通过一系列逆境信号分子,植物形成了非常复杂而完善的调控网络,锌指蛋白基因家族在此调控网络中,发挥着非常重要的调节作用[6]。许多研究表明,锌指蛋白在调节植物防卫基因表达和抗逆性起关键作用。研究利用优化的PCR 反应体系和反应,我们克隆到了野生大豆锌指蛋白命名为GsRNF12 基因cDNA 全长,属于C3HC4 型锌指蛋白基因。构建了该基因的植物表达载体,为下一步研究该基因功能奠定了基础。
[1] 张春宝,赵洪锟,李启云.野生大豆-吡咯琳-5-羧酸合成酶(P5CS)基因的克隆与序列分析[J].大豆科学,2008,27(6):915-920.
[2] 王丹阳,吴铭.对黑龙江省野大豆保护与利用的研究[J].林业勘察设计,2011(1):85-86.
[3] 张红梅.铝胁迫下大豆差异基因表达及GmRNF12、GmFDR3 基因功能分析[D].长春:吉林大学,2011.
[4] 韩毅强,张云超,张红梅.NaCl 胁迫对大豆种子萌发及其生理变化的影响[J].黑龙江八一农垦大学学报,2011,23(4):11-14.
[5] 刘强,张贵友,陈受宜.植物转录因子的结构与调控作用[J].科学通报,2000,45(14):1465-1474.
[6] Mukhopadhyay A,Vij S,Tyagi A K.Overexpression of a znic finger protein gene from rice confers tolerance to cold,dehydration and salt stress in transgenic tobacco[J].PNAS ,2004,101(16):6309-6314.
