基于机器学习方法的H1N1神经氨酸苷酶抑制剂的分类预测
2013-09-21孟庆伟
吕 巍 薛 英 孟庆伟,*
(1山东农业大学生命科学学院,作物生物学国家重点实验室,山东泰安271018;2山东农业大学生物学博士后科研流动站,山东泰安271018;3四川大学化学学院,教育部绿色化学与技术重点实验室,成都610064;4四川大学生物治疗国家重点实验室,成都610041)
1 引言
流行性感冒病毒(influenza virus)简称流感病毒,而流感是严重危害人类健康的一种急性病毒性呼吸道感染疾病,传播速度快,是导致人类死亡的主要病因之一.甲型H1N1流感病毒是流感病毒的一种,其外部的糖蛋白层含有血凝素(hemagglutinin,HA)和神经酰胺酶(nueraminidase,NA),对于流感病毒的入侵和传播起着非常重要的作用,从而为抗流感病毒的研究提供了两个可能的药物靶点.在发现神经酰胺酶抑制剂之前,流感治疗仅限于金刚烷胺、金刚乙胺和病毒疫苗等方法.1研究发现神经酰胺酶在H1N1病毒的复制、感染和致病过程中起重要作用,抑制神经酰胺酶活性,可以有效地控制H1N1型流感病毒感染引起的流感及其传播.2-5因此,以神经酰胺酶为靶点,筛选神经酰胺酶抑制剂,是研究和开发抗H1N1病毒药物的一个重要途径.6-9
机器学习(machine learning)是一门人工智能的科学,机器学习方法使用计算机程序来模拟人类的学习活动,它研究通过计算机程序来获取新知识和新技能、并识别现有知识,以数据或以往的经验优化计算机程序的性能.机器学习方法作为一种重要的计算方法和高效的计算工具,已经在计算机辅助药物设计和虚拟筛选中得到广泛的应用.它在药效团模拟、药代动力学计算和药物毒性预测等方面的研究中取得了巨大的成效.10-12现阶段得到广泛应用的机器学习方法主要有支持向量机(SVM)、k-最近相邻法(k-NN)和C4.5决策树(C4.5 DT).在化学分布多样性的小分子化合物的预测中,机器学习方法已经展示出其独特的效果.机器学习方法的预测精度与适当的分子描述符集密切相关,这些描述符在一定程度上代表了被预测化合物的物理化学性质.递归变量消除(REF)法13,14是一种被广泛应用的性质选择方法,可以通过优化预测化合物的性质集,从而进一步提高预测精度.另外在本研究中,我们使用两种方法对预测精度进行评价,包括五重交叉验证法和独立验证集法.
2 材料与方法
2.1 神经酰胺酶抑制剂的选取
本研究共选取237个NA抑制剂和非抑制剂(附表1,见Supporting Information),它们分别来自文献.15-25研究表明,当实验半数抑制浓度(IC50)值低于500 nmol·L-1时,化合物有较好抑制活性,而IC50值大于500 nmol·L-1时化合物基本无抑制活性,当化合物IC50值在500 nmol·L-1附近时,化合物有抑制活性但并不明显.26,27因此将237个化合物按其IC50值分成三个数据集:72个NA抑制剂(IC50≤400 nmol·L-1),155个非NA抑制剂(IC50≥600 nmol·L-1),10个化合物抑制活性非常弱(400 nmol·L-1<IC50<600 nmol·L-1).化合物二维分子结构由ChemDraw软件28绘制,并由Corina软件29将其转化为三维结构.
我们首先将所有化合物随机分为五个亚集进行交叉验证,以其中四个亚集作为SVM方法的训练集,剩余一个亚集则被用作测试集被SVM模型测试,这样重复进行五次测试,每个亚集都作为测试集被使用一次,最终得到五次预测结果的平均值.然后根据化合物的结构与化学性质在化学空间中的分布,将所有的化合物分为训练集、测试集与独立验证集(见附表2).训练集和测试集被用于开发和优化分类预测模型,独立验证集被用于评价分类模型的预测能力.
2.2 分子描述符的计算
分子描述符是一个分子的数学表征,可以定量表示分子的结构与物理性质,其在构效关系(SAR)、13定量构效关系(QSAR)30以及药效团模型31,32等相关研究领域得到广泛应用.在我们的研究中,通过手工方法,从相关文献中找出一千多个分子性质描述符,并根据化合物拓扑结构、电子结构和几何结构等性质,从中筛选出189个与化合物性质密切相关的描述符(见附表3).其中包括了18个简单分子性质描述符、27个分子连接和形状描述符、97个电拓扑态描述符、22个量子化学性质描述符及25个几何性质描述符.应用分子描述符计算程序,33通过化合物的三维结构计算所有分子描述符,并通过性质选择方法进一步筛选与NA抑制剂和非抑制剂最为相关的描述符.34
2.3 性质选择方法
研究显示,使用性质选择方法来消除数据集中多余描述符有助于提高机器学习方法的预测精度.递归特征消除(RFE)法是一种广泛应用并能高效发现相关特征的性质选择方法,它已在癌症的分类预测,35四膜虫毒性化合物预测36等领域获得广泛应用.本文使用递归性质消除法进行描述符的选择,通过与支持向量机相结合,获得更有利于预测NA抑制剂的分子描述符数据集,从而提高模型的预测精度.
RFE程序简述如下:用包含可调节参数σ的高斯核函数建立一个SVM分类系统,通过五重交叉验证来评定模型的预测能力.对一个确定的σ,第一步,用全部描述符建立SVM预测模型;第二步,通过打分函数计算当前描述符集中每个描述符对预测模型的贡献分值,并按降序排列;第三步,删除打分最低的m个描述符;第四步,用剩下的描述符重新训练SVM分类模型,相应的预测精度通过五重交叉法来计算.整个过程不断重复,直到剩下的描述符不多于m个.对其它的参数σ,重复同样的过程.预测总精度最高的模型为最优化模型,该模型所使用的描述符即为RFE选出的描述符.
m的选择影响到SVM的表现和变量选择的速度.每次消除一个变量对计算能力的要求太高,在某些情况下,每次消除多个变量显著提高计算速度,且对精度的影响不大.本课题组以前的工作表明,m=5相对于m=1,SVM体系的预测精度只有很小的损失.26,27,33考虑到计算效率,本研究选择m=5,另外,为了限制选出描述符的数量并避免过拟合,我们控制选出描述符的数量小于数据集个数的五分之一.37
2.4 机器学习方法
现在有大量的机器学习方法软件包可从网上免费下载,例如:PHAKISO(http://www.phakiso.com/index.htm)和 WEKA(http://www.cs.waikato.ae.nz/~ml/weka)包含了许多机器学习的方法,38NeuNet(http://www.cormactech.com/neunet/index.html)是关于神经网络方面的软件包,SVM-Light(http://svmlight.joachims.org)是应用很广泛的支持向量机方面的软件包.本研究中我们用自己建立的SVM模型33预测NA抑制剂与非抑制剂,同时用其它的机器学习方法(k-NN、C4.5)39,40进行预测,并对结果进行比较.
2.5 预测结果评价
像其它预测模型一样,41我们采用以下公式判断模型的预测精度.

其中TP,TN,FP,FN分别代表预测正确的NA抑制剂,预测正确的非NA抑制剂,预测错误的NA抑制剂以及预测错误的非NA抑制剂;SE用于评估NA抑制剂的预测正确率;SP用于评估非NA抑制剂的预测正确率;Q用于评估总体的预测正确率;C为马氏相关系数.
3 结果与讨论
3.1 机器学习方法对NA抑制剂的预测精度
我们通过五重交叉验证法对预测模型进行验证,所有的预测结果在表1中列出.通过性质选择方法,有19个描述符被选出,认为这19个描述符对NA抑制剂的分类预测起到至关重要的作用(表2).从中可见,当不使用性质选择方法时,SVM对NA抑制剂和非抑制剂的预测精度分别为40.5%和94.5%,平均预测精度为78.0%;当使用性质选择方法RFE时,SVM对NA抑制剂和非抑制剂的预测精度分别为72.0%和85.0%,平均预测精度为81.2%.由此可见,通过性质选择之后抑制剂的预测精度明显提高,虽然非抑制剂预测精度有所下降,但平均预测精度要高于前者.另外可以看出,当使用全部描述符进行预测时,抑制剂的预测结果非常低,与非抑制剂的预测结果相差太大.因此性质选择方法不但提高了抑制剂的预测结果,同时也提高了总的预测结果,由此可见性质选择方法更有利于提高支持向量机模型的预测精度,尤其是对抑制剂的预测精度.另外应用19个所选描述符不但降低支持向量机模型的过拟合现象以及多余描述符产生的噪音,提高预测精度,更重要的是显示了与NA抑制剂和非抑制剂相关的一些物理化学性质.
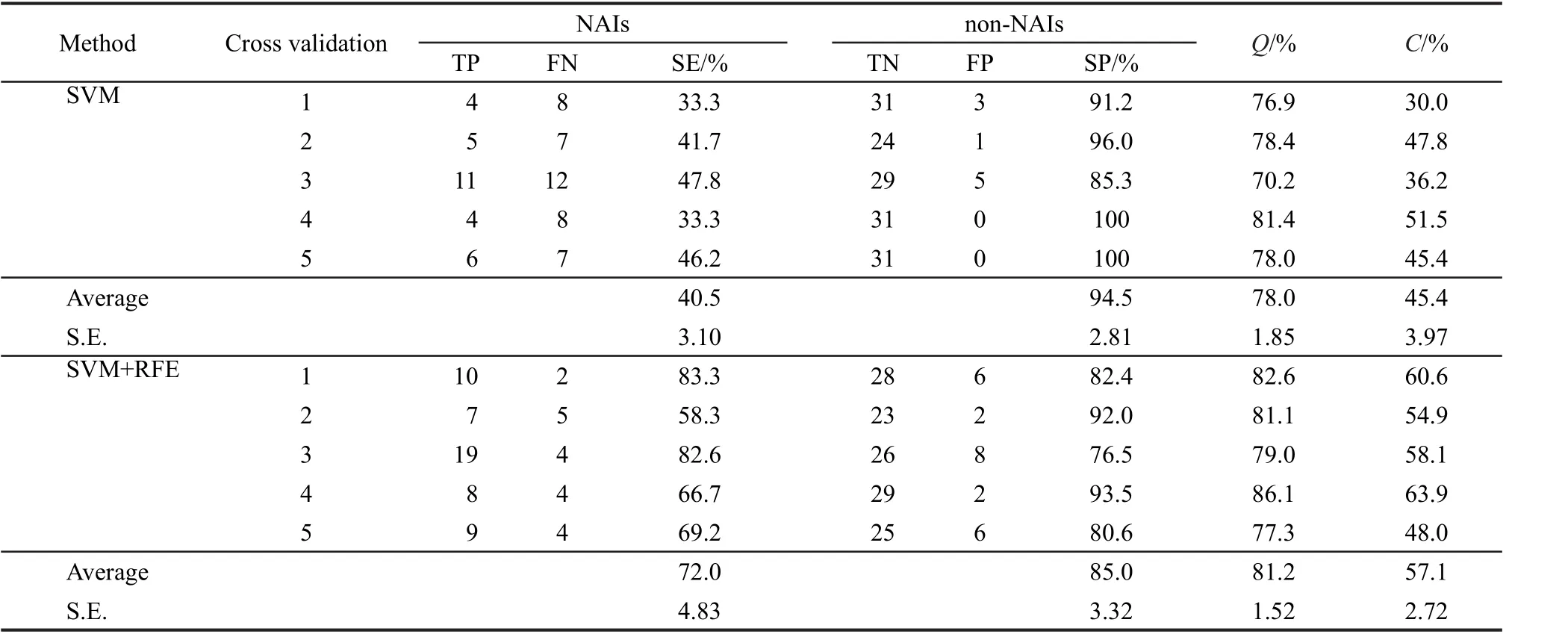
表1 SVM方法对NA抑制剂和非抑制剂的五重交叉验证aTable 1 Performance of SVM for predicting NAIs and non-NAIs as evaluated by five-fold cross validationa

表2 性质选择方法选出的19个与NA抑制剂分类预测相关的分子描述符Table 2 19 molecular descriptors selected from the RFE feature selection method for the classification of NAIs and non-NAIs
除了五重交叉验证法之外,独立验证集的方法也频繁地被使用在分类模型的预测上.在本论文中,根据化学空间,我们得到一个以14个NA抑制剂和40个NA非抑制剂构成的独立验证集(见附表2),通过k-NN、C4.5 DT和SVM的方法,用19个描述符对独立验证集进行了预测,结果见表3.如表所示,对NA抑制剂,其预测精度为64.3%-78.6%,其中SVM给出最好的预测结果为78.6%;对NA非抑制剂,其预测精度为77.5%-97.5%,其中C4.5 DT和SVM都给出最好的预测结果为97.5%;总预测精度为75.9%-92.6%,其中SVM给出最好的预测结果为92.6%,由此可见SVM对NA抑制剂的预测可以提供更精确的结果.

表3 不同机器学习方法对NA抑制剂和非抑制剂的独立验证集的预测Table 3 Comparison of the prediction accuracies of NAIs and non-NAIs derived from different machine learning methods by using independent validation sets
通过比较交叉验证方法和独立验证方法的预测结果,可以检查预测模型是否出现过拟合现象.由于描述符的选择是通过交叉验证测试集得到的,而过拟合的实际表现就是测试集的预测精度远高于独立验证集的预测精度.但是,通过表1和表3可以看出,测试集的预测精度稍微低于独立验证集的预测精度,但差别不大,因此证明我们的预测模型并没有出现过拟合现象.
3.2 神经酰胺酶抑制剂相关的分子描述符
选择与NA抑制剂相关的分子描述符是非常重要的,它不但可以对预测模型进行优化以提高预测精度,而且可以进一步阐明NA抑制剂的作用机理.在我们的研究中,通过递归性质消除的变量选择方法总共选择了19个与NA抑制剂最为相关的分子描述符,见表2.在这19个描述符中,包括4个分子连接与形状描述符,7个电拓扑态描述符和8个几何性质描述符.可见,电拓扑态和几何性质描述符在分类过程中起到重要作用.
3.3 预测错误的化合物
在我们的实验中通过独立验证集的SVM预测,总共有4个化合物预测错误.其中NA抑制剂的预测精度为78.6%,NA非抑制剂的预测精度为97.5%,平均预测精度为92.6%.14个NA抑制剂中有3个化合物被预测成NA非抑制剂,另一方面,40个NA非抑制剂中有1个化合物被预测成抑制剂.所有预测错误的分子见图1和图2,图1中的3个分子是NA的抑制剂,但通过预测得到的结果是非抑制剂;图2中的1个化合物是NA的非抑制剂,而预测模型将其预测为抑制剂.从图中我们可以看出预测错误的分子主要是一些含环化合物.预测错误的NA抑制剂都含有两个环,而预测错误的NA非抑制剂含有多个环.从这些预测错误的分子可以看出,当前使用的描述符还不足以正确地预测分辨那些带多环结构的化合物.因此我们应该探索研究新的描述符,并通过性质选择方法来筛选更适合机器学习方法模型的描述符.
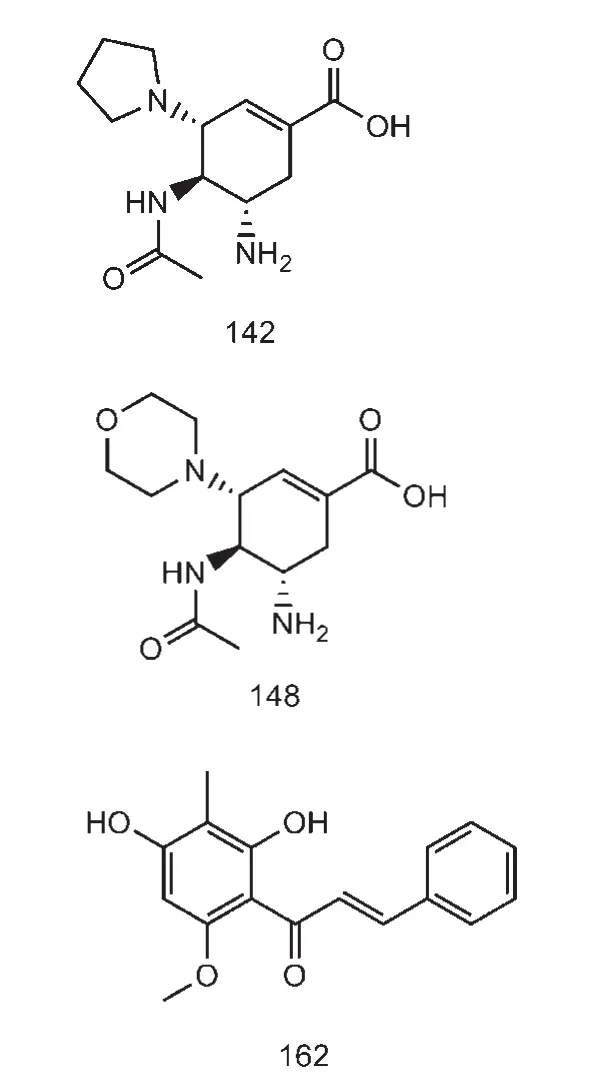
图1 预测错误的神经氨酸苷酶抑制剂Fig.1 Structures of the part of misclassified NAIs
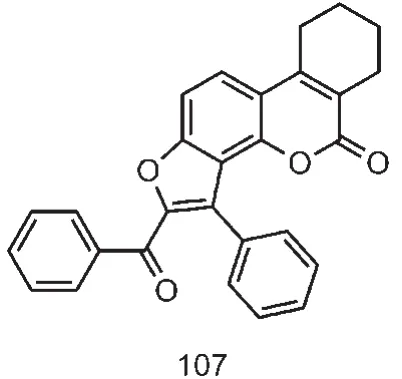
图2 预测错误的非神经氨酸苷酶抑制剂Fig.2 Structures of the part of misclassified non-NAIs
4 结论
研究显示,对于NA抑制剂,当其分子作用机理不明确时,可以用机器学习方法尤其是支持向量机方法对其预测,可以得到较高的预测精度.但我们也发现,由于描述符的限制,当前机器学习方法对化合物作用机理的预测能力还受到一定的制约;另外,由于预测样本的数量较少,不能够全面覆盖预测化合物,尤其是NA非抑制剂,因此造成预测结果与实际值有一定差距.不过我们相信在不久的将来,随着回归为基础的机器学习方法的不断发展,这些问题一定会被克服.总之在抗流感药物研发前期,如果能够更为广泛地了解NA抑制剂的相关信息与作用机理,将能够进一步提高机器学习方法对其抑制剂的预测精度,从而使机器学习方法在药物研发过程的前期成为预测NA抑制剂的更为实用的工具.
Supporting Information: available free of charge via the internet at http://www.whxb.pku.edu.cn.
(1) Erik,D.C.J.Clin.Virol.2001,22,73.doi:10.1016/S1386-6532(01)00167-6
(2) Palese,P.;Tobita,K.;Ueda,M.Virology 1974,61,397.doi:
10.1016 /0042-6822(74)90276-1
(3)Moscona,A.N.Eng.J.Med.2005,353,1363.doi:10.1056/NEJMra050740
(4) Erik,D.C.Nat.Rev.Drug.Disc.2006,5,1015.doi:10.1038/nrd2175
(5) Schmidt,A.C.Drugs 2004,64,2031.doi:10.2165/00003495-200464180-00003
(6) Suzuki,Y.;Sato,K.;Kiso,M.;Hasegawa,A.Glycoconjugate J.1990,7,349.doi:10.1007/BF01073378
(7) Hagiwara,T.;Kijima-Suda,I.;Ido,T.;Ohrui,H.;Tomita,K.Carbohydr.Res.1994,263,167.doi:10.1016/0008-6215(94)00133-2
(8)White,C.L.;Janakiraman,M.N.;Laver,W.G.;Philippon,C.Vasella,A.;Air,G.M.;Luo,M.J.Mol.Biol.1995,245,623.doi:10.1006/jmbi.1994.0051
(9) Meindl,P.;Bodo,G.;Palese,P.;Schulman,J.;Tuppy,H.Virology 1974,58,457.doi:10.1016/0042-6822(74)90080-4
(10)Mitchell,T.Machine Learning;McGraw-Hill:New York,1996.
(11) Kohavi,R.;John,G.H.Artif.Intell.1997,97,273.doi:10.1016/S0004-3702(97)00043-X
(12) Leach,A.R.;Gillet,V.J.An Introduction to Chemoinformatics;Springer:Heidelberg,2007;p 82.
(13)Yu,H.;Yang,J.;Wang,W.;Han,J.Proc.IEEE 2003,220.
(14) Furlanello,C.;Serafini,M.;Merler,S.;Jurman,G.Neural Networks 2003,16,641.doi:10.1016/S0893-6080(03)00103-5
(15)Lew,W.;Wu,H.W.;Mendel,D.B.;Escarpe,P.A.;Chen X.W.;Laver,W.G.;Graves,B.J.;Kim,C.U.Bioorg.Med.Chem.Lett.1998,8,3321.doi:10.1016/S0960-894X(98)00587-3
(16)Sun,C.W.;Huang,H.;Feng,M.Q.;Shi,X.L.;Zhang,X.D.;Zhou,P.Bioorg.Med.Chem.Lett.2006,16,162.doi:10.1016/j.bmcl.2005.09.033
(17)Wen,W.H.;Wang,S.Y.;Tsai,K.C.;Cheng,Y.S.E.;Yang,A.S.;Fang,J.M.;Wong,C.H.Bioorg.Med.Chem.2010,18,4074.doi:10.1016/j.bmc.2010.04.010
(18)Yeh,J.Y.;Coumar,M.S.;Horng,J.T.;Shiao,H.Y.;Kuo,F.M.;Lee,H.L.;Chen,I.C.;Chang,C.W.;Tang,W.F.;Tseng,S.N.;Chen,C.J.;Shih,S.R.;Hsu,J.T.A.;Liao,C.C.;Chao,Y.S.;Hsieh,H.P.J.Med.Chem.2010,53,1519.doi:10.1021/jm901570x
(19)Lew,W.;Wu,H.W.;Chen,X.W.;Graves,B.J.;Escarpe,P.A.;MacArthur,H.L.;Mendel,D.B.;Kim,C.U.Bioorg.Med.Chem.Lett.2000,10,1257.doi:10.1016/S0960-894X(00)00214-6
(20) Dao,T.T.;Tung,B.T.;Nguyen,P.H.;Thuong,P.T.;Yoo,S.S.;Kim,E.H.;Kim,S.K.;Oh,W.K.J.Nat.Prod.2010,73,1636.doi:10.1021/np1002753
(21) Kolocouris,N.;Kolocouris,A.;Foscolos,G.B.;Fytas,G.;Neyts,J.;Padalko,E.;Balzarini,J.;Snoeck,R.;Andrei,G.;Clercq,E.D.J.Med.Chem.1996,39,3307.doi:10.1021/jm950891z
(22) Brouillette,W.J.;Bajpai,S.N.;Ali,S.M.;Velu,S.E.;Atigadda,V.R.;Lommer,B.S.;Finley,J.B.;Luo,M.;Aird,G.M.Bioorg.Med.Chem.2003,11,2739.doi:10.1016/S0968-0896(03)00271-2
(23)Liu,A.L.;Wang,H.D.;Lee,S.M.Y.;Wang,Y.T.;Du,G.H.Bioorg.Med.Chem.2008,16,7141.doi:10.1016/j.bmc.2008.06.049
(24)Williams,M.A.;Lew,W.;Mendel,D.B.;Tai,C.Y.;Escarpe,P.A.;Laver,W.G.;Stevens,R.C.;Kim,C.U.Bioorg.Med.Chem.Lett.1997,14,1837.
(25) Zhang,L.J.;Williams,M.A.;Mendel,D.B.;Escarpe,P.A.;Kim,C.U.Bioorg.Med.Chem.Lett.1997,14,1847.
(26) Lv,W.;Xue,Y.Eur.J.Med.Chem.2010,45,1167.doi:10.1016/j.ejmech.2009.12.038
(27) Lü,W.;Xue,Y.Acta Phys.-Chim.Sin.2010,26,471.[吕 巍,薛 英.物理化学学报,2010,26,471.]doi:10.3866/PKU.WHXB20100125
(28) ChemDraw,Version 9.0;Cambridge Soft Corporation:Cambridge,USA,2004.
(29)Corina,Version 3.4;Molecular Networks GmbH Computerchemie:Erlangen,Germany,2006.
(30) Hasegawa,K.J.Chem.Inf.Comput.Sci.1999,39,112.doi:10.1021/ci980088o
(31) Byvatov,E.;Fechner,U.;Sadowski,J.;Schneider,G.J.Chem.Inf.Comput.Sci.2003,43,1882.doi:10.1021/ci0341161
(32) He,L.;Jurs,P.C.;Custer,L.L.;Durham,S.K.;Pearl,G.M.Chem.Res.Toxicol.2003,16,1567.doi:10.1021/tx030032a
(33) Lü,W.;Xue,Y.Acta Phys.-Chim.Sin.2011,27,1407.[吕 巍,薛 英.物理化学学报,2011,27,1407.]doi:10.3866/PKU.WHXB20110608
(34)Yang,X.G.;Lv,W.;Chen,Y.Z.;Xue,Y.J.Comput.Chem.2009,31,1249.
(35)Lin,H.H.;Han,L.Y.;Yap,C.W.;Xue,Y.;Liu,X.H.;Zhu,F.;Chen,Y.Z.J.Mol.Graph.Model.2007,26,505.doi:10.1016/j.jmgm.2007.03.003
(36)Xue,Y.;Li,H.;Ung,C.Y.;Yap,C.W.;Chen,Y.Z.Chem.Res.Toxicol.2006,19,1030.doi:10.1021/tx0600550
(37) Degroeve,S.;de Baets,B.;van de Peer,Y.;Rouze,P.Bioinformatics 2002,18,S75.
(38) Garner,S.R.Weka,version 3.4.12;University of Waikato:New Zealand,2005.
(39) Johnson,R.A.;Wichern,D.W.Applied Multivariate Statistical Analysis;Prentice Hall:New York,1982.
(40) Quinlan,J.R.C4.5,Programs for Machine Learning;Morgan Kaufmann:San Mateo,CA,1992.
(41) Baldi,P.;Brunak,S.;Chauvin,Y.;Andersen,C.A.;Nielsen,H.Bioinformatics 2000,16,412.doi:10.1093/bioinformatics/16.5.412
