马源禽I型副黏病毒HN基因的克隆与序列分析
2013-09-11梁丹洁李春英蒋家霞张欣明孙翔翔曾咏芳周师师
梁丹洁,欧 莹,李春英,蒋家霞,张欣明,孙翔翔,曾咏芳,周师师,吴 军,熊 毅
(1.广西大学动物科学技术学院,广西南宁 530004;2.广西区动物疫病预防与控制中心,广西南宁 530004)
禽I型副黏病毒(Avain Paramyxovirus I,APMV-I)属于副黏病毒科,副黏病毒亚科、腮腺病毒属,其基因组为无节段的负链单股RNA病毒,大小约为15kb,主要编码6种蛋白质[1],其中HN蛋白在病毒脂囊膜上形成纤突,主要负责细胞受体和病毒粒子之间的结合,以破坏这种结合的拮抗作用,它还通过促使F蛋白充分接近细胞,引起病毒囊膜与细胞融合,从而进一步启动感染过程[2]。有文献研究表明,HN的基因突变可能导致具有不同致病性的APMV-I变异株的不断产生,HN对毒力也有一定的影响[3]。近年来,APMV-Ⅰ宿主范围的不断扩大,不仅打破了“APMV-Ⅰ不感染水禽或感染不发病”的传统理论[4-5],而且还从最初的禽类宿主到现在的哺乳动物跨种间传播,感染谱的不断扩大,对人类公共卫生也存在潜在的威胁。本研究对从马身上分离获得M10株的HN基因进行克隆测序,并与已发表的HN基因序列进行同源性分析、绘制系统进化树,旨在从分子生物学水平为该病的预防和控制提供理论依据,并希望能够在分子水平上找出导致APMV-I病毒跨越种属宿主改变的可能原因,从而为同源或近源跨越种属引起人类疾病的病原体的研究提供参考依据。
1 材料与方法
1.1 主要仪器与试剂
总RNA抽提试剂盒、Marker 2000、DH5a大肠埃希菌感受态细胞、质粒抽提取试剂盒购自天根公司;胶回收试剂盒、ExTaqDNA 聚合酶、M-MLV反转录酶、HIR抑制酶、dNTPs、Mgcl2、PMD18-T质粒载体、限制性内切酶等购自大连宝生物有限公司。PCR反应仪、超速离心机、移液器等。
1.2 病毒株
马源禽I型副黏病毒M10株由广西动物疫病预防控制中心家畜诊断科分离、鉴定。
1.3 HN基因的克隆与测序
1.3.1 引物的合成与设计
根据GenBank中已发表的鹅副黏病毒GPVSF02株全基因组序列,通过Oligo软件设计了扩增HN基因的2对上下游引物(表1),引物由大连宝生物Takara公司合成。将其稀释成25 pmol/μL,-20 ℃分装,备用。

表1 HN基因扩增引物序列
1.3.2 病毒RNA的抽提
取病毒液按照天根公司总RNA抽提试剂盒说明书进行抽提取RNA。
1.3.3 RT-PCR反转录聚合酶链式反应
将上述抽提取所得的RNA为模板,HN1、HN2下游引物为反转录引物,合成第一链cDNA。取5μL cDNA并进行下一步PCR反应,PCR反应循环体系为:95 ℃预变性5 min;94 ℃变性50 s,55 ℃退火50 s,72 ℃延伸1 min,总共38个循环;72 ℃延伸10 min。反应后的PCR产物可以直接进行琼脂糖凝胶电泳。
1.4 目的基因的胶回收、连接、酶切鉴定以及克隆测序
胶回收按照Takara胶回收试剂盒进行。按DNA连接试剂盒说明书将回收纯化后的目的基因与PMD18-T载体连接,置4℃过夜。然后将连接产物转化至感受态细胞DH5ɑ中,涂布于添加有Amp(100 μg/mL)的LA培养基上,37℃培养12-16h,挑取上述生长良好的白色菌落,接至含有Amp(100 μg/mL)的LB液体培养基,37℃摇床培养16 h 。取1.0-1.5 ml培养物提取重组质粒,进行双酶切及质粒PCR鉴定,筛选出两者均为阳性的菌株送大连宝生物工程有限公司进行测序。
1.5 HN基因序列分析
根据M10的HN基因5’端与3’ 端cDNA的核苷酸序列测定结果,将HN基因5’端与3’端的核苷酸序列进行拼接,并将完整的HN基因序列通过NCBI blast与GeneBank中收录的APMV-I核苷酸序列进行类似性检索。用DNAStar.Lasergene.v7软件来分析其与参考株的同源性以及构建基因进化树。
2 结果
2.1 HN基因扩增结果
PCR扩增产物经1.0%琼脂糖凝胶电泳分析表明,利用鹅副黏病毒HN基因的特异性引物扩增出长度分别为1226、1202bp左右的2条带,与预期结果相符。
2.2 目的基因的重组质粒鉴定结果
本毒株HN基因的2个片段重组质粒用(Sal I和EcoRI双酶切),可分别见1226bp,2650bp;1202bp,2650bp左右的两个片段,分别为目的基因和pMD18-T载体,与预期结果相符。
2.3 马源禽I副黏病毒HN全基因各片段的克隆和测序
筛选出阳性克隆后进行序列测定,运用DNAStar软件包中SeqMan程序对所得序列进行拼接,该毒株HN基因约为2.0kb,ORF为1716个碱基,编码571个氨基酸。
2.4 HN基因的编码区序列和其推导的氨基酸序列同源性比较
利用DNAStar中Megalign程序对M10株的HN基因编码区与从Genbank下载的基因序列进行同源性分析比较可得:其编码区核苷酸序列的同源性在80.9-99.2%之间。其中与鸡源YG03(DQ228931.1)、NDV-03-044(GQ33831.1) 同源性最高,分别为99.2%与97.3 %;而与鸡源经典毒株B1(AF309418)、Lasota(AF077761)、强毒株F48E9(AY997298.1)同源性最低,分别为80.9%、81.2%、84.4%;与鸭源、鹅源、鹦鹉源、海鸥源、野鸟源相比较,同源性在91.4%~95.9%之间。而由M10株编码区推导的氨基酸序列与其他毒株相比,同源性在88.6%~98.6%之间(表2)。
2.5 编码区核苷酸序列进化树分析
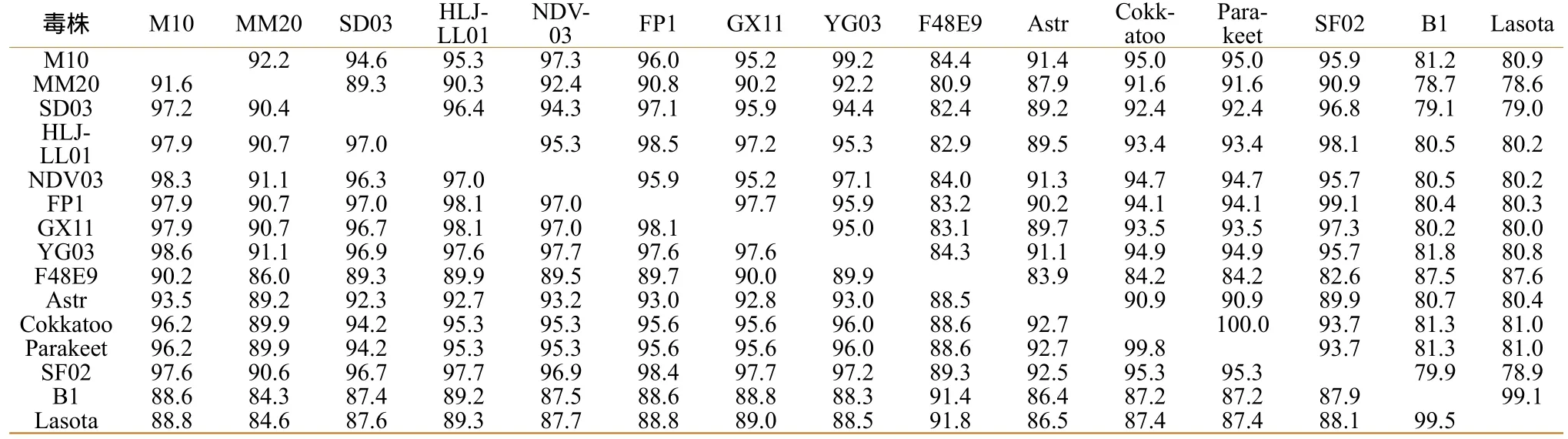
表2 M10分离株与标准株HN基因编码区核苷酸和氨基酸同源性比较(%)
根据HN基因的编码区核苷酸序列进行系统进化树分析(图1),从图中可以看出M10株与鸡源YG03、NDV-03-044,鸭源FP1,鹅源SF02等系统发育关系较近,而与传统经典疫苗株Lasota、B1,经典强毒株F48E9相距较远。

图1 HN基因比较的遗传进化树
2.6 HN蛋白的半胱氨酸位点与糖基化位点分析
由M10 株HN基因编码区推导出的氨基酸序列可得,M10株有13个半胱氨酸残基位点,分别 位 于 123、172、186、196、238、247、251、344、455、461、465、531、542位氨基酸位点处。
在糖基化位点分析上,M10株有5个保守的糖基化位点,分别位于115、119、341、433、508位氨基酸残基处。
3 讨论
副黏病毒HN基因预测的ORF框架长度与终止密码子的位置有差异,其翻译出的多肽长短不同,根据翻译出的多肽长度可将其分为3个型,相对应的片段长度为1713bp、1731bp以及1848bp,分别翻译出571、577、616个氨基酸[6]。研究表明多肽为571个氨基酸时为强毒株[7]。广西马源禽I型副粘病毒M10株的HN基因约为2.0kb,ORF为1716个碱基,共编码571个氨基酸,说明M10株HN基因蛋白的大小具备强毒株特征。HN基因同源性比较以及系统进化树分析发现,M10株与鸡源YG03、NDV-03株,鸭源FP1、鹅源SF02同源性最高,说明这些病毒有可能来自同一祖先,而与经典株Lasota、B1、F48E9相距甚远,说明与过去流行的传统毒株相比,M10株HN基因有了较大的变异。禽I型副粘病毒HN蛋白中的半胱氨酸(Cys)残基、糖基化位点对HN蛋白的结构功能起重要作用。国内外已报告的APMV-I型的强毒株一般含有13个半胱氨酸位点[8];而潜在的糖基化位点有6个,一般在119、341、433、481、508及538位,前4个可以被糖基化,而后两个不用于糖基化[9]。据报道,一些国内的APMV-I分离株缺少第538位的糖基化位点[10]。本研究的M10株HN基因含有13个半胱氨酸位点,与国内外毒株相比较,其数目和位置相对保守,糖基化位点有5个,第538-540位无糖基化位点特征,但是,这并不影响HN蛋白的结构特征。
近年来,禽I型副黏病毒的宿主范围变得越来越广泛,迄今已知能自然或人工感染的鸟类超过205多种,世界各地相继从猪、甚至人体内也分离到了禽I型副黏病毒[11-13]。出现这种情况与不健全的饲养模式有密切关系,家禽与家畜混养,饲养场地消毒不彻底,禽畜市场管理不严格等情况都有可能使得APMV-I病毒在家禽、家畜、人之间传播;另外,南方属于全球候鸟的迁徙线路上,有学者预测,鸽子、八哥等野生鸟类活动广泛,喜欢啄食猪背部蜱、蚊虫等,是猪感染APMV-I的可能途径之一[14]。而从人体分离到的新城疫病毒与先前在欧洲与北美从鸽子身上分离到的新城疫病毒有较高的同源性,并且具有强毒株的特点,可以考虑,鸽也可能是人类新城疫病毒的主要来源[15]。由此可推断,APMV-I也有可能通过以上途径传播至马身上并发生变异。越来越多的数据表明,APMA-I的宿主范围在不断的扩大,给人类健康带来了不可忽视的影响,因此加强APMA-I的分子流行病学调查,以及不同宿主源性APMA-I毒株演化分析具有重大意义。
[1]B w卡尔尼克.禽病学[M].9版 高福,刘文军,译.北京:北京农业大学出版社,1991,427-429.
[2]Sakaguchi T,Toyoda T,Gotoh B,et a1.Newcastle disease virus evolution I.Multiple lineages defined by sequence variability of the hemagglutinin—neuraminidase gene[J].Virol,1989,169(2):260-272.
[3]刘华雷,王永坤,严维巍,等.禽副粘病毒I型(APMV-1)分子生物学研究进展[J].动物医学进展,2001,22(1):16-21.
[4]潘群兴,王永山,杨建朋,等.新城疫不同宿主源分离株的生物学特性[J].江苏农业学报,2011,27(6):1325-1329.
[5]Abolnik C,Gerdes G H, Kitching J,et al.Characterization of pigeon paramyxoviruses (Newcastle disease virus) isolated in South Africa from 2001 to 2006.[J]Onderstepoort J Vet,2008,75(2):147-152.
[6]Mc Ginnes L W,Wilde A, Morrison T G.Nucleotide sequence of the gene encoding the Newcastle disease virus haemagglutin and comparisons of paramyxovirus HN protein sequence[J]Virus Res,1987,7(3):187-202.
[7]云涛,何永强,刘光清,等,鸭I型禽副粘病毒YH99V株HN基因的克隆和序列分析.[J].中国预防兽医学报,2006,28(6):664-667.
[8]McGinnes L W,Wilde A.The role of the individual cysteine residues in the formation of the mature,antigenic HN protein of Newcastle disease virus[J ]Virol,1994 ,200(2):470 -483 .
[9]McGinnes L W,Monison T G.Disul fi de bond formation is a determinat of glycosylation site usage in the hemaggltuininneuraminidase glycoportein of Newcastle disease virus [J].Virol, 1997,71(4):3083-3089.
[10]肖跃强,杨春富,管宇,等.鹅副黏病毒BZ02株F HN基因的克隆及分子特性分析[J]中国兽医杂志2009,45(6):14-17.
[11]刘海霞,敖艳华.几株野生鸟类来源的I型禽副粘病毒分子生物学特征的研究[J].中国兽药杂志, 2008,42(6):12-15.
[12]冯淑萍.广西猪源副粘病毒GuangXi株全基因的测序分析与生物学特性研究[D].广西大学硕士论文,2010.
[13]Goebei S J,Taylor J,Barr B C,et a1.Isolation of avian paramyxovirus I from a patient with a lethal case of pneumonia[J].J Virol,2007,81(22):12709-12714.
[14]鲁会军,金宁一,金扩世.猪副粘病毒的研究进展[A].中国畜牧兽医学会家畜传染病分会成立20周年庆典暨第十次学术研讨会讨论文集[C].杭州:中国畜牧兽医学会禽病学分会,2000,323-326.
[15]Ujvari D E,Wehmann E Kaleta F,et a1. Phylogenetic analysis reveals extensive evolution of avian paramyxovirus type I strains of pigeons(Columba livia)and suggests multiple species transmission[J].Virus Res,2003,96(1):63-73.
