拟南芥不定芽发生早期的数字基因表达谱分析
2013-09-03王兴春杨致荣张树伟李红英李生才
王兴春,杨致荣,张树伟,李红英,3,李生才
1 山西农业大学生命科学学院,山西 太谷 0308012 山西农业大学农业生物工程研究所,山西 太谷 0308013 农业部黄土高原作物基因资源与种质创制重点实验室,山西 太原 0300314 山西农业大学农学院,山西 太谷 030801
拟南芥不定芽发生早期的数字基因表达谱分析
王兴春1,2,杨致荣2,张树伟1,李红英2,3,李生才4
1 山西农业大学生命科学学院,山西 太谷 030801
2 山西农业大学农业生物工程研究所,山西 太谷 030801
3 农业部黄土高原作物基因资源与种质创制重点实验室,山西 太原 030031
4 山西农业大学农学院,山西 太谷 030801
王兴春, 杨致荣, 张树伟, 等. 拟南芥不定芽发生早期的数字基因表达谱分析. 生物工程学报, 2013, 29(2): 189−202.
Wang XC, Yang ZR, Zhang SW, et al. Digital gene expression profiling analysis of the early adventitious shoot formation inArabidopsis thaliana. Chin J Biotech, 2013, 29(2): 189−202.
目前,有关不定芽发生的研究主要集中在单基因的调控方面,缺乏转录组方面的系统研究。利用RNA-seq高通量测序技术在全基因组范围内检测了不定芽发生早期的基因表达谱,共检测到2 457个差异表达基因。这些基因参与了激素代谢和信号转导、愈伤组织和侧根的形成、茎顶端分生组织的发育和光合作用等过程。进一步的途径富集分析表明,不定芽发生早期苯丙氨酸代谢和苯丙胺素合成等途径相关的基因显著富集。并且苯丙氨酸可以显著抑制不定芽的发生,暗示了苯丙氨酸代谢和苯丙胺素的合成可能在不定芽发生过程起着重要的作用。
拟南芥,离体器官发生,不定芽形成,转录组,数字基因表达谱
作为植物再生完整植株的最主要方式之一,不定芽发生不仅广泛应用于植物发育调控机制等基础理论问题的研究,而且是植物快速繁殖和利用生物技术进行作物遗传改良的前提和基础[1-2]。因此,深入研究不定芽发生的机制既具有重要的理论价值又有广阔的应用前景。
植物激素尤其是细胞分裂素和生长素是影响不定芽发生的关键因素,因此调控激素代谢、转运和信号转导的基因通常也参与不定芽发生过程[3-6]。在无外源细胞分裂素的情况下,拟南芥功能获得性突变体植物生长激活子plant growth activator 22(pga22) 和细胞分裂素非依赖突变体cytokinin independent 1(cki1) 可以分化出大量不定芽[5]。PGA22基因编码一个异戊烯基转移酶,该酶催化细胞分裂素生物合成的一个限速步骤;而CKI1曾被认为是细胞分裂素的受体,但由于在体外不能结合细胞分裂素,故该推测还有待进一步证实。细胞分裂素信号转导的关键因子 B型细胞分裂素响应子 RESPONSE REGULATOR (ARR) 过量表达通常促进不定芽的再生,而A型ARR通常会抑制不定芽的再生[6-7]。
在模式植物拟南芥中,不定芽形成主要通过两步培养法进行:首先,将拟南芥的根或下胚轴在含有高浓度 2,4-二氯苯氧乙酸(2,4-dichlorophenoxyacetic acid,2,4-D) 的愈伤诱导培养基 (Callus induction medium, CIM) 中进行预培养;然后,再转移到含有高浓度细胞分裂素的芽诱导培养基 (Shoot induction medium,SIM) 继续培养,即可分化出不定芽[8]。深入探讨上述步骤相关基因的表达及其关系,对于阐明不定芽发生的分子机制具有重要的意义。Che等[9-10]利用基因芯片技术分析了拟南芥愈伤组织、不定芽和不定根再生过程中基因表达情况,发现了一系列在不同的发育过程中差异表达的基因。最近,Xu等[11]利用基因芯片技术研究了愈伤组织形成早期的基因表达情况,发现了一大批新的调控基因。其中转录因子HB52和CRF3的过量表达使外植体在无生长素的培养基上产生愈伤组织。这些研究虽然在一定程度上揭示了植物离体器官发生的基因调控网络,但由于所用芯片的探针数量有限,且基因芯片检测的灵敏度相对较低,不能对所有基因进行检测。与基因芯片相比,近年来发展起来的高通量测序技术有着诸多优势,已经广泛应用于基因表达调控等领域的研究[12]。但到目前为止,高通量测序技术仍未应用于不定芽发生早期转录组研究。为此,我们利用 RNA-Seq高通量测序技术在全基因组范围内检测了在不定芽发生早期差异表达的基因,为揭示不定芽发生的分子机制奠定了基础。
1 材料与方法
1.1 植物材料和培养基
本研究使用的野生型拟南芥Arabidopsis thaliana为Wassilewskija (WS) 生态型。
萌发培养基:1/2×MS (PhytoTechnology Laboratories,货号M519)、20 g/L蔗糖和8 g/L琼脂粉,pH 5.8。
CIM 培养基:1×MS (PhytoTechnology Laboratories,货号M519),10 g/L蔗糖,0.5 g/L MES,100 mg/L 肌醇,10 mg/L VB1, 1 mg/L VB3,1 mg/L VB6,0.5 mg/L 2,4-D,0.05 mg/L 激动素(Kinetin,KT),和2.5 g/L植物凝胶,pH 5.8。
SIM培养基:与CIM成分相似,只是将CIM中的2,4-D和KT替换为 0.2 mg/L吲哚-3-乙酸(Indole-3-acetic acid,IAA) 和0.5 mg/L反式玉米素 (trans-zeatin)。
1.2 方法
1.2.1 愈伤诱导和不定芽发生
拟南芥种子消毒后成簇播种在萌发培养基上,注意不能太分散,以便后期取材。4 ℃避光春化2~3 d后,置于温室培养。培养条件为22 ℃,16 h光照/8 h黑暗,光照强度为20~30 µmol/(m2·s)的弱光,以利于下胚轴的伸长。7 d后,取下胚轴放在富含生长素的CIM上培养7 d;然后再转移到SIM继续培养。
1.2.2 总RNA的提取和RNA-Seq
分别取CIM培养7 d (CIM7) 和SIM培养2 d(SIM2) 的材料提取总RNA。100 mg植物材料在液氮中研磨成粉末,然后利用康为世纪生物科技有限公司的RNA提取试剂盒 (含有DNase I,货号CW0559) 提取总RNA。总RNA利用Agilent 2100生物芯片分析系统检测合格后,利用带有Oligo (dT) 的磁珠富集mRNA。富集的mRNAs随机打断成 200 nt的片段,再以片段化后的mRNA为模板,用六碱基随机引物合成cDNA第一链;加入缓冲液、dNTPs、RNase H 和 DNA polymerase I合成cDNA第二链,经过QiaQuick PCR试剂盒纯化并加 EB缓冲液洗脱经末端修复、加碱基A,加测序接头,再经琼脂糖凝胶电泳回收目的大小片段,并进行 PCR扩增。建好的文库用Illumina HiSeq™ 2000进行测序。
1.2.3 数据分析
基因表达量的计算使用 RPKM 法 (Reads Per Kb per Million reads)[13],该方法能消除基因长度和测序深度差异对计算基因表达的影响,因此计算得到的基因表达量可直接用于比较不同样品间的基因表达差异。差异表达基因为 False Discovery Rate (FDR) ≤0.001且差异倍数不低于2倍 (即log2的绝对值≥1) 的基因。
Gene Ontology (GO) 功能显著性富集分析采用 Gene Ontology数据库 (http://www.geneontology.org/) 进行,将差异表达的基因向gene ontology数据库的各term映射。计算每个term的基因数目。P≤0.05为显著富集的 GO term。途径显著性富集分析 (Pathway enrichment analysis) 采用 KEGG数据库[14]进行,将差异表达基因向KEGG数据库映射,并统计基因在每个途径中的富集程度 (Pvalue)。
2 结果与分析
2.1 RNA-Seq高通量测序和评估
拟南芥下胚轴在富含生长素的 CIM 培养基培养 7 d后 (CIM7),中柱鞘部位膨大,形成大量愈伤组织 (图 1A)。将其转移到富含细胞分裂素的SIM培养基继续培养2 d (SIM2),愈伤组织继续膨大,颜色由白色变成淡黄色 (图 1B)。分别提取CIM7和SIM2的总RNA,构建cDNA文库,进行 RNA-Seq实验。CIM7和 SIM2样品RNA-Seq测序分别得到11 001 875和1 054 546 raw reads,其中clean reads分别为10 956 306和 10 956 306,分别占 raw reads的 99.55%和99.59%。去除杂质后的clean reads用作后续分析。

图1 RNA-Seq用外植体CIM7和SIM2Fig. 1 Explants of CIM7 and SIM2 for RNA-Seq. (A)Callus formation in CIM7. (B) Callus formation in SIM2.
我们使用短 reads比对软件 SOAPaligner/soap2[15]将得到的 clean reads分别比对到拟南芥参考基因序列 (ftp://ftp.arabidopsis.org/Sequences/blast_datasets/TAIR10_blastsets/TAIR10_cdna_20 101214)。结果表明,样品CIM7和SIM2得到的clean reads中,分别有92.96%和93.13%的clean reads匹配到拟南芥的2 3878个参考基因上 (表1和表2)。每个基因平均被426.52 (CIM7)和393.64(SIM2)个clean reads覆盖。
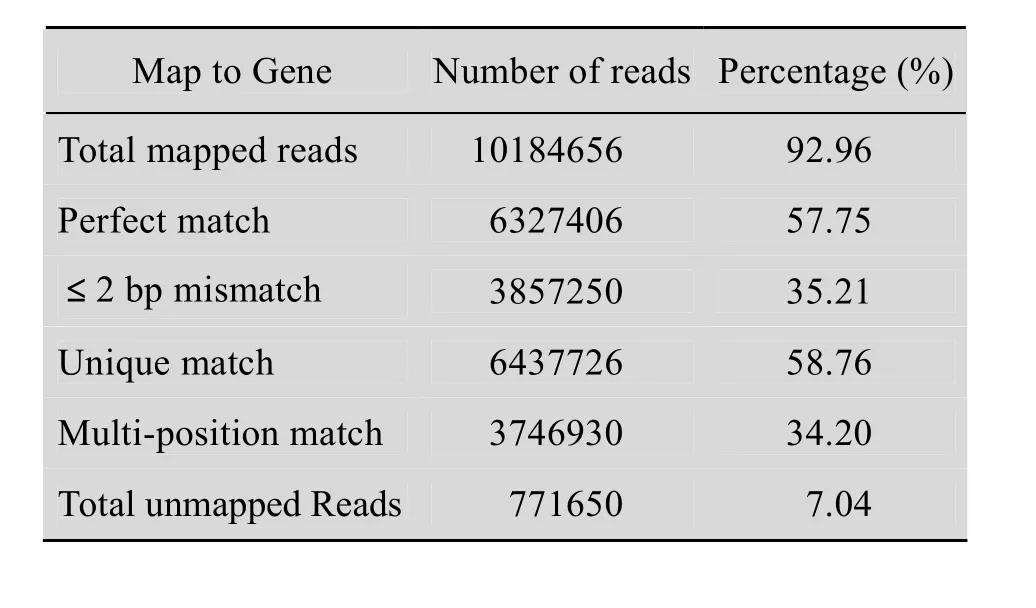
表1 样品CIM7和拟南芥参考基因比对的统计结果Table 1 Alignment statistics of CIM7 to Arabidopsis reference genes
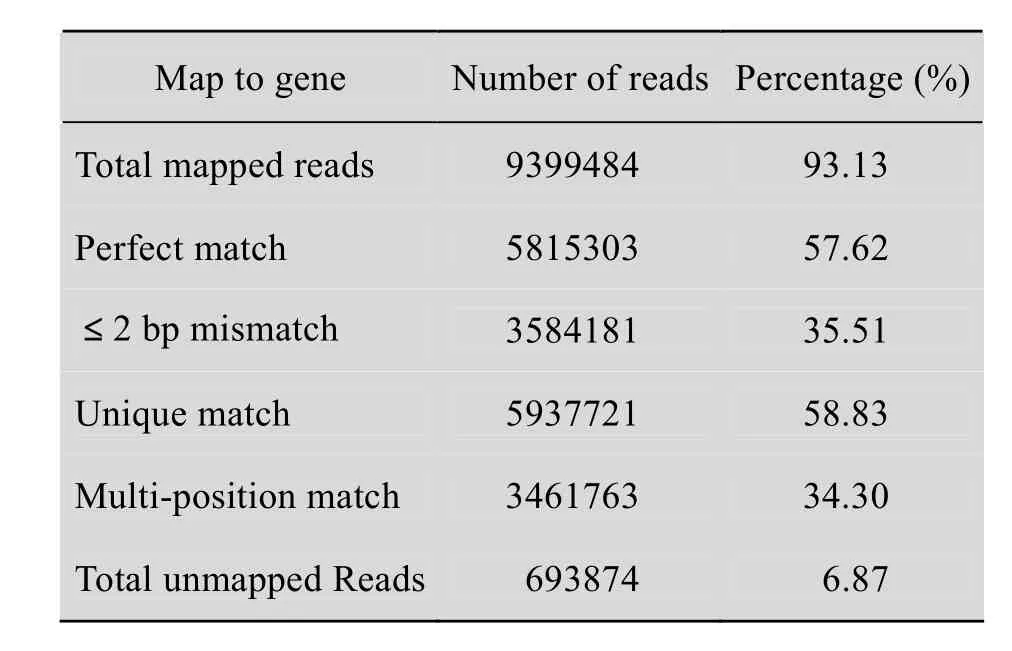
表2 样品SIM2和拟南芥参考基因比对的统计结果Table 2 Alignment statistics of SIM2 to Arabidopsis reference genes
测序饱和度分析表明,当clean reads在2 M以下时,检测到的基因数与测序量成正比;当达到2 M以上时,趋于平缓;超过8 M时检测到的基因数已经接近饱和 (图2A和2B)。样品CIM7和SIM2的测序量分别为10.18 M和9.40 M,已经接近饱和。随后,我们又对测序的随机性进行了统计,结果表明样品 CIM7和 SIM2的 reads在参考基因上基本成随机分布 (图 2C和 2D)。上述结果表明本次测序结果能够较好地反应细胞中基因表达的真实情况,可以用于后续基因差异表达分析。
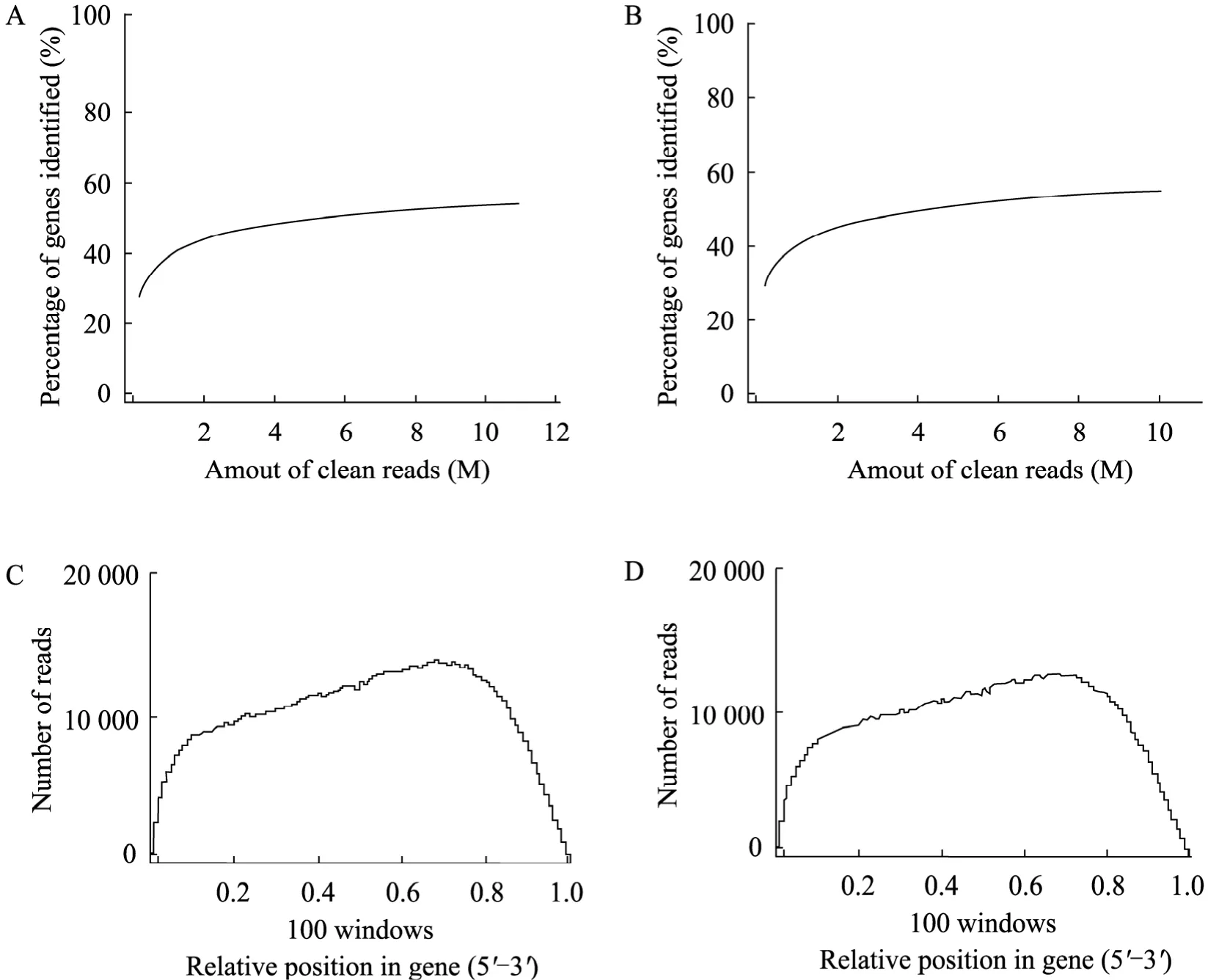
图2 测序饱和度和随机性评估Fig. 2 Sequencing saturation and randomness assessment. (A,B) Sequencing saturation analysis of CIM7 (A) and SIM2 (B). (C,D) Sequencing randomness analysis of CIM7 (C) and SIM2 (D).
2.2 不定芽发生早期差异表达的基因
在不定芽发生早期,我们共检测到2 457个差异表达基因,约占拟南芥整个基因组的10%左右。其中,有1 761个基因表达量上调,696个基因表达量下调。这些基因参与了植物激素的平衡和信号转导、愈伤组织的形成、侧根的发育、光合作用和细胞周期等。
2.2.1 激素平衡和信号转导与不定芽发生
在不定芽发生过程中,很多与细胞分裂素和生长素合成、代谢和信号转导相关的基因的表达量发生了显著改变。异戊烯基转移酶 (Isopentenyl transferase,IPT) 是细胞分裂素合成的关键酶,催化细胞分裂素合成的一个限速步骤。在拟南芥中过量表达IPT基因导致植物体内细胞分裂素含量上升,并促进了不定芽的再生[5,16]。然而,我们发现在不定芽发生早期IPT5基因的表达量下降了近1 500倍 (表3),而其他IPT基因的表达量没有显著变化,这暗示了IPT5基因可能在离体器官再生过程起着特殊的功能。作为细胞分裂素信号途径的下游组分,ARR是执行细胞分裂素生理功能的效应元件。在不定芽发生早期,有6个ARR基因的表达量显著升高 (表3)。这6个ARR全部为A型ARR,而B型ARR的表达量却没有明显变化。这与A型ARR基因的表达能被细胞分裂素快速诱导是相符的[16]。
外植体从含有高浓度生长素的 CIM 中转到含有低浓度生长素的SIM培养基中后,一些参与生长素信号转导的基因如IAA、GH3、SAUR和ARF等的表达量也发生了变化 (表3)。在拟南芥29个Aux/IAA中,有4个Aux/IAA基因 (IAA3、IAA17、IAA18和IAA28) 的表达量发生了上调;4个Aux/IAA基因 (IAA14、IAA19、IAA20和IAA30) 的表达量发生了下调。
有趣的是,虽然CIM和SIM都不含有乙烯、赤霉素和油菜素内酯等其他激素,但有些与这些激素平衡和信号转导相关基因的表达量发生了显著变化 (表 3)。乙烯反应元件结合蛋白(ETHYLENE-RESPONSIVE ELEMENT BINDING FACTOR,ERF) 是乙烯信号转导过程中的关键调控因子,在植物胁迫反应中起着重要的作用[17]。在拟南芥不定芽发生早期,有3个ERF基因的表达量发生显著上升,其中ERF13的表达量上升了21.21倍。这些结果暗示了乙烯等其他激素也可能在不定芽发生过程起着间接的调控作用。
2.2.2 愈伤形成和侧根发育相关基因的表达
在不定芽发生早期,有 4个 LATERAL ORGAN BOUNDARIES DOMAIN (LBD) 家族的基因 (LBD16、LBD18、LBD19和LBD29) 的表达量显著降低。其中LBD29基因的表达量降低幅度最大,达1 445.81倍,在不定芽发生早期完全关闭 (表3)。这4个LBD基因中,有3个(LBD16、LBD18和LBD29) 可以被CIM快速诱导,且它们中的任何一个异位表达都可以使外植体在无激素的情况下形成愈伤组织[18]。这表明,细胞由脱分化向再分化状态转变时,一些参与脱分化的基因将要被关闭。另外,由于愈伤组织和侧根起源相同[19],所以一些参与侧根形成或发育基因的表达量也有所改变 (表3)。
2.2.3 茎顶端分生组织相关基因的表达
外植体在SIM培养基培养2 d时,虽然还未出现明显的茎顶端分生组织 (SHOOT APICAL MERISTEM,SAM) (图1),但SAM相关基因的表达已经发生了变化 (表 3)。其中,CLAVATA3/ESR-RELATED 2(CLE2) 基因表达
上升幅度最大,上升了近 9 000倍。其次是CUP-SHAPED COTYLEDON 1(CUC1) 和CUC2基因,表达量分别提高了8.92倍和5.07倍。
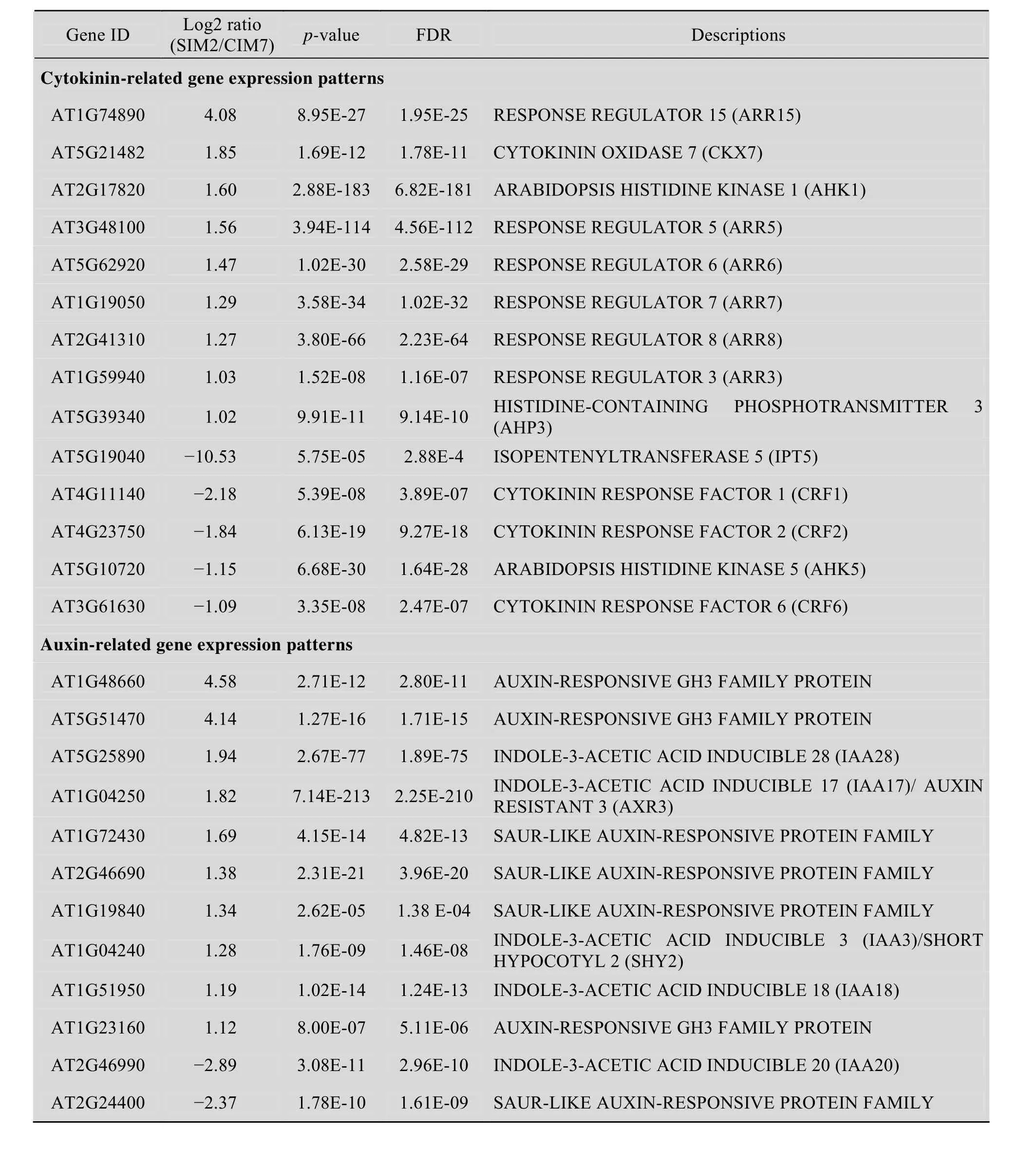
表3 不定芽发生早期差异表达的基因Table 3 Differential expressed genes at the early stage of adventitious shoot formation
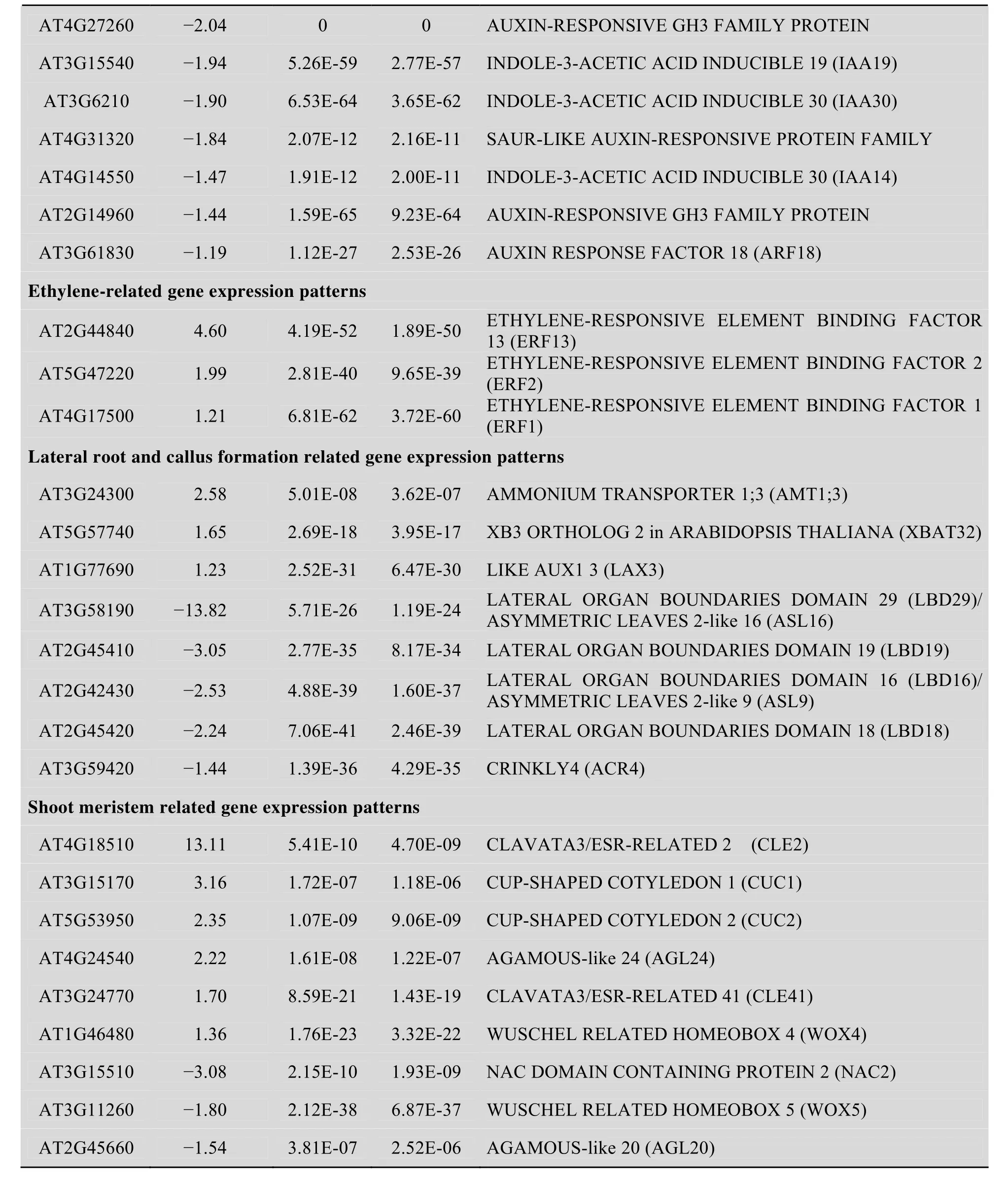
续表3
除了上述基因外,一些与细胞分化和细胞周期相关的基因的表达量也发生了显著变化 (数据未显示)。
2.3 不定芽发生早期代谢的变化
为了对差异表达基因的生物学功能有更清晰的了解,我们进行了途径富集分析。结果表明,差异表达的2 457个基因中有1 364个基因可以映射到111个不同的生物途径,显著富集的途径(Qvalue≤0.05) 有25个 (表4)。其中,苯丙胺素合成和苯丙氨酸代谢相关途径的基因显著富集,分别富集了3.38倍和3.84倍。为了进一步研究苯丙氨酸在不定芽发生过程的功能,我们将脱分化的下胚轴外植体放在含有不同浓度的苯丙氨酸的CIM培养基培养。结果表明,1 mmol/L的苯丙氨酸即可以完全抑制不定芽的再生(图 3),暗示了苯丙氨酸和苯丙胺素的生物合成可能在不定芽发生过程中起着重要的作用。
除了苯丙胺素和苯丙氨酸途径外,次生物质合成、植物激素信号转导和光合作用等相关途径的基因也显著富集了。进一步的分析表明,差异表达的基因中光合途径 (Photosynthesis pathway)、黄酮和黄酮醇生物合成途径 (Flavone and flavonol biosynthesis) 的基因的表达量全部升高了,暗示了不定芽发生早期外植体开始由异养向自养转变。而其余 23个途径中既有上调基因又有下调基因,表明不定芽发生早期转录水平调节的复杂性。
2.4 不定芽发生早期叶绿体相关组分显著富集
GO 功能显著性细胞组分 (Cellular component) 分析表明,在差异表达的2 457个基因中有 1 365个基因可以映射到 101个不同的term。其中富集的有20个term (表5)。进一步的分析发现,富集 term中有一半以上 (11个)term位于叶绿体/质体中 (表5),这暗示了在不定芽出现之前,外植体已经具备了部分光合作用的能力。
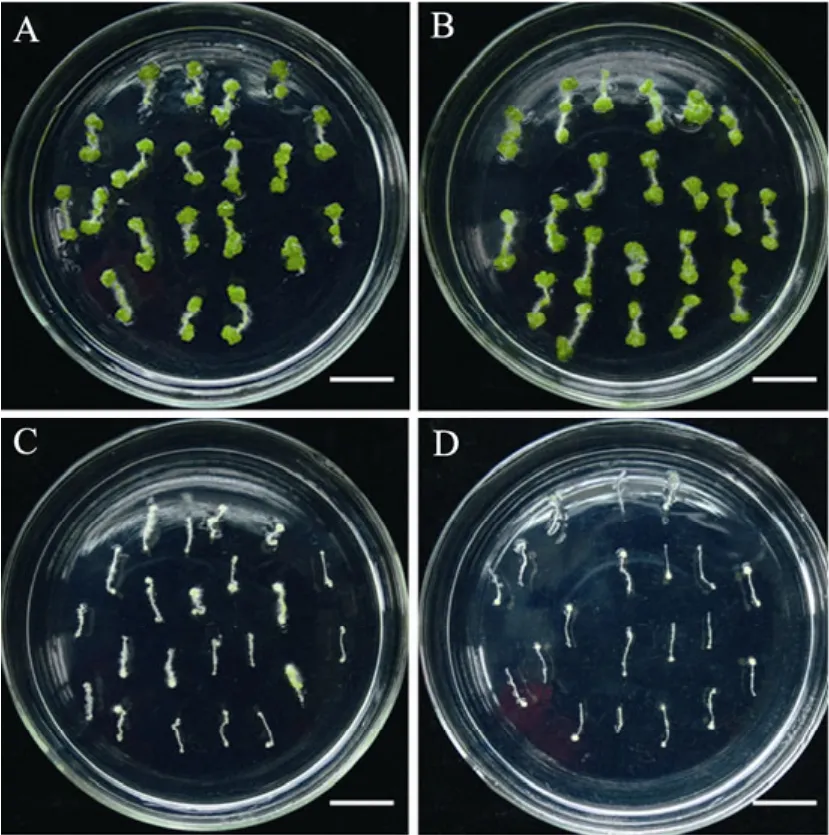
图3 苯丙氨酸抑制不定芽发生Fig. 3 Adventitious shoot formation was suppressed by phenylalanine. Hypocotyl explants were pre-cultured on CIM for 7 days and then cultured on SIM containing different concentration of phenylalanine for 12 days. (A)0 mmmol/L. (B) 0.1 mmol/L. (C) 1 mmol/L. (D)10 mmol/L. Bar= 1 cm.
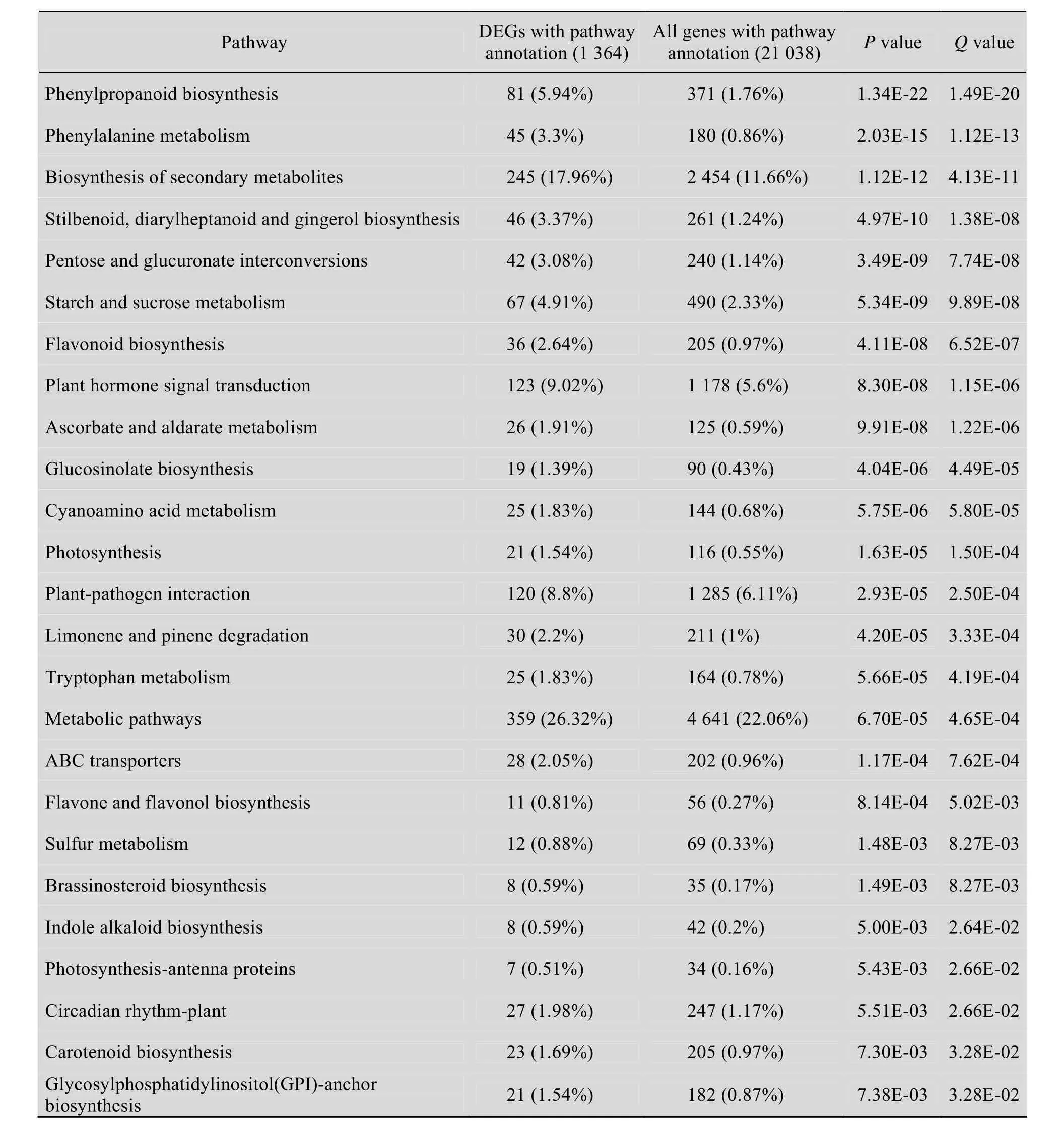
表4 不定芽发生早期显著富集的途径Table 4 Pathways significantly enriched at the early stage of adventitious shoot formation
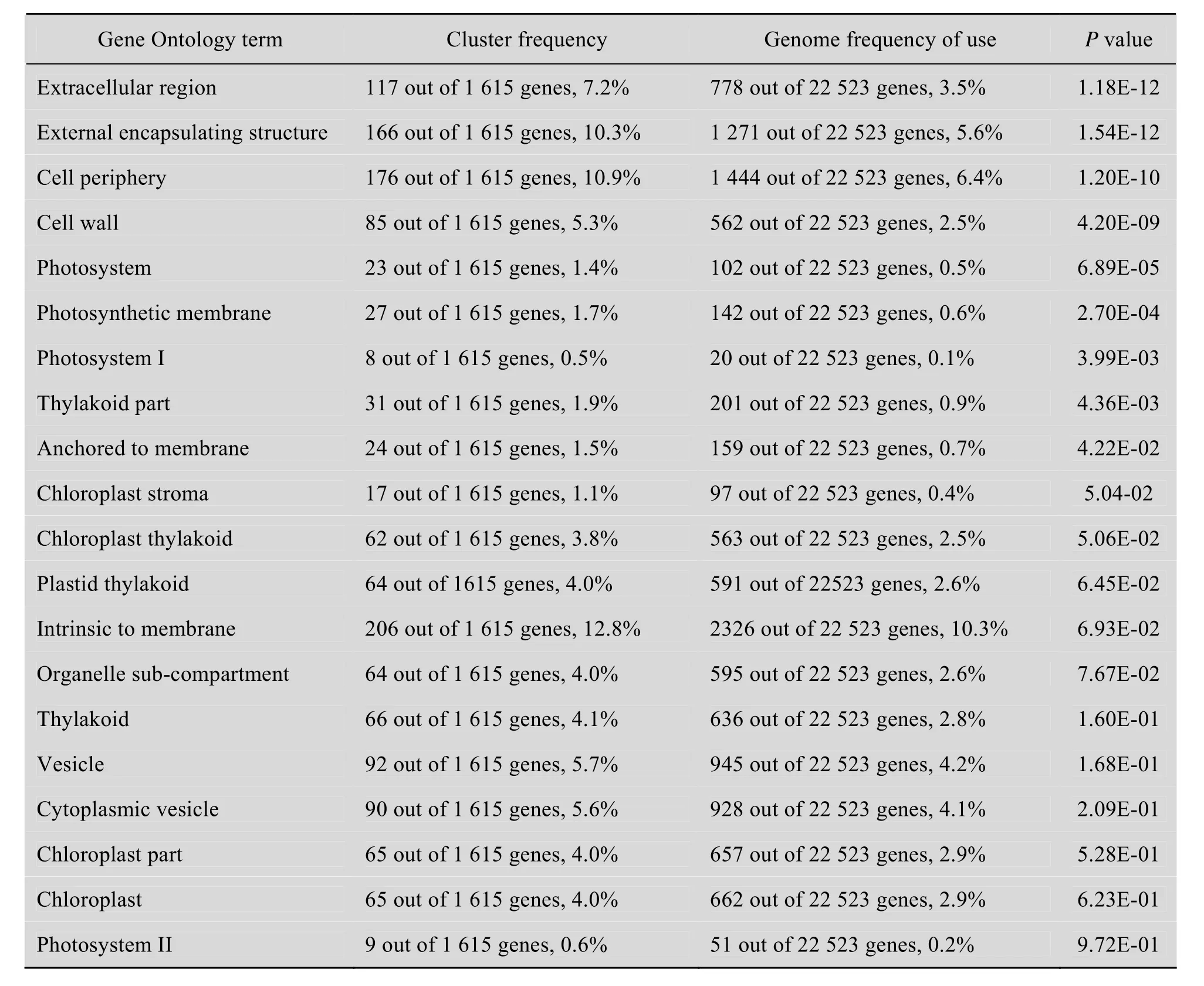
表5 不定芽发生早期差异表达基因的GO富集分析Table 5 GO analysis of the differential expressed genes at the early stage of adventitious shoot formation
3 讨论
植物不定芽发生是一个众多基因参与的复杂过程,在这一过程中许多基因的表达量发生了改变。Che等[9−10]曾利用基因芯片技术检测了不定芽发生时基因的表达情况。但由于基因芯片技术本身的局限性,检测的基因数目有限。另外,Che等用的是在SIM培养基培养3 d的材料,此时外植体已呈浅绿色,叶绿体已经开始分化。而我们利用的是在SIM培养基中培养2 d的材料,更有利于早期差异表达基因的研究。Che等[9−10]利用基因芯片进行的研究中,发现ARR5和RAP2.6L等基因的表达量显著上调。我们RNA-Seq技术检测结果表明,这两个基因分别上调了2.96和2.25倍。此外,本研究表明ARR15、CUC1、CUC2和LBD29等基因的表达量也明显上调 (表3),这些基因已经被证明与不定芽发生有关。这些结果从侧面证明本研究的 RNA-Seq检测结果是可信的。除了这些已知基因外,我们也新发现了一大批以前在不定芽发生过程未研究过的基因 (如IPT5、CLE2、ERF13和GH3家族的基因等),深入研究这些基因的功能将有助于我们对不定芽发生过程有更清晰的认识。同时,我们还发现苯丙氨酸和苯丙胺素的合成与代谢可能在不定芽发生过程起着极其重要的作用。
细胞分裂素是影响不定芽发生的关键因子,也是SIM培养基中添加的主要外源激素。当将外植体从富含生长素的 CIM 培养基转移到富含细胞分裂素的SIM培养基后,IPT5基因的表达量大幅下降,培养2 d时已经检测不到该基因的表达,而其他8个IPT基因的表达却没有明显变化(表 3)。细胞分裂素在 4 h内即可负反馈抑制IPT1、IPT3、IPT5和IPT7基因的表达[20]。因此,我们推测不定芽分化早期IPT5基因的表达量下降可能是由于 SIM 培养基上高浓度细胞分裂素负反馈调节的结果。但我们不排除IPT5基因在脱分化时起着重要作用的可能性,因此在脱分化时高表达,在再分化时表达量降低。为此,我们将深入研究IPT5基因在外植体脱分化阶段的功能。除了IPT基因外,细胞分裂素的氧化分解是调节细胞分裂素含量动态平衡的一种重要方式。这一反应受到细胞分裂素氧化酶 (Cytokinin oxidase/dehydrogenase,CKX) 的催化。在不定芽发生早期,CKX7基因的表达量略有上升 (表3),这可能是由于 SIM 中高浓度的细胞分裂素负反馈调控的结果。A型ARRs是细胞分裂素途径的负调控因子,其中ARR15基因的表达可以被细胞分裂素快速诱导,且该基因可以作为细胞获得全能性具备不定芽分化能力的标记基因[21−22]。与该报道相一致,本实验表明在 SIM 培养基培养2 d时,ARR15的表达量提高了16.90倍。除了ARR15基因外,在本研究中,我们发现ARR3、ARR5、ARR6、ARR7和ARR8等5个其他的A型ARR基因的表达量也都有所升高。除了上述细胞分裂素相关的基因外,生长素相关的一些基因的表达量也发生了显著变化 (表3)。生长素调控着愈伤的形成,在细胞脱分化过程起着关键的作用。最近的一项研究表明,LBD16、LBD17、LBD18和LBD29等4个LBD基因位于生长素响应因子ARF的下游,调控着愈伤组织的形成[18]。我们发现这些基因在不定芽形成早期表达量都有所下降,其中LBD29基因的表达量降幅最大为14 415.81倍 (表3)。这表明,这些基因虽然在愈伤组织形成过程起着重要作用,但在不定芽发生早期却不再发挥功能。
SAM 的发育和分生能力的维持与不定芽的再生关系密切,许多SAM发育相关的基因突变也会影响不定芽的再生[2,23]。不定芽发生早期有9个和 SAM相关的基因的表达量发生了改变,其中上调的有6个,下调的3个。上调幅度较大的3个基因分别为CLE2、CUC1和CUC2。有关CUC1和CUC2基因在不定芽发生过程的功能已有报道[24],但CLE2基因在不定芽发生过程的功能还不清楚。这些结果表明,虽然在SIM培养基培养2 d时,还未出现不定芽,但此时一些芽顶端分生组织相关的基因已经开始表达。
途径富集分析表明,不定芽发生早期苯丙氨酸代谢和苯丙胺素合成的基因显著富集 (表4)。进一步的实验表明,苯丙氨酸可以抑制不定芽的形成 (图3)。苯丙胺素是一类由苯丙氨酸合成的重要的次生代谢物质,在植物生长发育过程起着极其重要的作用[25]。但苯丙氨酸和苯丙胺素在不定芽发生过程的功能还不清楚,深入研究二者合成和代谢相关突变体不定芽的再生情况将有助于这一问题的解决。
[1]Guan CM, Zhang XS. Advances in the molecular mechanism ofin vitroplant organogenesis. Chin Sci Bull, 2006, 23(5): 595−602 (in Chinese).
关春梅, 张宪省. 植物离体器官发生控制机理研究进展. 植物学通报, 2006, 23(5): 595−602.
[2]Duclercq J, Sangwan-Norreel B, Catterou M, et al.De novoshoot organogenesis: from art to science.Trends Plant Sci, 2011, 16(11): 597−606.
[3]Inoue T, Higuchi M, Hashimoto Y, et al.Identification of CRE1 as a cytokinin receptor fromArabidopsis. Nature, 2001, 409(6823): 1060−1063.
[4]Sun J, Hirose N, Wang X, et al.Arabidopsis SOI33/AtENT8gene encodes a putative equilibrative nucleoside transporter that is involved in cytokinin transportin planta. J Integr Plant Biol,2005, 47(5): 588−603.
[5]Sun J, Niu QW, Tarkowski P, et al. TheArabidopsisAtIPT8/PGA22gene encodes an isopentenyl transferase that is involved inde novocytokinin biosynthesis. Plant Physiol, 2003,131(1):167−176.
[6]Ren B, Liang Y, Deng Y, et al. Genome-wide comparative analysis of type-AArabidopsisresponse regulator genes by overexpression studies reveals their diverse roles and regulatory mechanisms in cytokinin signaling. Cell Res, 2009,19(10): 1178−1190.
[7]Hwang I, Sheen J. Two-component circuitry inArabidopsiscytokinin signal transduction. Nature,2001, 413(6854): 383−389.
[8]Valvekens D, Montagu MV, Van Lijsebettens M.Agrobacterium tumefaciens-mediated transformation ofArabidopsis thalianaroot explants by using kanamycin selection. Proc Natl Acad Sci USA,1988, 85(15): 5536−5540.
[9]Che P, Gingerich DJ, Lall S, et al. Global and hormone-induced gene expression changes during shoot development inArabidopsis. Plant Cell,2002,14(11): 2771−2785.
[10]Che P, Lall S, Nettleton D, et al. Gene expression programs during shoot, root, and callus development inArabidopsistissue culture. Plant Physiol, 2006,141(2): 620−637.
[11]Xu K, Liu J, Fan M, et al. A genome-wide transcriptome profiling reveals the early molecular events during callus initiation inArabidopsismultiple organs. Genomics, 2012,100(2): 116−124.
[12]Wang XC, Yang ZR, Wang M, et al.High-throughput sequencing technology and its application. China Biotechnol, 2012, 32(1):109−114 (in Chinese).
王兴春, 杨致荣, 王敏, 等. 高通量测序技术及其应用. 中国生物工程杂志, 2012, 32(1):109−114.
[13]Mortazavi A, Williams BA, McCue K, et al.Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nat Meth, 2008, 5(7): 621−628.
[14]Kanehisa M, Araki M, Goto S, et al. KEGG for linking genomes to life and the environment.Nucleic Acids Res, 2008, 36(Database issue):D480−484.
[15]Li R, Yu C, Li Y, et al. SOAP2: an improved ultrafast tool for short read alignment.Bioinformatics, 2009, 25(15): 1966−1967.
[16]Deng Y, Wang XC, Yang SH, et al. New insights into cytokinins: metabolism, signal transduction,cross talks and potentials in agricultural applications. Chin Sci Bull, 2006: 23(5): 478−498(in Chinese).
邓岩, 王兴春, 杨淑华, 等. 细胞分裂素: 代谢、信号转导、交叉反应与农艺性状改良. 植物学通报, 2006, 23(5): 478−498.
[17]Onate-Sanchez L, Singh KB. Identification ofArabidopsisethylene-responsive element binding factors with distinct induction kinetics after pathogen infection. Plant Physiol, 2002, 128(4):1313−1322.
[18]Fan M, Xu C, Xu K, et al. LATERAL ORGAN BOUNDARIES DOMAIN transcription factors direct callus formation inArabidopsisregeneration.Cell Res, 2012, 22(7): 1169−1180.
[19]Sugimoto K, Jiao Y, Meyerowitz EM.Arabidopsisregeneration from multiple tissues occurs via a root development pathway. Dev Cell, 2010,18(3):463−471.
[20]Miyawaki K, Matsumoto-Kitano M, Kakimoto T. Expression of cytokinin biosynthetic isopentenyltransferase genes inArabidopsis: tissue specificity and regulation by auxin, cytokinin, and nitrate. Plant J, 2004, 37(1): 128−138.
[21]Che P, Lall S, Howell SH. Developmental steps in acquiring competence for shoot development inArabidopsistissue culture. Planta, 2007,226(5):1183−1194.
[22]Brenner WG, Romanov GA, Kollmer I, et al.Immediate-early and delayed cytokinin response genes ofArabidopsis thaliana identified by genome-wide expression profiling reveal novel cytokinin-sensitive processes and suggest cytokinin action through transcriptional cascades. Plant J,2005, 44(2): 314−333.
[23]Tamaki H, Konishi M, Daimon Y, et al.Identification of novel meristem factors involved in shoot regeneration through the analysis of temperature-sensitive mutants ofArabidopsis. Plant J, 2009, 57(6): 1027−1039.
[24]Cary AJ, Che P, Howell SH. Developmental events and shoot apical meristem gene expression patterns during shoot development inArabidopsis thaliana.Plant J, 2002, 32(6): 867−877.
[25]Weisshaar B, Jenkins GI. Phenylpropanoid biosynthesis and its regulation. Curr Opin Plant Biol, 1998, 1(3): 251−257.
September 17, 2012; Accepted: November 6, 2012
Xingchun Wang. Tel: +86-354-6287191-306; Fax: +86-354-6289318; E-mail: wxingchun@163.com
国家自然科学基金 (Nos. 31100235, 31171181),山西省青年科技研究基金 (No. 2010021030-1),中国博士后研究经费 (No. 80839) 资助。
Digital gene expression profiling analysis of the early adventitious shoot formation inArabidopsis thaliana
Xingchun Wang1,2, Zhirong Yang2, Shuwei Zhang1, Hongying Li2,3, and Shengcai Li4
1College of Life Sciences,Shanxi Agricultural University,Taigu030801,Shanxi,China
2Institute of Agricultural Bioengineering,Shanxi Agricultural University,Taigu030801,Shanxi,China
3Key Laboratory of Crop Gene Resources and Germplasm Enhancement on Loess Plateau,Ministry of Agriculture,Taiyuan030031,Shanxi,China
4College of Agriculture,Shanxi Agricultural University,Taigu030801,Shanxi,China
Most current research in the field of adventitious shoot formation is focused on the regulatory function of a single gene. However, a systematic transcriptomic analysis of the early adventitious shoot formation is still lacking. Here,we analyzed the transcriptome profiling of the early adventitious shoot formation inArabidopsisby RNA-seq high throughput sequencing technology, and identified 2 457 differentially expressed genes. Detailed categorization revealed that these genes were mainly involved in hormone homeostasis or signal transduction, callus and lateral root formation,shoot apical meristem development and photosynthesis. Further pathway enrichment analysis showed that genes involved in phenylalanine metabolism and phenylpropanoid biosynthesis were significantly enriched. Moreover, exogenous phenylalanine could repress adventitious shoot formation, indicating that phenylalanine metabolism and phenylpropanoid biosynthesis might be important for adventitious shoot formation.
Arabidopsis thaliana,in vitroorganogenesis, adventitious shoot formation, transcriptome, digital gene expression profiling
Supported by: National Natural Science Foundation of China (Nos. 31100235, 31171181), Natural Science Foundation of Shanxi Province(No. 2010021030-1), Postdoctoral Research Foundation (No. 80839).
Shengcai Li. Tel/Fax: +86-354-6288324; E-mail: sxaulisc@126.com
(本文责编 郝丽芳)
