中国石榴(Punica granatum L.)居群遗传多样性
2013-08-02赵丽华
赵丽华
(西昌学院农业科学学院,四川 西昌 615000)
石榴(Punica granatum L.)为石榴科(Punicaceae)石榴属(Punica)落叶灌木或小乔木,原产伊朗、阿富汗、印度等中亚一带[1],石榴属有3个种:P.granatum、P.nana 及 P.protopunica,(2n=2x=16,18),因为P.granatum具有经济、观赏及药用价值而被广泛栽培[2-3]。从西汉张骞将石榴引入中国,至今已有2000多年的栽培历史,经长期天然杂交、基因突变、嫁接等,在中国产生了复杂多样的品种和类型。据不完全统计,中国现有石榴品种资源230多个,形成了山东、新疆、陕西、安徽、河南、云南和四川7个主栽培区[4]。由于石榴是小杂果,国内外对其研究的总体水平较落后,基因组信息非常有限,进而影响了石榴的生产以及国内、国际间的交流。近年来国内外学者应用 RAPD[5-6]、SRAP[7]、SSR[8]、ISSR[9-10]、AFLP[11]等分子标记对石榴进行了遗传分析,为石榴种质资源研究提供分子依据。目前鲜于利用RAMP(Random am-plified microsatellite polymorphism)标记对石榴进行遗传研究的报道,RAMP是利用ISSR引物和RAPD引物组合对基因组进行随机扩增,其适合遗传背景尚不清楚的物种的遗传分析[12]。本研究旨在利用RAMP对中国石榴居群进行遗传分析,为石榴资源进行有效保护及可持续利用提供遗传信息。
1 材料与方法
1.1 材料
供试46个石榴品种(表1)采自云南蒙自县石榴研究所、四川会理县农业局石榴品种资源圃。每个品种从3~5株健壮、无病虫害石榴树采30~50片嫩叶,冰壶运回实验室,于-80℃冰箱保存。
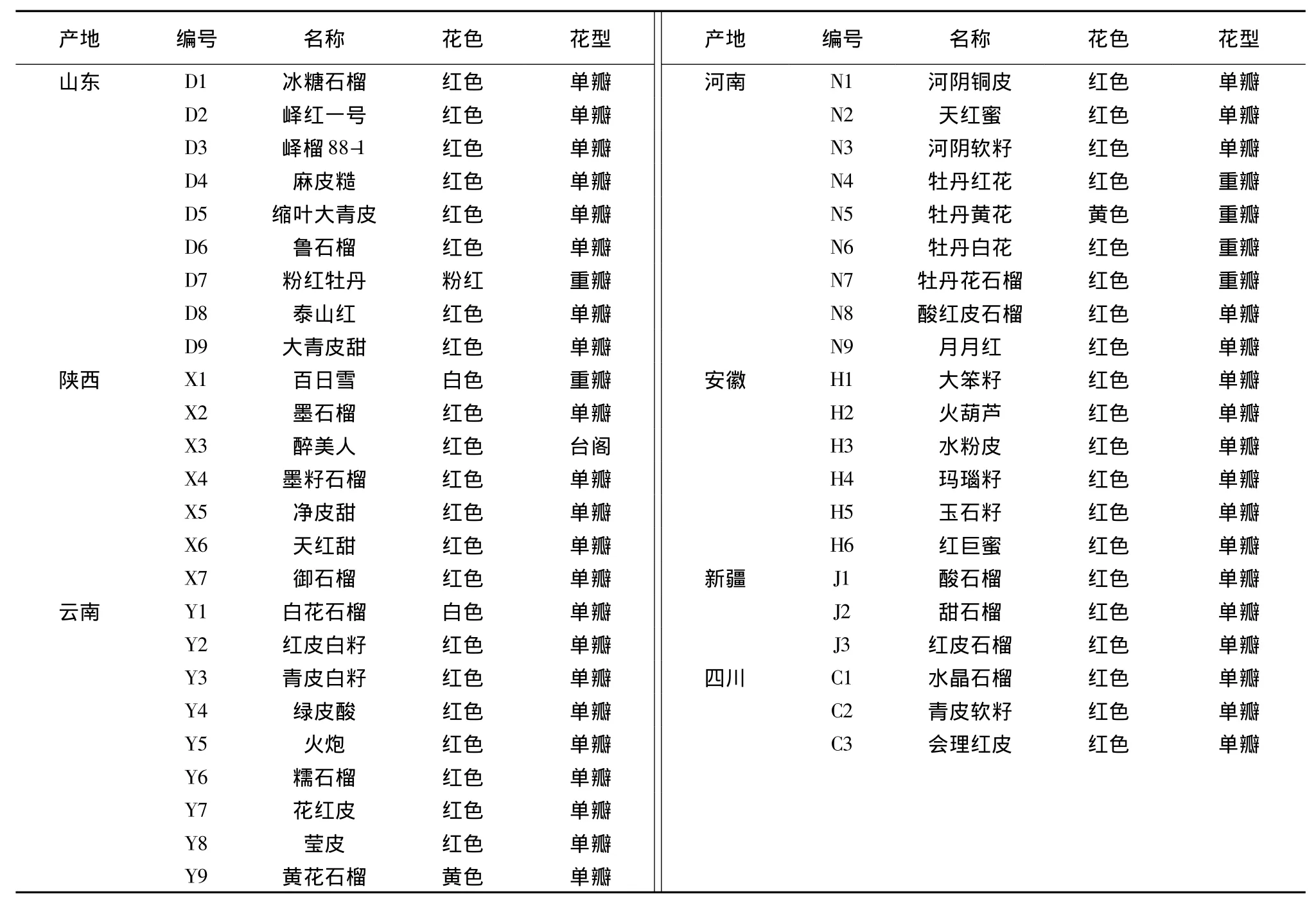
表1 供试46个石榴品种产地、编号、名称及特性Table 1 Origin,code,cultivar and main characteristics of 46 pomeg ranate cultivars
1.2 DNA 提取
参见赵丽华等[13]改良CTAB法从石榴幼叶中提取基因组DNA,0.80%琼脂糖凝胶电泳及核酸蛋白质分析仪测定DNA产量及质量,然后将其稀释为50 ng/μl工作液,放入4℃冰箱里备用。
1.3 引物筛选及PCR扩增
引物由上海英骏生物技术有限公司合成,由5条ISSR引物和20条RAPD引物组成100对引物,用3个形态差异较大的石榴品种从100对引物中筛选出DNA条带清晰、多态性较高的引物对(表2),用选出的多态性引物对对46个石榴品种基因组DNA进行PCR扩增;用梯度PCR仪(Eppendorf)确定每对引物的最适退火温度。用2.00%琼脂糖凝胶电泳(含0.1‰GV)分离PCR扩增产物,电压为2 V/cm,电泳1.5~2.0 h,用紫外与可见分析装置观察并拍照记录。
参照沈洁等[12]PCR 反应体系,优化 25 μl的PCR反应体系为:Mg2+1.5 mmol/L、dNTPs 0.2 mmol/L、Taq DNA 聚合酶1 U、模板DNA 25 ng、引物各0.15 mmol/L、1×PCR buffer。扩增程序为:94℃预变性4 min;94℃变性45 s,退火(温度见表2)1 min,72℃延伸2 min,45个循环;72℃延伸10 min,4℃保存。
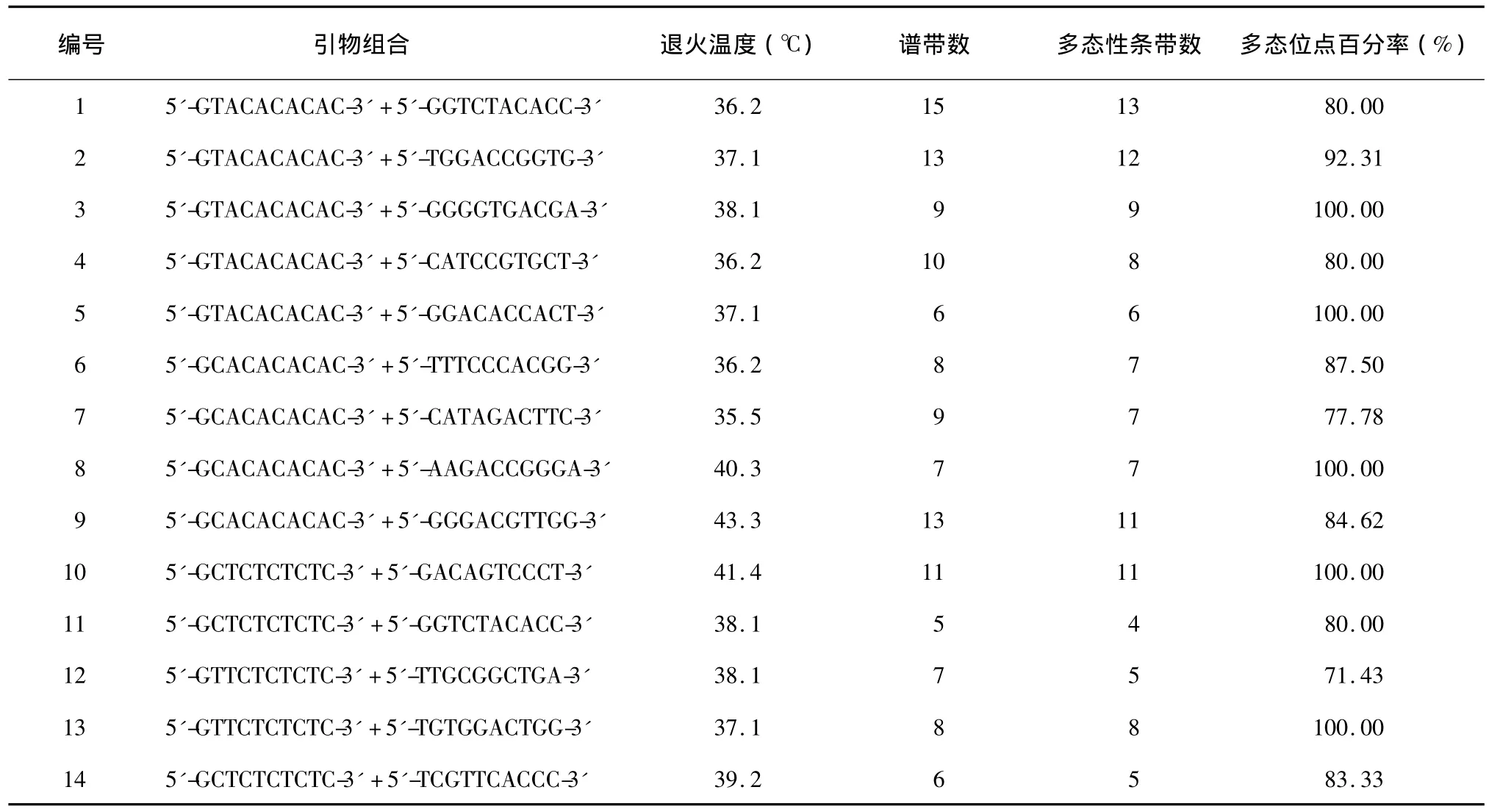
表2 RAMP引物序列、退火温度和扩增结果Table 2 List of RAMP primers,annealing temperatures and amplification results
1.4 数据处理
将RAMP引物扩增产物进行DNA带多态性位点统计,在相同迁移位置上有带记录为“1”,无带记录为“0”,亮度相差2倍的记录为不同的条带,建立由“0、1”组成的二元数据矩阵。采用 PopGen-1.32软件计算多态位点百分率(P)、等位基因数(Na)、有效等位基因数(Ne)、Nei’s氏基因多样性指数(H)、Shannon 多样性指数(I)、Nei’s遗传距离(Dg)、Nei’s遗传相似系数(Sg)、遗传分化系数(Gst)、基因流(Nm)。
2 结果
2.1 RAMP 多态性
14对RAMP引物对46个石榴品种进行PCR扩增,共扩出127条DNA条带,平均每条引物扩增出8.57条DNA条带,其中113条显示多态性,平均每条引物扩增出7.53条多态性条带,多态位点百分率(P)为88.98%(表2)。14对RAMP引物扩增的DNA条带数为5~15条,多态位点百分率为71.43%~100.00%(表2);其中引物对5'-GTACACACAC-3'+5'-GGTCTACACC-3'扩增出的DNA谱带数最多,为15条,多态位点百分率为80.00%,引物对5'-GTACACACAC-3'+5'-GGGGTGACGA-3'、5'-GTACACACAC-3'+5'-GGACACCACT-3'、5'-GCACACACAC-3'+5'-AAGACCGGGA-3'、5'-GCTCACTCTCTC-3'+ 5'-GACAGTCCCT-3'和5'-GTTCTCTCTC-3'+5'-TGTGGACTGG-3'扩增DNA谱带多态性比率均为100.00%。
2.2 居群遗传多样性
用PopGen-1.32软件对7个石榴居群DNA谱带进行分析,结果(表3)显示:7个居群多态位点百分率(P)分布范围为32.28% ~65.63%,其中山东居群多态位点百分率最大,为65.63%,其对应的等位基因数、有效等位基因数、Nei’s遗传多样性指数、Shannon多样性指数也最大,分别为:1.653 5、1.314 3、0.191 2、0.296 6,表明该居群遗传多样性最为丰富;陕西、云南、河南、安徽、四川、新疆多态位点百分率由大到小依次为:64.57%、62.30%、60.63%、57.48%、36.22%、32.28%,表明各居群遗传多样性依次为:山东居群>陕西居群>云南居群>河南居群>安徽居群>四川居群>新疆居群。

表3 居群间遗传多样性Table 3 Genetic diversity of pomegranate populations
2.3 居群间的遗传距离及遗传分化
用PopGen-1.32对7个石榴居群DNA谱带进行分析,得出各居群间的Nei’s遗传相似系数(Sg)和遗传距离(Dg)(表4)显示,各居群间的Nei’s遗传相似系数很高,变化范围为0.912 0~0.980 6,各居群间的遗传距离很近,变化范围为0.019 6~0.092 1。其中山东居群与四川居群的亲缘关系最远,遗传距离为0.092 1,遗传相似系数为0.912 0;云南居群与陕西居群亲缘关系最近,遗传相似系数为0.980 6,遗传距离为0.019 6。

表4 7个石榴居群间的遗传距离及遗传相似系数Table 4 Coefficient of genetic similarity and genetic distance among seven pomegranate populations
7个石榴居群总基因多样性系数(Ht)为0.188 7,居群内多样性系数(Hs)为0.154 1,遗传分化系数(Gst)为0.183 0,表明分布在居群间的遗传变异占总遗传变异的18.30%,居群间遗传分化较弱;居群间基因流的估测值(Nm)为2.231 5,Nm>2,表明居群间存在较强的基因流。
3 讨论
居群是生活在一定时间与空间上的所有同种个体的总和,是进化的基本单位。本研究结果表明,石榴在中国7个主产区居群的遗传多样性依次为:山东居群﹥陕西居群﹥云南居群﹥河南居群﹥安徽居群﹥四川居群﹥新疆居群。山东居群遗传多样性最丰富,其地理位置临近陕西,从陕西引种后,石榴可能对山东生态环境因子各异的驱动力及人为选择压力等因素产生应答,在较短的时间尺度内促进生物物种的多样化进程[14-15],因而具有丰富的遗传多样性。石榴在陕西的栽培历史最为悠久,发生突变、选择等时间也较长,其遗传多样性位于其次。由于基因流更容易从遗传多样性较高的居群进入遗传多样性较低的居群[16-17],因此,山东和陕西可能是中国石榴传播中心。
在基因流相关的适应性进化和多样化进程中,新的或变化的生态环境因子为不同的遗传来源提供了新的选择压力和适应性进化机会,一旦种内、种间的基因流积极应答复杂的生态环境因子及生态各异的选择压力与驱动力,在较短的时间尺度内也会促进生物物种的适应性进化和多样化进程[15]。本研究中7个居群石榴间的遗传分化系数(Gst)为0.183 0,表明分布在居群间的遗传变异占总遗传变异的18.30%,居群内遗传变异占总变异的81.70%,可能是中国石榴的7个主栽培区自然环境多样化,为居群内变异提供了有利条件,因而遗传变异主要存在与居群内。生物界普遍认为居群间基因流(Nm)>1的基因流就足以抵制居群由遗传漂变引起的遗传结果,维持遗传变异的多样性[14,17],本试验7个石榴居群间基因流的估测值(Nm)为2.231 5,表明居群间存在较强的基因流。石榴为异花授粉,从栽培石榴的7个主产区地理位置看,花粉传播形成基因流的可能性很低,应主要为人工引种完成遗传物质的迁移,形成基因流,而过强的基因流可能会导致当地基因型的同质化或更换,并破坏当地物种的适应性[18-20],因此,各地在进行引种石榴丰富当地遗传多样性时,同时要注意地方品种资源的保护。
[1]张建成,曲红征,张晓伟.中国石榴的研究进展[J].河北林果研究,2005,20(3):265-268.
[2]AWAMLEH H,HASSAWI D,MIGDADI H,et al.Molecular characterization of pomegranate(Punica granatum L.)landraces grown in Jordan using amplified fragment length polymorphism markers[J].Biotechnol,2009,8:316-322.
[3]PIRSEYEDI S M,VALIZADEHGHAN S,MARDI M,et al.Isolation and characterization of novel microsatellite markers in pomegranate(Punica granatum L.)[J].Int J Mol Sci,2010,11:2010-2016.
[4]汪小飞.石榴品种分类研究[D].南京:南京林业大学,2007.
[5]杨荣萍,龙雯虹,张 宏,等.云南25份石榴资源的RAPD分析[J].果树学报,2007,24(2):226-229.
[6]康美玲.田忠景石榴种质资源RAPD反应体系的引物筛选[J].江苏农业科学,2012,40(5):16-18.
[7]RANADE S A,RANA T S,NARZARY D.SPAR profiles and genetic diversity amongst pomegranate(Punica granatum L.)genotypes[J].Physiol Mol Biol Plants,2009,15(1):61-69.
[8]SAEIDEH E,BADRALDIN E,BAHRAM S.Microsatellite isolation and characterization in pomegranate(Punica granatum L.)[J].Iranian Journal of Biotechnology,2010(3):156-163.
[9]NARZARY D,RANA T S,RANADE S A.Genetic diversity in inter-simple sequence repeat profiles across natural populations of Indian pomegranate(Punica granatum L.) [J].Plant Biol(Stuttg),2010,12:806-813.
[10]赵丽华,李名扬,王先磊,等.石榴种质资源遗传多样性及亲缘关系的 ISSR 分析[J].果树学报,2011,1:66-71.
[11]苑兆和,尹燕雷,朱丽琴,等.山东石榴品种遗传多样性与亲缘关系的荧光 AFLP 分析[J].园艺学报,2008,35(1):107-112.
[12]沈 洁,徐慧君,袁英惠,等.铁皮石斛野生居群基于RAMP标记的遗传多样性评价[J].药学学报,2011,46(9):1156-1160.
[13]赵丽华,王先磊.成熟石榴叶片DNA提取方法研究[J].安徽农业科学,2009,37(31):15141-15143.
[14]刘义飞,黄宏文.植物居群的基因流动态及其相关适应进化的研究进展[J].植物学报,2009,44(3):351-362.
[15]CARROLL S P,HENDW A P,REZNICK D N.Evolution on ecological time-scales[J].Functional Ecology,2007(21):387-389.
[16]ALLEAUME-BENHARIRA M,PEN IR,RONCE O.Geographical patterns of adaptation within a species'range:Interactions between drift and gene flow[J].J Evol Biol,2006,19:203-215.
[17]TEMPLETON A R.Nested clade analyses of phylogeographic data:testing hypotheses about gene flow and population history[J].Mol Ecol,1998,7:381-397.
[18]石 欣,李 亚,杨如同,等.中国楸树(Catalpa bungei C.A.Mey)种质资源遗传多样性的ISSR分析[J].江苏农业学报,2011,27(3):634-639.
[19]GARANT D,FORDE S E,HENDRY A P.The multifarious effects of dispersal and gene flow on contemporary adaptation[J].Funct Ecol,2007,21:434-443.
[20]SCHAAL B A,HAYWORTH D A,OLSEN K M,et al.Phylogeographic studies in plants:problems and prospects[J].Mol Ecol,1998,7:465-474.
