翘嘴鳜EST-SSR标记的开发及3个群体遗传多态性分析
2013-07-03梁旭方彭敏燕于海静皮达峰
朱 滔, 梁旭方, 彭敏燕, 于海静, 皮达峰
(暨南大学生命科学技术学院生物工程学系,广东 广州 510632)
翘嘴鳜(Siniperca chuatsi)隶属于鲈形目鲈亚目,俗称桂鱼、桂花鱼、胖鳜、季花鱼等,是淡水底栖的典型肉食性鱼类.由于它具有体型大,生长快,味道鲜美等特点,深受国内外消费者喜爱,有“淡水石斑鱼”之称,目前翘嘴鳜养殖规模不断扩大,已经成为我国重要的名贵经济鱼类,具有很大商业养殖价值[1].近年来,鱼类疾病不断暴发,翘嘴鳜水产养殖业遭受严重的损失.此外,过度捕捞、干旱,尤其是水替污染导致翘嘴鳜野生种群数量明显下降.生产中近亲繁殖导致种质退化,已影响到翘嘴鳜养殖业的正常发展[2-3].因此,完善遗传标记体系对翘嘴鳜的遗传多样性,种质资源的保护以及翘嘴鳜水产养殖业的持续稳定发展具有重要意义.
表达序列标签(expressed sequence tag,EST)中存在一定数量的微卫星或简单重复序列(ESTSSR),它不仅具有在基因组中均匀分布、共显性和实验重复性好等优点,而且开发成本较低,已成功应用于群体遗传结构分析、QTL定位和分子标记辅助选择育种等研究领域[4-7].目前,用于翘嘴鳜遗传多样性研究的分子标记主要是微卫星和单核苷酸多态性 (single nucleotide polymorphism,SNP)标记.Yang等[8]、Qu 等[9]开发 EST-SSR 引物用于鳜鱼的遗传多样性研究;杨宇辉等[10]利用SNP标记翘嘴鳜的脂蛋白酯酶和胃蛋白酶并进行食性驯化分析.但是,用于群体遗传结构研究的高效微卫星标记开发目前还在初步阶段,供选择使用的微卫星标记较少,在研究野外翘嘴鳜群体结构方面则更少.因此,本研究拟从翘嘴鳜EST数据库中开发微卫星序列,筛选更多具有多态性的可供翘嘴鳜研究使用的微卫星引物,并应用于陆水水库和怀化的翘嘴鳜群体遗传结构的研究.以期为野生翘嘴鳜种质资源的保护,遗传育种和人工养殖群体的扩充等方面研究提供理论基础.
1 材料与方法
1.1 试验群体和基因组DNA的提取
试验鱼取自赤壁(湖北省)和沅江流域(湖南省)的野生翘嘴鳜群体,赤壁翘嘴鳜群体27尾,沅江流域(常德16尾;怀化18尾)翘嘴鳜群体共34尾.采样后取鳍条保存在体积分数95%的酒精之中,然后按照TIANGEN DNA提取试剂盒推荐方法提取基因组DNA.
1.2 微卫星分子标记开发及引物设计
利用本实验室自行开发的鳜鱼EST序列,从中选取重复单元为二核苷酸至六核苷酸,重复次数不少于5次的微卫星位点共120个,筛去不能设计引物的序列,使用NCBI/Primer-BLAST为剩余的91个的微卫星序列设计PCR扩增引物,相关的参数均设为默认值(引物长度18~27 bp,最适长度为20 bp;扩增退火温度57~63℃,最适温度为60℃;鸟嘌呤(G)胞嘧啶(C)含量 20% ~80%,最适含量为50%;扩增产物长度100~1 000 bp,最适长度为200 bp).所有引物均由上海生工生物工程技术服务有限公司合成.
1.3 PCR扩增和多态性检测
对筛选后的具有多态性的微卫星引物进行PCR扩增.25 μL PCR反应体系如下:10×Buffer溶液2.5 μL,每对引物0.5 μL,dNTP 0.25 μL,ddH2O20.25 μL,模版 DNA 0.5 μL 和 0.5 μL Taq 酶.PCR扩增反应条件:94℃预变性5 min,94℃变性30 s,tm℃退火1 min,72℃延伸45 s,35个循环,随后72℃延伸10 min.PCR扩增产物用琼脂糖凝胶电泳检测,获得特异的PCR扩增产物进行聚丙烯酰胺凝胶电泳银染检测,然后采用全自动凝胶成像分析系统(AlphalmagerTM)记录电泳结果.
1.4 数据统计与分析
微卫星引物扩增结果中每条带表示一个等位基因,根据微卫星扩增产物银染后显示的等位基因来统计两个翘嘴鳜群体的基因型.微卫星多态性参数由种群遗传变异分析软件POP2GENE(Version 1131)计算,包括等位基因频率、每个位点的等位基因数(Na)、有效等位基因(Ne)、观测杂合度(Ho)、期望杂合度(He)和哈迪-温伯格平衡,进行多态信息含量(PIC)的计算.
2 结果与分析
2.1 多态性EST-SSRs标记的序列信息和电泳检测结果
从合成的91对EST-SSRs引物中共筛选出22对能扩增稳定、条带清晰并且具有多态性的引物,序列信息见表1.在两个翘嘴鳜群体中共检测到134个等位基因,平均等位基因数为6.090 1个.微卫星基因座SC19电泳情况见图1.
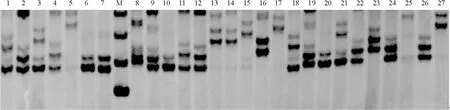
图1 引物SC19的扩增电泳图Fig.1 Electrophoresis pattern of locus SC19
2.2 群体遗传多样性分析
经分析得到翘嘴鳜群体的遗传多样性参数,结果见表2和表3.由表2可知,除了位点SC2(Ho=0.283 3)、SC3(Ho=0.203 4)和 SC15(Ho=0.169 5)以外,大部分翘嘴鳜微卫星位点遗传多样性较高,其平均观测杂合度为0.675 4,平均期望杂合度0.748 7;平均多态信息含量为0.701 0,大于0.5的多态信息含量具有高度的多态性,表明等位基因分布均匀,多态性较高,适用于进行翘嘴鳜遗传多样性的研究.根据Nei等[11]计算群体间的遗传距离和遗传相似系数,结果表明,翘嘴鳜的22个微卫星位点的Fst值为0.011 7 ~0.160 6,平均 Fst值是 0.038 3(Fst<0.05),说明翘嘴鳜群体间的分化程度较小.
由表3可知,赤壁群体的等位基因数最多(6.000 0),常德(5.409 1)与怀化(5.454 5)群体的等位基因数相当;平均观测杂合度为0.656 6~0.658 9,平均期望杂合0.710 0 ~0.744 6;多态信息含量均高于0.5,表明3个群体具有高度的遗传多样性.
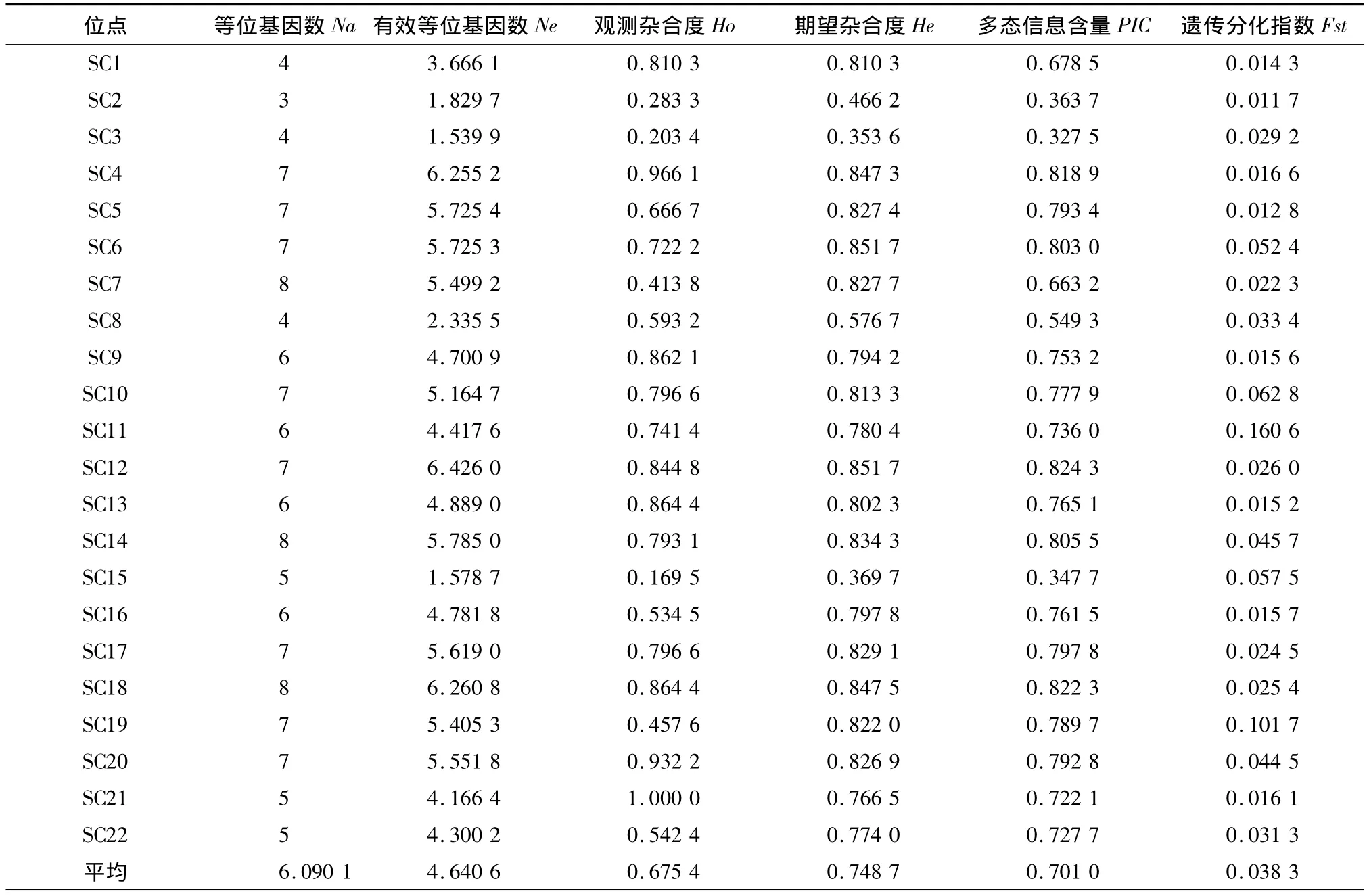
表2 22个EST-SSRs位点的遗传多样性参数Table2 Genetic diversity parameters of 22 EST-SSRs markers

表3 3个翘嘴鳜群体的遗传多样性参数Table3 Genetic diversity parameters of 3 Siniperca chuatsi populations
2.3 群体间的遗传相似度和遗传距离
遗传相似性指数和遗传距离是衡量群体亲缘关系的重要的指标.一级亲缘关系的个体间遗传相似指数为0.5,二级亲缘关系为0.25[11].根据 Nei指数法计算三个翘嘴鳜群体之间的遗传相似性指数和遗传距离见表4.由表中可知,3个群体的遗传相似度均大于0.5,说明它们之间的亲缘关系较近.其中,常德和怀化两个群体间的遗传相似度最大(0.884 2),遗传距离最短(0.123 1),这两个群体亲缘关系最近;而常德与赤壁的遗传相似度最小(0.844 0),遗传距离最大(0.169 6).
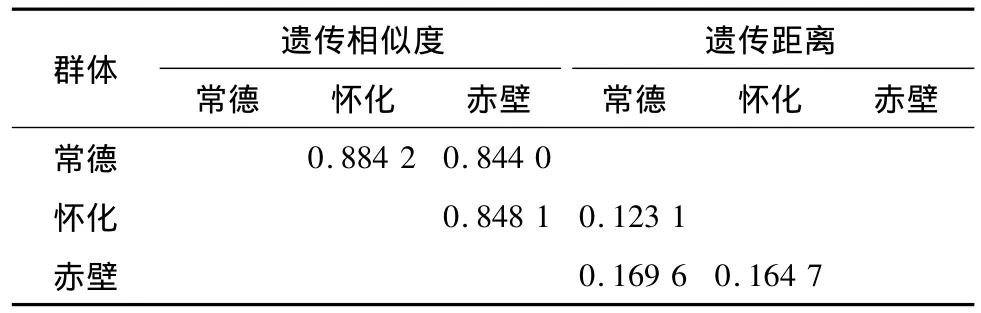
表4 3个翘嘴鳜群体间的遗传相似度和遗传距离Table4 Genetic idenity and genetic distance of 3 Siniperca chuatsi population
3 讨论
EST-SSR序列大多数与已知和未知的功能基因连锁,在生物进化中起到编码氨基酸和蛋白质的重要作用,比基因组所受到的选择压力大,其变异频率偏低、变异类型也会偏少[12].本研究所用的 ESTSSRs引物中双碱基所占的比例较大,重复序列以(AC)n/(GT)n为主(占70.4%),该结论与许多学者的研究一致.宋春妮等[13]研究发现(AC)n/(GT)n类型微卫星序列在日本蟳中含量非常丰富.李偲等[14]在草鱼EST-SSRs标记中开发的(AC)n/(GT)n重复位点也最多,占二核苷酸重复位点总数的50.3%.Wang和鲁翠云等[15-16]在鲤鱼 ESTs中筛选出EST-SSRs,(AC)n/(GT)n占双碱基重复类型的67.57%.
等位基因数、观测杂合度、期望杂合度和多态信息含量等都是反映群体遗传基础多样性水平的指标,是基因丰富度的体现,其数值越大,说明基因丰富度越高,遗传潜力越大[17].多态信息含量(PIC)起衡量基因片段多态性的作用,当 PIC>0.5时,该座位为高度多态性座位;0.25<PIC<0.5时,为中度多态性座位;PIC<0.25时,为低度多态性座位[18].本实验中22个微卫星位点的遗传多样性较为丰富,其观测杂合度和期望杂合度分别是0.675 4和0.748 7.所有位点中除了位点SC2、SC3和SC15的PIC值较低,属于中度多态性座位,其余的位点均为高度多态性座位,平均多态信息含量为0.701 0,表明其等位基因分布较均匀,多态性较高,适合用于进行翘嘴鳜群体的遗传多样性研究.对3个翘嘴鳜群体的遗传参数进行统计,各群体的杂合度较高,PIC值也显示了高度多态性.
遗传分化指数(Fst)是反映亚群间遗传分化的重要指标.本研究中群体平均Fst值为0.038 3,揭示了翘嘴鳜群体遗传分化程度较低.根据Nei's指数法得到翘嘴鳜群体间的遗传相似性指数和遗传距离,常德和怀化两个群体间的遗传相似度最大,遗传距离最短;而赤壁与常德和怀化两个群体的遗传相似度较小,遗传距离较大,数值相当.说明了常德和怀化群体的亲缘关系较近,而与赤壁的亲缘关系较远.这与常德和怀化群体来自湖南省的沅江流域,而赤壁群体来自湖北省的赤壁有关.王解芳等[19]根据5个草鱼群体的遗传距离构建了UPGMA树,结果长江水系的长沙群体、石首群体与监利群体聚成一支;珠江水系的肇庆群体与清远群体聚成一支,这也恰好证明了遗传距离与其地理分布情况基本一致.此外,运用F-检测分析群体间的亲缘关系可避免生产养殖中近亲繁殖导致的种质退化.
本研究从翘嘴鳜的EST文库中开发并筛选出具有多态性的微卫星标记共22个,绝大多数位点在3个翘嘴鳜群体中表现出了较高的遗传多样性,并适用于进行群体间的亲缘关系分析.这为以后翘嘴鳜的遗传育种和人工养殖群体的扩增提供了理论依据,且对翘嘴鳜野生种质资源的保护具有重要的意义.
[1] 梁旭方.国内外鳜类研究及养殖概况[J].水产科技情报,1996,23(1):13-17.
[2] 黄志坚,何建国.鳜鱼疾病的研究概括[J].水产科技情报,1999,26(6):268-271.
[3] HE J G,ZENG K,WENG S P,et al.Experimental transmission pathogenicity and physical-chemical properties of infectious spleen and kidney necrosis virus[J].Aquaculture(ISKNV),2002,204(1/2):11-24.
[4] POWELL W,MACHRAY G C,PROVAN J.Polymorphism revealed by simple sequence repeats[J].Trends in Plant Science,1996,1(7):215-222.
[5] SUBRAMANIAN S,MISHRA R K,SINGH L.Genome wide analysis of microsatellite repeats in humans:their abundance and density in specific genomic regions[J].Genome Biology,2003,4(2):1301-1310.
[6] SKAALA O,HOYHEIM B,GLOVER K,et al.Microsatellite analysis in domesticated and wild Atlantic salmon(Salmosalar L):allelic diversity and identification of Individuals[J].Aquaculture,2004,240(1/4):131-143.
[7] 林 琳,李月琴,谢卫兵,等.中国人群eNOS基因内含子13微卫星多态性的初步研究[J].暨南大学学报:医学版,2000,21(4):20-23.
[8] YANG Min,LIANG Xufang,TIAN Changxu,et al.Isolation and characterization of fifteen novel microsatellite loci in golden mandarin fish(Siniperca scherzeri)Steindachne[J].Conservation Genetics Resources.2012,4(3),599-601.
[9] QU C M,LIANG X F,HUANG W.Isolation and characterization of 46 novel polymorphic EST-simple wequence repeats(SSR)markers in two sinipercine fishes(siniperca)and cross-species amplification[J].International Journal of Molecular Sciences,2012,13(8):9534-9544.
[10] 杨宇辉,梁旭方,方荣,等.鳜脂蛋白酶基因SNP及其与食性驯化相关性分析[J].遗传,2011,33(3):996-1002.
[11] NEI M,LI W H.Mathematical model for studying genetic variation in terms of restriction endonucleases[J].Proceedings of the National Academy of Sciences of the United States of America,1979,76(10):5269-5273.
[12] 张琼,刘小林,李喜莲,等.EST-SSR分子标记在水生动物遗传研究中的应用[J].水产科学,2010,29(5):302-306.
[13] 宋春妮,李 健,刘 萍,等.日本蟳微卫星富集文库的建立与多态性标记的筛选[J].水产学报,2011,35(1):35-42.
[14] 李 偲,刘 航,黄 容,等.草鱼Ⅰ型微卫星标记的发掘及其多态性检测[J].水生生物学报,2011,35(7):681-686.
[15] WANG D,LIAO X L,CHENG L,et al.Development of novel EST-SSR markers in common carp by data mining from public EST sequences[J].Aquaculture,2007,271(1/4):558-574.
[16] 鲁翠云,全迎春,李大宇,等.用鲤鱼EST-SSRs分子标记分析长江黑龙江鲤种群结构[J].农业生物技术学报,2007,15(6):947-952.
[17] 侯 宁,张 研,鲁翠云,等.微卫星DNA标记分析德国镜鲤的遗传潜力[J].遗传,2007,29(12):1509-1518.
[18] NEI M.Molecular evolutionary genetics[M].New York:Columbia University Press,1987:121-134.
[19] 王解放,于凌云,白俊杰,等.草鱼EST-SS标记5个不同地域群体的遗传结构分析[J].动物性杂志,2011,46(5):24-32.
