雌激素受体α36 亚型配体结合区的结构模建、优化及验证
2013-03-08金宏威刘振明张亮仁
王 东,金宏威,刘振明 ,张亮仁
(北京大学 药学院 天然药物及仿生药物国家重点实验室,北京100191)
雌激素是一类重要的内分泌调节分子,在体内通过与雌激素受体(estrogen receptor,ER)作用,而与组织发育、生殖功能中的多种生理过程相关,是重要的内分泌调节分子[1]。已知的研究表明,雌激素与乳腺癌、卵巢癌、结肠癌等多种癌症的发生与治疗关系密切[2-6]。雌激素替代物、雌激素受体选择性调节剂(selective estrogen receptor modulator,SERM)等可以调控雌激素功能的分子,在乳腺癌以及更年期后妇女骨质疏松的防治方面有着广泛的应用,同时SERM 还具有一定的心血管保护能力[5-8]。目前已经有多个SERM上市或处于研究中,如他莫昔芬[5]、雷洛昔芬等[7]。
雌激素受体可以分为α、β 两个亚型[9],上市或处于研究中的SERM 主要通过分子量为66 kDa 的雌激素受体亚型ER-α66 发挥药理作用。2005 年,王兆一教授课题组发现了一种新的雌激素受体亚型ER-α36[10]。该亚型与ER-α66在染色体上的编码基因相同,但经过不同的mRNA 剪辑而形成了新的分子量为36 kDa 的受体蛋白。不同于主要分布于胞浆中的ER-α66,ER-α36 主要分布在细胞膜上[10-11]。已知研究结果表明,ER-α36 具有独特的配体结合谱及配体响应机制[11];它对目前已有的雌激素受体激动剂和拮抗剂均有响应,可快速激活ERK(extracellular signal-regulated kinase)信号通路,被认为介导了雌激素受体的非基因组快反应机制[11-13]。同时,ER-α36 可以通过非配体依赖的方式,抑制ERα66 和ER-β 的活性[11,14-15]。生理学研究发现,在ER-α66 阳性或者阴性的乳腺癌患者体内,都可以发现ER-α36[16],已有研究结果表明,ER-α36也与子宫内膜癌、食道癌、白血病以及骨质疏松存在着密切关联[17-19]。ER-α36 在SERMs 的耐药性效应产生中也有重要影响[20]。
目前,已有针对ER-α36 的调控分子处于临床Ⅱ期研究阶段(进展未发表),但这些分子在体内如何通过选择性或者多靶点相互作用产生治疗效果的分子机制有待进一步的研究。由于ERα36 的三维结构尚未被解析,制约了选择性雌激素调控分子的深入研究。因此,对ER-α36 的模建、分析其蛋白结构特征以及配体结合特征、模拟其配体结合模式对ER-α36 作用机制的研究以及新配体设计有重要意义。
本研究工作希望通过同源模建方法,建立ER-α36 的配体结合区域(ligand binding domain,LBD)三维结构,并进一步对其结构特征以及与雌激素受体亚型ER-α66 之间的异同进行比较分析。由于已知的ER-α66 LBD 存在与激动剂和拮抗剂结合的两种空间结构,本文将分别建立对应的ER-α36 LBD 同源模型。与此同时,将采用动力学模拟和分子对接分析方法对建立的受体-配体复合物模型进行进一步的优化与验证。
1 材料与方法
1.1 同源模建
雌激素受体亚型ER-α36 的序列来自王兆一教授发表的论文[10]。由于本次模建的主要目标是探索ER-α36 的配体结合区域特征,参照已知的ER-α66 LBD 的晶体结构[21-22],截取了ERα36 LBD 序列区域进行模建,包括Leu133 ~Gly284 共152 个氨基酸。
ER-α66 LBD 已解析得到的晶体结构较多,按配体作用分类,存在与激动剂和拮抗剂结合的两大类晶体复合物结构。本文作者同时采用了激动剂雌二醇(17β-estradiol,E2)复合物(PDB 编码:1ERE)[21],以及拮抗剂4-羟基-他莫昔芬(4-hydroxy-tamoxifen,OHT)复合物(PDB 编码:3ERT)[22]作为同源模建的模板。两个模板均用Discovery Studio 中的Clean Protein 功能修补了残缺氨基酸,1ERE 模板中缺失的Leu462 ~Ser464 段不在对比序列范围内,Tyr331 ~Pro336最近处与结合口袋距离超过1.3 nm,且本身为无规则卷曲,因而由建模程序自行优化最终模型中该段氨基酸结构。
序列比对工作采用Discovery Studio 2.5 软件包完成[23]。由于ER-α36 与ER-α66 的高度同源性,采用手动的方法对齐了两个受体序列中的对应氨基酸残基。ER-α36 C 端的26 个氨基酸Ile285 ~Val310 无法对齐到可用的模板,且该段氨基酸对138 个氨基酸缺失已经形成的结构改变影响较小,故被排除在模建结构以外,详细讨论见结果与讨论部分。同源模建通过Discovery Studio软件包中的Build Homology Model 模块完成。优化参数“Optimization Level”设置为High,禁止软件自动优化Loop 区。分别针对激动剂和拮抗剂结合生成5 个同源模建模型,取DOPE(discrete optimized protein energy)分数[24]绝对值最大的模型,进行下一步的配体-受体复合物动力学优化研究。
1.2 分子动力学优化
以同源模建得到的雌激素受体亚型ER-α36的三维结构为基础,使用Gromacs 3.3.3 分子动力学模拟软件包[25]对结构模型进行进一步的优化处理。以Discovery Studio 软件包完成分子坐标叠合操作。配体的构象优化以及电荷分布的计算采用Gaussian 09 程序完成[26],其结果用于建立配体的Gromos96 力场参数[27]。PRODRG Server v 2.5 beta[28]用来生成配体力场的拓扑文件。
配体力场的准备:在Gaussian 09 程序中导入激动剂E2、拮抗剂OHT 的结构,通过半经验方法AM1 分别对两个配体进行结构优化和电荷计算。通过Amber[29]中的Xleap 对照导出配体的电荷分布。以PRODRG Server v2.5 beta 生成配体的Gromos96 力场文件,并导入Gaussian 09 计算得到的电荷分布结果。
受体文件的准备:模建得到的结构在Discovery Studio 中。以口袋附近氨基酸Glu180 ~Arg221 为参照,与已知ER-α66 模板对应位置Glu353 ~Arg394 对齐,并加入ER-α66 中参与配体结合作用的水分子。然后,选用Gromacs 的pdb2gmx 模块设定Gromos96 力场,并通过Discovery Studio 软件加入配体。复合物结构用genbox 模块加入适量的水分子,水分子采用简单点电荷(SPC)模型[30]。采用genpr 模块加入抗衡离子,保证模拟体系的电中性。
对于初始模建结构,使用最陡下降法(steepest descend)对体系进行能量优化。然后在300 K 恒定温度下进行束缚分子动力学模拟计算:首先束缚配体-受体复合物的所有原子,平衡体系中的溶剂分子;溶剂分子平衡后,依次对配体与蛋白侧链、α 碳原子之外的蛋白质骨架原子、α碳原子解开束缚。各阶段分别进行50 ps 的动力学模拟,降低体系能量。
具体参数设置:步长2 fs,使用Berendsen等[31]提出的压力-温度耦合,耦合时间0.1 ps。体系压强保持在105Pa,采用0.5 ps 耦合时间模拟自由水。模拟水分子的等温压缩系数设定为4.5 ×10-10Pa-1。用LINCS(linear constraint solver)算法[32]约束所有原子的键长,计算距离小于1 nm 的带电基团之间的静电相互作用。用PME(particle-mesh ewald)方法[33]计算长程静电相互作用,格点宽度设为0.12 nm;Lennard-Jones 相互作用的截断距离设为1 nm。
最终,对模建的E2-ER-α36 复合物和OHTER-α36 复合物结构分别进行了总长度5 ns 的分子动力学模拟。采用Discovery Studio 的Verify Protein(MODELLER)模块计算DOPE(discrete optimized protein energy)分值[24],Verify Protein(Profiles-3D)模块计算Profiles-3D 分值[34],并用拉氏图(Ramachandran)功能[35-36]对生成的蛋白质结构进行验证。本研究中用到的模型命名见表1。

Table 1 Protein structure nomenclature in current study
1.3 分子对接验证及条件摸索
为了对ER-α36 的模建结构进行验证并对配体结合口袋特征进行分析,研究采用AutoDock 4.0[37]对雌激素受体亚型ER-α36 目前已知的4 个配体E2、OHT、genistein 以及G-1 与模建受体结构进行了分子对接研究。各分子结构式见图1。
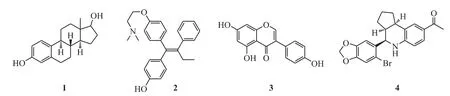
Figure 1 Ligands used in current study
配体分子通过AutoDockTools[37]工具加氢加Gasteiger 电荷[38],并分配原子类型。对接受体为t3g(3ERT 作为模板的ER-α36 结构),并同样在AutoDock Tools 中加氢加Gasteiger 并分配原子类型。对接格点中心以配体坐标定位,x、y、z各轴上的对接格点边长均为40 个单位,每单位为0.0375 nm,组成1.5 nm ×1.5 nm ×1.5 nm 的对接格点。通过AutoGrid 程序进行对接格点计算后,由AutoDock 程序完成对接以及相互作用能评价。ga_num_evals 参数设为25 000 000,ga_num_generatoins 设为27 000,其他参数取默认值。对接结果在Discovery Studio 中进行结合模式的评价,并讨论ER-α36 的配体口袋特征。
2 结果与讨论
2.1 ER-α36 同源模建及结构优化
图2 显示了ER-α36 LBD 与ER-α66 LBD 序列的比对结果。由于基因序列本身与ER-α66 序列同源,仅是mRNA 剪辑有所差异,ER-α36 LBD N 端的大部分一级序列结构与ER-α66 并无区别,仅有的区别在于ER-α36 C 端的26 个氨基酸。由于剪辑变异,这26 个氨基酸取代了原ER-α66 LBD 中C 端的138 个氨基酸,其序列来源为ERα66 基因ESR1 下游64 141 bp 处[10]。从Val458对应的位置开始,ER-α36 中的这部分序列被Ile285 ~Val310 共26 个氨基酸取代,所以无法形成ER-α66 原有的四段α 螺旋结构。
Ile285 之前的Gly284 是最后一个有可靠模板位置信息的氨基酸。该氨基酸位于蛋白H8 的C 端,与口袋内壁相隔H8 以及H5 两条α 螺旋结构,距离较远。在模型结构中测量则发现,Gly284中最靠近结合口袋的α-C 原子与结合口袋壁中最近的Trp210 侧链末端距离约为1.5 nm。相对维系蛋白质的非键相互作用,该距离较大[39]。另外,对于26 个氨基酸本身,以BLAST(basic local alignement search tool)方法无法找到对应的模板构建其结构[40],PSIPRED 方法也无法通过一级序列特征预测其二级结构[41],其形成规则二级结构的可能性极低。综上,作者认为ER-α36 LBD 中该段氨基酸对138 个氨基酸缺失已经形成的结构改变影响较小,难以影响结合口袋中的几何以及电性环境,因此,模建中排除了末端的26 个氨基酸残基。通过同源模建,得到了E2-ER-α36(t1)和OHT-ER-α36(t3)两个结构。图3 为t1 和1ERE 的结构示意图。

Figure 2 Sequence alignment of ligand binding domain(LBD)of ER-α36 and ER-α66(PDB∶1ERE)
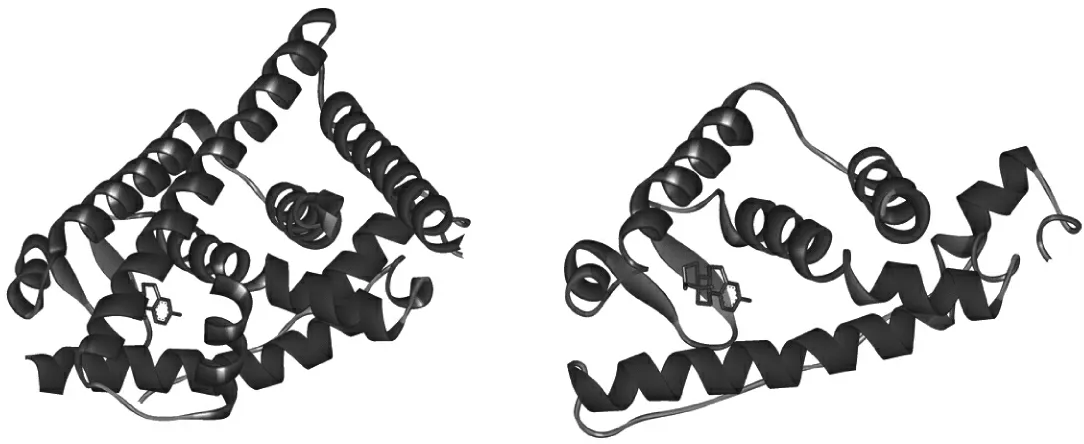
Figure 3 Structures of ER-α36 and ER-α66 LBD
以t1 和t3 作为初始结构,作者对两个分子进行了5 ns 的分子动力学模拟研究。从计算得到的RMSD 曲线(图4)可以看到,两个体系的RMSD 值分别在0.5 ns 和1.5 ns 前持续上升,然后趋于稳定,分别在0.2 nm 和0.25 nm 附近上下波动,说明得到了稳定结构。图中显示RMSD 值很小,说明动力学结构相较于初始结构,并无较大差异。作者将动力学后3 ns 模拟时间内的结构进行平均,并通过能量优化得到最终的ER-α36复合物结构(t1g 和t3g),供下一步分析。
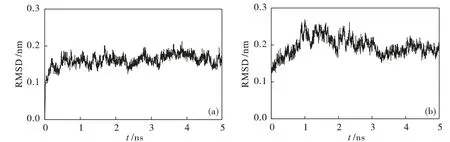
Figure 4 Root mean square deviation(RMSD)of(a),t1g(b)t3g during 5 ns molecular dynamics simulation
通过拉氏图可以考察模建结构中各氨基酸残基的二面角分布情况,从而判断蛋白结构的合理性[35-36]。在图5 中可以看到,模型t1g 和t3g 的拉氏图中,仅有个别氨基酸位于非最优区,分别占激动构象的1.3%及拮抗构象的2.6%,说明该模建的结构较为合理。
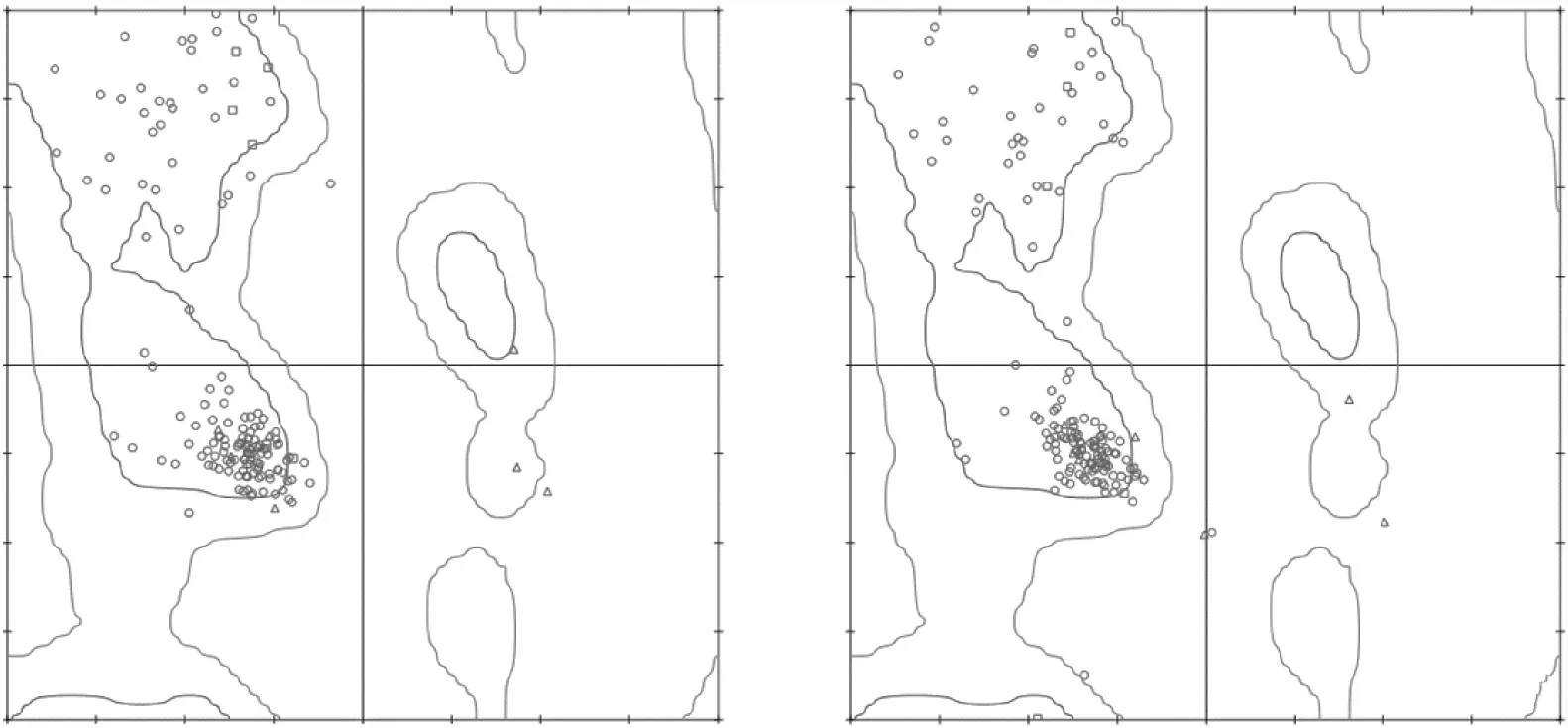
Figure 5 Ramachandran plot of agonist-and antagonist-template modeled ER-α36 LBD
DOPE 打分以及Verify Protein 模块评价同样也支持了当前模建蛋白结构的合理性。各评价模型的打分情况见表2。
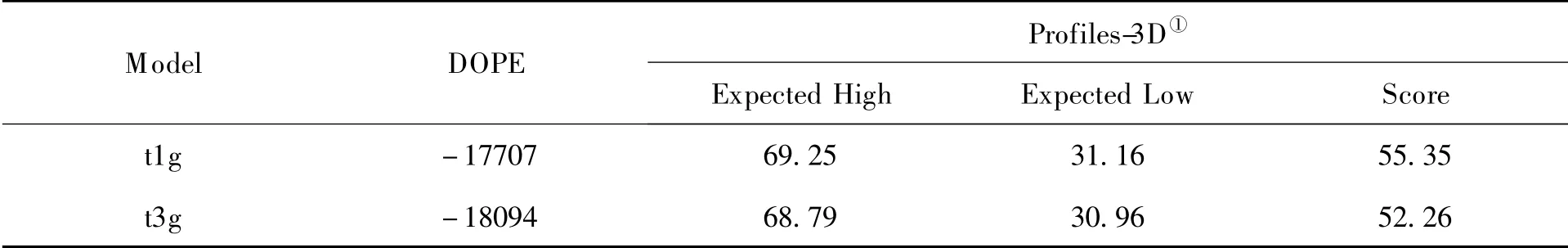
Table 2 Quantitative structure evaluation of different ER-α36 LBD models after molecular dynamics simulation
2.2 ER-α36 结合口袋的特征分析
关于ER-α66 LBD 已有大量深入的结构研究[21,42-44]。在与不同配体结合时,其结合口袋内的残基构象变化很小。ER-α66 LBD 口袋内部为H3、H6、H8、H11 和H12 等α 螺旋所包围的强疏水结构。配体一般包含一个甾体骨架,以及两个其间隔相距约1.1 nm 的羟基。其中一侧为酚羟基,苯环平面与口袋一端的疏水夹子相吻合[43],另一端His524 与甾体骨架另一侧的羟基发生极性相互作用。在结合激动剂和拮抗剂的不同情况下,口袋附近H11-H3 处的氢键网络发生变化,引发口袋盖子H12 构象改变[45],形成不同的蛋白相互作用平面,与下游信号分子发生蛋白-蛋白相互作用[46-47]。
图6 展示了作者构建的ER-α36 模建结构。可以看到在ER-α36 的配体口袋附近,H3、H6 和H8 得以保留,但H11 和H12 则随着转录剪辑变异消失(图3),这对ER-α36 的结构产生了较大的影响。图6b 展示了ER-α36 结合位点附近的氨基酸,可见位于H11 的有着重要氢键作用的氨基酸His524 以及另外两个氨基酸Gly521 和Met528从ER-α36 的结构中消失。随之而来的是整个口袋的一端失去了特征性的氢键作用中心以及位阻特征,从具有氢键定位特征的疏水口袋端点转化为暴露于溶剂之中的裸露环境,这样的结构导致ER-α36 的结构牢固性降低、柔性增大。
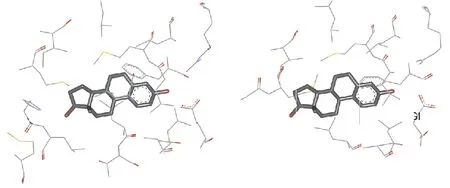
Figure 6 Binding pocket comparison of ER-α66 and ER-α36
使用LigBuilder V2.0 软件[48]中的Cavity 模块进行口袋分析,可以给出关于口袋特征更详细的定量数据以及形状分析。t1、t1g、t3 和t3g 配体结合口袋的定量信息列于表3。图7 展示了4 个受体模型经过Cavity 软件分析后生成的口袋特征图。
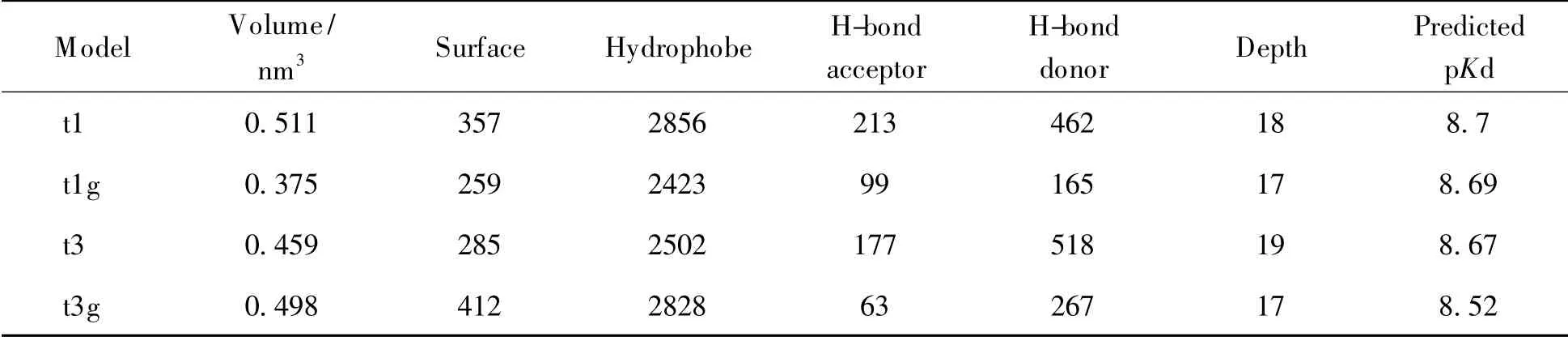
Table 3 Quantitative measures of binding pockets of different ER-α36 models by Cavity module in LigBuilder V2.0
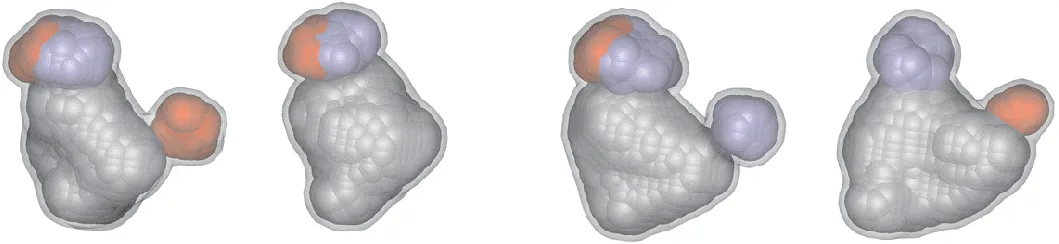
Figure 7 3D fill-in of different ER-α36 models' binding cavity produced by Cavity module in LigBuilder V2(a)t1,(b)t1g,(c)t3,(d)t3g
从口袋的定量数据对比中可以发现,虽有相同的一级结构,但复合物配体的不同导致了ER-α36 口袋结构在动力学模拟后发生了较大变化。在口袋体积方面,激动构象由0.511 nm3降低到0.375 nm3,拮抗构象由0.459 nm3变化为0.498 nm3。在氢键作用点体积方面,激动构象和拮抗构象的各项氢键作用点体积都减少了50%以上。口袋填充模型图上也可以直观地发现,两个受体结构通过动力学模拟的过程,发生了不同形式的形变。原本相似的口袋形状以及药效团特征,在动力学模拟之后变得有较大差异,体现了结合口袋很大的柔性。由于没有了ERα66 中的H11,结合口袋的一端失去了这一α 螺旋的覆盖和支撑,原本被氨基酸占据的空间现被溶剂填充,两侧氨基酸活动的自由度显著增大,口袋该侧的残基间极性相互作用显著减少。对于结合拮抗构象的ER-α36 模型t3,其结合的配体分子OHT 含有拮抗剂所特有的侧链。因此其在动力学模拟中,口袋边缘的氨基酸被OHT 的侧链不断碰撞而不致显著缩小,反而在5 ns 的动力学模拟之后发生了显著增大。
由于一端缺失了3 个重要的氨基酸His524、Gly521 和Met528,结合口袋这一侧的开口显著变大。在OHT 与口袋开口边缘天冬氨酸的相互作用中,明显体现了开口变大带来的影响。作为典型ER-α66 拮抗剂,OHT 的骨架中部延伸出含有季铵氮原子的侧链,与口袋边缘的极性残基发生作用。该作用在ER-α66 晶体结构以及ER-α36模建结构中分别对应Asp351(图8a)以及Asp178(图8b)。未经分子动力学优化的ER-α36 结构中,这一相互作用与ER-α66 相比没有明显变化。但经过动力学优化,ER-α36 配体结合口袋边缘逐渐松散。由于缺乏H11 的支撑以及H12 提供的相互作用,自由活动的配体很难保持与Asp178的紧密相互作用,因而该氢键特征在动力学优化后的ER-α36 模型中逐渐消失。

Figure 8 Comparison of pocket rim environment between ER-α66 and ER-α36
2.3 分子对接验证
这一工作是将已知的ER-α36 配体对接入现有的ER-α36 模建结构中,通过评价对接构象和对接打分,验证模型的合理性并探索ER-α36 特有的结合模式。本文中用于进行对接的配体是E2、OHT、genistein、G-1 四个分子,之前的研究均表明可以通过ER-α36 发生配体依赖的作用[11-12]。其中E2、OHT 分别是经典的ER-α66激动剂和拮抗剂的配体[21-22]。genistein 为ER-β选择性激动[49],G-1 则是新报道的ER-α36 膜转导激活配体[12-13,50]。对接模拟中用到的受体为拮抗构象模板的ER-α36 结构t3g。2.2 节中已讨论,与激动构象相比,拮抗构象的口袋边缘更宽大,易于容纳不同大小的配体。故在本部分中使用该构象进行对接。
配体分子与ER-α36(t3g)的结合模式示意图见图9。如图所示,各配体酚羟基均与结合口袋内部的Arg221、Glu180 发生了氢键相互作用,同时疏水性骨架部分也与口袋中的疏水性残基有合理的相互作用。各配体的另一端,则由于缺少了ERα66 中的H11 和H12,位置有一定差异。这源于ER-α36 结构变化所造成的结合位点开口变大。OHT 可与口袋边缘的Asp178 产生一定的极性相互作用。这点是目前其他已知配体所不具备的。

Figure 9 Interaction pattern of different ligands with ER-α36
以上的对接结果与目前已报道的实验结果相符,这支持了本研究中所建立的ER-α36 结构。已知分子与ER-α36 的结合模式、其配体取向以及口袋内侧相互作用情况与ER-α66 中基本一致,但由于口袋外侧的结构变化,配体另一端取向较不固定。
3 结论
本文通过同源模建方法初步建立了ER-α36 LBD 结构模型。现有的蛋白结构打分、动力学模拟方法以及已发表的配体结合实验数据,为本模型提供了支持。通过该模型,作者发现了相对ER-α66 的一级结构差异所导致的H9-H12 等重要的二级结构在ER-α36 中缺失。ER-α36 结合口袋内侧的化学环境没有改变,但口袋另一端的α 螺旋结构H11 消失,产生了较大的溶剂暴露面。ER-α36 LBD 部分的结构刚性下降,在不同配体的结合下可能有较大的结合口袋构象差异。通过初步探究ER-α36 的配体结合模式发现,口袋内部的氢键相互作用以及口袋中的疏水环境与ERα66 很相似,但口袋外侧允许配体残基更大,或进行更自由的活动。以上结果为进一步研究ERα36 提供了基础。
[1] DEROO B J,KORACH K S.Estrogen receptors and human disease[J]. J Clin Invest,2006,116(3):561 -570.
[2] CALLE E E,MIRACLE-MCMAHILL H L,THUN M J,et al. Estrogen replacement therapy and risk of fatal colon cancer in a prospective cohort of postmenopausal women[J].J Natl Cancer Inst,1995,87(7):517 -523.
[3] HENDERSON B E,FEIGELSON H S.Hormonal carcinogenesis[J]. Carcinogenesis,2000,21(3):427 -433.
[4] LEVIN E R.Invited review:cell localization,physiology,and nongenomic actions of estrogen receptors[J].J Appl Physiol,2001,91(4):1860 -1867.
[5] WICKERHAM D L,FISHER B,WOLMARK N,et al. Association of tamoxifen and uterine sarcoma[J].J Clin Oncol,2002,20(11):2758 -2760.
[6] FABIAN C J,KIMLER B F. Selective estrogen-receptor modulators for primary prevention of breast cancer[J].J Clin Oncol,2005,23(8):1644 -1655.
[7] ETTINGER B,BLACK D M,MITLAK B H,et al.Reduction of vertebral fracture risk in postmenopausal women with osteoporosis treated with raloxifene[J].JAMA,1999,282(7):637 -645.
[8] WOOD A J,RIGGS B L,HARTMANN L C.Selective estrogen-receptor modulators-mechanisms of action and application to clinical practice[J].New Engl J Med,2003,348(7):618 -629.
[9] HELDRING N,PIKE A,ANDERSSON S,et al. Estrogen receptors:how do they signal and what are their targets[J]. Physiol Rev,2007,87(3):905 -931.
[10] WANG Z-Y,ZHANG X-T,SHEN P,et al. Identification,cloning,and expression of human estrogen receptor-α36,a novel variant of human estrogen receptor-α66[J]. Biochem Biophys Res Commun,2005,336(4):1023 -1027.
[11] WANG Z-Y,ZHANG X-T,SHEN P,et al.A variant of estrogen receptor-α,hER-α36:transduction of estrogen-and antiestrogen-dependent membrane-initiated mitogenic signaling[J]. Proc Natl Acad Sci,2006,103(24):9063 -9068.
[12] KANG L-G,ZHANG X-T,XIE Y,et al.Involvement of estrogen receptor variant ER-α36,not GPR30,in nongenomic estrogen signaling[J].Mol Endocrinol,2010,24(4):709 -721.
[13] NOTAS G,KAMPA M,PELEKANOU V,et al. Interplay of estrogen receptors and GPR30 for the regulation of early membrane initiated transcriptional effects:a pharmacological approach[J]. Steroids,2012,77(10):943 -950.
[14] ZHANG J,LI G-L,LI Z-Q,et al. Estrogen-independent effects of ER-α36 in ER-negative breast cancer[J].Steroids,2012,77(6):666 -673.
[15] ZOU Y,DING L,COLEMAN M,et al. Estrogen receptor-alpha(ER-α)suppresses expression of its variant ER-α36[J].FEBS Lett,2009,583(8):1368 -1374.
[16] LEE L M J,CAO J,DENG H,et al.ER-α36,a novel variant of ER-α,is expressed in ER-positive and-negative human breast carcinomas[J]. Anticancer Res,2008,28(1B):479 -483.
[17] CHAUDHRI R, OLIVARES-NAVARRETE R,SCHWARTZ Z. Estrogen receptor alpha-36 expression and function in osteoblast plasma membranes[C].IADR 86th General Session & Exhibition,Toronto,Canada,2008.
[18] JIANG H-P,TENG R-Y,WANG Q,et al.Transcriptional analysis of estrogen receptor alpha variant mRNAs in colorectal cancers and their matched normal colorectal tissues[J]. J Steroid Biochem Mol Biol,2008,112(1/3):20 -24.
[19] LIN S-L,YAN L-Y,LIANG X-W,et al.A novel variant of ER-alpha,ER-alpha36 mediates testosteronestimulated ERK and Akt activation in endometrial cancer Hec1A cells[J]. Reprod Biol Endocrinol,2009,7:102 -110.
[20] LIN S-L,YAN L-Y,ZHANG X-T,et al. ER-a36,a variant of ER-a,promotes tamoxifen agonist action in endometrial cancer cellsviathe MAPK/ERK and PI3K/Akt pathways[J/OL]. PLoS One,2010,5(2):e9103.
[21] BRZOZOWSKI A M,PIKE A C,DAUTER Z,et al.Molecular basis of agonism and antagonism in the oestrogen receptor[J]. Nature,1997,389(6652):753 -757.
[22] SHIAU A K,BARSTAD D,LORIA P M,et al. The structural basis of estrogen receptor/coactivator recognition and the antagonism of this interaction by tamoxifen[J].Cell,1998,95(7):927 -937.
[23] Accelrys Software Inc. Discovery studio release 2.5[M].San Diego:Accelrys Software Inc,2007.
[24] SHEN M,SALI A.Statistical potential for assessment and prediction of protein structures[J]. Protein Sci,2009,15(11):2507 -2524.
[25] LINDAHL E,HESS B,van der SPOEL D. GROMACS 3.0:a package for molecular simulation and trajectory analysis[J]. J Mol Model,2001,7(8):306 -317.
[26] Gaussian Inc. Gaussian 09 revision A.1[M]. Wallingford CT:Gaussian Inc,2009.
[27] DAURA X,MARK A E,van GUNSTERN W F.Parametrization of aliphatic CHnunited atoms of GROMOS96 force field[J]. J Comput Chem,1998,19(5):535 -537.
[28] SCHUTTELKOPF A W,van AALTEN D M. PRODRG:a tool for high-throughput crystallography of protein-ligand complexes[J]. Acta Crystallogr Sect D:Biol Crystallogr,2004,60(8):1355 -1363.
[29] PEARLMAN D A,CASE D A,CALDWELL J W,et al.AMBER,a package of computer programs for applying molecular mechanics,normal mode analysis,molecular dynamics and free energy calculations to simulate the structural and energetic properties of molecules[J].Comput Phys Commun,1995,91(1):1 -41.
[30] BERENDSEN H J,POSTMA J P M,van GUNSTEREN W F,et al.Interaction models for water in relation to protein hydration[J]. Intermol Forces,1981,11(1):331 -342.
[31] BERENDSEN H J,POSTMA J P M,van GUNSTEREN W F,et al. Molecular dynamics with coupling to an external bath[J].J Chem Phys,1984,81(8):3684 -3690.
[32] HESS B,BEKKER H,BERENDSEN H J,et al.LINCS:a linear constraint solver for molecular simulations[J]. J Comput Chem,1997,18(12):1463 -1472.
[33] DARDEN T,YORK D,PEDERSEN L.Particle mesh Ewald:an N·log(N)method for Ewald sums in large systems[J]. J Chem Phys,1993,98(12):10089 -10092.
[34] LIITHY R,BOWIE J U,EISENBERG D. Assessment of protein models with three-dimensional profiles[J].Nature,1992,356(6364):83 -85.
[35] RAMACHANDRAN G T, SASISEKHARAN V.Conformation of polypeptides and proteins[J]. Adv Protein Chem,1968,23:283 -437.
[36] LOVELL S C,DAVIS I W,ARENDALL W B,et al. Structure validation by Cα geometry:ψ and Cβ deviation[J]. Proteins:Struct,Funct,Bioinfo,2003,50(3):437 -450.
[37] MORRIS G M,HUEY R,LINDSTROM W,et al.AutoDock4 and AutoDockTools4:Automated docking with selective receptor flexibility[J].J Comput Chem,2009,30(16):2785 -2791.
[38] GASTEIGER J,SALLER H. Calculation of the charge distribution in conjugated systems by a quantification of the resonance concept[J].Angew Chem(Int Ed Engl),2003,24(8):687 -689.
[39] HOOFT R W,SANDER C,VRIEND G. Positioning hydrogen atoms by optimizing hydrogen-bond networks in protein structures[J]. Proteins:Struct,Funct,Bioinfo,1998,26(4):363 -376.
[40] ALTSCHUL S F,MADDEN T L,SCH FFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Res,1997,25(17):3389 -3402.
[41] MCGUFFIN L J,BRYSON K,JONES D T. The PSIPRED protein structure prediction server[J].Bioinformatics,2000,16(4):404 -405.
[42] MORAS D,GRONEMEYER H.The nuclear receptor ligand-binding domain:structure and function[J].Curr Opin Cell Biol,1998,10(3):384 -391.
[43] BOURGUET W,GERMAIN P,GRONEMEYER H.Nuclear receptor ligand-binding domains:three-dimensional structures,molecular interactions and pharmacological implications[J]. Trends in Pharmacol Sci,2000,21(10):381 -388.
[44] ELLMANN S,STICHT H,THIEL F,et al. Estrogen and progesterone receptors:from molecular structures to clinical targets[J]. Cell Mol Life Sci,2009,66(15):2405 -2426.
[45] CELIK L,LUND J D D,SCHI TT B.Conformational dynamics of the estrogen receptor α:molecular dynamics simulations of the influence of binding site structure on protein dynamics[J]. Biochemistry,2007,46(7):1743 -1758.
[46] WÄRNMARK A,TREUTER E,GUSTAFSSON J
A,et al. Interaction of transcriptional intermediary factor 2 nuclear receptor box peptides with the coactivator binding site of estrogen receptor α[J].J Biol Chem,2002,277(24):21862 -21868.
[47] LEDUC A-M,TRENT J O,WITTLIFF J L,et al.Helix-stabilized cyclic peptides as selective inhibitors of steroid receptor-coactivator interactions[J]. Proc Natl Acad Sci,2003,100(20):11273 -11278.
[48] YUAN Y,PEI J,LAI L.LigBuilder 2:a practical de novo drug design approach[J]. J Chem Inf Model,2011,51(5):1083 -1091.
[49] MANAS E S,XU Z B,UNWALLA R J,et al. Understanding the selectivity of genistein for human estrogen receptor-β using X-ray crystallography and computational methods[J].Structure,2004,12(12):2197 -2207.
[50] BOLOGA C G,REVANKAR C M,YOUNG S M,et al.Virtual and biomolecular screening converge on a selective agonist for GPR30[J]. Nat Chem Biol,2006,2(4):207 -212.