耐受高体积分数丁醇乳酸菌的筛选及其鉴定
2013-03-03刘洪儒曹龙奎孙大庆杨春宇
刘洪儒,曹龙奎,,*,孙大庆,宋 亮,杨春宇
(1.黑龙江八一农垦大学食品学院,黑龙江 大庆 163319;2.黑龙江省农产品加工工程技术研究中心,黑龙江 大庆 163319)
耐受高体积分数丁醇乳酸菌的筛选及其鉴定
刘洪儒1,曹龙奎1,2,*,孙大庆2,宋 亮1,杨春宇1
(1.黑龙江八一农垦大学食品学院,黑龙江 大庆 163319;2.黑龙江省农产品加工工程技术研究中心,黑龙江 大庆 163319)
从不同样品中分离和筛选在较高丁醇体积分数下生长的菌株,通过不断提高培养基中的丁醇添加量测定菌株的相对生长率,经过初测和复测,选择相对生长率较高的菌株绘制其耐受曲线,分析其能够耐受丁醇的体积分数。对菌株进行常规生理生化鉴定和16S rDNA序列分析,结合菌体形态及菌落特征确立菌株的种属。筛选得到2株耐受丁醇的体积分数均达到了3%的菌株,分别为A37和A50。经鉴定,序列分析与生理生化实验结果是一致的,A37为Enterococcus faecalis,A50为Lactobacillus fermentum。结果表明两株菌具有较高的丁醇耐受性。
乳酸菌;丁醇耐受性;相对生长率;筛选;鉴定
丁醇除了作为重要的有机化工原料外,还是一种极具潜力的可再生的生物液体燃料,其热值、辛烷值与汽油相当,具有低亲水性以及不易挥发等特点[1-2]。随着石油化工生产丁醇成本的增长以及其造成的环境污染问题,使得生物发酵法生产丁醇成为新的研究热点[3]。进行丁醇发酵的微生物主要是梭状芽孢杆菌Clostridium acetobutylicum、C.beijerinckii、C.saccharoperbutylacetonicum和C.saccharobutylicum[4-5]。然而,由于丁醇对细胞的毒性,导致在发酵中其产丁醇或者耐受丁醇的体积分数均无法超过20g/L[6-8]。在很大程度上制约了丁醇发酵工业的发展,而且梭菌(Clostridium)从产酸过程到产溶剂过程的过渡中,多复合因子对该细胞代谢过程进行着复杂的调节和控制,不管是通过菌株突变还是通过代谢工程改造的途径获得发酵高产的梭菌菌株都是非常困难的[9-11]。因此有必要寻找新的菌株和进行新的发酵丁醇生产途径的研究。
目前梭菌的丁醇合成及代谢网络的研究比较清晰[12],通过构建其他菌株表达系统表达梭菌丁醇合成基因的研究已经获得成功[13-15]。但由于这些菌株本身对丁醇的耐受性不高并没有明显提高丁醇产量,因而寻找耐受高体积分数丁醇微生物是丁醇异源重组生产的关键因素。在筛选能够耐受丁醇微生物的研究中,Knoshaug等[16]通过比较24种微生物对丁醇的耐受性,发现大多数菌株耐丁醇的体积分数在1%~2%之间,几株酵母菌可以耐丁醇体积分数为2%,只有两株乳酸菌能耐丁醇体积分数达到3%,说明各种微生物对丁醇的耐受性是不尽相同的。Li等[17]筛选得到能够在丁醇体积分数为2.5%的培养基中生长的Enterococcus faecium,丁醇体积分数为3%时生长的Lactobacillus plantarum E4,说明乳酸菌可能具有固有的丁醇耐受性。左文朴等[18]得到能够耐受丁醇体积分数为25g/L的菌株Lactobacillusm ucosae btpz-4-1和Pediococcus pentosaceus btpz-6-3。
本实验通过自然筛选,从20个样品中分离能够耐受高体积分数丁醇的菌株,得到两株耐受丁醇的体积分数可达到3%的菌株,并对其进行鉴定,确定种属,为利用这些高耐受菌株构建新的丁醇生产途径提供参考。
1 材料与方法
1.1 样品采集
正常人的粪便、果园新鲜及腐烂水果、水果土壤、腌制蔬菜等可能富含乳酸菌的样本,实验室4℃保存。
1.2 培养基
MRS液体培养基(g/L):蛋白胨10.0、牛肉膏5.0、酵母膏4.0、葡萄糖20.0、乙酸钠5.0、柠檬酸二铵2.0、磷酸氢二钾2.0、MgSO4·7H2O 0.2、MnSO4·H2O 0.05,吐温-80 1.0mL,pH 6.2;MRS固体培养基:液体培养基中加入15g琼脂;筛选培养基Ⅰ:在MRS液体培养基中添加体积分数3%的正丁醇;筛选培养基Ⅱ:在MRS固体培养基中添加3%正丁醇。
1.3 试剂与仪器
正丁醇(分析纯) 天津市大茂化学试剂厂;细菌DNA提取试剂盒K713、PCR反应试剂盒 上海英潍捷基生物科技有限公司;TaqDNA聚合酶 日本TaKaRa公司。
PTC-200型PCR仪 东胜创新生物科技有限公司;电泳仪、电泳槽 北京六一仪器厂;UVP-8000凝胶成像系统 美国Ultra-Violet公司;Thermo酶标仪 美国热电公司;BH-2光学显微镜 日本Olympus公司。
1.4 筛选方法
1.4.1 分离及纯化
将采集的各种样品用生理盐水打散后,取上清液2%接入筛选培养基Ⅰ中,于37℃恒温静置培养24h。将能够生长的样品管中的菌液进行梯度稀释在MRS固体培养基和筛选培养基Ⅱ上涂布,37℃恒温倒置培养48h,获得单菌落。采用平板划线法纯化,并将纯化的菌株接种到MRS液体培养基中培养12h后加适量甘油保藏。
1.4.2 耐受丁醇菌株的筛选
菌株2%接种量37℃活化培养24h,确定此时菌液的OD600nm值。2%转接含有体积分数为1%丁醇的液体培养基中,37℃培养24h,取200µL菌液加入96孔板中用酶标仪测得OD600nm值,计算其相对生长率,选取相对生长率较高的菌株进行下一步实验,同样步骤测定在含有体积分数为2%和3%丁醇的液体培养基中的相对生长率。不接种的培养基作为空白对照,所有实验重复3次。相对生长率[18]为在含有丁醇的培养基中的OD600nm值与不加丁醇的培养基中的OD600nm值的比值。
1.4.3 菌株耐受丁醇曲线测定
为了防止丁醇挥发,在带橡皮胶塞的三角瓶中进行菌株耐受丁醇培养和耐受曲线测定。在MRS液体培养基中添加不同体积分数的丁醇,分别为0、1%、1.5%、2%、2.5%、3%、3.25%、3.5%,取过夜培养的菌液以2%的接种量接至含有各个不同体积分数丁醇的培养基中,37℃静置培养,通过测定OD600nm值来检测菌株的生长情况。
1.5 菌株的鉴定
1.5.1 菌株形态学观察
将筛选得到的耐受丁醇体积分数较高的菌株分别接种于MRS固体培养基上,37℃培养24h,观察菌落形态,并进行革兰氏染色,荚膜染色,于光学显微镜下观察。
1.5.2 菌株的生理生化鉴定
根据常规生理生化鉴定,参照《伯杰细菌鉴定手册》和《乳酸细菌分离鉴定及实验方法》[19],对菌株进行生理生化实验。
1.5.3 菌株16S rDNA序列的测定与分析
1.5.3.1 基因组DNA的提取
经传代培养15h的菌液用K713细菌试剂盒提取DNA,用化学裂解法[20]提取菌株的基因组DNA。
1.5.3.2 16S rDNA的扩增
采用细菌通用引物27F和1492R扩增16S rRNA基因。引物序列为27F:5’-GAGAGTTTGATCCTGGCTCAG-3’,1492R:5’-CTACGGCTACCTTGTTACGA-3’。
反应体系:取DNA模板1µL,5×PCR缓冲液10µL,引物1µL,dNTPs 4µL,TaqDNA聚合酶0.25µL,加超纯水补至总反应体积为50µL。循环参数:98℃预变性30s;98℃变性10s,55℃退火30s,72℃延伸90s,共30个循环;72℃延伸2min。取扩增产物5µL 用1%琼脂糖凝胶电泳检测,紫外灯下观测结果。
1.5.3.3 16S rDNA序列测定及同源性分析
16S rDNA测序由上海英俊生物工程技术服务有限公司完成,将测定的16S rDNA序列在NCBI核酸数据库进行BLAST搜索,与已有的16S rDNA序列进行相似性比较分析。从GenBank中选择近缘菌株的16S rDNA基因序列,应用MEGA4.1和ClustalX软件,采用 Neighbor-Joining方法构建系统发育树,并进行 Bootstrap分析,重复次数为1000次。
2 结果与分析
2.1 菌株的分离纯化结果
采集的样品中只有处理后的正常人的粪便能在筛选培养基Ⅱ中生长,获得了461个能在丁醇体积分数为3%的培养基中生长的单菌落,初步认为其具有丁醇耐受性,具体结果见表1。

表1 在筛选培养基Ⅱ中分离纯化得到的菌株数及其编号Table1 Strains purified in the screening medium and corresponding number
2.2 耐受丁醇菌株筛选结果
将得到的461个单菌落进行复筛,得到22株在含有丁醇体积分数为1%的液体培养基中的相对生长率大于60%,在丁醇体积分数为2%的培养基中相对生长率大于30%的菌株。22株菌的具体数据结果见表2。在菌株筛选的过程中,22株菌在含有丁醇体积分数为1%、2%的培养基中相对生长率均较高,基本处于同一水平,并且实验重复性良好;当丁醇体积分数继续提高到3%时,只有A37和A50能够较好生长且相对生长率达到10%以上,比其他的菌株相对生长率都要高。对比Li等[17]研究中报道的耐受3%丁醇菌株的相对生长率,故选择A37和A50作为耐受丁醇菌株继续研究。
2.3 耐受丁醇菌株在含不同体积分数丁醇培养基中的生长曲线
在37℃条件下,菌株A37和A50在12h内的丁醇耐受情况如图1所示。可以看出,当丁醇体积分数为2%以下时,菌株都能够非常良好的生长,当丁醇体积分数达到3%时,菌株的生长受到一定程度的抑制,但是依然能够有一定的生长,当丁醇体积分数达到3%以上时,A37菌株的生长基本受到明显的抑制。A50在丁醇体积分数为3.25%时还有一定的生长,但在3.5%是基本陷于停滞,这表明,菌株A37和A50都能够耐受3%的丁醇。

图1 37℃、培养基中添加不同体积分数丁醇时对A37(A)和A50(B)菌株的生长曲线Fig.1Growth curves of the isolated tolerant strains A37 (A) and A50 (B) in medium containing butanol with various concentrations at 37 ℃
2.4 耐受丁醇菌株的鉴定
2.4.1 菌株A37和A50的菌落和形态特征观察
菌株A37在MRS培养基上生长成卵圆形,凸起、表面光滑、全缘、乳白色,直径约为0.5~1.0mm; A50在MRS培养基平板上菌落呈圆形或不规则,扁平、表面光滑、透明,直径约为0.5~1.0mm。经革兰氏染色后用显微镜油镜观察,见图2,A37菌体形态为球状,单独、成对或短链排列,革兰氏染色呈阳性,无荚膜,无芽孢,不形成孢子。A50菌体为短杆状,单个排列,有时成对或成链,形态微小,革兰氏染色呈阳性,无芽孢。初步判定两株菌为乳酸菌,A37为球菌,A50为杆菌。

表2 菌株在含不同丁醇体积分数的培养基中的相对生长率(x±s, n=3)Table2 Relative growth rates of the strains in medium containing butanol at various concentrations(x±s, n=3)
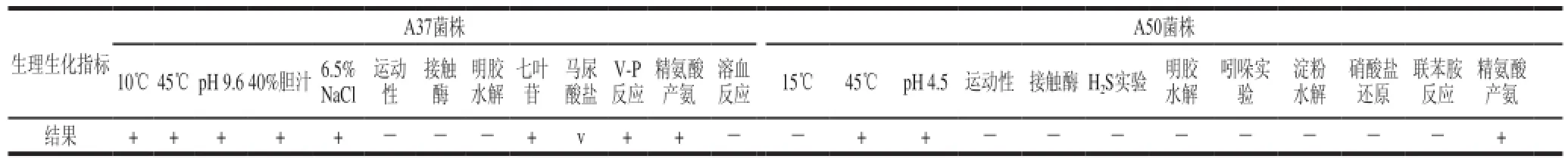
表3 菌株A37和A50的生理生化特征Table3 Physiological-biochemical properties of the strain A37 and A50

表4 菌株A37和A50的糖发酵实验结果Table4 Sugar fermentation results of strains A37 and A50

图2 菌株A37(a)和A50(b)的显微镜照片(10×100)Fig.2 The images of A37 (a) and A50 (b) strain examined by microscope (10×100)
2.4.2 菌株A37和A50的生理生化及糖发酵实验
根据镜检结果先确定其在乳酸菌中属的地位,结果见表3,然后再根据属内种间的主要特征进行鉴定,结果见表4。通过各种生理生化实验,再结合各菌株的形态结构和培养特性分析,将A37鉴定为粪肠球菌,A50为发酵乳杆菌。
2.4.3 菌株16S rDNA鉴定及进化树分析

图3 菌株的16S rDNA PCR扩增产物Fig.3 PCR amplified products of 16S rDNA gene from the strains
由图3可知,测序结果表明扩增菌株的16S rDNA序列全长A37为1446bp,A50为1462bp。在1500bp左右处出现亮带,无其他杂带。
BLAST与GenBank中的核酸数据进行比对分析,结果A37与粪肠球菌属Enterococcus faecalis(AB154827)的序列相似性为99%。A50与发酵乳杆菌属Lactobacillus fermentum (JF812168)的序列相似性为99%。将序列提交GenBank数据库,得到登录号分别为JF903802和JF903803,根据1.5.3.3节的方法进行鉴定,建立系统进化树(图4),结果表明,菌株A37与Enterococcus faecalis聚在同一分支,1000次 Bootstrap分析完全支持该分支,同源性达100%,与其他种的发育关系相对较远。菌株A50在系统发育地位上Lactobacillus fermentum在同一分支,与其相邻菌株亲缘关系较远。结合菌落特征和生理生化鉴定结果将菌株A37鉴定为粪肠球菌,A50鉴定为发酵乳杆菌。

图4 菌株A37(A)和A50(B) 的16S rDNA基因序列系统发育树Fig.4 Phylogenentic tree of strain A37 (A) and A50 (B) based on the 16S rDNA gene sequences
3 讨 论
采用有一定生长压力的平板法从采集样品中分离,限制了很多细菌的生长,减轻筛选的工作量。同时结合Li等[17]建立的以测定菌株的相对生长率为指标对其进行筛选,利用酶标仪96孔板检测菌株的OD600nm值,提高了筛选的效率,得到较理想的目的菌株,而且本研究中得到的耐受3%丁醇的菌株比左文朴等[18]筛选得到的菌株的耐受体积分数高,基本达到研究中报道的耐受水平,但已报道的可以耐受3%丁醇的菌种中未见发酵乳杆菌。
菌株A37和A50鉴定为粪肠球菌和发酵乳杆菌,属于乳酸菌类,拓展了乳酸菌的应用范围。在进行菌种分类鉴定时,仅仅根据表型特征来划分是不可取的。在进行常规鉴定时,有时会出现表型不够稳定、或弱反应等现象,这些都直接影响了实验结果的判断,而且很多表型特征不同的菌株通过16S rDNA基因序列比对后,结果却为同一种。所以,要具体鉴定某一未知菌种,在依据表型特征鉴定的同时,要结合基因型鉴定,才能够比较准确的进行分类鉴定。
筛选得到的耐受丁醇的菌株为构建其他耐受丁醇基因工程菌株生产丁醇提供了新的菌种资源,具有良好的工业应用价值。研究表明丁醇产量具有很大的提升空间,但对摸清其遗传背景,通过分子生物学技术改造使其生产丁醇仍需进一步研究。
[1] SCHWAR W H, DOMINIK A. Biofuels from microbes[J]. Applied Microbiology Biotechnology, 2007, 77: 23-35.
[2] ZVERLOV V, BEREZINA O, VELIKODVORSKAYA G, et al. Bacterial acetone and butanol production by industrial fermentation in the Soviet Union: use of hydrolyzed agricultural waste for biorefinery[J]. Applied Microbiology and Biotechnology, 2006, 71(5): 587-597.
[3] 姜岷, 韦萍, 卢定强, 等. 后化石经济时代工业生物技术发展的若干思考[J]. 化工进展, 2006, 25(10): 1119-1123.
[4] KEIS S, SHAHEEN R, JONES D T. Emended descriptions of Clostridium acetobutylicum and Clostridium beijerinckii, and descriptions of Clostridium saccharoperbutylacetonicum sp.nov. and Clostridium saccharobutylicum sp.nov.[J]. Int J Syst Evol Microbiol, 2001, 51(Pt6): 2095-2103.
[5] PAPOUTSAKIS E T. Engineering solventogenic clostridia[J]. Curr Opin Biotechnol, 2008, 19: 420-429.
[6] NASIB Q, BADAL S, MICHAEL C. Butanol production from wheat straw hydrolysate using Clostridium beijerinckii[J]. Bioprocess and Biosystems Engineering, 2007, 30(6): 419-427.
[7] BORDEN J R, PAPOUTSAKIS E T. Dynamics of genomiclibrary enrichment and identification of solvent tolerance genes for Clostridium acetobutylicum[J]. Applied and Environmental Microbiology, 2007, 73(9): 3061-3068.
[8] EZEJI T, QURESHI N, BLASCHEK H P. Butanol production from agricultural residues: impact of degradation products on Clostridium beijerinckii growth and butanol fermentation[J]. Biotechnol Bioengineering, 2007, 97: 1460-1469.
[9] SCOTCHER M C, RUDOLPH F B, BENNETT G N. Expression of abrB310 and sinR, and effects of decreased abrB310 expression on the transition from acidogenesis to solventogenesis, in Clostridium acetobutylicum ATCC 824[J]. Applied and Environmental Microbiology, 2005, 71(4): 1987-1995.
[10] THORMANN K, FEUSTEL L, LORENZ K, et al. Control of butanol formation in Clostridium acetobutylicum by transcriptional activation[J]. Journal of Bacteriology, 2002, 184(7): 1966-1973.
[11] DURRE P. New insights and novel developments in clostridial acetone/butanol/isopropanol fermentation[J]. Applied Microbiology and Biotechnology, 1998, 49(6): 639-648.
[12] 杨明, 刘力强, 牛昆, 等. 丙酮丁醇发酵菌的分子遗传改造[J]. 中国生物工程杂志, 2009, 29(10): 109-114.
[13] ATSUMI S, CANN A F, CONNOR M R, et al. Metabolic engineering of Escherichia coli for 1-butanol production[J]. Metabolic Engineering, 2008, 10(6): 305-311.
[14] INUI M, SUDA M, KIMURA S, et al. Expression of Clostridium acetobutylicum butanol synthetic genes in Escherichia coli[J]. Appl Microbiol Biotechnol, 2008, 77(6): 1305-1316.
[15] STEEN E J, CHAN R, PRASAD N, et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol[J]. Microbial Cell Factories, 2008, 7: 36.
[16] KNOSHAUG E, ZHANG Min. Butanol tolerance in a selection of microorganisms[J]. Applied Biochemistry and Biotechnology, 2009, 153(1): 13-20.
[17] LI J, ZHAO J B, ZHAO M, et al. Screening and characterization of butanol-tolerant micro-organisms[J]. The Society for Applied Microbiology, 2010, 50: 373-379.
[18] 左文朴, 裴建新. 两株高丁醇耐受细菌的分离鉴定及其丁醇耐受特性的研究[J]. 生物技术通报, 2009(12): 155-159.
[19] 凌代文, 东秀珠. 乳酸菌细菌分类鉴定及实验方法[M]. 北京: 中国轻工业出版社, 1999.
[20] AUSUBEL F M, KINGSTON R E. 精编分子生物学实验指南[D]. 北京: 科学技术出版社, 2001: 36-39.
Screening and Identification of Butanol-Tolerant Lactic Acid Bacteria
LIU Hong-ru1,CAO Long-kui1,2,*,SUN Da-qing2,SONG Liang1,YANG Chun-yu1
(1. College of Food Science, Heilongjiang Bayi Agricultural University, Daqing 163319, China;2. Agri-food Processing and Engineering Technology Research Center of Heilongjiang Province, Daqing 163319, China)
Bacterial strains that can grow well in the presence of butanol at high concentration were isolated and screened from different samples. Their relative growth rates in the medium with the addition of butanol were monitored. Subjected to repeated screening, the strains with higher relative growth rates were chosen to make the butanol-resistant curve. Meanwhile, the species of the selected strains were identified by physiological and biochemical experiments and 16S rDNA analysis coupled with bacterial colony characteristics. Two bacterial strains (A37 and A50) revealed the butanol tolerance up to 3%. The 16S rDNA sequence analysis exhibited the consistent results with biochemical analysis. Moreover, A37 and A50 were identified as Enterococcus faecalis and Lactobacillus fermentum.
lactic acid bacteria;butanol tolerance;relative growth rate;screening;identification
Q939.97
A
1002-6630(2013)03-0212-05
2011-10-18
黑龙江省研究生创新科研项目(YJSCX2011-265HLJ)
刘洪儒(1985—),女,硕士研究生,研究方向为农产品加工与贮藏。E-mail:liuhongru886@126.com
*通信作者:曹龙奎(1965—),男,教授,博士,研究方向为农产品加工与贮藏。E-mail:longkuicao@yahoo.com.cn
