Atp11c基因诱捕小鼠遗传背景分析
2013-02-05陈科肖君华周宇荀李凯
陈科,肖君华,周宇荀,李凯
(东华大学生物科学与技术研究所,上海 201620)
基因诱捕(gene trapping)技术是一种卓越的突变策略,适用于小鼠胚胎干细胞的大规模突变研究和细胞系中靶基因的完全失活[1-3];结合胚胎技术生产的嵌合体突变小鼠,可广泛应用于功能基因的研究[4]。
Atp11c 基因位于小鼠基因组X 染色体上的33.5 厘摩(centimorgan,cM)处,由369,406 个碱基组成。其编码的蛋白作为脂类的翻转酶,可能对生长发育、新陈代谢和机体免疫等多种生命活动的信号传递起关键作用[5-10]。通过基因诱捕技术将Atp11c 基因完全失活,借此研究Atp11c 基因在体内的功能和表达特征。令人有些担忧的是基因诱捕可能引起内源基因的重组或删除,造成基因序列的缺失或者损伤。Graziotto 等[11]发现诱捕载体插入导致被诱捕基因出现了12 kb 左右的删除;Niwa 等[12]也发现了类似事件,他发现在诱捕载体3'插入区域出现了多达10 kb 的删除。这发生在诱捕载体3'端的删除,可能是因为人工合成的载体原件在体内不稳定造成的,而基因诱捕个体出现野生型蛋白表达和报告基因活性缺失的现象可能就是外源载体片段的删除或者重组导致的[13,14]。与之相似的,诱捕载体的多拷贝整合也会引起报告基因或筛选基因失效,引起转录异常导致基因诱捕失败。Dhara[15]利用3'端cDNA 末端快速克隆技术(3'-rapid amplification of cDNA ends,3'-RACE)确定诱捕载体在转录本上的整合位点,继而辅助逆转录PCR (reverse transcription polymerase chain reaction,RT-PCR)技术筛选正确克隆用于构建突变小鼠模型。然而Galy 等[16]证实即使利用体外筛选正确克隆建成的小鼠,其表达图谱也可能不正确。因此分析Atp11c诱捕小鼠的遗传背景是研究Atp11c 在体内生物学功能的必要前提。
本研究以Atp11c 基因诱捕嵌合体小鼠与纯合129P2/OlaHsd 小鼠构建Atp11c 基因诱捕小鼠模型。以此模型为样本,利用荧光竞争性PCR 技术,建立一种快速、准确的诱捕载体拷贝数检测方案;同时根据诱捕载体特异性设计引物对后代小鼠分型,并针对被诱捕基因序列的特异性设计检测引物,以确定诱捕载体的插入位点及其对宿主染色体造成的损伤。
1 材料与方法
1.1 实验动物
编号RRX137 的Atp11c 基因诱捕小鼠ES 细胞购自BayGenomics,并委托南京模式动物中心利用胚胎技术培育出的Atp11c 基因诱捕嵌合体小鼠。纯和?129P2/OlaHsd 小鼠购买自英国Harlan 实验室。嵌合体小鼠与纯和?129P2/OlaHsd 小鼠回交构建基因诱捕小鼠模型。动物实验遵守中国1988年实验动物管理条例,实验在东华大学生物科学与技术研究所屏障动物实验设施进行[SYXK(沪)2008-0059]。
1.2 DNA 提取
DNA 提取采用Axygen(杭州爱思进生物技术有限公司,中国)基因组DNA 小量抽提试剂盒,操作步骤依说明书进行。以0.8%琼脂糖凝胶电泳和NanoDrop 2000c 超微量分光光度计(Thermo Fisher Scientific,美国)确定DNA 质量和浓度。
1.3 引物的设计
Atp11c 的基因序列信息来源于NCBI,诱捕载体序列信息来源于BayGenomics。所有引物用Primer3 在线软件(http://frodo.wi.mit.edu/primer3/)设计,并由上海生工生物工程股份有限公司合成。
如图1 所示,针对pGT2lxf 载体PA 序列设计诱捕载体检测引物vector-F 和vector-R;针对Atp11c的第一个内含子设计特异区段检测引物1-F 和1-R、N-F 和N-R、54-F 和54-R 共54 对;针对载体插入两端设计的缺失区段检测引物E-F 和E1-R、E2-R、E3-R 与P1-F、P2-F、P3-F 和P-R。

图1 Atp11c 基因诱捕小鼠遗传背景检测引物示意图Fig.1 Schematic diagram of the genetic background of Atp11c gene trap mice and the detection primers
1.4 测序分析
所有测序反应由上海生工生物工程技术服务有限公司完成,PCR 产物纯化后均用双脱氧末端终止法进行测序(ABI 3730XL 测序仪,美国),两对测序引物分别为E-F 和E3-R、P2-F 和P-R。
1.5 荧光竞争性PCR 确定诱捕载体拷贝数
本研究根据竞争性PCR 定量原理[17],利用基因组中已知拷贝数的Rfx3 基因作为检测拷贝数的内对照。由上海翰宇生物科技有限公司全基因组合成该基因和载体lacZ 上的核心序列,分别在其序列中插入两个CT 碱基,将该段合成序列转入质粒中,质粒经过限制性酶切处理后作为内对照样本[18],利用内对照样本和待测样本进行竞争性PCR 反应。
1.6 PCR
1.6.1 单重PCR
PCR 反应终体积(15 μL)包括4.1 μL 双蒸水,1.5 μL 10 × PCR buffer (含34 mmol/L Mg2+),3 μL dNTPs (2.5 mmol/L),2.4 μL 上下游引物(2 μmol/L),1U Taq DNA 聚合酶(1 μL)和3 μL DNA模板(10 ~15 ng/μL)。PCR 反应程序为:93℃变性1 min 30 s,93℃变性30 s,57℃退火30 s,65℃延伸2 min,40 个循环;65℃保持10 min。反应产物经琼脂糖凝胶电泳检测特异性。PCR 产物4℃保存。
1.6.2 两重荧光竞争性PCR
PCR 反应终体积(15 μL)包括1.5 μL 双蒸水,1.5 μL 10× PCR buffer(含34 mmol/L Mg2+),3 μL dNTP s(2.5 mmol/L),3 μL 上下游引物(0.05 μmol/L),3 μL 通用荧光引物上下游引物(4 μmol/L),1 U Taq DNA 聚合酶(1 μL),1 μL DNA 模板(30 ng/μL),1 μL合成模板(3×10-4ng/μL)同时设阴性对照。PCR 反应程序为:95℃变性,2 min;94℃变性30 s,62℃复性2 min,72℃延伸1 min,10 个循环;95℃变性2 min;94℃变性30 s,52℃复性1 min 30 s,68℃延伸1 min 30 s,20个循环;68℃补齐20 min。反应产物经琼脂糖凝胶电泳检测特异性,PCR 产物4℃保存。
2 结果
2.1 诱捕载体插入位置的确定
如图2 所示,部分插入区段检测引物的PCR 结果显示:雄性诱捕个体在区段45 和46 没有条带,作为对照的129 雄性个体没有区段缺失。基因诱捕载体位于区段45 -46 之间。
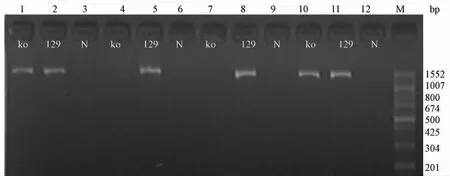
图2 PCR 检测诱捕载体插入位点Fig.2 Detection of gene trap vector insertion site by PCR
2.2 宿主染色体和载体缺失序列的检测
如图3-A 所示,P-R 和P2-F 的测序结果显示质粒骨架端插入导致基因组丢失了ATCTC 5 个碱基并伴随着载体上359 bp 的缺失,图3-B 显示,诱捕载体En2 端缺失421 bp,而基因组出现了跳跃式删除,距离插入位点543 bp 后出现了418 bp 的删除。区段46 的检测引物上游位于该区段,解释了载体插在区段45,而区段46 的检测引物无目的条带的结果。
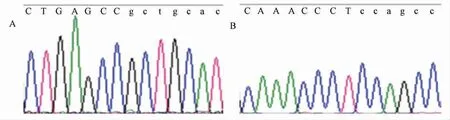
图3 Atp11c 基因诱捕小鼠染色体缺失序列引物测序结果Fig.3 Primer sequencing results of fragments losing of the Atp11c gene trap mice
2.3 荧光竞争性PCR 确定诱捕载体拷贝数
如图4 所示,基因诱捕雄性、基因诱捕雌性杂合子和基因诱捕雌性纯合子三个样本,分别与内对照模板进行荧光竞争性PCR 反应,通过ABI 3730 测序仪进行产物检测。产物浓度可由荧光强度标示,将内对照和检测样本的荧光数据进行换算,鉴定出基因诱捕雄性和基因诱捕雌性杂合子中Rfx3 与lacZ 的拷贝数比值分别为1.94∶1和2.02∶1。由于Rfx3 基因已知为2 拷贝基因,说明的在雄性和雌性杂合子中基因诱捕载体为单拷贝整合;相对的,基因诱捕雌性纯和子中Rxf3 与lacZ 的拷贝数比值为0.99∶1;因此,基因诱捕雌性纯合子中诱捕载体为两拷贝整合。
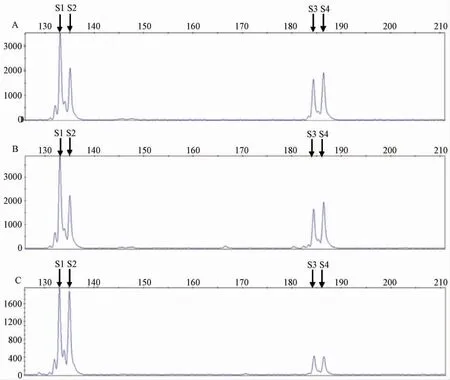
图4 Atp11c 基因诱捕小鼠载体拷贝数检测峰形图Fig.4 Peak shape diagram of vector copy number detection of Atp11c gene trap mice
3 讨论
基因诱捕正在走向成熟的时代,尽管有分歧,但越来越多成功的事例证明基因诱捕是一种富有成效的研究方法[19]。近来,全世界主要的基因诱捕组织联合成立了国际基因诱捕联盟(International Gene Trap Consortium,IGTC)[20],旨在努力建立ES细胞资源库,将小鼠基因组中全部基因诱捕,这些基因诱捕产生的突变小鼠被用于功能分析并作为表型激活筛选的标记[21]。由此可见,基因诱捕将不断发展并最终成为小鼠遗传学研究的一种经典技术[21]。但是现今的基因诱捕技术仍有一些不足:诱捕载体可能激活已失活或者关闭的基因,或者造成某些必需原件的缺失,损害相关基因,导致表型假阳性等。Voss[22]研究了209 个基因诱捕事件,发现35%的没有报告基因活性,39%的报告基因活性无限制,26%的报告基因在某些组织和细胞中有限制性活性,这些情况与诱捕小鼠的遗传背景密切相关。因此,对基因诱捕个体的遗传背景进行分析是研究功能基因在体内作用时的必要补充。
基因诱捕载体在基因组中插入位点的确定,常用反向PCR(inverse PCR)这一经典技术。本研究曾使用识别4 bp 的限制性内切酶对载体尾端进行酶切反应,利用反向PCR 进行基因组整合位点的鉴定,未得到理想结果(数据未展示)。后经PCR 定位检测确定诱捕载体的插入会导致诱捕载体两端和宿主染色体出现不规律的删除。因此,利用常规的反向PCR 技术很难确定基因诱捕载体在基因组的整合位点。谭晓红等[23]利用质粒拯救技术克服了载体两端序列缺失的限制,成功获得了基因组整合位点的序列。然而,质粒拯救技术在操作上复杂,费时费力;最重要的是假阳性克隆比例高、筛选繁琐、工作量大,如果宿主染色体出现大片段的删除或重组,则很难快速准确的确定诱捕载体整合位点。
本研究结果显示PCR 定位可以准确定位基因诱捕载体插入区段,进一步利用精细定位引物可检测宿主染色体和诱捕载体的缺失状态。该方案利用基因诱捕小鼠和纯和对照小鼠作为样本,进行PCR反应,两种样本的结果相互对照,不仅克服假阳性问题,并且具有结果精确、操作简单、筛选迅速和工作量小等特点,最主要的是不受诱捕载体和宿主染色体缺失状态的影响。
基因诱捕载体在基因组中的拷贝数,可以使用Southern 杂交技术检测,但是难以精确计算出拷贝数数值[23]。而针对诱捕载体核心序列设计Taqman探针,结合荧光定量PCR 技术虽然能够准确计算出诱捕载体拷贝数[24]。但是因为Taqman 探针过于昂贵的造价,致使该方法不具备通用性。
本研究使用荧光竞争性PCR,对Atp11c 基因诱捕小鼠雄性、雌性纯合子和雌性杂合子三种基因型小鼠进行拷贝数鉴定,结果显示Atp11c 基因诱捕雄性小鼠和雌性杂合子中诱捕载体为单拷贝,Atp11c基因诱捕雌性纯合子小鼠中诱捕载体为两拷贝。荧光竞争性PCR 检测拷贝数,操作简单、重复性强、结果准确,内对照模板一经合成可永久使用,因此价格低廉,并对不同诱捕基因都具有适用性,说明基于荧光竞争性PCR 可建立一种快速、准确和通用性强的载体拷贝数检测方案。
本实验成功的分析了Atp11c 基因诱捕小鼠的遗传背景,建立了快速、准确检测诱捕小鼠遗传背景的方案,为研究Atp11c 突变小鼠表型形成机理和Atp11c 在体内功能提供了必要的补充。
[1]Gossler A,Joyner AL,Rossant J,et al.Mouse embryonic stem cells and reporter constructs to detect developmentally regulated genes[J].Science,1989,244:463 -465.
[2]Skarnes WC,Auerbach BA,Joyner AL.A gene trap approach in mouse embryonic stem cells:the lacZ reported is activated by splicing,reflects endogenous gene expression,and is mutagenic in mice[J].Genes Dev,1992,6:903 -918.
[3]Wurst W,Rossant J,Prideaux V,et al.A large scale gene trap screen for insertional mutations in developmentally regulated genes in mice[J].Genetics,1995,139:889 -899.
[4]Stanford WL,Caruana G,Vallis KA,et al.Expression trapping:identification of novel genes expressed in hematopoietic and endothelial lineages by gene trapping in ES cells[J].Blood,1998,92(12):4622 -4631.
[5]Kühlbrandt W.Biology,structure and mechanism of P-type ATPases[J].Nat Rev Mol Cell Biol,2004,5:282 -295.
[6]Mukhopadhyay S,Linstedt AD.Identification of a gain-of-function mutation in a Golgi P-type ATPase that enhances Mn2+efflux and protects against toxicity[J].PNAS,2011,108(2):858 -863.
[7]Clark MR.Flippin’lipids[J].Nature Immunol,2011,12:373-375.
[8]Bowl MR,Nesbit MA,Harding B,et al.An interstitial deletioninsertion involving chromosomes 2p25.3 and Xq27.1,near SOX3,causes X-linked recessive hypoparathyroidism[J].J Clin Invest,2005,115(12):2822 -2831.
[9]Siggs OM,Arnold CN,Huber C,et al.The P4-type ATPase Atp11c is essential for B lymphopoiesis in adult bone marrow[J].Nature Immunol,2012,12(5):434 -441.
[10]Yabas M,Teh CE,Frankenreiter S,et al.Atp11c is critical for the internalization of phosphatidylserine and differentiation of B lymphocytes[J].Nature Immunol,2011,12(5):441 -451.
[11]Graziotto JJ,Inglehearn CF,Pack MA,et al.Decreased levels of the RNA splicing factor prpf3 in mice and zebrafish do not cause photoreceptor degeneration[J].Invest Ophthalmol Visual Sci,2008,49(9):3830 -3838.
[12]Niwa H,Araki K,Kimura S,et al.An efficient gene-trap method using polyA trap vectors and characterization of gene-trap events[J].J Biochem,1993,113:343 -349.
[13]Voss AK,Thomas T,Gruss P.Compensation for a gene trap mutation in the murine microtubule 2 associated protein 4 locus by alternative polyadenylation and alternative splicing[J].Dev Dynamics,1998,212:258 -266.
[14]Evans MJ.Gene trapping:a preface[J].Dev Dynamics,1998,212:167 -169.
[15]Dhara SK,Benvenisty N.Gene trap as a tool for genome annotation and analysis of X chromosome inactivation in human embryonic stem cells[J].Nucleic Acids Res,2004,32(13):3995 -4002.
[16]Galy B,Ferring D,Benesova M,et al.Targeted mutagenesis of the murine IRP2 and IRP1 genes reveals context-dependent RNA processing differences in vivo[J].RNA,2004,10:1019 -1025.
[17]杜迪军,缪竞智,张秀珍.竞争性PCR 内参照菌的构建及应用[J].中华微生物学和免疫志,2000,20(5):442 -446.
[18]刘泽文,徐涤平,周平华,等.竞争性PCR 定量检测鸡传染性贫血病毒核酸[J].中国预防兽医学报,2005,27(3):227 -229.
[19]Stanford WL,Cohn JB,Cordes SP.Gene trapping mutagenesis:past,present and beyond[J].Nature Rev Genet,2001,2:756-768.
[20]Austin CP,Battey JF,Bradley A,et al.The knockout mouse project[J].Nature Genet,2004,36:921 –924.
[21]党素英,王铸钢.基因诱捕技术[J].国际遗传学杂志,2006,29(1):20 -25.
[22]Voss AK,Thomas T,Gruss P.Efficiency assessment of the gene trap approach[J].Dev Dynamics,1998,212:171 -180.
[23]谭晓红,程萱,毛春明,等.利用基因诱捕技术进行小鼠基因剔除的初步研究[J].生物化学与生物物理进展,2004,31(7):606 -610.
[24]Floss T,Uez N,Frey S,et al.Genotyping gene-trap mutant mice by real-time PCR[J].Tech Tips Online,2002,7:6 -8.
