水牛、大额牛和牦牛抑制素α亚基编码基因外显子1多态性分析
2012-12-25苗永旺高华山李大林袁跃云
苗永旺, 哈 福 , 高华山, 袁 峰, 李大林, 袁跃云
(1. 云南农业大学 动物科学技术学院分子遗传学实验室, 云南 昆明 650201; 2. 云南省家畜改良工作站, 云南 昆明 650225)
水牛、大额牛和牦牛抑制素α亚基编码基因外显子1多态性分析
苗永旺1,#,*, 哈 福1,#, 高华山1, 袁 峰1, 李大林2, 袁跃云2
(1. 云南农业大学 动物科学技术学院分子遗传学实验室, 云南 昆明 650201; 2. 云南省家畜改良工作站, 云南 昆明 650225)
为了揭示牛科物种INHA基因的遗传特征, 该文采用PCR产物直接测序法对水牛、大额牛和牦牛INHA基因外显子1及其侧翼序列进行多态性检测, 并结合已发表的包括牛科物种在内的一些哺乳动物数据进行了比较分析。结果表明, 在水牛INHA基因外显子1中存在c.73C>A替换, 为同义替换, 河流型和沼泽型水牛编码产物一致; 在大额牛的INHA基因外显子1中发现c.62 C>T、c.187 G>A替换, 分别引起INHA中氨基酸发生p. P21L、p. V63M改变, 两者均为相同性质氨基酸的替换; 在牦牛中发现c.62 C>T、c.129A>G替换, 前者也引起编码氨基酸发生p. P21L替换, 后者为同义替换。在INHA基因5'侧翼区所测出的序列中, 水牛、大额牛和牦牛等物种内均未发现SNP位点, 但在种间发现存在c. -6T>G的替换, 大额牛、牦牛和普通牛均为c. -6G, 而水牛为c. -6T。在INHA基因内含子中, 水牛的第31~36位核苷酸处发现有6个碱基的缺失, 即c. 262+31_262+36delTCTGAC; 该位点在河流型水牛中野生型(+/+)占主体, 而在沼泽型水牛中则缺失型(-/-)占主体。在大额牛、牦牛和普通牛等其它牛科物种的内含子中均未发现该缺失, 但与水牛相比, 大额牛、牦牛和普通牛内含子中发现缺失c. 262+78_262+79delTG。序列比对显示,INHA基因外显子1序列中c. 43A和c. 67G为水牛中所特有, 而c. 173A和c. 255G 为大额牛、牦牛和普通牛所共有, c. 24C、c. 47G、c. 174T和c. 206T为山羊所特有。大额牛、牦牛和普通牛间INHA基因外显子1序列差异较小, 而山羊和水牛与它们间的差异相对较大。
水牛; 大额牛; 牦牛; 抑制素α亚基基因(INHA)外显子1; 多态性
抑制素(inhibin, INH)是一种主要由性腺细胞分泌的糖蛋白激素, 由α亚基和β亚基通过二硫键接合而成(Thompson et al, 1994; Xue et al, 2004)。α亚基为抑制素的固有亚基, β亚基包含两种结构相似的β A亚基和β B亚基。α亚基分别与β A亚基和β B亚基结合, 被称作抑制素 A(inhibin-A)和抑制素B(inhibin-B)(Thompson et al, 1994; Xue et al, 2004)。
抑制素能够对促卵泡素(follicle-stimulating hormone, FSH)的含量起到抑制作用(Thompson et al,1994; Xue et al, 2004), 从而调控雌性动物的卵泡生成。在雄性动物中, 抑制素α亚基(inhibin α-subunit,INHA)主要存在于精原细胞、精母细胞和早期的精细胞中, 是睾丸间质细胞和支持细胞增殖、分化和类固醇合成潜在的旁分泌和自分泌调节因子(Sang et al, 2011)。因此, INHA编码基因为控制家畜繁殖性状的重要候选基因, 具有很高的研究和利用价值。过去, 对INHA基因的研究主要集中在人、猪、普通牛、山羊和绵羊等动物上(Thompson et al, 1994;Jeong et al, 2004; Xue et al, 2004; Zhou et al, 2007;Fallahian et al, 2009; Wu et al, 2009; Ding et al, 2011;Sang et al, 2011; Tang et al, 2011)。Ensemble 数据库(http://www.ensembl.org/index.html)中公布的普通牛INHA基因序列共含有2个外显子和1个内含子, 第一外显子编码区长为262 bp, 第二外显子编码区长为821 bp, 内含子长1 836 bp; 该基因编码序列长1 083 bp, 共编码360个氨基酸。
家养水牛分为河流型和沼泽型两种类型(Zhang,2000), 有关水牛抑制素α亚基完整的编码区序列虽已有报道(Xie et al, 2009), 但目前有关两类水牛INHA基因的遗传差异及群体遗传特征还未见报道。在牛科物种中, 有关牦牛和大额牛INHA基因的研究也未见报道。本研究采用PCR产物直接测序法, 对水牛、牦牛和大额牛的INHA基因外显子 1进行了群体变异检测, 并结合已发表的包括普通牛和山羊等牛科物种在内的一些哺乳动物同源序列进行了比较分析, 以揭示牛科物种INHA基因外显子1的序列差异及其群体遗传特征。
1 材料与方法
1.1 材料
148头水牛样品分别采自3个河流型和11个沼泽型水牛群体, 河流型水牛样品包括槟榔江水牛35头、摩拉水牛12头、尼里−拉菲水牛10头, 共57头; 沼泽型水牛样品包括湖南滨湖水牛4头、恩施水牛7头、盐津水牛8头、福安水牛9头、东流水牛7头、信丰水牛6头、滇东南水牛8头、江西滨湖水牛7头、富钟水牛10头、德昌水牛18头、德宏水牛7头, 共91头。8头大额牛样品采自云南省怒江州; 牦牛样品共12头, 其中8头采自西藏自治区, 4头采自云南省迪庆州中甸县。每头牛取耳组织或肌肉样, 低温带回实验室,-80 ℃冻存。
由 NCBI数据库(http://www.ncbi.nlm.nih.gov/)中下载了不同哺乳动物INHA基因第1外显子序列,用于比较分析。序列包括: 水牛: EU884446; 普通牛:U16237、M13273、NM_174094、L20601; 山羊:EF602161、HQ699620、JN620334、JN620335; 牦牛: JN572112; 猪: AK235034、AK234503、AK347411、DQ356013、M13980、NM_214189; 人:NM_002191、X04445、AK292340、BC006391、BC039076、BT006954、CU675381、M13144、M13981;猴: NM_001032955和黑猩猩: XM_001148064。由Ensembl数据库中下载了人和普通牛INHA基因全序列(ENSG00000123999, ENSBTAG00000009972)也用于比较分析。
1.2 基因组DNA的提取
采用酚/氯仿抽提法(Joseph & David, 2002)提取基因组 DNA, 经紫外分光光度法检测其纯度和浓度, TE缓冲液稀释为50 ng/μL, 保存于4 ℃备用。
1.3 PCR引物设计和PCR扩增
根据普通牛INHA基因全序列(登录号:ENSBTAG00000009972)并参考 GenBank数据库(http://www.ncbi.nlm.nih.gov/)中水牛和普通牛该基因 mRNA 序列(登录号: EU884446、BC109837、M13273、NM_174094), 采用Oligo 6软件设计扩增INHA基因外显子1及其旁侧序列的引物。PCR引物也作为测序引物。上游引物: 5'-GTAGAAGAG GGCGGTGGT-3', 下游引物: 5'-TCCTGAAATGTA GGCGTTAT-3'。PCR 反应体系为 25 μL, 包含10×buffer 2.5 μL(Mg2+为 1.5 mmol/L), 上、下游引物各 0.4 μmol/L, dNTP 2.5 mmol/L, Taq 酶 1.25 U,DNA模板100 ng。PCR程序为: 95 ℃预变性5 min;然后, 94 ℃ 35 s, 57 ℃ 35 s, 72 ℃ 40 s, 30 个循环;72 ℃后延伸10 min终止反应。
1.4 PCR产物回收及测序
PCR产物用2%琼脂糖凝胶电泳检测效果, PCR扩增效果好的样品, 经回收纯化后, 送北京百泰克生物技术有限公司进行正、反链双向测序。
1.5 数据分析
使用DNAStar 6.0软件包(DNAstar Inc.)对测序数据进行核对编辑后, 用Clustal X软件(Thompson et al, 1994)对序列进行比对。各物种序列一致性分析采用DNAStar软件包进行, 再用MEGA 4.0软件(Tamura et al, 2007)进行突变位点输出, 并采用Kimura双参数模型构建系统发育树。
2 结果与分析
2.1 物种内序列变异分析
本研究获得水牛INHA基因外显子1及其两侧序列长度为496 bp, 包括外显子1序列262 bp, 5'侧翼序列40 bp和内含子序列194 bp。经比对, 各牛科物种INHA基因外显子1序列长度相同, 各物种内序列一致性在99.2%~99.6%之间。
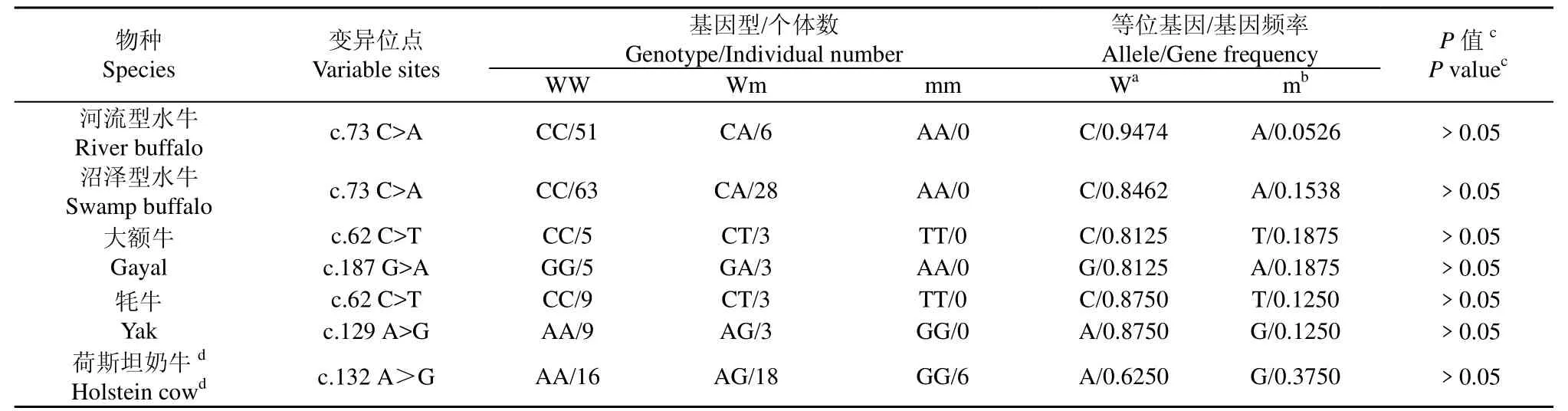
表1 牛科物种INHA基因外显子1序列的SNP位点及其基因型、基因频率Tab. 1 Bovine species SNPs, corresponding genotypes and gene frequencies in INHA exon 1
在INHA基因外显子1序列中, 发现水牛INHA基因编码区第73位核苷酸处(本文以水牛序列起始密码子第一位核苷酸为+1记录突变位点)存在一个SNP位点, 即为c.73C>A颠换, c.73C和c.73A基因频率分别为0.8851和0.1149, 但此SNP并没有引起编码氨基酸的改变。在沼泽型水牛中, 发现该位点存在c.73C和c.73A两个等位基因, 但c.73A等位基因仅以杂合状态存在。在河流型水牛中, 摩拉水牛和尼里−拉菲水牛只存在等位基因c.73C, 而槟榔江水牛却含有c.73C和c.73A两个等位基因, 基因频率分别为0.90和0.10, c.73A也仅以杂合状态存在。
在大额牛中, 检测到INHA基因编码区第62、187位核苷酸处存在c.62 C>T、c.187 G>A替换, 前者引起了INHA中相应位置氨基酸由脯氨酸(Pro, P)变为亮氨酸(Leu, L), 即 p. P21L; 后者引起了相应氨基酸由缬氨酸(Val, V)变为甲硫氨酸(Met, M), 即p. V63M, 两者均为相同性质氨基酸的替换。INHA基因编码区 c.62 C>T的非同义替换也发现存在牦牛中。此外, 在牦牛该基因编码区第129位核苷酸处发现存在c.129A>G替换, 但该替换为同义替换。在大额牛群体中发现的c.62 C>T、c.187 G>A及在牦牛群体中发现的c.62 C>T、c.129A>G替换均只存在于杂合个体中。各SNP位点及其群体遗传组成信息见表1。
在所得到的40 bp的 5'侧翼序列中, 水牛、大额牛和牦牛物种内均未发现 SNP位点; 在种间, 发现大额牛与牦牛序列完全一致, 但它们与水牛的 5'侧翼序列在-6位核苷酸上存在差异, 在水牛中该位置为 c. -6T, 在大额牛与牦牛中为 c. -6G。与已发表的普通牛序列相比, 发现普通牛中该位点也为 c.-6G。
在INHA基因内含子所测出的序列中, 水牛的第31~36位核苷酸处发现有 6个碱基的缺失, 即 c.262+31_262+36delTCTGAC, 将之命名为等位基因“-”;而将水牛中无 6个碱基缺失的类型命名为等位基因“+”。在河流型水牛中, 摩拉水牛和尼里-拉菲水牛该位置均为无缺失类型, 对应的基因型为+/+, 而槟榔江水牛却含有+/+、+/-和-/-三种基因型个体, 所占比例分别为 71.43%(25/35)、20.00%(7/35)和 8.57%(3/35); 在沼泽型水牛中该位置存在+/+、+/-和-/-三种基因型, 所占比例分别为 2.20%(2/91)、29.67%(27/91)和68.13%(62/91)。在大额牛、牦牛和已发表的普通牛INHA基因内含子中均未发现该缺失, 但与水牛序列相比, 发现它们在内含子的第78~79位核苷酸处有2个碱基的缺失, 即c. 262+78_262+79delTG。
2.2 物种间INHA基因外显子1序列差异
本文所测序列与NCBI数据库上已发表的牛科物种和人、猪等物种的同源序列进行比对, 确定各物种的单倍型。由水牛INHA基因外显子1序列变异定义两种单倍型: buffaloC和 buffaloA; 由水牛INHA基因外显子1及其内含子序列变异共同定义4种单倍型: buffaloC(+),、buffaloC(-),、buffaloA(+)和buffaloA(-)。依据INHA基因外显子1序列变异,在大额牛中定义两个单倍型: gayalI1和 gayalI2; 在牦牛中定义两个单倍型: yakI1和yakI2; 在普通牛中,定义3个单倍型: cattleI1、cattleI2和cattleI3; 山羊中也定义3个单倍型: goatI1、goatI2和goatI3。各单倍型及其相应的蛋白质编码序列差异见图1和图2。
牛科物种INHA基因外显子1与猪和灵长类同源序列差异较大。牛科物种与猪的序列一致性在89.7%~90.9%之间, 与灵长类序列一致性在83.3%~85.2%之间。而各牛科物种间INHA基因外显子1序列一致性在95.8%~100%之间, 其中, 大额牛、牦牛和普通牛间序列INHA基因外显子1序列差异较小, 序列一致性在 98.9%~100%之间, 单倍型cattleI3与yakI1序列一致; 而山羊、水牛与大额牛、牦牛和普通牛间序列差异相对较大, 山羊与它们间序列一致性在 95.8%~97.3%之间; 水牛与它们间序列一致性在 97.0%~98.5%之间; 水牛与山羊之间序列一致性在96.6%~97.3%之间。
通过物种间序列比对, 发现INHA基因外显子1序列中c. 43A、c. 67G和c. 73A为水牛所特有, 其中c. 43A、c. 67G导致水牛INHA第15、23位相应氨基酸分别为精氨酸(Arg, R)和缬氨酸(Val, V), 即p. 15R和p. 23V, 而在其他物种中该位置核苷酸均为c. 43G、c. 67C, 相应的编码氨基酸分别为甘氨酸(Gly, G)和亮氨酸(Leu, L), 即p. 15G和p. 23L。其中, p. G15R为非相同性质氨基酸的替换, p. L23V为相同性质氨基酸的替换。值得注意的是,INHA基因外显子1序列中c. 173A和c. 255G 为普通牛、大额牛和牦牛所共有, 而在其他牛科物种中为 c.173C和c. 255A, 前者差异引起了普通牛、大额牛和牦牛的INHA第58位编码氨基酸由脯氨酸(Pro, P)变为组氨酸(His, H), 为非相同性质氨基酸的替换;而后者为同义替换。在山羊中,INHA基因外显子1序列中c. 24C、c. 47G、c. 174T和c. 206T为山羊所特有, 在其他物种中该位置分别为c. 24G、c. 47A、c. 174C和c. 206G, 其中, c. 24G>C、c. 47A>G和c.206G>T替换分别引起了在山羊INHA第8、16和69位编码氨基酸改变为p. L8F(F为苯丙氨酸, Phe)、p. H16R和p. R23M, p. L8F和p. H16R替换为相同性质氨基酸的替换, 而p. R23M为非相同性质氨基酸的替换。
2.3 系统发育分析
基于各物种INHA基因外显子1不同单倍型序列构建的系统发育树, 见图3。在系统发育树上, 普通牛、大额牛和牦牛间表现出较近的遗传关系, 它们聚在一个较大的支系上, 而水牛和山羊等物种分别聚在各自独立的分支上, 且有较高的支持率, 表现出水牛、山羊与大额牛、牦牛和普通牛间遗传关系较远。
3 讨 论
本文在水牛群体中, 尽管检测出INHA基因外显子1存在c.73C>A替换, 但为同义替换, 河流型和沼泽型水牛在编码产物上相一致。对于该基因内含子序列中的 c. 262+31_262+36delTCTGAC缺失而言, 河流型水牛倾向于+/+基因型, 而沼泽型水牛则倾向于-/-基因型。在槟榔江水牛INHA基因外显子1中存在c.73C>A替换以及在其内含子中存在c.262+31_262+36delTCTGAC缺失, 提示槟榔江水牛与国外著名的摩拉水牛和尼里-拉菲水牛相比, 选育程度低, 并存在沼泽型水牛基因渗入, 这与以往对该水牛的报道相一致(Miao et al, 2011)。在大额牛群体中发现的c.62 C>T、c.187 G>A及在牦牛群体中发现的c.62 C>T、c.129A>G替换, 在遗传上是连锁的, 且处于 Hardy-Weinberg平衡状态。由于在水牛、大额牛和牦牛群体中发现的SNPs为同义替换或相同性质氨基酸的替换, 揭示各牛科物种INHA基因外显子1的不同单倍型在功能上差异不大, 提示牛科物种内INHA基因在功能上具有一定的保守性。

图1 INHA基因外显子1及其两侧翼序列各单倍型核苷酸差异Fig. 1 Nucleotide differences of haplotypes detected in INHA exon 1 and its bilateral sequences

图2 INHA基因外显子1序列各单倍型相应的编码氨基酸差异Fig. 2 Amino acid differences encoded by haplotype sequences of INHA exon 1 among various species

图3 基于各物种INHA基因外显子1单倍型序列构建的系统发育树Fig. 3 Phylogenetic tree based on haplotype sequences of INHA exon 1 in various species
各物种序列比对及系统发育分析结果表明, 大额牛、牦牛和普通牛间INHA基因外显子1序列差异较小, 序列一致性较高, 而山羊和水牛与它们的差异相对较大, 这揭示大额牛、牦牛和普通牛间遗传关系较近, 它们的INHA基因在功能上具有相似性; 而山羊和水牛与它们间遗传关系较远,INHA基因在功能上可能与大额牛、牦牛和普通牛存在一定差异性。这种差异是否与水牛的繁殖率普遍偏低,以及山羊较高的繁殖率有关, 需进一步深入研究。该基因5'侧翼序列第-6位核苷酸, 在大额牛、牦牛和普通牛中均为c. -6G型, 而在水牛中为c. -6T型,也揭示大额牛、牦牛和普通牛间较近的遗传关系。以往研究揭示, 该区域在普通牛中为转录因子Ap-2(activator protein 2)的作用位点(Thompson et al,1994), 以上差异是否反映水牛INHA基因在表达上与大额牛、牦牛和普通牛有差异需进一步研究。在水牛内含子序列中发现的 c. 262+31_262+ 36del TCTGAC缺失, 在其他物种中都不存在, 说明该缺失可能是水牛中独有的。另外, 在大额牛、牦牛和普通牛发现的 c. 262+78_262+79delTG缺失可作为区分水牛的分子标记; 在INHA基因外显子1序列中为普通牛、大额牛和牦牛所特有的核苷酸c. 173A和c.255G, 在水牛中特有的核苷酸c. 43A、c. 67G, 在山羊中特有的核苷酸c. 24C、c. 47G、c. 174T、c. 206T,可作为分子标记进行水牛、山羊与普通牛、大额牛和牦牛3个物种间的区分。以上标记可用于畜产品种类等方面的鉴定, 在畜牧生产上加以利用。
已有研究结果揭示, 体内 INHA水平与成年男性的精子数量和睾丸体积呈正相关(Sang et al, 2011),与女性卵巢早衰相关(Jeong et al, 2004; Fallahian et al,2009)。在中国荷斯坦牛中,INHA基因外显子 1的c.132A>G替换(参考文献中为c.192A>G)与超数排卵效果相关, 与精子顶体完整率有显著的相关, 即 GG基因型较 AA基因型个体具有较高的精子顶体完整率和较好的超数排卵效果(Sang et al, 2011; Tang et al,2011)。本研究在水牛、大额牛和牦牛群体中未发现c.132A>G型个体, 即群体中均为GG基因型个体。在小尾寒羊INHA基因外显子1中发现c.231T>C替换与繁殖力性状具有一定关联性, CC型个体比 TT个体具有较高的繁殖力(Zhou et al, 2007)。本研究在水牛、大额牛和牦牛群体中未发现c. 231C型, 即它们群体中均为TT基因型个体。以上结果是否在一定程度上揭示了水牛、大额牛和牦牛的精子顶体完整率高和超数排卵效果好及它们的繁殖力(它们多为单胎)比绵羊低, 尚需进一步验证。有人在波尔山羊INHA基因外显子1中发现SNP c.129G>A, 但并未发现该SNP与繁殖性状有直接关系(Wu et al, 2009)。本研究在大额牛、牦牛和普通牛中也发现存在 c.129A型, 但在水牛中只有c.129G。
本文揭示了牛科物种INHA基因外显子1的遗传特征, 可为从分子水平开展牛科物种繁殖性状的选育提供参考。
Ding CY, Chen B, Liu XQ, He CQ. 2011. Association analysis of polymorphisms inINHAgene with litter size in pigs [J].Chn J Anim Sci, 47(1): 6-9. [丁朝阳, 陈斌, 柳小春, 贺长青. 2011.INHA基因多态性与猪产仔数的相关性分析. 中国畜牧杂志, 47(1): 6-9.]
Fallahian M, Pouresmaeili F, Azizi F, Zali MR, Samani EM, Kharaziha P.2009. Existence of inhibin α-subunit gene mutation in a population of Iranian women with premature ovarian failure [J].Int J Endocrinol Metab, 7(2): 67-71.
Jeong HJ, Cho SW, Kim HA, Lee SH, Cho JH, Choi DH, Kwon H, Cha WT,Han JE, Cha KY. 2004. G769A variation of inhibin α-gene in Korean women with premature ovarian failure[J].Yonsei Med J,45(3):479-482.
Joseph S, David WR. 2002. Molecular Cloning: A Laboratory Manual.[M].3rd ed. Cold Spring Harbor: CSHL Press, 483-485.
Miao YW, Li DL, Yuan F, Liang JP, Li WZ, Liu W, Li ZR, Zhang CX. 2011.Analysis of genetic characterization of Binglangjiang buffalo using mtDNA D-loop sequences [J].J Yunnan Agric Univ: Nat Sci,26(5):625-633. [苗永旺, 李大林, 袁峰, 梁建平, 李卫真, 刘伟, 李卓然,章纯熙. 2011. 利用mtDNA D-loop序列为遗传标记分析槟榔江水牛的群体遗传特征. 云南农业大学学报: 自然科学版, 26(5): 625-633.]
Sang L, Du QZ, Yang WC, Tang KQ, Yu JN, Hua GH, Zhang XX, Yang LG.2011. Polymorphisms in follicle stimulation hormone receptor, inhibin alpha, inhibin bata A, and prolactin genes, and their association with sperm quality in Chinese Holstein bulls [J].Anim Reprod Sci,126(3-4):151-156.
Tamura K, Dudley J, Nei M, Kumar S. 2007. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0 [J].Mol Biol Evol24(8): 1596-1599.
Tang KQ, Li SJ, Yang WC, Yu JN, Han L, Li X, Yang LG. 2011. An MspI polymorphism in the inhibin α gene and its associations with superovulation traits in Chinese Holstein cows [J].Mol Biol Rep, 38(1):17-21.
Thompson DA, Cronin CN, Martin F. 1994a. Genomic cloning and sequence analyses of the bovine α-, βA- and βB-Inhibin/activin genes [J].Eur J Biochem,226(3): 751-764.
Thompson JD, Higgins DG, Gibson TJ. 1994b. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice.Nucl Acids Res,22(22): 4673-4680.
Wu WS, Hua GH, Yang LG, Wen QY, Zhang CY, Zoheir KM, Chen SL.2009. Association analysis of theINHAgene with litter size in Boer goats [J].Small Rumin Res,82(2-3): 139-143.
Xie TS, Nong W, Zhang XM, Huang YQ, Cui KQ, Shi DS. 2009. Cloning and prokaryotic expression of swamp buffalo Inhibin alpha subunit gene [J].Heilongjiang Anim Sci Vet Med,(8): 14-17. [谢体三, 农微,张新民, 黄雅琼, 崔奎青, 石德顺. 2009. 水牛抑制素α亚基基因的克隆与原核表达. 黑龙江畜牧兽医, (8): 14-17.]
Xue Y, Chu MX, Zhou ZX. 2004. Advances on inhibin genes[J].Hereditas,26(5): 749-755. [薛昱, 储明星, 周忠孝. 2004. 抑制素基因的研究进展. 遗传, 26(5): 749-755.]
Zhang CX. 2000. Chinese Buffalo Science [M]. Nanning: Guangxi Science and Technology Press. [章纯熙. 2000. 中国水牛科学[M]. 南宁: 广西科学技术出版社.]
Zhou WR, Chu MX, Sun SH, Fang L, Ye SC. 2007. A candidate geneINHAfor prolificacy of small Tail Han sheep [J].J Agric Biotechnol, 15(1):32-36. [周文然, 储明星, 孙少华, 方丽, 叶素成. 2007. 小尾寒羊高繁殖力候选基因INHA的研究[J]. 农业生物技术学报, 15(1): 32-36.]
Polymorphisms of inhibin α gene exon 1 in buffalo (Bubalus bubalis),gayal (Bos frontalis) and yak (Bos grunniens)
MIAO Yong-Wang1,#,*, HA Fu1,#, GAO Hua-Shan1,YUAN Feng, LI Da-Lin2, YUAN Yue-Yun2
(1.Faculty of Animal Science and Technology,Yunnan Agricultural University,Kunming650201,China;
2.Domestic Animal Breeding and Crossbreed-improvement Station of Yunnan Province,Kunming650225,China)
To elucidate the genetic characteristics of the bovine Inhibin α subunit (INHA) gene, the polymorphisms in exon 1 ofINHAand its bilateral sequences were assayed using PCR with direct sequencing in buffalo, gayal and yak. A comparative analysis was conducted by pooled the results in this study with the published data ofINHAon some mammals including some bovine species together. A synonymous substitution c.73C>A was identified in exon 1 ofINHAfor buffalo, which results in identical encoding product in river and swamp buffalo. In gayal, two non-synonymous but same property substitutions in exon 1 ofINHA, viz. c.62 C>T and c.187 G>A, were detected, which lead to p. P21L, p.V63M changes in INHA, respectively. In yak, nucleotide substitution c.62C>T, c.129A>G were found in exon 1 ofINHA,the former still causes p. P21L substitution and the latter is synonymous. For the sequence of the 5'-flanking region ofINHAexamined, no SNPs were found within the species, but a substitution, c. -6T>G, was found. The nucleotide in this site in gayal, yak and cattle was c. -6G, whereas in buffalo it was c. -6T. Meanwhile, a 6-bp deletion, namely c.262+31_262+36delTCTGAC, was found in the intron of buffaloINHAgene. For this deletion, wild types (+/+) account for main part in river buffalo while mutant types (-/-) are predominant in swamp buffalo. This deletion was not found in gayal, yak and cattle, though these all have another deletion in the intron ofINHA, c. 262+78_262+79delTG. The results of sequence alignment showed that the substitutions c. 43A and c. 67G in exon 1 ofINHAare specific to buffalo, whereas the substitutions c. 173A and c. 255G are exclusive to gayal, yak and cattle, and c. 24C, c. 47G, c. 174T and c. 206T are specific to goat. Furthermore, there are few differences among gayal, yak and cattle, but there relatively great differences between buffalo, goat and other bovine species regarding the sequences ofINHAexon 1.
Buffalo; Gayal; Yak; Inhibin α gene(INHA)exon 1; Polymorphism
S823.8;Q75
A
0254-5853-(2012)04-0402-07
10.3724/SP.J.1141.2012.04402
2012-02-16; 接受日期: 2012-04-13
云南省应用基础研究重点项目(2007C0003Z); 国家自然科学基金项目(30660024); 云南省应用基础研究计划面上项目(2006C0034M);国家高技术研究发展计划(“863”计划)项目(2008AA101001); 云南省财政厅基金项目(槟榔江水牛种质特性研究)
∗通信作者(Corresponding author), E-mail: yongwangmiao999@yahoo.com.cn
#共同第一作者(Authors contributed equally to the work)
苗永旺(1964—), 男, 博士,教授,从事动物遗传学教学和研究工作; 哈福(1974— ), 男, 博士, 副教授, 主要从事动物繁殖学教学和科研工作
猜你喜欢
杂志排行
Zoological Research的其它文章
- 中国蛇类物种丰富度地理格局及其与生态因子的关系
- Genetic diversity and differentiation of masu salmon (Oncorhynchus masou masou) between and within cultured populationsinferred from microsatellite DNA analysis
- 具资源效应的非对称“鹰鸽博弈”进化稳定分析
- Tssk1和Tssk2的克隆、表达及其在成熟精子中的分布与亚细胞定位
- 抵抗素对NAFLD肝纤维化的影响及可能的体内外机制
- 文昌鱼tropomyosin基因的克隆、进化分析及其胚胎发育与成体中的表达模式
