棉花GhFTL1基因启动子的克隆及序列分析
2012-11-10张定国黄先忠东锐郑银英崔百明
张定国,黄先忠,东锐,郑银英,崔百明
(石河子大学生命科学学院/农业生物技术重点实验室,石河子832003)
FT(Flowering Locus T)基因是光周期途径中决定植物开花的重要因子。CO和FT基因在植物开花中起着关键作用,CO基因在长日照下能够诱导叶片FT基因的表达,当FT基因在茎尖特异表达时可以促进花芽分化。FT蛋白(诱导开花的成花素信号分子)从叶片经过韧皮部长距离运输到达茎顶端分生组织后,与顶端组织特异表达的Flowering Locus D(FD)基因编码产物之间结合,形成一种蛋白复合体,共同促进下游成花基因Apetala 1(AP1)、Leafy(LFY)等的表达,从而促进植物开花[1]。棉花Gh FTL1基因已经被克隆,系统进化分析表明Gh FTL1属于FT亚家族成员[2]。
染色体步移技术(Genome walking)是一种重要的分子生物学研究技术,使用这种技术可以有效获取与已知序列相邻的未知序列。热不对称交错PCR(Thermal asymmetric interlaced PCR,TAILPCR)技术[3]因快速、简单、高效等优点而备受青睐,目前已有大量成功报道[4]。Wang 等[5]利用 TAIL-PCR法分离了小球藻硝酸还原酶基因5′端侧翼序列,将其与GUS基因融合并表达,结果显示该侧翼序列能启动GUS基因的表达,该片段含有硝酸还原酶基因转录表达所必要的启动子元件。财音青格乐等[6]通过此方法,成功地克隆了大豆种子特异性启动子序 列 (约 700 bp)。李 秋 莉 等[7]也 应用TAIL-PCR法成功地克隆了辽宁碱蓬BADH基因的启动子片段。Liu等[8]报道,TAIL-PCR 方法通常可以扩增到0.2~2.0 kb的片段。因此,TAILPCR法是克隆未知启动子的一种有效、简便的方法。
本实验室已经获得GhFTL1基因的序列[2],为了研究Gh FTL1基因自身启动子的功能,利用TAIL-PCR方法从棉花基因组中分离了长度为715 bp的该基因序列片段,并对已得到的序列片段序列进行分析,旨在为进一步研究该启动子的功能奠定基础。
1 材料与方法
1.1 材料
1.1.1植物材料
实验材料为新疆陆地棉品种新陆早33。
1.1.2菌株和质粒
大肠杆菌DH5α、根癌农杆菌GV3101、植物表达载体pCAMBIA1301、p35S::Gh FTL1为本实验室保存。
1.2 方法
1.2.1 DNA的提取
棉花DNA提取采用CTAB法。
1.2.2引物的设计及合成
根据已知棉花FT同源(登录号HM631972)基因的序列,设计引物(华大基因合成)(表1)用作PCR扩增。

表1 TAIL-PCR引物Tab.1 Primers of TAIL-PCR
1.2.3 pGhFTL1启动子片段的克隆
TAIL-PCR第1轮扩增:以棉花叶片DNA为模板,在50μL的反应体系中加入DNA模板2μL,5μL 10×LA Buffer,8μL d NTP Mixture,0.5μL 5U LA Taq DNA聚合酶,以AD1为正向引物,SP1为反向引物各1μL,dd H2O补齐。第2轮扩增:将第1轮PCR反应液稀释100倍后,取1μL作为第2轮PCR反应的模板,5μL 10×LA Buffer,8μL d NTP Mixture,0.5μL 5 U LA Taq DNA聚合酶,以AD1为正向引物,SP2为反向引物各1μL,dd H2O补齐。第3轮扩增:将第2轮PCR反应液稀释100倍后,取1μL作为第3轮PCR反应的模板,反应体系与第2轮的相同。取上述各步PCR反应液在1%的琼脂糖凝胶上检测,割下目的条带,利用凝胶回收试剂盒纯化后,将回收产物与载体p GEM-T Easy连接。连接产物转化DH5α感受态细胞,在涂有IPTG/X-gal及氨苄抗生素的LB平板上通过蓝白斑筛选,挑取阳性克隆于LB培养基中过夜摇菌,小量提取质粒经酶切鉴定后,将阳性克隆交由北京华大基因公司测序。
第1轮反应程序:94℃1 min,98℃1 min 1个循环;94℃30 s,61℃1 min,72℃2 min 5个循环;94℃30 s,25℃3 min,72℃2 min 1个循环;94℃30 s,61℃1 min,72℃2 min 94℃30 s,61℃1 min,72℃2 min 94℃30 s,44℃1 min,72℃2 min 15个循环;72℃10 min 1个循环。
第2轮反应程序:94℃30 s,61℃1 min,72℃2 min 94℃30 s,61℃1 min,72℃2 min 94℃30 s,44℃1 min,72℃2 min 15个循环;72℃10 min 1个循环。
第3轮反应程序:同第2轮反应程序。
1.2.4 GhFTL1启动子的验证
为了验证所克隆的片段是GhFTL1的启动子,从已克隆的启动子片段中设计2个引物P-Jian-F-1:5′-TGGGGTGTGTATGCAGTTGT-3′,P-Jian-F-2,5′-GAATCACAGAAAG GGCAAGC-3′分 别和TAIL-PCR中所用到的SP1引物进行PCR验证,以棉花叶片DNA为模板,在10μL反应体系中加入DNA模板0.5μL,1μL 10×Hotmaster Buffer,1μL d NTP Mixture,0.1μL 2.5 U Hotmaster Taq DNA 聚合酶,以 P-Jian-F-1和 P-Jian-F-2为正向引物,SP1为反向引物各0.5μL,dd H2O补齐。
PCR反应程序为:94℃30 s,61℃1 min,72℃2 min 94℃30 s,61℃1 min,72℃2 min 94℃30 s,44℃1 min,72℃2 min 15个循环;72℃10 min 1个循环。
2 结果与分析
2.1 GhFTL1启动子片段的克隆与测序
利用引物 AD1、AD2、AD3和SP1、SP2、SP3分别进行3组3轮PCR,第3轮扩增出约为800 bp的产物(图1),将第3轮PCR产物回收纯化后连接到p GEM-T载体上,转化大肠杆菌DH5α,挑取单克隆提取质粒经EcoRⅠ酶切鉴定(图2)。挑选其中阳性克隆进行测序,将800 bp的测序结果经DNAstar软件分析,和已知的FT序列比对,该序列确实为Gh FTL1上游启动子序列。
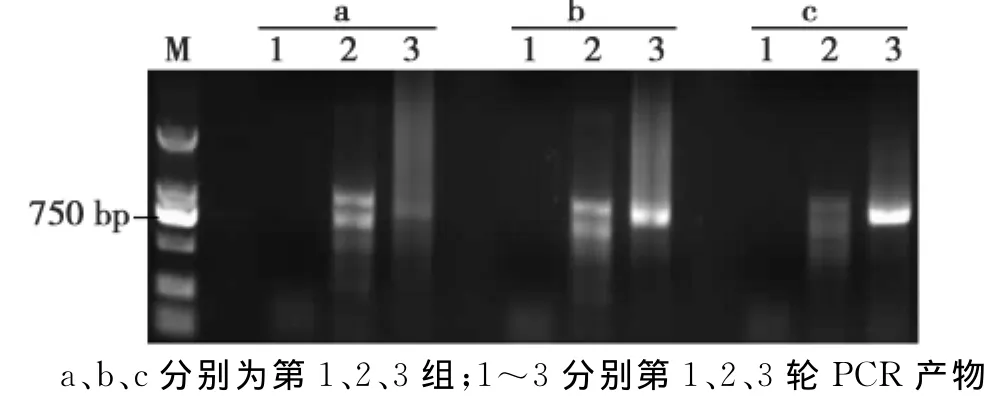
图1 Genome Walking PCR方法经过三轮扩增后PCR片段Fig.1 PCR fragments amplified by using genome Walking PCR
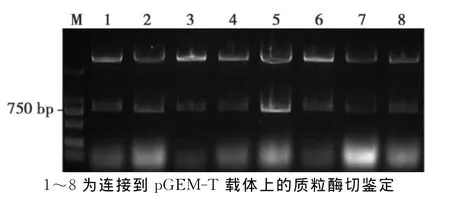
图2 pGEM-T载体上的质粒酶切鉴定Fig.2 The restriction enzyme digestion analysis of pGEM-T vector
2.2 GhFTL1启动子的验证
从已克隆的启动子片段中设计2个引物,分别与TAIL-PCR中所用到的SP1引物进行PCR验证。将PCR产物用1%的琼脂糖凝胶进行电泳。电泳结果(图3)与预期结果一致。
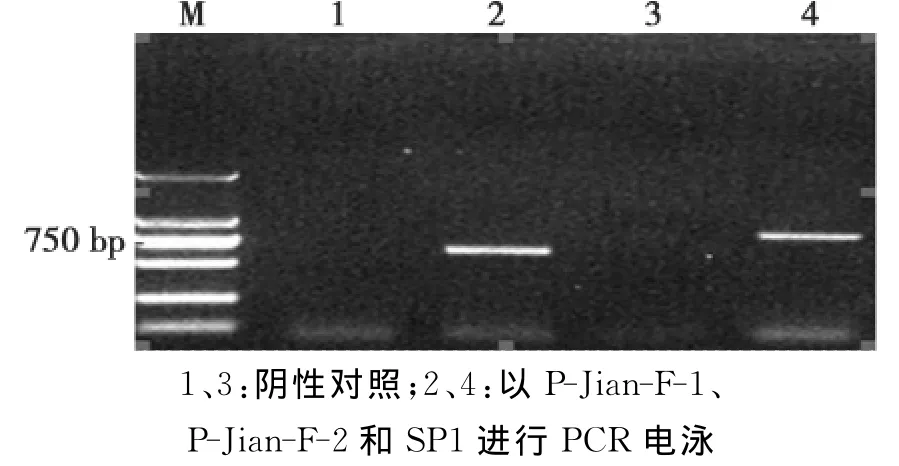
图3 PCR验证启动子Fig.3 PCR validation promoter
2.3 GhFTL1启动子序列分析
通过TAIL-PCR,获得715 bp的启动子片段,经Program NSITE(Softberry Inc.)序列分析表明,在该片段起始密码子104 bp处有1个典型的TATA box,预测的转录起始位点在起始密码子前69 bp处,除了有TATA box、CAAT box等基本元件外,还含有与光调节有关的元件,GT-1和SP1,另外还有如 CAr G box、GT-1#Box7、telo-box、GS1b AC box 1等其他一些元件(图4)以及逆境胁迫相关等的顺式作用元件,通过同源克隆法先后从小拟南芥中克隆了一些耐逆基因,这些基因的启动子中有许多逆境胁迫元件[9-11]。
植物开花受环境因素和发育状态的控制,在拟南芥中几种开花信号,包括光周期应答,集中在FT基因的转录调节水平上。当拟南芥FT在长日照条件的诱导下FT在叶片的维管组织中转录,几种转录抑制子参与了FT的调节,尽管这些元件对FT调节的重要性没有完全得到证明,但FLC的MADS box与SVP形成的复合物与FT启动子近端区域和包含有CAr G boxe的第1个内含子有一定的联系[12]。FT的表达受FLC表达的抑制,这种抑制受FT启动子的介导,在FT的启动子中有1个CArG box。拟南芥FT基因编码的蛋白产物是可以长距离转运的成花激素,可从叶片运输到芽顶端,优先在芽顶端表达的FD,对FT促进开花是必需的,FD和FT通过蛋白互作,激活花分生组织特异性基因AP1,从而促进开花[13]。
GT元件是一个光应答元件。GT元件最初是在豌豆叶片rbcS-3A基因启动子中被鉴定出来,其核心序列为5′-GTGTGGTTAATATG。GT-1是一种蛋白,其可与光调节基因的启动子相互作用,分布在cab-E启动在的7个不同为点上。在cab-E基因的启动子内GT-motifs是通过GT-1把他们结合在一起,rbcS-3A基因启动子与GT-1的结合位点序列和cab-E基因的启动子与GT-1的结合位点序列一致[14]。紫外光能能增强Tdc启动子的表达,通过紫外光证明了GT-1在Tdc中调节的功能。PsGS1b在整个植物的维管组织中表达,在不同的新陈代中铵盐的重新利用中起重要作用,还可在酵母、拟南芥和松树的细胞中激活转录[15]。分析了PsGS1b的启动子的403 bp的序列在该启动子转录起始位点上游的1个区域含有大量的AC元件,这个区域被称为GS1b AC box 1,其包含3种AC元件,AC元件能调节基因的表达[16]。siteⅡ和telobox是一种顺式调节元件,与基因的分生表达有关。细胞循环的众多基因启动子中siteⅡ和telo-box之间存在保守关联,包括几种细胞循环有关的基因和编码核糖体蛋白的拟南芥153基因。植物在拟南芥启动siteⅡ元件的控制下可观测到GUS基因的分生表达,而启动子中的telo-box能增强这一表达。telo-box在拟南芥中编码延长因子基因的启动子中首次被观测到,后来在几种植物的RP启动子中也发现了这一元件[17],已经确定telo-box可激活细胞循环中一组基因的诱导表达或过量表达[18]。telobox不能完全依赖自身来激活基因的表达,但要同其他顺式激活序列协同作用才能发挥作用。因此,初步推断此序列片段可能是Gh FTL1基因的启动子。

图4 pGhFTL1启动子的序列分析Fig.4 Sequence analysis of Cotton pGhFTL1 gene promoter
3 结论与讨论
在转基因棉工程中,有关启动子在棉花中的研究较多,但是大多数采用组成型启动子。其中所应用的表达载体基本上是利用外源型启动子Ca MV 35S。Tail-PCR、SiteFinding-PCR 等克隆技术的发展,加快了启动子克隆的进程。近年来,世界各国正在加快棉花基因组的测序,并且随着克隆方法的不断改进,棉花基因组中各种类型的启动子已被大量克隆测序,其功能也在一定程度上得到验证。在棉花基因工程研究中,除利用外源启动子外,越来越多的棉花内源启动子被开发出来,利用棉花内源启动子本身的特性来改良基因在棉花中的表达。
TAIL-PCR技术[13]因快速、简单、高效等优点而备受青睐,本实验采用TAIL-PCR方法克隆了Gh FTL1启动子片段。特异性引物设计基本遵循一般PCR 引物设计原则[18],为了提高 TAIL-PCR扩增的特异性,本实验进行了两处重要改进:1)3条特异引物退火温度均高于60℃,低于72℃,且3条特异引物sp1、sp2、sp3退火温度依次升高,使第1轮及第2轮TAIL-PCR中的非特异扩增片段不能进一步有效扩增,进而提高终产物扩增的特异性;2)特异性引物3′末端5~10个碱基中适当提高G、C含量,尤其是3′末端5个碱基设计时不要出现二联体或三连体的A或T,同时增加G或C碱基的个数,这样可大幅减少特异引物的自身非特异扩增。本实验采用改良的TAIL-PCR法取代5′RACE方法分离基因的5′端序列,为全长基因克隆提供了一种新选择。改良后的TAIL-PCR技术很大程度地弥补了原TAIL-PCR技术非特异扩增高、成功率低的缺陷,通过随机引物的筛选及特异引物设计的改进,极大地提高了TAIL超级循环中特异扩增的效率,操作简单,3轮超级循环在1 d内就可完成,因此该技术省时省力、快速高效。改良后的TAIL-PCR技术对试剂、仪器等的要求相对较低,一般实验室均可满足要求,因此该技术是进行全长基因克隆的理想选择,具有较广阔的应用前景。
对克隆的启动子片段进行序列分析,在此启动子上不仅含有TATA box和CAAT box基本元件,而且含有与光调节有关的元件,如GT-1和SP1,另还外存在一些其他元件,在该片段在起始密码子104 bp处有1个典型的TATA box,预测的转录起始位点在起始密码子前69 bp处。
[1]ABE M,Kobayashi Y,Yamamoto S,et al.FD,a bZIP protein mediating signals from the floral pathway integrator FT at the shoot apex[J].Science,2005,309:1052-1056.
[2]东锐,院海英,顾超,等.棉花GhFTL1基因的克隆及初步功能分析[J].棉花学报,2011,23(6):515-521.
[3]许锋,称水源,王燕,等.TAIL-PCR方法快速克隆银杏查尔酮合成酶基因及序列分析[J].果树学报,2007,24(2):237-243.
[4]应革,武威,何朝族.TAIL-PCR方法快速分离Xcc致病相关基因序列[J].生物工程学报,2002,28(2):182-186.
[5]Wang P,Sun Y,Li X,et al.Rapid isolation and functional analysis of promoter sequence of the nitrate reductase gene from Chlorella ellipso idea[J].J Appl Phycol,2004,16(11):11-16.
[6]财音青格乐,李明春,蔡易,等.大豆种子特异性启动子的分离及结构分析[J].中国农业科学,2005,38(3):454-461.
[7]李秋莉,张毅,尹辉,李丹.辽宁碱蓬甜菜碱醛脱氢酶(BADH)基因启动子分离及序列分析[J].生物工程学报,2006,22(1):77-81.
[8]Liu Y G,Huang N.Effecient amplification of insert and sequences from bacterial artificial chromosome clones by thermal asymmetric interlaced PCR[J].Plant Molecular Biology Reporter,1998,16:175-181.
[9]崔百明,李予霞,乐锦华,等.小拟南芥COR15α基因的克隆及序列分析[J].石河子大学学报:自然科学版,2003,7(2):178-179.
[10]任艳利,崔百明,张秀春,等.小拟南芥ApCBF基因的克隆及转化烟草的初步研究[J].石河子大学学报:自然科学版,2006,26(5):591-595.
[11]黄先忠,张鹏,吕新华,等.新疆小拟南芥ApCBF1基因的克隆和过量表达转基因的研究[J].石河子大学学报:自然科学版,2009,27(3):265-267.
[12]Searle I,He Y H,Turck F,et al.The transcription factor FLC confers a flowering response to vernalization by repressing meristem competence and systemic signaling in Arabidopsis[J].GenesDev,2006,20:898-912.
[13]Corbesier L,Vincent C,Jang S,et al.FT Protein movement contributes to long-distance signaling in floral induction of Arabidopsis[J].Science,2007,316:1030-1033.
[14]Schindler U,Cashmore A R.Photoregulated gene expression may involve ubiquitous DNA binding proteins[J].EMBO J,1990,9:3415-3427.
[15]Josefa Gomez-Maldonado,Concepcion Avila1,Fernando dela Torre.Functional interactions between a glutamine synthetase promoter and MYB proteins[J].The Plant Journal,2004,39:513-526.
[16]Go′mez-Maldonado J,Ca′novas F M,Avila C.Molecular analysis of the 5′upstream region of a gibberel lininduciblecytosolic glutamine synthetase gene(GS1b)expressed in thepine vas cular tissue[J].Planta,2004,218:1036-1045.
[17]Axelos M,Bardet C,Liboz T,et al.The gene family encoding the translation elongation factor eEF1A:molecular cloning,characterization and expression[J].Mol Gen Genet,1989,219:106-112.
[18]Manevski A,Bertoni G,Bardet C,et al.In synergy with various cis-acting elements,plant interstitial telomere motifs regulate gene expression in Arabidopsis root meristems[J].FEBS Lett,2000,483:43-46.
