三株H9N2亚型禽流感病毒全基因组序列分析
2012-09-26刘国乾冯军祥张长辉焦培荣亓文宝
刘国乾,曹 蓝,和 君,冯军祥,张长辉,田 进,焦培荣,亓文宝,廖 明
(华南农业大学兽医学院,人兽共患病防控制剂国家地方联合工程实验室,农业部兽用疫苗创制重点实验室,广东省动物源性人兽共患病预防与控制重点实验室,广东广州510642)
禽流感病毒(Avian influenza virus,AIV)变异频繁,血清亚型众多,迄今已发现16种HA亚型和9种NA亚型。1966年,H9N2亚型AIV首次分离于北美洲,1994年在中国大陆首次分离到,并逐渐成为中国大陆流行的AIV亚型[1]。近年的研究结果表明,H9N2亚型AIV的HA基因大部分来自于Ck/BJ-like,并且通过与 G1-like、G9-like(代表毒株为 A/Chicken/Hong Kong/G9/97)、Y439-like(代表毒株为 A/duck/Hong Kong/Y439/97)及SH/F/98-like(代表毒株为 A/Chicken/Shanghai/F/98)基因重组,形成了多基因型H9N2 AIV[2]。
H9N2亚型AIV为低致病力病毒,分布广泛,能引起呼吸道症状和产蛋下降,并造成宿主的免疫抑制,以及与其他病原微生物通过协同作用引起禽类发病[4-5]。该病毒不仅对家禽具有危害,还可以跨宿主传播,感染哺乳动物,具有可能产生抗原特性与遗传特性都完全有别于禽流感的新型流感病毒风险[6]。一些H9N2亚型AIV获得人流感病毒的一些特异性受体,可以感染人类,引起轻微的呼吸道疾病[7-8]。而且,H9N2亚型 AIV 对 H5N1亚 型 AIV病毒的基因多样性具有一定贡献,1997年流行的H5N1亚型禽流感的内部基因可能来源于G1-like[9]。这些发现表明H9N2亚型AIV存在在人与家禽中流行的风险。因此,对H9N2亚型AIV的研究不仅有助于了解其在畜禽中的流行特点,而且还具有重要的公共卫生意义。
本研究对3株华南地区不同年代分离的H9N2 AIV分离株的全基因组进行了序列测定,并进行了详细的系统进化分析,为解析华南地区H9N2 AIV的分子流行病学特点提供科学依据。
1 材料与方法
1.1 材料
1.1.1 病 毒 CK/GD/162/03、Francolin/GD/298/05、Ck/GD/A8/10 3株 H9N2亚型禽流感病毒毒株,由华南农业大学兽医学院传染病教研室分离、鉴定并保存。
1.1.2 试剂 Trizol试剂为Invitrogen公司产品;DNA Marker DL2000、d NTPs(each2.5 mmol/L)、RNA酶抑制剂和反转录酶M-MLV均为宝生物工程(大连)有限公司产品;EZNA Gel Extraction Kit纯化试剂盒为Omega公司产品。
1.2 方法
将含有病毒的鸡胚尿囊液利用Trizol试剂提取病毒的RNA,再用M-MLV酶将所得到的RNA反转录成cDNA,然后利用针对禽流感病毒8个基因片段的特异性引物[10]进行PCR扩增,并按照EZNA Gel Extraction Kit纯化试剂盒说明书回收目的片段,送上海英潍捷基生物有限公司测序。
利用软件 DNA Star(version 5.07,DNA Star公司,USA)和 MEGA4(version 4.0,biodesign institute of center for evolutionary functional genomics,USA)对所得到的序列和部分具有代表性的参考序列进行分析。系统进化树是用 MEGA 4中“neighbor-joining”算法所得。用于计算核苷酸序列相似性和绘制系统进化树的各基因片段见表1。

表1 3株H9N2亚型AIV分离株8个基因片段Table1 Genome profile of the 3 H9N2 isolates and gene region for phylogenetic analysis
2 结果
2.1 核苷酸序列相似性分析
应用DNAStar软件的Meg Align功能,将CK/GD/162/03、Francolin/GD/298/05、Ck/GD/A8/10 3个毒株各基因片段与部分具有代表性的参考毒株(图1~图8)进行比对,发现CK/GD/162/03毒株PB2、PB1基因与 A/Hong Kong/156/97(H5N1)相似性分别达到96.9%和96.7%,而Francolin/GD/298/05毒株M基因与 A/Hong Kong/156/97(H5N1)相似性达到96.0%;CK/GD/162/03、Francolin/GD/298/05、Ck/GD/A8/10 3 个毒株的 NP基因与 A/VN/1203/04(H5N1)的相似性最高,分别 为97.9%、97.4%、96.3%;CK/GD/162/03、Francolin/GD/298/05 2个毒株 PA 基因亦与 A/VN/1203/04(H5N1)的相似性分别达到95.6%与94.5%。其余基因均与H9N2亚型禽流感的相似性最高,其中 CK/GD/162/03 HA 基因与 Ck/BJ/1/94 的相似性最高,Francolin/GD/298/05、Ck/GD/A8/10 HA基因与Dk/HK/Y280/97相似性最高,都属于 BJ/1/94-like;CK/GD/162/03、Francolin/GD/298/05两毒株 NA基因与Ck/SH/F/98的相似性最高,Ck/GD/A8/10 NA 基因与 Dk/HK/Y280/97相似性最高,并且Ck/GD/A8/10在62~64位缺失3个氨基酸。
2.2 遗传进化分析
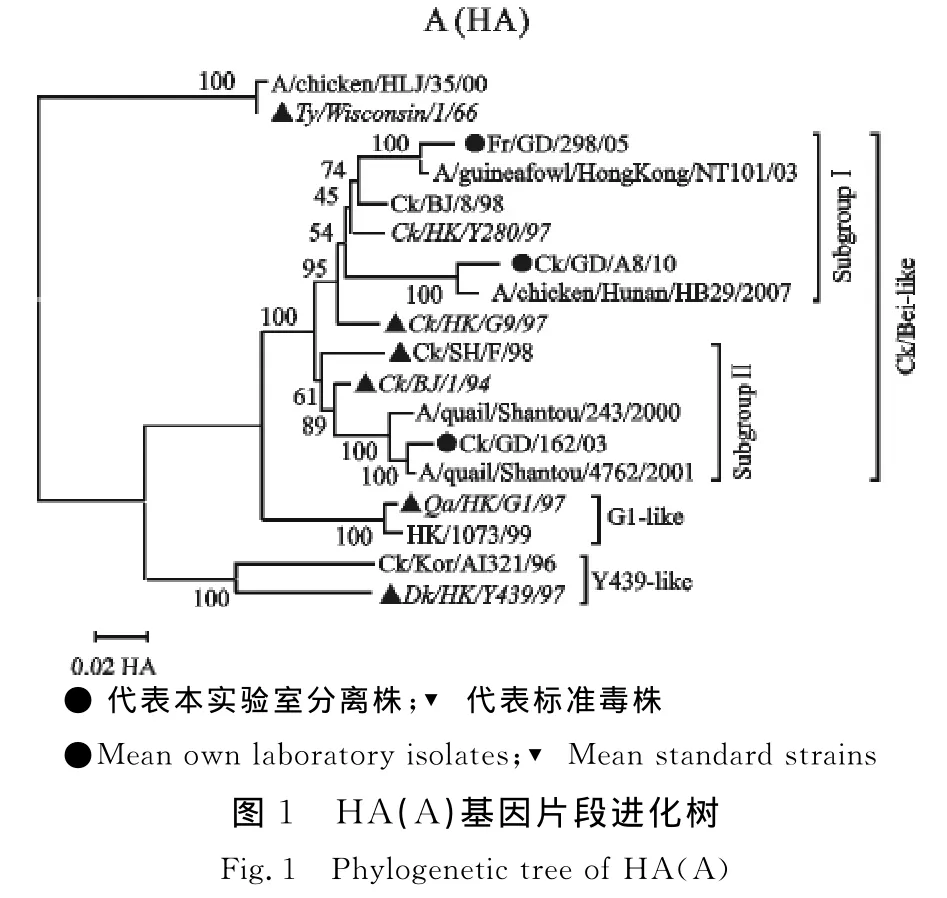
根据H9亚型AIV HA基因和NA基因的序列特点,将其分为欧亚系和北美系两个大分支。HA基因欧亚系又分为三大亚系:Y280-like,代表毒株为 Dk/HK/Y280/97或 Ck/BJ/1/94;G1-like,代表毒株为 Qa/HK/G1/97;Y439-like,代表毒株为DK/HK/Y439/97;北美型代表毒株为 Ty/Wisconsin/1/66[3,11]。NA 基因欧亚系又分为三大亚系,代表毒株分别是 Ck/HK/G9/97和 Qa/HK/G1/97,Dk/HK/Y439/97[12]。进化分析结果表明3个毒株HA基因位于欧亚系中的Ck/Bei-like分支,而Ck/Bei-like又分为亚群Ⅰ与亚群Ⅱ两个亚枝,其中以Dk/HK/Y280/97为代表毒株的亚群Ⅰ主要流行于鸡,以Qa/ST/243/00为代表毒株的亚群Ⅱ为鹌鹑等珍禽类流行株[3,11],本试验分离株Francolin/GD/298/05、Ck/GD/A8/10属于亚群Ⅰ,Ck/GD/162/03属于亚群Ⅱ(图1)。NA基因位于系统发生树的Ck/Bei-like分支,其中CK/GD/162/03、Francolin/GD/298/05属于 G9-like,Ck/GD/A8/10 属于Y280-like(图2),这个结果与 Guan[3,11]等报道的华南地区流行的H9N2亚型AIV相一致。
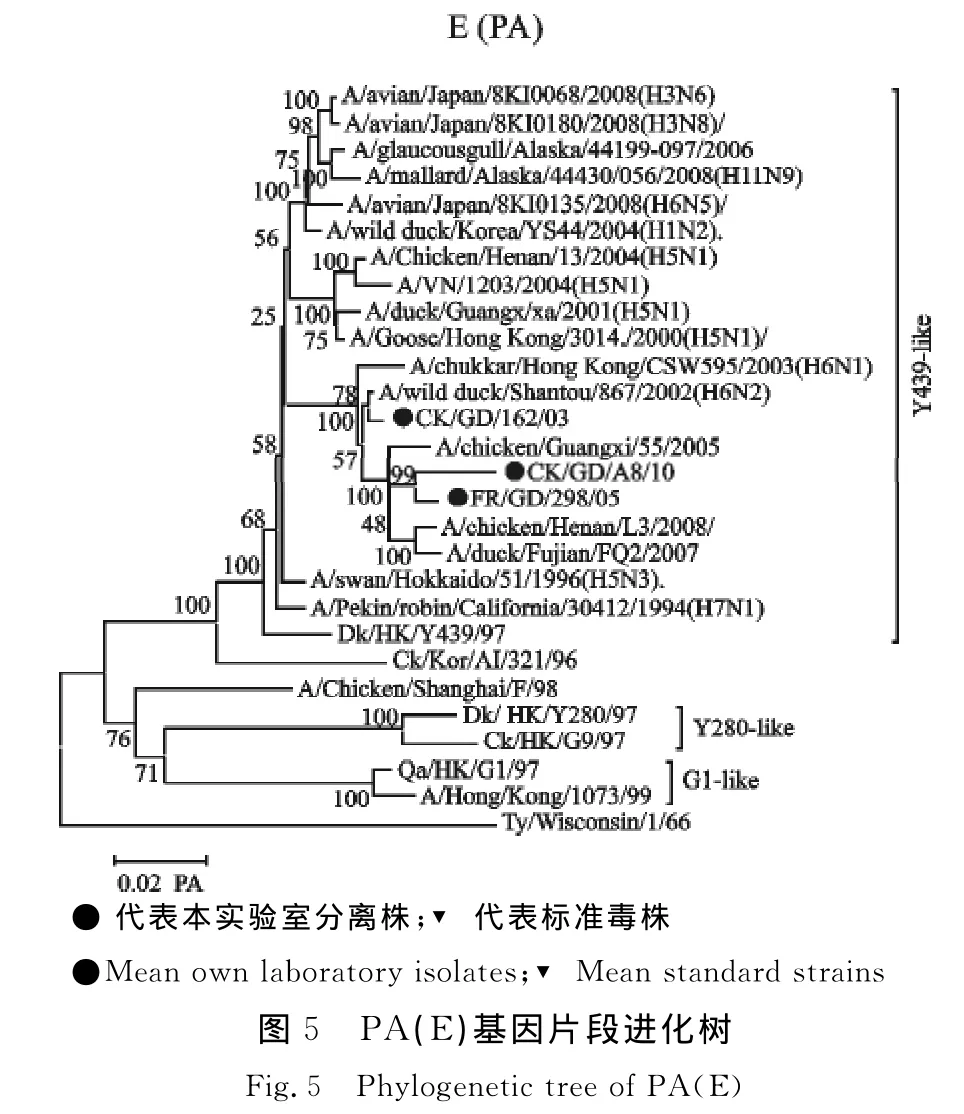
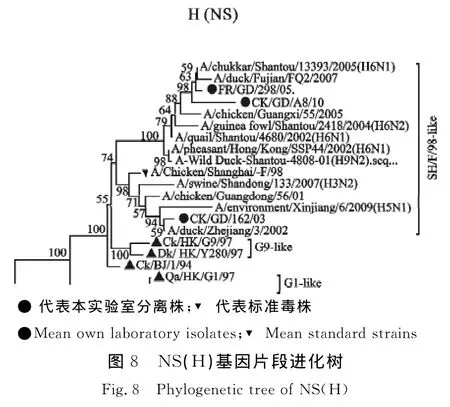
进化分析结果显示,内部基因(PB2、PB1、PA、NP)基因来源具有多样性,出现不同程度重组现象。Francolin/GD/298/05、Ck/GD/A8/10 PB2基因位于系统发生树的SH/F/98-like分支,CK/GD/162/03 PB2基因于系统发生树的G9-like分支(图3);CK/GD/162/03 PB1基因位于系统发生树的 G1-like 分 支,Francolin/GD/298/05、Ck/GD/A8/10PB1基因位于系统进化树的 BJ/1/94-like分支(图4);PA基因位于系统进化树的Y439-like分支,但与Y439-like分支已经有一定的遗传距离(图5);NP、NS基因均位于系统进化树的SH/F/98-like分支,但NP基因与H5N1亚型AIV在同一分支,Francolin/GD/298/05、Ck/GD/A8/10 NS 基因与H6亚型 AIV在同一分支,CK/GD/162/03 NS与H5亚型AIV在同一分支(图6和图8);CK/GD/162/03 M 基因位于系统进化树的 Ck/Bei-like分支,与 A/swine/Shandong/2/03(H5N1)在同一分支,Francolin/GD/298/05、Ck/GD/A8/10 M 基因位于系统进化树的G1-like分支,与H6亚型AIV在同一分支(图7)。


2.3 各基因型分子特征分析
3个分离株HA基因推导出的氨基酸长度均为560aa,其中包括信号肽(1 aa~18 aa)、HA1(19 aa~337 aa)和 HA2(339 aa~560 aa)3个区域,3个分离株HA裂解位点均为RSSR↓GLF,符合低致病性禽流感病毒的分子特征。对HA 226位受体位点进行分析,CK/GD/162/03 HA蛋白的受体结合位点的第226位氨基酸为 Q(Gln),Francolin/GD/298/05和Ck/GD/A8/10这2个毒株 HA蛋白的受体结合位点的第226位氨基酸均由Q(Gln)变为了L(Leu),使病毒由识别唾液酸α2-3(SAα2-3)受体变为识别哺乳动物唾液酸α2-6(SAα2-6)受体。Francolin/GD/298/05分离毒株HA上共有7个相对保守的糖基化位点,分别位于第29~31、218~220、305~307、551~553、141~143、298~300、492~494位氨基酸,而 CK/GD/162/03、Ck/GD/A8/10分离株在218~220位氨基酸失去一个潜在糖基化位点。
属于G9-like分支的CK/GD/162/03 Francolin/GD/298/05分离株具有全长 NA(569 aa),而属于Y280-like分支的 Ck/GD/A8/10 在 NA 颈部 区 域(62~64位)缺失3个氨基酸,但是处在Y280-like分支根部的Ck/Bei-like没有这3个氨基酸缺失,这说明这种缺失是在进化过程中发生的。


2.4 基因型分析
[13]对H9N2 AIV基因型分型,可将本试验获得的3株H9N2 AIV进一步分为3个不同的基因型,其中 CK/GD/162/03 与 Ck/GD/A8/10的6个内部基因中有4个基因发生重组,例如CK/GD/162/03的 PB2、PB1 来 自 于 Qa/HK/G1/97,HA、NS来自于 CK/BJ/1/94,PA 来自于 A/quail/Shantou/5663/2001,NP 来自于 CK/SH/F/98,NA来自 于 CK/HK/G9/97,故 CK/GD/162/03 属 于B34。Francolin/GD/298/05有5个基因发生了重组。Ck/GD/A8/10与Francolin/GD/298/05的7个基因分别来自相同的分支,其PB1和NP基因来源于CK/SH/F/98;PB1和 NS基因来源于 CK/BJ/1/94;PA 基 因 来 源 于 A/quail/Shantou/5663/2001;HA 基因来源于 DK/HK/Y439/97;但 NA 不同,FR/GD/298/05的 NA 基 因 来 源 于 CK/HK/G9/97,CK/GD/A8/10的 NA 基因来源于 CK/BJ/1/94,故FR/GD/298/05属于B47,CK/GD/A8/10属于B54,具体结果见(表2、表3、图9)。

表2 标准参考毒株基因型Table2 Genotypes of classic H9N2 reference viruses

表3 3株H9N2分离株各基因片段来源Table3 Genotype description of 3 H9N2 isolates in South China

3 讨论
本试验的3株H9N2亚型AIV分离株属于Ck/BJ-like分支,符合我国大陆 H9N2亚型 AIV HA基因的遗传进化特点,但各基因出现不同程度重组现象。
CK/GD/162/03、 Francolin/GD/298/05 与CK/GD/A8/10分别属于 B34、B47和 B54基因亚型,而这3个亚型是从2002年开始流行于中国大陆的,具有各基因片段高重组的特点,B34和B47宿主也具有多样性,分布在鸡、鸭等家禽以及鹌鹑、鹧鸪等珍禽,B54主要流行于鸡[14]。从鸡分离的毒株CK/GD/162/03的 HA 处在以 Qa/ST/243/00为代表毒株的亚群Ⅱ(为鹌鹑等珍禽类流行株),从中华鹧 鸪 分 离 的 Francolin/GD/298/05HA 处 在 以Dk/HK/Y280/97为代表毒株的亚群Ⅰ(主要流行于鸡),也充分说明了这一点。Xu K M 等[11]对华南地区H9N2亚型的分子流行病学调查表明,鹌鹑可能对H9N2亚型AIV流行发挥重要作用,从以上分析推测,CK/GD/162/03分离株可能是从鹌鹑等珍禽传播到鸡群,Francolin/GD/298/05分离株可能是从鸡群传播到鹧鸪等珍禽,而CK/GD/A8/10可能为鸡群流行株。
Qa/HK/G1/97与CK/SH/F/98的 RNP基因可能为H5N1亚型禽流感内部基因的供体,本试验中3个分离株RNP基因来源于Qa/HK/G1/97与CK/SH/F/98分支,其中3个分离株的NP基因与A/VN/1203/04(H5N1)核苷酸相似性达到96.3%以上,氨基酸相似性更是高达98.8%以上,PA基因与A/VN/1203/04(H5N1)的核苷酸相似性达到94%以上,CK/GD/162/03毒株 PB2、PB1基因与A/Hong Kong/156/97(H5N1)相似性分别达到96.9%与96.7%,这说明此3个分离株内部基因与H5N1-like毒株可能有密切的遗传进化关系。遗传进化树显示,3株H9N2亚型AIV分离株PB2、PB1、PA 及 M 基因遗传演化与 H5N1、H1N1、H3N8、H4N2、H6N1、H6N2、H1N2、H3N6、H5N3、H7N1、H11N9等亚型的病毒有一定联系,NP基因与H5N1亚型,NA基因与H6N2亚型的病毒也具有一定联系,进一步表明H9N2亚型AIV遗传进化的复杂性与多样性,这与在中国其他地区流行的H9N2亚型 AIV 情况相一致[2,12,16-17]。
HA基因226位点(H3上位置)为区分人流感病毒与禽流感病毒的受体位点,研究表明,中国分离的H9N2亚型AIV 226L呈上升趋势,在20世纪90年代所占比例为34.3%,在21世纪初所占比例达到76%[18-19]。Zhang等的调查结果也显示在1998年-2003年中国流行的H9N2亚型AIV主要为226Q,在2005年-2006年[23]主要为226L。本试验的 3 个 分 离 株 中,CK/GD/162/03 为 226Q,Francolin/GD/298/05与 CK/GD/A8/10为226L,符合中国流行的H9N2亚型AIV特点,这提示H9N2亚型AIV有可能适应于宿主人并且可能存在在人与人之间传播的风险。糖基化位点的突变能够影响病毒的细胞融合能力和受体结合能力[20],分离株HA上糖基化位点分析显示,CK/GD/162/03、Ck/GD/A8/10分离株在218~220位氨基酸失去一个潜在糖基化位点,这种糖基化位点的缺失是否影响病毒的致病力,需要进一步试验验证。
PA、PB1、PB2基因为流感病毒内部基因,对其致病力有重要影响作用。研究发现,PB1蛋白与PA蛋白结合位点(1~25位氨基酸残基)处的第13位氨基酸为Pro,与PB2蛋白结合位点(600~757位氨基酸残基)处的第678位氨基酸为Asn时,能够促进聚合酶亚单位与NP蛋白在新宿主环境中的相互作用,通过对聚合酶活性的调控来影响流感病毒对哺乳类动物的致病性[21]。研究亦发现,在鸭上,PA基因在S224P与N383D发生突变时能够增加其致病力[22]。在PB2基因,通过反向遗传技术等已被证明K627E或D701Q或S714I(G)突变都能增强毒株对小鼠的致病性[23-24],本研究毒株在这些位点没有发生突变,这表明本试验分离株可能对鸭及小鼠致病性不强。
PB1-F2蛋白能够增强 A型流感的致病性[25-26],本研究中的3株 H9N2亚型 AIV CK/GD/162/03、Francolin/GD/298/05 及 CK/GD/A8/10所推导的PB1-F2氨基酸序列长度各不相同,分别为57、79与11位氨基酸,而编码11位氨基酸的PB1-F2多见于典型猪流感HIN1,2009年流行的HIN1也都为11位氨基酸,在H9N2 AIV中不多见[27]。在 A/Vietnam/1203/04(H5N1)毒 株 上N66S突变能增强其对小鼠的致病性[28],Francolin/GD/298/05编码的79位氨基酸PB1-F2上没有N66S突变。3株H9N2亚型AIV分离株M2基因在26L、27V、30A、31S、34G、37H 或41W 都没有发生任何氨基酸的突变,这表明所分离毒株对金刚烷胺仍然敏感[29]。
综上所述,本研究中的3株不同年代H9N2亚型AIV分离株为重组毒株,具有来源多样性。因此,在我国,特别是华南地区应更加重视对H9N2亚型AIV的监控及分子流行病学调查,防止该亚型病毒在选择压力下形成新的危害畜禽及公共卫生安全的病毒。
参考文献:
[1]Guo Y J,Krauss S,Senne D A,et al.Characterization of the pathogenicity of members of the newly established H9N2 influenza virus lineages in Asia[J].Virology,2000,267(2):279-288.
[2]Sun Y,Pu J,Jiang Z,et al.Genotypic evolution and antigenic drift of H9N2 influenza viruses in China from 1994 to 2008[J].Vet Microbiol,2010,146(3-4):215-225.
[3]Xu K M,Smith G J,Bahl J,et al.The genesis and evolution of H9N2 influenza viruses in poultry from southern China,2000 to 2005[J].J Virol,2007,81(19):10389-10401.
[4]Xu X J,Xu G Y,Zhou H B,et al.Evolutionary characterization of influenza virus A/duck/Hubei/W1/2004(H9N2)isolated from central China[J].Virus Genes,2008,36(1):79-83.
[5]Liu J H,Okazaki K,Shi W M,et al.Phylogenetic analysis of hemagglutinin and neuraminidase genes of H9N2 viruses isolated from migratory ducks[J].Virus Genes,2003,27(3):291-296.
[6]Yu H,Zhou Y J,Li G X,et al.Genetic diversity of H9N2 influenza viruses from pigs in China:a potential threat to human health?[J].Vet Microbiol,2011,149(1-2):254-261.
[7]Lin Y P,Shaw M,Gregory V,et al.Avian-to-human transmission of H9N2 subtype influenza A viruses:relationship between H9N2 and H5N1 human isolates[J].Proc Natl Acad Sci U S A,2000,97(17):9654-9658.
[8]Peiris M,Yuen K Y,Leung C W,et al.Human infection with influenza H9N2[J].Lancet,1999,354(9182):916-917.
[9]Guan Y,Shortridge K F,Krauss S,et al.H9N2 influenza viruses possessing H5N1-like internal genomes continue to circulate in poultry in southeastern China[J].J Virol,2000,74(20):9372-9380.
[10]齐 岩,袁润余,张贺楠,等.H9N2亚型禽流感病毒广东分离株的全基因克隆及序列分析[J].病毒学报,2010(3):176-182.
[11]Xu K M,Li K S,Smith G J,et al.Evolution and molecular epidemiology of H9N2 influenza A viruses from quail in southern China,2000 to 2005[J].J Virol,2007,81(6):2635-2645.
[12]Huang Y,Hu B,Wen X,et al.Diversified reassortant H9N2 Avian influenza viruses in chicken flocks in northern and eastern China[J].Virus Res,2010,151(1):26-32.
[13]Li J,Ishaq M,Prudence M,et al.Single mutation at the amino acid position 627 of PB2 that leads to increased virulence of an H5N1 avian influenza virus during adaptation in mice can be compensated by multiple mutations at other sites of PB2[J].Virus Res,2009,144(1-2):123-129.
[14]Dong G,Luo J,Zhang H,et al.Phylogenetic diversity and genotypical complexity of H9N2 influenza A viruses revealed by genomic sequence analysis[J].PLoS One,2011,6(2):e17212.
[15]Sun Y,Qin K,Wang J,et al.High genetic compatibility and increased pathogenicity of reassortants derived from avian H9N2 and pandemic H1N1/2009 influenza viruses[J].Proc Natl Acad Sci U S A,2011,108(10):4164-4169.
[16]Bi Y,Lu L,Li J,et al.Novel genetic reassortants in H9N2 influenza A viruses and their diverse pathogenicity to mice[J].Virol J,2011,8(1):505.
[17]Ji K,Jiang W M,Liu S,et al.Characterization of the hemagglutinin gene of subtype H9 avian influenza viruses isolated in 2007-2009 in China[J].J Virol Methods,2010,163(2):186-189.
[18]Lin Y P,Shaw M,Gregory V,et al.Avian-to-human transmission of H9N2 subtype influenza A viruses:relationship between H9N2 and H5N1 human isolates[J].Proc Natl Acad Sci U S A,2000,97(17):9654-9658.
[19]Zhang Y,Yin Y,Bi Y,et al.Molecular and antigenic characterization of H9N2 avian influenza virus isolates from chicken flocks between 1998 and 2007 in China[J].Vet Microbiol,2011.
[20]Kaverin N V,Rudneva I A,Ilyushina N A,et al.Structural differences among hemagglutinins of influenza A virus sub-types are reflected in their antigenic architecture:analysis of H9 escape mutants[J].J Virol,2004,78(1):240-249.
[21]Ohtsu Y,Honda Y,Sakata Y,et al.Fine mapping of the subunit binding sites of influenza virus RNA polymerase[J].Microbiol Immunol,2002,46(3):167-175.
[22]Song J,Feng H,Xu J,et al.The PA protein directly contributes to the virulence of H5N1 avian influenza viruses in domestic ducks[J].J Virol,2011,85(5):2180-2188.
[23]Mok C K,Yen H L,Yu M Y,et al.Amino Acid Residues 253 and 591 of the PB2 Protein of Avian Influenza Virus A H9N2 Contribute to Mammalian Pathogenesis[J].J Virol,2011,85(18):9641-9645.
[24]Li Z,Chen H,Jiao P,et al.Molecular basis of replication of duck H5N1 influenza viruses in a mammalian mouse model[J].J Virol,2005,79(18):12058-12064.
[25]Gocnikova H,Russ G.Influenza a virus PB1-F2 protein[J].Acta Virol,2007,51(2):101-108.
[26]Chen W,Calvo P A,Malide D,et al.A novel influenza A virus mitochondrial protein that induces cell death[J].Nat Med,2001,7(12):1306-1312.
[27]Krumbholz A,Philipps A,Oehring H,et al.Current knowledge on PB1-F2 of influenza A viruses[J].Med Microbiol Immunol,2011,200(2):69-75.
[28]Conenello G M,Zamarin D,Perrone L A,et al.A single mutation in the PB1-F2 of H5N1(HK/97)and 1918 influenza A viruses contributes to increased virulence[J].PLoS Pathog,2007,3(10):1414-1421.
[29]Suzuki H,Saito R,Masuda H,et al.Emergence of amantadine-resistant influenza A viruses:epidemiological study[J].J Infect Chemother,2003,9(3):195-200.
