大豆SBP基因家族的序列特征、表达及进化分析
2012-09-20朱红霞胡利宗邓小莉
朱红霞,胡利宗,邓小莉*,蔺 芳
(1.新乡学院生命科学与技术系,河南 新乡 453003;2.中国科学院遗传发育所,北京 100101;3.中国科学院研究生院,北京 100039)
转录因子是参与转录调控的一类蛋白因子,通过与启动子区域的顺式元件相互作用,能够激活或者抑制多个下游基因表达[1]。SBP基因家族是植物所特有的重要转录因子,属于一类锌指蛋白,由多个成员组成,主要参与植物生长、发育以及多种生理生化过程[2]。自首次在金鱼草(Antirrhinum majus)中发现SBP基因[3]以来,在拟南芥[4]、水稻[5]、葡萄[6]和白桦树[7]等植物物种中均发现该基因家族成员。就模式植物而言,拟南芥SPL3基因在花和叶片中高度表达[8];拟南芥SPL8很可能参与花粉发育的调控[9];拟南芥SPL3、SPL4和SPL5中具有microRNA156的调控位点[10-11]。此外,玉米SBP转录因子Liguleless1(LG1)能够影响舌叶和叶耳的发育,具体表现为其缺失突变体不能形成舌叶和叶耳[12]。白桦BpSPL1基因通过特异结合BpMADS5启动子参与花发育过程[7]。
随着模式植物和一些重要农作物基因组数据的快速积累,生物信息学方法已经成为剖析基因家族序列特征和进化关系的有效手段,例如大白菜乙烯受体和大豆SBP蛋白家族的研究[13-14]。尽管朱命喜等已对大豆SBP基因家族的成员数目、启动子及保守结构域的三维结构等进行预测分析,但大豆SBP基因家族的保守基序、功能结构域以及表达情况仍未见报道。本文挖掘到大豆SBP基因家族的49个成员,并分析各成员基因结构、染色体定位、蛋白保守序列、亚细胞定位、表达情况及亲缘进化关系。为大豆SBP基因结构和功能分析提供参考信息,并为阐明SBP基因家族在大豆生长发育中的调控作用奠定理论基础。
1 材料与方法
1.1 SBP基因序列检索及分析
为收集拟南芥、水稻和大豆3个物种基因组中所有的SBP基因,利用SBP结构域的一致性序列,通过BLAST对拟南芥基因组序列数据库TAIR(http://www.arabidopsis.org/),水稻基因组序列数据库TIGR(http://rice.plantbiology.msu.edu/)和JGI大豆基因组序列数据库(http://www.phytozome.net/search.php?show=blast&method=Org_Gmax)进行搜索。利用Pfam工具对候选蛋白质序列进行检索[15],若存在SBP结构域,则将其作为该蛋白质家族成员。针对鉴定得到的SBP基因,按照下列标准进行分类:如果SBP基因位于不同基因座位时,该基因被认为是1个成员;同一基因座位的多个剪接体被认为是1个成员,选择最长剪接体为代表。基于已知大豆SBP基因的DNA序列,利用BLAST在线工具和Phytozome提供的基因相关信息确定其染色体定位(http://www.phytozome.net/cgi-bin/gbrowse/soybean/)。此外,为阐明SBP基因家族扩增模式,本研究检索PGDD数据库(http://chibba.agtec.uga.edu/duplication/)中所有SBP同源基因对。
1.2 蛋白保守基序与亚细胞定位
利用MEME工具对大豆SBP蛋白的保守基序进行分析[16]。参数设置如下:同一基序在一条序列中出现的次数为0或者1,基序长度范围10~300个氨基酸残基,基序最大发现数目5个,其他参数为默认值。利用PROlocalizer在线服务器对大豆SBP蛋白的细胞内定位进行预测[17]。
1.3 基因组织表达谱分析
表达序列标签(Expressed sequence tag,EST)为在mRNA水平上研究目的基因表达提供有效工具。通过对不同数据库中目的基因对应的EST或cDNA出现频率可了解其组织特异性表达情况[18]。针对有对应UniGene的SBP基因,对其不同组织来源的EST数目进行数字化表示,具体情况如下:EST数目小于25条的记为1;EST数目在25~50 范围内的记为 2;同理,50~100、100~200、200~500、500~1 000、大于1 000分别记为3、4、5、6、7、8。最后,对SBP基因EST数字化处理后的矩阵进行聚类分析,以此来表示其基因组织表达谱。
1.4 系统进化分析
由于拟南芥、水稻和大豆等物种SBP蛋白的氨基酸序列具有高度保守的功能结构域,因此,可以进行多序列的比对和构建进化树,用于研究它们的差异以及系统进化关系。SBP蛋白序列多重比对由Clustal X软件完成,参数为默认值[19]。采用邻接法(Neighbor-Joining Method)构建系统发生树[20],其输出借助于MEGA软件完成[21]。
2 结果与分析
2.1 大豆SBP基因的鉴定与分析
结合BLAST和HMMER搜索方法,加上Pfam工具鉴定,最终在大豆基因组中鉴定出49个SBP基因,分别命名为GmSBP1~49(见表1)。从表1可以看出,大豆SBP基因家族可分为两类:一类为有内含子的SBP基因,例如GmSBP1~3、GmSBP5~21、GmSBP23、GmSBP26~38、GmSBP40~42和GmSBP44~49等,另外一类为没有内含子,包括GmSBP21和GmSBP42。外显子数目分析显示:该基因家族外显子数目具有较大变化,即由1~14个外显子组成。其中外显子最少的基因为GmSBP21和GmSBP42,仅有1个外显子;最多的是GmSBP43,为14个外显子;大多数基因含有2~4个外显子。
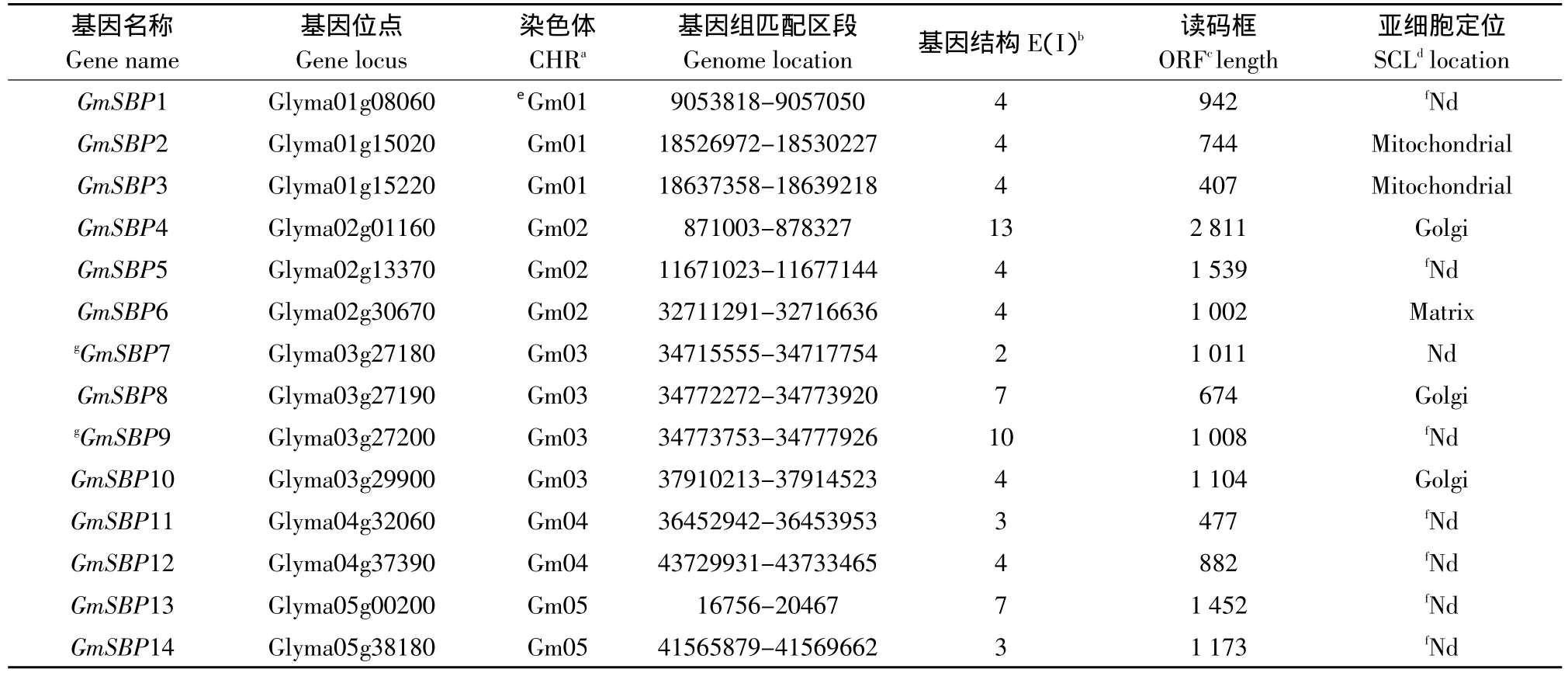
表1 大豆SBP基因家族信息Table1 Information of SBP-domain gene family in soybean(Glycine max)
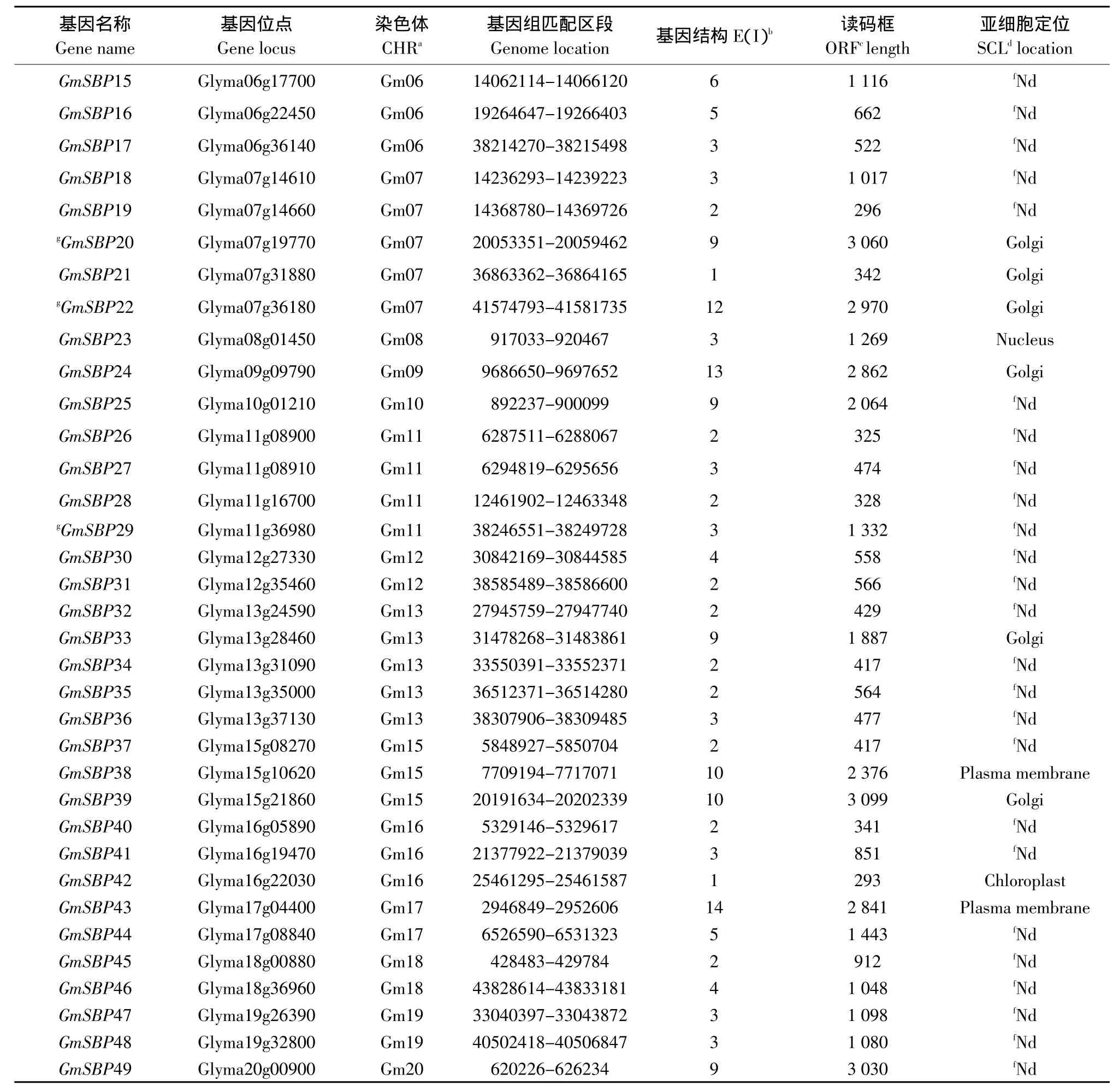
续表
表1显示大豆SBP基因家族成员在染色体上的位置和分布情况。从表1可以看出,除第14条染色体以外,SBP基因锚定于所有染色体上,几乎呈均匀分布情况。就基因数目而言,第7和13号染色体上SBP数目最多,为5个成员;第8、9、10和20号染色体上SBP基因数目最少,为1个成员;其余染色体上有SBP基因2~4个成员。此外,第13号染色体上的5个SBP基因分布极不均匀,主要集中分布在27.9~38.3 Mb之间。共线性分析显示,在SBP基因家族中,有16对基因具有较高的微共线性(见表2)。这充分说明大豆在进化过程中经历多次多倍化过程,此外,多倍化产生的SBP基因已经被保留下来,并以同源基因对形式存在。
2.2 蛋白序列特征与亚细胞定位
保守基序分析显示:大豆SBP蛋白具有5个保守基序,这5个保守基序通过不同组合方式最终形成6种组织模式,不同组织模式所占比例不同(见图1A和表3)。从图1B可以看出,基序组织模式4占比例最大,约为55%,该模式包含基序1、2和5,线性排列顺序为Motif5-Motif1-Motif2;此外,其他5种基序组织模式,有3种是在模式4基础上丢失特定的保守基序形成,而有两种是在模式4基础上获得特定的保守基序形成。结构域分析结果表明:绝大多数SBP蛋白包括完整的功能结构域,但也有的成员具有不完整的SBP结构域,例如GmSBP3、GmSBP27和GmSBP42等(见图2和表3)。蛋白保守基序与功能结构域的比较分析显示,保守基序1、2、5和功能结构域相互重叠。基序模式1、2和3不具有完整的SBP保守结构域,这意味着它们很可能丧失功能结构域活性,最终功能发生变化。同理可知,获得新基序的组织模式5和6很可能增加该家族所不具有的新功能。此外,利用 PROlocalizer(http://bioinf.uta.fi/PROlocalizer/)在线工具对大豆SBP蛋白的细胞内定位进行预测。亚细胞定位结果显示:GmSBP38和GmSBP43都定位于质膜;GmSBP23、GmSBP43和GmSBP6分别被定位于核、叶绿体与基质中;GmSBP2和GmSBP3定位于线粒体中;GmSBP4、GmSBP8、GmSBP10和GmSBP20等9个蛋白定位于高尔基体(见表1)。尽管大约67.3%的SBP蛋白不能确定其亚细胞的定位情况,但不难看出,SBP蛋白定位于多个细胞器,这就意味着其功能具有多样性。

表2 大豆SBP同源基因对Table2 Homologous pairs found in the SBP gene family of soybean
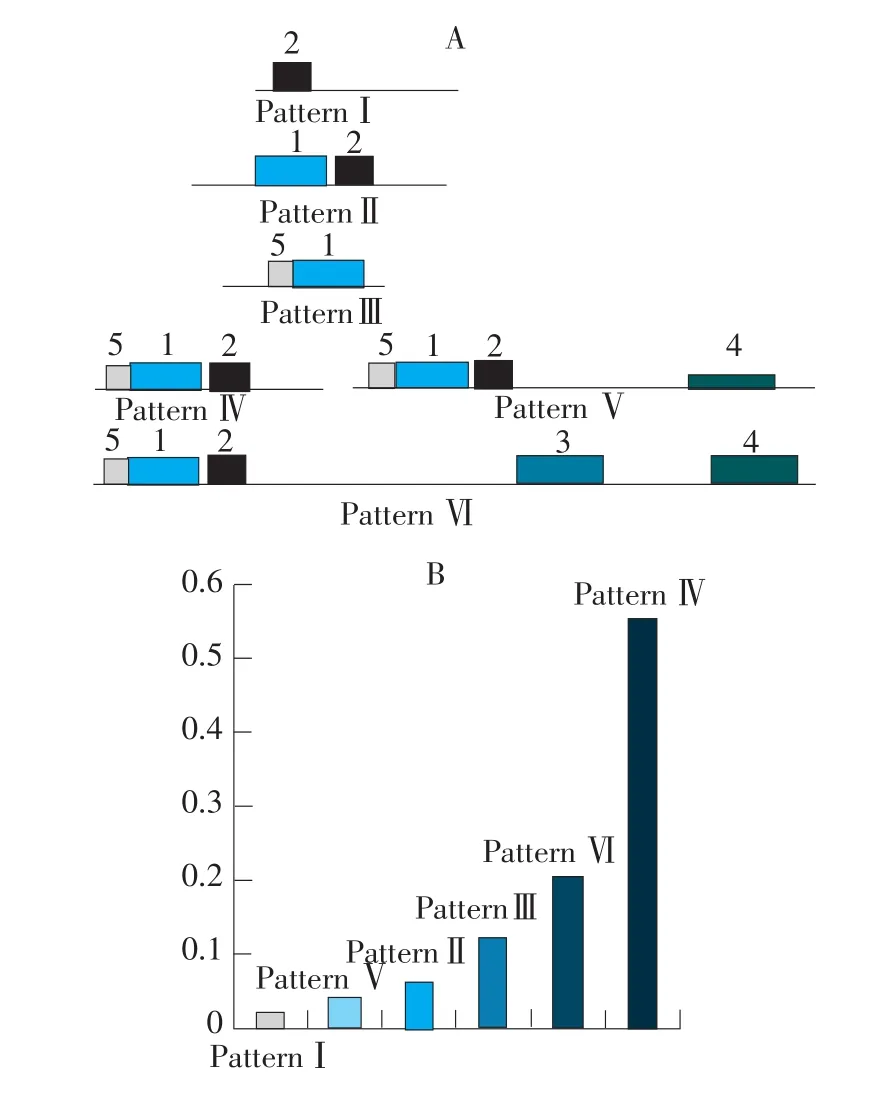
图1 大豆SBP蛋白保守基序的组织结构模式(A)和比例(B)Fig.1 Organization pattern(A)and ratio(B)for conserved motifs and phylogenic tree of SBP-domain gene family in soybean
2.3 大豆SBP基因组织表达谱
基于EST数据及统计数目,可以推测基因组织特异性表达。在EST数据库中GmSBP2、GmSBP-11、GmSBP16、GmSBP26、GmSBP27 和 GmSBP28没有相应的EST序列发现,说明这些基因很可能没有转录活性,以假基因形式存在于大豆基因组中;GmSBP24、GmSBP39,GmSBP32、GmSBP34和GmSBP37虽然能够找到相应的EST序列,但是并不能确定这些EST序列来源于哪种类型的组织,故这些EST数据只能说明它们有转录活性。此外,对剩余的大豆SBP基因组织电子表达谱进行预测,结果见图3。由图3可看出,GmSBP46是一种组成型表达基因,在子叶、上胚轴、花、下胚轴、叶子、分生组织、豆荚、根、种皮、体细胞胚、茎和芽中均表达,并且表达量较高;GmSBP41在叶片中特异表达;GmSBP1、GmSBP3、GmSBP5和GmSBP15等在分生组织中特异性表达;GmSBP-13、GmSBP21和GmSBP44在根中特异表达;GmSBP17、GmSBP30和GmSBP31等基因在果荚中特异表达;此外,多数SBP基因都可以在多个组织中表达,表现出多样化的组织表达谱(见图3)。

表3 大豆SBP蛋白的保守基序Table3 Conserved motif found in the SBP protein of soybean

图2 大豆SBP蛋白的功能结构域Fig.2 Functional domain of the SBP proteins in soybean(Glycine max)

图3 大豆SBP基因家族成员的表达谱Fig.3 Expression pattern of members of SBP-domain gene family in soybean
2.4 SBP基因的进化分析
根据方法中提供的步骤,分别从拟南芥和水稻基因组中分离到18和25个SBP基因,与以往报道的相一致[22-23]。为揭示SBP基因家族成员间的亲缘关系,18个拟南芥SBP蛋白、25个水稻SBP蛋白与49个大豆SBP蛋白用于该蛋白的系统发生分析。以支持率15%为阀值,3个物种的SBP蛋白被划分为8个类群即A、B、C、D、E、F、G和H类群。从进化树可以看出:水稻的一个SBP蛋白属于孤儿基因,单独形成一个分支;大豆的4个SBP基因落入类群外,以基因对形式单独形成进化支;F类群成员数目少,仅包括1个水稻SBP蛋白和2个大豆SBP蛋白;其他类群由拟南芥、水稻和大豆SBP蛋白共同组成(见图4)。进化树末端同一分支的两个外部结点,很可能就是亲缘关系比较近的同源基因对。进化树末端分析显示:5对拟南芥/大豆SBP蛋白位于系统发生树的同一分支,4对水稻/大豆SBP蛋白位于系统发生树的同一分支,3对拟南芥/水稻SBP蛋白处于进化树的同一分支,这说明该基因家族的祖先基因起源于3个物种分化之前。此外,大豆中有15对SBP基因位于系统发生树的同一分支,而拟南芥和水稻分别有3和4对SBP基因处于同一进化分支中,这充分说明,大豆物种形成后SBP基因发生了多次重复事件。
3 讨论
利用BLAST和HMMER工具,在全基因组水平对SBP基因进行挖掘。利用Pfam和MEME工具进行过滤,最终在拟南芥、水稻和大豆中分别得到18、25和49个SBP基因。前人研究揭示拟南芥、水稻和大豆中分别有16、20、44个SBP基因[13,22-23]。两者比较显示,本文鉴定的SBP基因较多。与拟南芥和水稻相比,大豆基因组中有更多SBP基因,说明大豆SBP基因家族经历更为复杂的扩增、丢失以及进化过程。朱命喜等从PlnTFDB,PlantTFDB,RiceTFDB直接下载并筛选44个大豆SBP基因,详细分析该家族成员启动子序列特征,并认为该转录因子家族参与生长发育、逆境胁迫响应、激素应答、抗霉菌应答、光合作用等调控过程。此外,对个别SBP蛋白还进行三维结构模建[13]。但大豆SBP基因的保守基序模式、共线性、表达谱和亚细胞定位等问题仍然没有被阐明,本研究填补大豆SBP基因这些方面的空白。大豆是典型的古四倍体植物,其基因组在进化过程中经过加倍的过程,所以很多基因在大豆基因组中都是以多拷贝形式存在。Schmutz等研究结果显示,在59和13MYA前,大豆分别发生2次全基因组重复事件,这也支持基因是以多拷贝形式存在的观点[24]。本研究发现多拷贝的SBP基因散落分布于不同染色体上。微共线性角度分析表明,在大豆基因组中存在16对SBP旁系同源基因,这为大豆全基因组加倍事件提供有利证据(见表2)。
从系统进化角度看,SBP基因在大多数植物中都是由多成员组成。例如在拟南芥、水稻和大豆基因组中分别鉴定出3、4和15对SBP同源基因对,它们属于横向同源基因,处于同一进化分支中,形成于物种发生后。与拟南芥和水稻相比,大豆具有更多SBP同源基因对,这充分说明,大豆物种形成后SBP基因发生更多重复事件。一般而言,具有完整SBP功能结构域的SBP基因往往能够找到EST序列,也就是说这些SBP基因具有转录活性。相反,GmSBP26和GmSBP27不包括典型SBP功能结构域,同时也没有EST数据支持其转录活性,因此,推测其为假基因。总之,大豆SBP基因家族的进化过程很可能受不等价交换、局部片段重复多倍化的全基因组重复以及转座子等多种因素影响,导致其进化错综复杂。因此,在生物信息预测分析基础上,有必要进行大量验证,最终弄清其起源和进化关系。本研究可为下一步研究大豆SBP转录因子的生物学功能提供参考。
[1]FRiechmann J L,Heard J,Martin G,et al.Arabidopsis transcription factors:Genome-wide comparative analysis among eukaryotes[J].Science,2000,290(5499):2105-2110.
[2]Guo A Y,Zhu Q H,Gu X,et al.Genome-wide identification and evolutionary analysis of the plant specific SBP-box transcription factor family[J].Gene,2008,418(1-2):1-8.
[3]Klein J,Saedler H,Huijser P.A new family of DNA binding proteins includes putative transcriptional regulators of the Antirrhinum majus floral meristem identity gene SQUAMOSA[J].Mol Gen Genet,1996,250(1):7-16.
[4]Unte U S,Sorensen A M,Pesaresi P,et al.SPL8,an SBP-box gene that affects pollen sac development in Arabidopsis[J].Plant Cell,2003,15(4):1009-1019.
[5]Shao C X,Takeda Y,Hatano S,et al.Rice genes encoding the SBP domain protein,which is a new type of transcription factor controlling plant development[J].Rice Genet Newsl,1999,16:114.
[6]曹雪,上官凌飞,于华平,等.葡萄SBP基因家族生物信息学分析[J].基因组学与应用生物学,2010,29(1):791-798.
[7]Lannenpaa M,Janonen I,Holtta-Vuori M,et al.A new SBP-box gene BpSPL1 in silver birch(Betula pendula)[J].Physiol Plant,2004,120(3):491-500.
[8]Cardon G H,Hohmann S,Nettesheim K,et al.Functional analysis of the Arabidopsis thaliana SBP-box gene SPL3:A novel gene involved in the floral transition[J].Plant,1997,12(2):367-377.
[9]Schmid M,Uhlenhaut N H,Godard F,et al.Dissection of floral induction pathways using global expression analysis[J].Development,2003,130(24):6001-6012.
[10]Wu G,Poethig R S.Temporal regulation of shoot development in Arabidopsis thaliana by miR156 and its target SPL3[J].Development,2006,133(18):3539-3547.
[11]Gandikota M,Birkenbihl R P,Hthmann S,et al.The miRNA 156/157 recognition element in the 3'UTR of the Arabidopsis SBP box gene SPL3 prevents early flowering by translational inhibition in seedlings[J].The Plant Journal,2007,49(4):683-693.
[12]Moreno M A,Harper L C,Krueger R W,et al.Liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis[J].Genes&Development,1997,11(5):616-628.
[13]朱命喜,刘洋,吴琼,等.大豆SBP转录因子家族的预测分析[J].大豆科学,2011,30(2):178-183.
[14]朱红霞,胡利宗,邓小莉.大白菜乙烯受体基因家族:分子特征、微同线性与进化分析[J].生物技术通报,2011(7):88-94.
[15]Sonnhammer E L,Eddy S R,Durbin R.Pfam:A comprehensive database of protein domain families based on seed alignments[J].Proteins,1997,28(3):405-420.
[16]Bailey T L,Williams N,Misleh C,et al.MEME:Discovering and analyzing DNA and protein sequence motifs[J].Nucleic Acids Res,2006,34:369-373.
[17]Kirsti L,Mauno V.PROlocalizer:Integrated web service for protein subcellular localization prediction[J].Amino Acids,2011,40:975-980.
[18]Ohlrogge J.Benning C.Unraveling plant metabolism by EST analysis[J].Curr Opin Plant Biol,2000,3(3):224-228.
[19]Thompson J D,Gibson T J,Plewniak F.The CLUSTAL_X windows interface:Flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res,1997,25:4876-4882.
[20]Saitou N,Nei M.The neighbor-joining method:A new method for reconstructing phylogenetic trees[J].Mol Biol Evo,1987,4(4):406-25.
[21]Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular evolutionary gengtics analysis(MEGA)software version 4.0[J].Mol Biol and Evo1,2007,24(8):1596-1599.
[22]Cardon G,Hohmann S,Klein J,et al.Molecular characterisation of the Arabidopsis SBP-box genes[J].Gene,1999,237:91-104.
[23]王翊,胡宗利,杨妤欣,等.水稻SBP基因家族的生物信息学分析[J].生物信息学,2011,9(1):82-87.
[24]Schmutz J,Cannon S B,Schlueter J,et al.Genome sequence of the palaeopolyploid soybean[J].Nature,2010,463:178-183.
