亚侧耳RAPD反应体系的优化
2012-09-18张跃新闫宝松马凤胡伟
张跃新,闫宝松,马凤,胡伟
(黑龙江省林副特产研究所,黑龙江省非木质林产品研发重点实验室,黑龙江 牡丹江157011)
亚侧耳(Hohenbuehelia serotina)[1-2]商品名元蘑,又名冻蘑,是黑龙江省特有的山珍之一。亚侧耳肉质细腻,柔嫩鲜美,营养价值极高,含有人体所需的多种氨基酸、维生素和微量元素,经常食用具有加强肌体免疫,增强机体抵抗能力,益智开心,益气不饥,延年轻身等作用。
RAPD(Random Amplified Polymorphic DNA,随机扩增多态性DNA)技术,是Williams和Welsh等1990年发明的一种新型DNA分子标记技术,该技术具有简单、快速、准确、灵敏度高和多态性丰富等优点,广泛应用于遗传图谱构建、基因定位和遗传多样性的检测[3]。本文在参考一般食用菌RAPD试验的基础上[4-5],建立了亚侧耳 RAPD基本反应体系,并对基本反应体系进行了优化,为亚侧耳种质资源遗传多样性的RAPD分析奠定基础。
1 材料和方法
1.1 材料
1.1.1 供试菌株
亚侧耳3号,为黑龙江省林副特产研究所培育菌株。
1.1.2 试剂
随机引物购自上海生工,基因组DNA提取试剂盒购自天根生化科技有限公司,Taq酶及相配套的反应缓冲液购自TaKaRa公司。
1.2 亚侧耳基因组DNA提取
采用天根生化科技有限公司的“快捷型植物基因组提取试剂盒”进行DNA提取。
1.3 亚侧耳RAPD反应体系设计
1.3.1 基本反应体系及基本反应条件
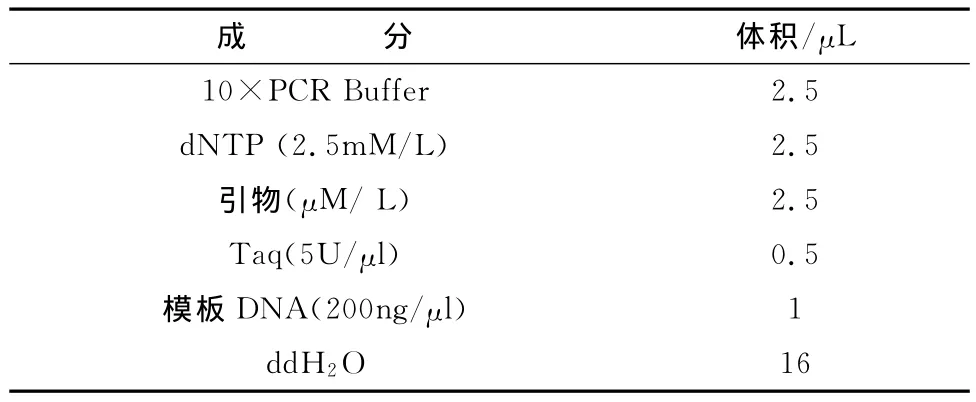
表1 基本反应体系(25μl体系)
基本反应程序
1.3.2 各反应因素梯度设计
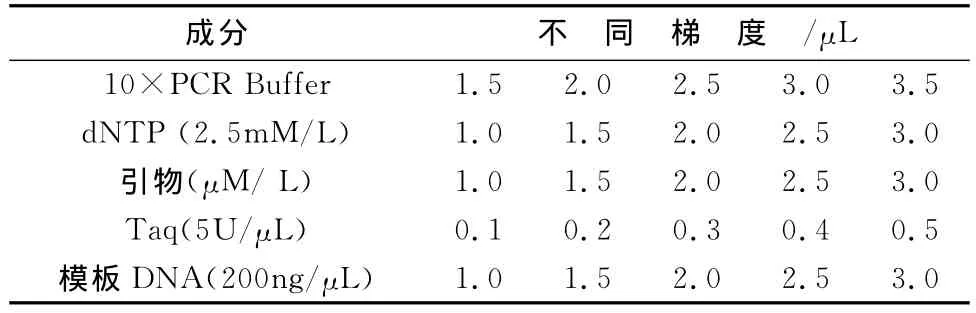
表2 各反应因素梯度设计
2 结果与分析
2.1 亚侧耳模版DNA提取结果
将提取的亚侧耳DNA进行琼脂糖凝胶电泳(图1)。DNA呈现出一条明亮的谱带且无明显的拖尾现象,说明所提取的DNA比较完整,质量较高,可用于RAPD分析。
2.2 引物浓度优化结果
经引物筛选试验,在100个随机引物中筛选出S167、S467、S475、S460四个引物,其中引物以S167扩增效果最好,因此选用S167进行RAPD反应体系构建。在引物浓度优化试验中(图2)可知,5个引物浓度梯度中,除1号外,其它梯度扩增效果多态性均非常丰富,且条带清晰。因此,出于成本考虑,选择2号为最佳引物浓度,即25μL反应体系中添加1.5μL引物。
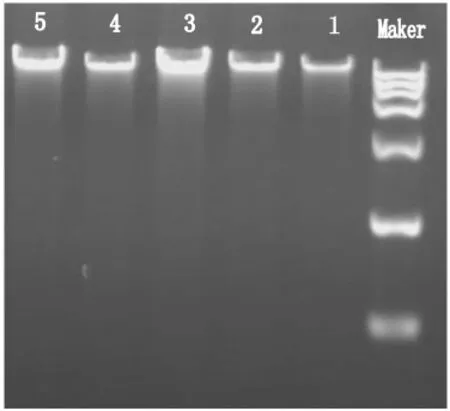
图1 亚侧耳DNA电泳结果
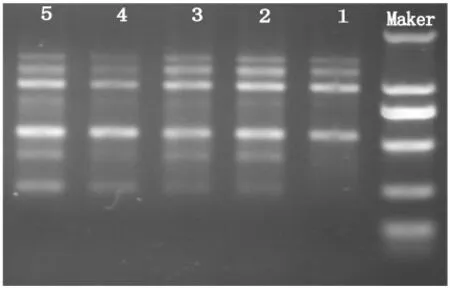
图2 引物浓度优化试验
2.3 模版DNA浓度优化结果
模版DNA浓度优化结果如图3,其中1号、2号扩增条带不清晰,扩增效果不好;3号、4号、5号扩增条带清晰,且呈多态性,出于成本考虑,选择3号浓度,即25μL反应体系中添加2.0μL模版DNA。
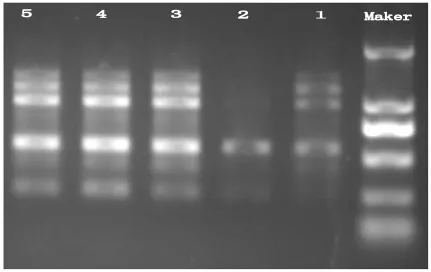
图3 模版DNA浓度优化试验
2.4 Taq酶浓度优化结果
Taq酶浓度优化结果如图4,各浓度条带均比较清晰,且多态性丰富,其中以2号、3号浓度条件下最为清晰,出于成本考虑,选择2号浓度,即25μL反应体系中添加0.2μL Taq酶。
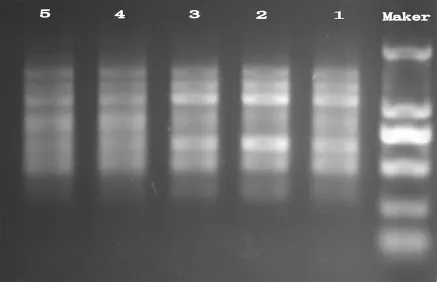
图4 Taq酶浓度优化试验
2.5 10×PCR Buffer浓度优化结果
10×PCR Buffer浓度优化结果如图5,以2号浓度最为清晰,因此25μL反应体系中添加2.0μL10×PCR Buffer溶液。
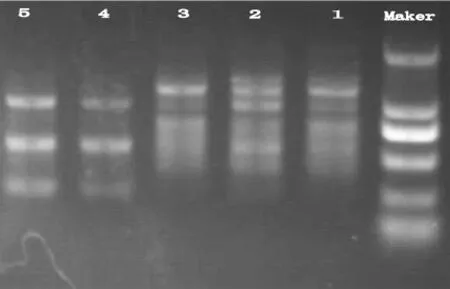
图5 10×PCR Buffer浓度优化试验
2.6 dNTP浓度优化结果
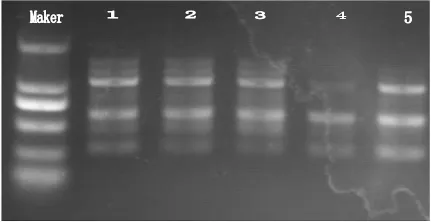
图6 dNTP浓度优化试验
dNTP浓度优化如图6,其中1号、2号、3号浓度条件下扩增效果均比较清晰,且条带呈多态性,其中1号浓度条件下扩增效果最为清晰,因此25μL反应体系中添加3.0μL dNTP溶液。
3 结论
在RAPD反应中,影响PCR反应的因素很多,其中包括PCR缓冲液、dNTP的浓度、循环参数(退火温度、变温时间、PCR仪),Taq DNA聚合酶的来源及型号、模板DNA的质量和含量、引物纯度及浓度、DNA污染的共抽提和扩增干扰等。本文对RAPD反应体系中的10×PCR Buffer浓度 、dNTP浓度、模板DNA浓度、引物浓度、Taq酶浓度这5个主要影响因素进行了优化,最终确立了亚侧耳RAPD-PCR反应体系和反应程序。反应体系(25μL):10×PCR Buffer 2.0μL、2.5mM/L dNTPs3.0μL、5U/μL Taq酶0.20μL、5μM/L引物1.5μL、200ng/μL模板 DNA 2.0μL、去离子水16.30μL。PCR 反应程序为:94℃预变性3min,94℃变性30s,37℃退火30s,72℃延伸2min,40个循环,72℃延伸10min,4℃forever。
[1] 戴芳澜.中国真菌总汇[M].科学出版社,1979.
[2] Liu Y,Bau Tolgor.A new species of Hohenbuehelia from China[J].Mycotaxon.2009.
[3] 刘明,王继华.DNA分子标记技术.东北林业大学学报,2003,31,(6):65-67.
[4] 李三署,林新坚.姬松茸和双胞蘑菇不同菌株的RAPD扩增研究[J].食用菌学报,2002,9(3):1-4.
[5] 詹才新,朱兰宝.RAPD技术在金针菇杂交育种中的应用[J].食用菌学报,1995,2(1):7-11.
