一种基于DNA自组装模型求解最大团问题的算法*
2012-08-14周炎涛李肯立黎福海
周炎涛,李肯立,罗 兴,黎福海,朱 青
(1.湖南大学 电气与信息工程学院,湖南 长沙 410082; 2.湖南大学 信息科学与工程学院,湖南 长沙 410082)
最大团问题(Maximum clique problem,MCP)又称为最大独立集问题,是图论中的一个经典组合优化问题,也是一类NP完全问题[1].求解MCP算法主要有二类:确定性算法和启发式算法,前者有回溯法、分支限界法等 .随着问题规模的增大(顶点增多和边密度变大),求解问题的时间复杂度越来越高,确定性算法显得无能为力,不能有效地解决这些NP完全问题 .后者有蚁群算法、顺序贪婪算法、DLS-MC算法和智能搜索算法等,大部分确定性算法所不能解决的图,用启发式算法都能得到有效解决,但启发式算法不一定能找到最优解,有时只能找到近似值[2].近年来,常借鉴算法之间优势互补策略,形成新的混合启发式算法来求解最大团问题[3].
DNA计算是应用分子生物技术进行计算的新方法,具有高度并行性、大容量、低能耗等特点,为解决NP完全问题开辟了一条新途径[4].自Adleman首次运用DNA计算来解决NP问题以来[5],研究者对最大团问题的分子求解方法进行了很多有益的尝试,如基于粘贴模型的最大团问题算法[6]、质粒DNA算法[7]、闭环求解最大团问题算法[8]等,但这些方法均存在实验操作步骤过多、活体内不易操作以及环化效率不高等弊端.此外,Brun采用自组装DNA计算给出了路径寻找问题[9],以及可满足性问题的自组装计算模型的解决方案[10].文献[11]在分布式系统中建立自组装模型解决自由等待一致性问题 .文献[12]将AuNP自组装聚合色变与DNA计算相结合,构建了系列基本逻辑计算模型,等等.
在DNA计算机研究中,所建模型的好坏直接影响着DNA计算中诸多问题,如编码的难易程度、整个生物操作或生化反应的设计、解空间的大小、计算时间多少、应用范围以及通用性的程度等.目前DNA计算模型有很多[13],其中自组装DNA计算模型是通过DNA分子间的相互作用形成特定的构型来完成计算过程 .它组合了DNA计算、Ting理论和DNA纳米技术,成为目前备受关注的模型之一[14-15].在计算过程中,它避免了其他DNA计算模型所需要的众多实验操作次数,减少了操作带来的时间消耗和误差倾向.分子自组装体系是一个高度组织、高度有序、结构化、功能化和信息化的复杂系统,对研究分子自组装特性并求解最大团问题极有价值.本文采用DNA自组装模型,设计出相应的DNA tiles分子,利用基因工程的生物操作,提出一种基于DNA自组装模型求解最大团问题的算法.该算法实验操作复杂度为常数级,既大大减少了因为人为实验操作所带来的误差,又保证了实验结果的准确性.
1 DNA自组装模型
DNA的自组装可形成一维、二维、三维结构.本文主要利用其形成的二维平面结构来解决最大团问题.1998年,根据 Wang的tiles理论,Winfree设计了双交叉分子DX[16],这个DX就充当了tiles的作用,通过自组装就能实现所需的计算.理论上有5种可能的DX结构,但通过大量实验证明,只有DAO和DAE这2种结构是稳定的[16].抽象的DAO和DAE结构如图1所示.
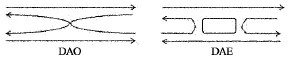
图1 DAO,DAE抽象结构Fig.1 Abstractive structures of DAO and DAE
观察图1可知,DAO结构包含4条DNA链,其中有2条链分布在2条双螺旋上,另外2条分布在一条双螺旋上 .相比之下,DAE结构包含5条DNA链,其中有3条链分布在2条双螺旋上,另外2条链分布在一条双螺旋上.由于DX可有4个粘性末端,故可利用这些粘性末端来实现所需要的计算.Winfree使用了带颜色的tiles来模拟双交叉分子的计算过程,规定只有具有相同颜色的边才能够结合形成较大的格局,其反应过程如图2所示.

图2 DX计算过程模拟Fig.2 Process simulation of DX computing
根据Wang的tiles理论模型的形式化描述以及Yuriy Brun引入DNA 的tiles自组装模型[17],结合最大团问题的特点可将基于DAE结构的自组装模型表示为S=〈T,g,τ〉,其中T为DAE块的集合,g为能量函数,τ为常数.相应的符号描述如下:
1)Σ:DAE块中所有粘性末端所表示符号的集合(即所要参与计算的符号的集合);
2)D′= {UL,DL,UR,DR}:分别表示DAE块的左上角、左下角、右上角和右下角4个位置;
3){σUL,σDL,σUR,σDR}∈Σ4:DAE块的形式化描述,代表DAE块4个位置分别所表示的符号;
4)(x,y)∈Z2:表示位置;
5)D={N,E,S,W}:表示4个方向函数,且满足:
●∀(x,y)∈Z2,N(x,y)=(x,y+1),E(x,y)=(x+1,y),S(x,y)=(x,y-1),W (x,y)=(x-1,y);
●若位置(x,y)与(x’,y’)相邻,则∃d∈D,使得d(x,y)= (x’,y’).
6)bdd(t)∈Σ,其中t∈T,d∈D’:表示t的位置d所表示的符号;
7)函数g:Σ×Σ→R,此函数定义如下:
●∀σ∈Σ,则g(null,σ)=0;
●若g(σ,σ’)=0(其中σ,σ’∈Σ),则σ≠σ’;
●∀σ≠null,有g(σ,σ)=1;
●∀σ≠σ’,有g(σ,σ’)=0.
8)函数A:Z×Z→T,此函数有如下特点:
●若A(x,y)≠empty,则(x,y)∈A;
●若只有有限个不同的(x,y)∈A,则A为一个有限集.
9)A是一个格局,若一个DAE块t能在位置(x,y)产生一个新的格局A’,需要满足以下条件:
●(x,y)∉A;
●Σd∈D’,d’∈Dg(bdd(t),bdd-1(A(d’(x,y))))≥τ,其中d与d-1为相对的方向,如:UR与DL相对.
●∀(u,v)∈Z2,(u,v)≠ (x,y),则A’(u,v)=A(u,v);
●A(x,y)=t.
2 算法设计步骤
2.1 算法设计思路
基于DNA自组装模型的算法设计利用的是“堆积木”的思想,其具体算法设计思路如下:
1)设计初始分子,使之组装成所有可能结果;
2)分析问题,找出问题正确结果所需满足的条件;
3)根据2)条件限制,设计规则分子,判定1)中结果正确性;
4)搜索正确结果,得到所需答案.
本文按照上述步骤设计相应的DAE块,然后通过基因工程的相应生物操作,利用凝胶电泳和荧光标记技术提取得到最终结果.相应生物操作描述[18]如下:
1)合并(merge):给定试管P1和P2,操作∪(P1,P2)=P1∪P2表示将试管P1和P2合并到一个试管中而不改变P1和P2中的任何链.
2)提取(extract):给定试管P1和P2,操作P2=extract(P1)表示提取试管P1中具有某种特征的DNA分子到试管P2.
3)退火(anneal):给定试管P,操作Anneal(P)将试管P中的所有DNA单链变成双链.
4)荧光标记(fluorescence labeling):给定试管P,该操作将具有某种特征的DNA分子作标记.
5)凝胶电泳(gel electrophoresis):给定试管P,该操作将试管中DNA分子按照分子大小进行分离.
2.2 设计初始分子
设G={V,E},其中V={v1,v2,… ,vn},则任何顶点子集都可能是图G的团,则初始分子设计如图3所示.其中,s和e是控制自组装反应的始端和终端,与图G无关;i,j∈{1,2,…,n},且i<j.分析可知:所有初始分子的种类为2n2-7n+8.
若顶点子集为{1,2},则退火反应之后则形成如图4所示结构.若顶点子集为{1,2,3},则退火后反应之后形成如图5所示结构.
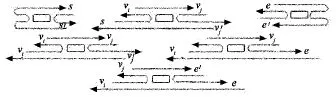
图3 初始分子Fig.3 Initial moleculars

图4 顶点子集{1,2}形成的结构Fig.4 Forming structure of vertex subset{1,2}
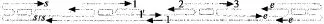
图5 顶点子集{1,2,3}形成的结构Fig.5 Forming structure of vertex subset{1,2,3}
引理1 若Σ1={s,e,v1,v2,…,vn},T1为按上述方式设计的初始分子DAE块的集合,τ=1,则形成的自组装系统S1=〈T1,g,τ〉.不失一般性,比如顶点子集为{v1,v2,… ,vk},经过退火反应后则会形成如下格局A1:
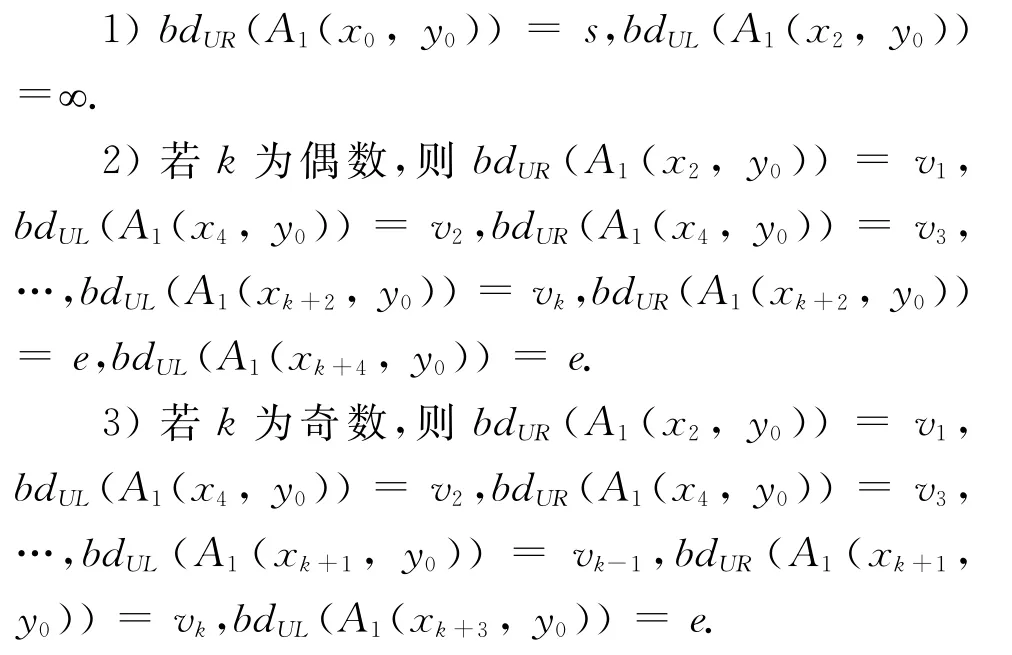
证 根据T1中的DAE块的特点可知,起始端为{null,null,s,s},所以bdUR(A1(x0,y0))=s.已知τ=1,所以只需要一边能够结合就能够产生新的格局.又已知顶点子集为{v1,v2,… ,vk},所以∃DAE块{∞,s,v1,v1},所以{null,null,s,s}与{∞,s,v1,v1}能够结合产生新的格局.则此时bdUL(A1(x2,y0))=∞,bdUR(A1(x2,y0))=v1.
同理可知:当k为偶数时,bdUL(A1(x4,y0))=v2,bdUR(A1(x4,y0))=v3,…,bdUL(A1(xk+2,y0))=vk,bdUR(A1(xk+2,y0))=e,bdUL(A1(xk+4,y0))=e.
当k为奇数时,bdUL(A1(x4,y0))=v2,bdUR(A1(x4,y0))=v3,…,bdUL(A1(xk+1,y0))=vk-1,bdUR(A1(xk+1,y0))=vk,bdUL(A1(xk+3,y0))=e.
(2)要根据运行方式来调整好继电保护装置的保护功能、保护范围以及相关的定制等等,对电网中所有有关的信息进行综合,再实时修正好保护定值。
证毕.
2.3 设计规则分子
我们把规则分子的DAE块设计成如图6所示.

图6 两顶点邻接时DAE块Fig.6 DAE block with adjoining vertexes
若输入顶点a,b在图中是邻接的,则存在上述DAE块,使其输出时交换顶点变成b,a.考虑到边界条件,则可以把规则分子设计成如图7.

图7 规则分子Fig.7 Regular moleculars
其中s为始端,e为终端,与图G无关.节点a,b邻接,且a,b,c,d∈{v1,v2,…,vn-1,vn,∞},所需DAE块种类为|E|+2n+1.
引理2 在格局A1的基础上,若T2为按上述方式设计的规则分子的集合,并且τ=2,则形成的自组装系统S2=〈T2,g,τ〉.若G对于生成的格局A1中的所有顶点都相邻,则系统S2会生成格局A2,其中bdUR(A2(xk+1,yk+1))=∞,bdUL(A2(xk+3,yk+1))=e.若G对于生成的格局A1中存在两个顶点不相邻,则系统S2不会生成上述格局A2.
证k为偶数且图G对于生成的格局A1中的所有的顶点都相邻,则形成的A1如引理1中所述.由于τ=2,所以需要两边能够结合才能产生新的格局.又因为所有的顶点都相邻,所以对于任意两个顶点vi,vj都存在 DAE 块{vj,vi,vi,vj},即每经过一轮比较∞都会向后移动,所以∞终会移动到末尾,即bdUR(A2(xk+1,yk+1))= ∞,且bdUL(A2(xk+3,yk+1))=e.同理可知:若k为奇数时,bdUR(A2(xk+1,yk+1))=∞,bdUL(A2(xk+2,yk+1))=e.故系统S2会生成格局A2.
反之,若A1中存在两个顶点不相邻.不失一般性,假设存在两个顶点vi,vj不相邻,则不会存在DAE块{vj,vi,vi,vj}.由于τ=2,当反应进行到此后,格局就不会再增高,∞也就不会移动到末尾.故不能形成格局A2.
证毕.
2.4 设计检测分子

图8 检测分子Fig.8 Detected moleculars
i,j∈{1,2,…,n-1,n},并且利用荧光标记技术在上图中最左边的DNA分子加上与其他DAE块不同颜色的荧光.则这些DAE块种类共计n2+1种.
若加入规则分子后能够形成格局A2,则加入检测分子后就能够形成一个完整的格局,否则就不能形成一个完整稳定的格局(即该格局没有粘性末端可提供给其他DAE块结合,并且该格局中拥有带有荧光标记的检测分子).
综上所述,设计初始分子所需的DAE块种类为2n2-7n+8;设计规则分子所需的DAE块种类为|E|+2n+1;设计检测分子所需的DAE块的种类为n2+1.总共需要的DAE块种类为3n2-5n+|E|+10.所以需要的DAE块的种类为Θ(n2+|E|),说明设计这些DAE块是可行的.
3 基于DNA自组装模型求解最大团问题
3.1 算法步骤
算法基于自组装模型的最大团问题DNA算法.
输入:图G={V,E},以及根据图G并按上述方式设计的tiles集T1,T2,T3.
输出:图G的最大团.

7)T’end= Extract(T),提取在6)中所观测到的分子块,将其放入试管T′end;
8)使用凝胶电泳技术将T中的分子块按照大小进行分离,通过激光共焦距显微镜观察分子块最大的DNA块;
9)Tend=Extract(T′end),提取8)中所观察的最大的DNA块,将其放入试管Tend.
引理3 有a1,a2,a3,…,an个顶点,按照以下方式进行n轮比较,则这n个顶点每两个顶点都会比较一次,即总共比较Cn2=n(n-1)/2次.

证 当n为偶数时,奇数轮比较(n/2)-1次,偶数轮比较n/2次,则总共比较的次数为(n/2)(n/2-1)+ (n/2)(n/2)=n(n-1)/2=Cn2.
同理可知:当n为奇数时,总共比较次数为n(n-1)/2=Cn2.
证毕.
定理1 若算法中Tend不为空,则Tend中所代表的团即为所求图的最大团;否则该图的任意两个顶点都不邻接.
证 若Tend不为空,则Tend中的顶点集是按照引理1中的方式进行比较.由引理1可知:Tend中的顶点集共进行了Cn2次比较.又由于两个顶点比较之后一个顶点向左移动,另外一个顶点向右移动,则这两个顶点不会重复比较,所以每两个顶点都比较过一次.说明这n个顶点两两邻接,即此顶点子集的导出子图就为此图的一个团.又由算法1的8)可知Tend中所表示顶点集都规模最大,所以Tend中所代表的团即为所求图的最大团.反之,若Tend为空,则说明图G的任意顶点子集的导出子图都不是团,即该图的任意两个顶点都不邻接.
证毕.
3.2 算法复杂性
文中2.2设计初始分子所需的DAE块种类为2n2-7n+8;2.3设计规则分子所需的DAE块种类为|E|+2n+1;2.4设计检测分子所需的DAE块的种类为n2+1.则总共需要的DAE块种类为3n2-5n+|E|+10.所以需要的DAE块的种类为Θ(n2+|E|),即算法设计tiles的种类为Θ(n2+|E|).算法1(基于自组装模型的最大团问题DNA算法)生物操作为9步,即其生物操作复杂性为Θ(1).
3.3 示 例
给定图G={V,E},其中V={1,2,3,4,5},E={(1,2),(2,3),(3,4),(4,5),(2,5),(2,4),(3,5)},如图9所示.

图9 5个顶点无向图Fig.9 Graph of 5vertexes
结合3.1的算法步骤,则求得最大团的步骤为如图10所示 .在试管T1中加入连接酶,并使用退火操作,使其初始分子充分反应,则会形成不同的顶点子集,如图10(a)形成的顶点子集为{2,3,4,5}.
将试管T1,T2中的DAE块合并到试管T,之后在试管T中加入连接酶,并使用退火操作.此步骤实际上是加入规则分子,用以判断前面的顶点子集是否为团.如图10(b)即为加入规则分子去判断顶点子集{2,3,4,5}是否为团.
最后加入检测分子,便于检测顶点子集的导出子图是否为团.经过上述步骤后,凡是能够形成如图10(c)所示的结构,则说明该导出子图即为团,即该图中存在有荧光标记的DNA分子.

图10 图G最大团计算Fig.10 Computing maximum clique of G
4 结 论
自组装技术能最底层揭示DNA计算的本质,它是一种由简单到复杂、从无序到有序、由多组分收敛到单一组分不断自我修正、自我完善的过程.本文采用DNA自组装模型,设计出相应的DNA tiles分子,利用基因工程的生物操作,提出一种基于DNA自组装模型求解最大团问题的算法 .该算法实验操作复杂度为常数级,既大大减少了因为人为实验操作所带来的误差,又保证了实验结果的准确性,对DNA计算和DNA计算机的研究来说,是一次有意义的实践.目前DNA计算模型的DNA编码已经从一维结构发展到了三维结构 .相对于其他的计算模型,从理论上已表明,使用三维DNA图结构可以明显减少解决最大团问题所需的时间和步骤,而且由于它能直观地反映图结构,用它建立计算模型可能会使一些传统的难解问题得以解决.
本文主要利用其形成的二维平面结构来解决最大团问题 .在技术层面上,将这种二维DNA分子组装成三维DNA图结构模型还存在很多需要解决的问题.目前对复杂三维DNA结构进行普通的实验操作还少有研究,且与它相关联的酶处理操作也容易发生错误.随着DNA计算研究的深入,三维DNA计算也将会有更好的发展 .此外,将分子计算与量子计算紧密地结合在一起,为未来的新型计算技术提供了发展的可能.伴随分子操控技术的发展,相信DNA计算在信息处理、密码技术、纳米智能机器和纳电子学等多个领域将会有更多的潜在应用.
[1] OUYANG Q,KAPLAN P D ,LIU S,et al.DNA solution of the maximal clique problem[J].Science,1997,278:446-449.
[2] PULLAN W,HOOS H H.Dynamic local search for the maximum clique problem[J].Journa l of Artificial Intelligence Research,2006,25:159-185.
[3] KATAYAMA K,KOHMURA A,KOHMOTO K,et al.Memetic algorithm with strategic controller for the maximum clique problem[C]∥Taiwan:TaiChung:Proceedings of the 2011ACM Symposium on Applied Computing,2011:1062-1069.
[4] SHARRMA J,CHHABRA R,CHENG A,et al.Control of self-assembly of DNA tubules through integration of gold nanoparticles[J].Science,2009,323:112-116.
[5] ADLEMAN L M.Molecular computation of solutions to combinatorial problems[J].Science,1994,266(11):1021-1024.
[6] 周康,刘朔,覃磊,等.基于粘贴模型的最大团问题算法[J].华中科技大学学报:自然科学版,2010,38(9):89-92.ZHOU Kang,LIU Shuo,QIN Lei,et al.Sticker model-based DNA algorithm of maximum clique problem[J].Huazhong Univ of Sci &Tech:Natural Science Edition,2010,38(9):89-92.(In Chinese)
[7] HEAD T,ROZENBERG G,BLADERGROEN R B,et al.Computing with DNA by operating on plasmids[J].Bio Systems,2000,57:87-93.
[8] 张成,杨静,许进,等.DNA缩短法计算模型求解最大独立集问题[J].科学通报,2009,54(24):3913-3919.ZHANG Cheng,YANG Jing,XU Jin,et al.A DNA length reducing computing model for maximum independent set problem[J].Chinese Sci Bull,2009,54(24):3913-3919.(In Chinese)
[9] BRUN Y,REISHUS D.Path finding in the tile assembly model[J].Theoretical Computer Science,2009,410(15):1461-1472.
[10] BRUN Y.Solving satisfiability in the tile assembly model with a constant-size tileset[J].Journal of Algorithma,2008,63(4):151-166.
[11] STERLING A.Distributed agreement in tile self-assembly[J].Natural Computing:an International Journal,2011,10(1):337-355.
[12] 张成,杨静,许进.自组装DNA/纳米颗粒分子逻辑计算模型[J].科学通报,2011,56(27):2276-2282.ZHANG Cheng,YANG Jing,XU Jin.Molecular logic computing model based on self-assembly of DNA nanoparticles[J].Chinese Sci Bull,2011,56(27):2276-2282.(In Chinese)
[13] XU Jin,TAN Gang-jun,FAN Yue-ke,et al.DNA computer principle,advances and difficulties(Ⅳ):on the models of DNA computer[J].Chinese Journal of Computers,2007,30(6):881-893.
[14] 张勋才.自组装DNA计算模型的研究及应用[D].武汉:华中科技大学计算机工程与技术学院,2009.5.ZHANG Xun-cai.The reserch and applications of DNA computing by self-assembly[D].Wuhan:Huazhong Univ of Sci&Tech,2009.5.(In Chinese)
[15] HE Yu,MAO Cheng-de,et al.Hierarchical self-assembly of DNA into symmetric supramolecular polyhedral[J].Nature,2008,452:198-202.
[16] WINFREE E.Design and self-assembly of two-dimensional DNA crystals[J].Nature,1998,394:1223-1226.
[17] YURIY B.Solving NP-complete problems in the tile assembly model[J].Theoretical Computer Science,2008,395(1):31-46.
[18] 李肯立,姚凤娟,许进,等.子集和问题的O(1.414n)链数DNA计算机算法[J].计算机学报,2007,30(11):1948-1953.LI Ken-li,YAO Feng-juan,XU Jin,et al.An O(1.414n)volume molecular solutions for the subset-sum problem on DNA-based supercomputing [J].Chinese Journal of Computers,2007,30(11):1948-1953.(In Chinese)
