包涵体变复性技术研究进展
2012-04-13罗莉李坤王保成王明蓉
罗莉,李坤,王保成,王明蓉
·综述·
包涵体变复性技术研究进展
罗莉,李坤,王保成,王明蓉
现代生物技术的迅猛发展使大规模表达异源重组蛋白成为现实,这极大地促进了蛋白类产品的开发,尤其是重组蛋白药物的研发。目前非糖基化的重组蛋白表达多采用大肠杆菌的原核表达系统,该系统的突出特点是表达产物多以包涵体形式存在。与蛋白可溶性表达相比,包涵体具有产量高、蛋白稳定、容易纯化等优点,对毒性蛋白制备尤其适宜。因此,采用包涵体形式已成为规模制备重组蛋白产品的主要途径之一。
包涵体形成的原因普遍认为主要是外源蛋白在细胞内的高表达使蛋白合成速度过快,没有时间形成正确折叠;另外原核表达系统由于缺乏蛋白翻译后修饰,也容易形成包涵体;培养条件如温度、pH 值、培养基成分等也是影响包涵体形成的因素。由于包涵体绝大多数是不溶的,没有生物活性的,因而需要经过蛋白质变性-复性的过程以恢复其生物活性。目前重组蛋白变复性过程中的技术难点主要在提高蛋白浓度和生物活性两方面,而寻找高效、快速、低成本的变复性方法是该领域研究的重点。随着重组蛋白产品研发的异军突起,包涵体变复性技术的研究再次成为了国内外关注的焦点。
1 包涵体的变性
包涵体因具有高密度(1.3 mg/ml)和高抗剪切力,在变性前常采用高压匀浆或超声的方法破碎细菌,然后再洗涤除去破碎后样品中含有的脂类、杂蛋白、核酸、脂多糖等杂质。经过洗涤的包涵体,需要进一步加入变性溶液,以破坏维持包涵体蛋白结构的分子内和分子间作用力,从而使多肽链伸展达到溶解包涵体的作用。
1.1 变性溶液的改进
一般常用的变性溶液中含有去垢剂或离液剂。常用的去垢剂如 SDS、十二烷基肌氨酸等,可破坏蛋白内的疏水键,溶解包涵体。常用的离液剂如 6 ~ 8 mol/L 的尿素或盐酸胍,可破坏蛋白间的氢键,使蛋白增溶。强变性剂或变性剂浓度过高可能造成蛋白的不可逆失活,因此采用降低变性剂浓度的温和变性条件,甚至非变性的条件去溶解包涵体成为了该研究的新方向。Tsuji 等[1]报道,在 3.5 mol/L 盐酸胍变性液中加入 8 mmol/L 半胱氨酸可有效溶解包涵体且对其后续的蛋白复性有极大的促进作用。但如果在复性前透析除去半胱氨酸则该变性蛋白复性率又降回到变性液中没有半胱氨酸存在时的复性率。同样,如果将半胱氨酸只加入到复性液中也不能提高复性效率。这为含二硫键蛋白变性条件的优化提供了一个新思路。最近 Li 等[2]比较了 7 mol/L盐酸胍、8 mol/L 尿素、2% Sarkosyl 与 2 mol/L 尿素加高pH 值(通常为 12.5,过高可能引起蛋白降解)的变性液对rhG-CSF 包涵体蛋白的溶解和复性的影响,认为高 pH 环境能有效溶解该蛋白,低浓度的尿素具有协同作用,并且2 mol/L 尿素加高 pH 值的条件没有破坏蛋白二级结构,又能有效减少聚集体的形成,因而更有利于蛋白的复性。另外,Singh 等[3]研究了含有不同羟基的醇类,如甲醇、乙醇、丙醇、乙二醇、丙三醇、丁醇对 hGH 包涵体蛋白的溶解,认为 6 mol/L 正丙醇加 2 mol/L 尿素的溶解效果最好。虽然此条件溶解的蛋白量比用尿素、盐酸胍强变性剂溶解的蛋白量少,但此法溶解的蛋白复性率较后两者高,推测可能与该包涵体蛋白在这种非变性条件下仍保有类天然态的二级结构有关。
1.2 变性方法的改进
包涵体蛋白的溶解通常采用一步变性法,即用含有高浓度的盐酸胍或尿素的变性溶液一次变性。2005 年,Wang 等[4]采用两步变性法对 hIL-2/GM-CSF 包涵体蛋白进行变复性研究。首先用 7 mol/L 盐酸胍进行第一次变性(也可用含 L-精氨酸的碱性溶液代替盐酸胍进行第一次变性[5]),变性后包涵体中错误折叠的蛋白被完全展开,然后用 10 mmol/L HCl 透析除去变性液,蛋白重新聚集,再用8 mol/L 尿素溶解,这样得到的蛋白溶液在之后的复性过程中更有可能形成正确结构。而且经历了一次变性析出过程后,目的蛋白的纯度更高,有利于提高复性效率。2011 年Yang 等[5]对这种两步变性一步复性方法(two-step-denaturing and refolding method,2DR)在蛋白变复性中的应用进行了深入研究。首先采用该方法对 3 种不同折叠特性的蛋白(EGFP、MMP12、NRSF/REST DBD)变复性后的生物活性进行检测,表明 2DR 法比传统的一步变性一步复性方法更有效;接着对 88 种包涵体蛋白采用 2DR 方法进行变复性研究,其中 76% 的蛋白平均复性率高于 75%(图 1),表明此方法可广泛应用于包涵体蛋白的变复性研究和工业应用。
2 包涵体的复性
变性后的蛋白不具有生物活性,因此需要进行蛋白的重折叠,以恢复其正确空间结构,从而获得生物活性。
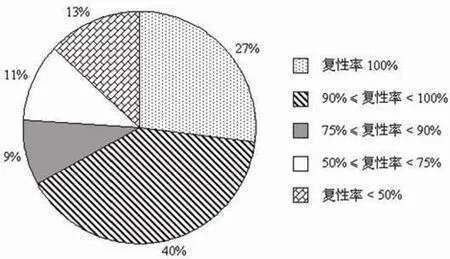
图1 88种包涵体蛋白 2DR 复性率
2.1 复性方法的研究进展
稀释复性法和透析复性法是两种经典的蛋白复性方法,主要是通过降低变性剂浓度,使蛋白缓慢重折叠,从而实现复性。这两种方法需要的稀释液或透析液体积较大,且复性时间长,因此在工业应用上有一定的限制。
近几年发展起来一种高流体静压复性(HHP)方法,其基本原理是利用 0.25 ~ 3.5 kbar (1 bar = 100 000 Pa)高压(1.5 ~ 3.0 kbar 更优)[6]使变性蛋白压缩到最紧致的状态(最少的疏水区暴露,最大的分子内静电作用),当压力消失的时候,就朝向最稳定的构象伸展,实现变性蛋白的复性。此方法已成功用于重组人 DNA 多聚酶 v[7]、OmpA70[8]等包涵体蛋白的复性。
此外,近年来发展最为迅速的复性方法是蛋白质折叠液相色谱(PFLC)[9],主要包括尺寸排阻色谱(SEC)、亲和色谱(AFC)、离子交换色谱(IEC)和疏水相互作用色谱(HIC)。SEC 是利用变性蛋白随着流动相进入凝胶颗粒内部的孔隙时,由于变性剂浓度降低促使蛋白重折叠,其分子Stokes 半径逐渐减少,当蛋白完全复性时,Stokes 就是一个常数,蛋白以天然状态被洗脱下来,此法对蛋白生物活性影响较小。AFC 是利用色谱柱上特异配体与目的蛋白间可逆的亲和作用使变性蛋白保留在柱上,与变性剂分离,而后在洗脱过程中进行复性。它具有高选择性、高分辨率和高容量的特点。近年来固定化金属亲和色谱(immobilized metal affinity chromatography,IMAC)是这类技术中应用最为广泛的。IEC 是利用变性蛋白与固定相分子间电荷作用而被吸附到固定相表面,在洗脱过程中进行吸附-解吸附-再吸附的复性。HIC 作用基础是溶质在高浓度盐中保留,低浓度盐中洗脱的色谱模式,疏水作用有效抑制变性蛋白的相互聚集,有利于蛋白的折叠。目前 PFLC 因为具有有效移除变性剂,同时进行目的蛋白的复性和纯化,以及变性剂易回收的优点,被广泛应用于重组蛋白的复性研究。
2.2 复性效率提高的改进
变性蛋白在复性过程中遇到的主要困难有复性浓度低、复性过程中容易重新形成聚集体,以及复性蛋白的生物活性低等问题。因此人们在研究蛋白变复性技术时针对这3方面的问题提出了不少的改进方法和建议。
2.2.1提高复性浓度 蛋白浓度是影响复性效率的一个主要因素,由于蛋白在高浓度条件下容易形成聚集或错误折叠,因此一般采用低浓度(< 0.1 mg/ml)复性,但这又会延长工艺流程,降低生产效率,不适合大规模生产。多次、间断的脉冲式方法与各种复性技术结合,可以显著提高复性浓度,如 Chen 和 Leong[10]采用脉冲式 SEC(pulsed fed SEC,PF-SEC),通过 3 次上样,使 AFP(α-胎球蛋白)的上样量从 5 mg 提高到 17.7 mg,复性浓度达到 0.9 mg/ml,而复性收率只从 60% 轻微下降到 53%。此外他们还比较了PF-SEC 和脉冲式稀释复性 2 种方法,结果表明前者复性收率及纯度(53% 和 40%)都显著高于后者(7% 和5%)。
2.2.2减少聚集体形成 在复性过程中除去变性剂或降低其浓度后蛋白容易重新聚集,尤其是在蛋白浓度提高后更为明显,这对复性过程非常不利。目前防止蛋白聚集体产生的措施有:①在稀释和透析复性中可通过添加一些小分子物质来减少聚集体形成,主要有两类:一类目的是增加蛋白稳定性,如 MgCl2、甘氨酸、硫酸铵、多元醇、低浓度尿素或盐酸胍等;一类目的是减少聚集体形成,如精氨酸、脯氨酸、聚乙二醇等[11]。②PFLC 复性过程中,蛋白的聚集可能会引起柱压升高、堵柱等问题。人工分子伴侣可帮助蛋白折叠和减少聚集,目前已与 PFLC 法结合,广泛应用于蛋白复性。样品进入 SEC 柱前先经过分子伴侣,可以改善上样过程和柱上复性时易出现的变性蛋白聚集的情况,但进样时间和体积非常重要[12]。在 IMAC 基础上引入人工分子伴侣(artificial chaperone-assisted IMAC,AC-IMAC),常用的是CTAB-βCD(cetyltrimethylammonium bromide-β-cyclodextrin)体系。首先将蛋白和 CTAB 混合形成复合物,再通过 His标签结合到 IMAC 柱上,然后用含有 βCD 的复性液移除CTAB,从而实现蛋白的柱上复性。Dong 等[13]报道利用此法获得了纯度达 99%,活性达 80% 的 EGFP 重组蛋白,比稀释复性(活性 14%)、分子伴侣辅助复性(活性 26%)、IMAC 复性(活性 60%)的效率都高;2010 年,Wang 等[14]在对 lysozyme 重组蛋白的复性中采用人工分子伴侣与 IEC 结合的方法,即 AMC-IEC(artificial molecular chaperone-IEC),表明蛋白起始浓度在 200 mg/ml 时,CTAB也可有效抑制蛋白聚集,并且蛋白浓度为 180 mg/ml 时的AMC-IEC 复性效率(76.6%)比单独的 IEC 复性效率(42.4%)高的多。③其他方法。Yamamoto 等[15]认为在复性液中加入去污剂和水溶性有机溶剂对蛋白复性有很好的协同作用,一方面去污剂阻止蛋白聚集,另一方面有机溶剂可以影响蛋白-蛋白、蛋白-去污剂之间的相互作用。他们通过对溶菌酶复性条件的研究,表明复性液中加入 2 mmol/L 的 CTAB 和 20% 的 DMSO 的复性效率(34%)比不加CTAB 和 DMSO 的复性效率(10%)和只加 CTAB(13%)或 DMSO(20%)的复性效率都高。
2.2.3提高蛋白生物活性 复性最终的目的是要获得有生物活性的蛋白,所以复性蛋白量的提高并不意味着有活性蛋白的含量就提高了。在复性过程中蛋白所处环境的改变是影响其生物活性的重要因素,因而缓慢改变复性环境(尤其是变性剂的浓度)将有利于蛋白生物活性的提高。具体方法有如下两种:①一元梯度。缓慢降低变性剂浓度能为蛋白重折叠提供一个较温和的条件,有利于提高蛋白生物活性。这种技术与 SEC 结合已在 rhG-CSF[16]复性中获得成功,但这种方法中变性剂的最终浓度和变性剂改变的梯度长度与蛋白复性效率息息相关[17]。②二元梯度。在一元梯度的基础上又提出了二元梯度的复性方式,它主要是通过缓慢降低变性剂浓度的同时逐渐增加帮助折叠的添加剂的浓度,使复性效率进一步得到提高。如 pH-尿素二元梯度(DT389-hIL13重组蛋白[18],pH 3.0 → 8.0,尿素 8 → 0.5 mol/L)、精氨酸-尿素二元梯度(NTA 重组蛋白[19],精氨酸 0 → 0.6 mol/L,尿素 8 → 2 mol/L)、甘油-盐酸胍二元梯度(rhG-CSF[20],甘油0 → 15%,盐酸胍 6 → 0 mol/L)等。
2.3 复性条件优化的新方法
蛋白复性是一个十分复杂的过程,影响复性的因素也有很多,比如蛋白浓度、离子强度、温度、溶液 pH 值、添加剂的浓度和组合等,这无疑增加了研究人员的工作量,因此高通量筛选技术在这种情况下迅速发展起来。Nara 等[21]报道了使用沸石(zeolite)的高通量蛋白复性筛选方法,沸石是一种多孔的晶状铝硅酸盐,不同的 SiO2/Al2O3比例表现了不同的离子交换特性和疏水性。首先用 6 mol/L 盐酸胍溶解包涵体,然后吸附到 96 孔板的沸石上,分别加入含有不同 pH值、盐浓度、添加剂等的复性溶液,从而筛选出最适的复性条件。它最大的优点是在高浓度盐存在的情况下,蛋白也可以被很好地吸附。Dechavanne 等[22]提出将高通量复性筛选和实验设计(design of experiments,DOE)软件相结合的两步复性条件筛选。首先在 96 孔板上进行96 种不同条件的复性,以确定影响该蛋白复性的因素,接着用 DOE 软件设计第二次复性筛选方案,每个因素都有较广的浓度范围,从而确定各种添加物的最优浓度,此法需要的蛋白量低(只需要 40 mg 变性蛋白)、耗时短(少于2 周)。
3 展望
要获得以原核系统表达的活性重组蛋白可以从以下几个方向考虑:①从上游基因构建(如同义密码库、融合蛋白构建)、蛋白表达(培养基、温度等条件优化)出发,尽可能使重组蛋白以可溶的活性形式存在,减少包涵体形成。②包涵体变复性技术路线。此法难点在于如何将无生物活性、错误折叠的不溶蛋白转变成有活性的可溶蛋白。由于变性过程中强变性剂易导致蛋白不可逆失活,因而人们越来越关注寻找温和、甚至非变性的条件来代替强变性条件,并且在变性方法上也在寻求新的突破。蛋白变性后的复性方法有很多种,但由于每种蛋白都有自己特有的折叠方式和途径,因而至今没有一种对所有包涵体蛋白都通用的复性方法。PFLC法具有复性和纯化同步进行的显著优点,因而发展最为迅速。另外在复性过程中提高蛋白浓度和生物活性是复性技术的瓶颈,因此近年来人们在这方面做了很多研究,并取得了一定的进展。③过去认为包涵体是无活性、错误折叠的蛋白,但 2005 年,García-Fruitós 等[23]提出不是所有包涵体都是无生物活性的。同年,Jevsevar 等[24]也报道他们从包涵体中不通过变性-复性过程就提取出了有活性的 hG-CSF蛋白,后来人们陆续证实了这种非传统包涵体[25-26]的存在。2012 年,Sans 等[27]报道通过优化表达 FucA 醛缩酶的培养条件获得了具有生物活性的包涵体,并且其生物活性比用传统培养条件获得的可溶性蛋白的生物活性更高。无疑这种非传统包涵体的提出给原核系统表达重组蛋白带来了新的曙光,但由于其形成机制以及获得条件和途径都不清楚,因而还处在研究的初级阶段,但其潜力巨大,不容忽视。
[1] Tsuji I, Mastui H, Ito T, et al. L-cysteine-enhanced renaturation of bioactive soluble tumor necrosis factor ligand family member LIGHT from inclusion bodies in Escherichia coli. Protein Expres Purif, 2011, 80(2):239-245.
[2] Li M, Fan H, Liu J, et al. High pH solubilization and chromatography-based renaturation and purification of recombinant human granulocyte colony-stimulating factor from inclusion bodies. Appl Biochem Biotech, 2012, 166(5):1264-1274.
[3] Singh SM, Sharma A, Upadhyay AK, et al. Solubilization of inclusion body proteins using n-propanol and its refolding into bioactive form. Protein Expres Purif, 2012, 81(1):75-82.
[4] Wang QR, Ma L, Zhou MQ, et al. Expression, refolding, purification, and bioactivity of recombinant bifunctional protein, hIL-2/GM-CSF. Protein Expres Purif, 2005, 39(2):131-136.
[5] Yang Z, Zhang L, Zhang Y, et al. Highly efficient production of soluble proteins from insoluble inclusion bodies by a two-stepdenaturing and refolding method. PLoS One, 2011, 6(7):e22981.
[6] Randolph TW, Carpenter JF, John RS. High pressure refolding of protein aggregates and inclusion bodies: US, 2010/0255536 A1. 2010-10-07.
[7] Arana ME, Powell GK, Edwards LL, et al. Refolding active human DNA polymerase v from inclusion bodies. Protein Expres Purif, 2010, 70(2):163-171.
[8] Fraga TR, Chura-Chambi RM, Goncales AP, et al. Refolding of the recombinant protein OmpA70 from Leptospira interrogans from inclusion bodies using high hydrostatic pressure and partial characterization of its immunological properties. J Biotechnol, 2010, 148(2-3):156-162.
[9] Geng X, Wang C. Protein folding liquid chromatography and its recent developments. J Chromatogr B Analyt Technol Biomed Life Sci, 2007, 849(1):69-80.
[10] Chen Y, Leong SSJ. High productivity refolding of an inclusion body protein using pulsed-fed size exclusion chromatography. Process Biochem, 2010, 45(9):1570-1576.
[11] Gerami SMN, Farajnia S, Mahboudi F. Co-solute assistance in refolding of recombinant proteins. Afr J Biotechnol, 2011, 10(53): 10811-10816.
[12] Chen YC, Liu HS. Chaperon solvent plug design in size-exclusion chromatography protein refolding process. Enzyme Microb Tech, 2011, 49(2):203-208.
[13] Dong XY, Chen LJ, Sun Y. Refolding and purification ofhistidine-tagged protein by artificial chaperone-assisted metal affinity chromatography. J Chromatogr A, 2009, 1216(27):5207-5213.
[14] Wang C, Zhang Q, Cheng Y, et al. Refolding of denature/reduced lysozyme at high concentrations by artificial molecular chaperone-ion exchange chromatography. Biotechnol Prog, 2010, 26(4):1073-1079.
[15] Yamamoto E, Yamaguchi S, Nagamune T. Synergistic effects of detergents and organic solvents on protein refolding: control of aggregation and folding rates. J Biosci Bioeng, 2011, 111(1):10-15.
[16] Wang C, Wang L, Geng X. High recovery refolding of rhG-CSF from Escherichia coli, using urea gradient size exclusion chromatography. Biotechnol Prog, 2008, 24(1):209-213.
[17] Wang C, Cheng Y. Urea-gradient protein refolding in size exclusion chromatography. Curr Pharm Biotechno, 2010, 11(3):289-292.
[18] Sun W, Dai X, Zheng Y, et al. On-column refolding purification of DT389-hIL13 recombinant protein expressed in Escherichia coli. Protein Expres Purif, 2011, 75(1):83-88.
[19] Fan X, Xu D, Lu B, et al. Improving the refolding of NTA protein by urea gradient and arginine gradient size-exclusion chromatography. J Biochem Bioph Meth, 2008, 70(6):1130-1138.
[20] Wang F, Liu Y, Ma G, et al. Glycerol-assisted hydrophobic interaction chromatography improving refolding of recombinant human granulocyte colony-stimulating factor. Appl Biochem Biotech, 2009, 159(3):634-641.
[21] Nara TY, Togashi H, Sekikawa C, et al. High-throughput protein refolding screening method using zeolite. Biotechnol Prog, 2009, 25(4):1071-1077.
[22] Dechavanne V, Barrillat N, Borlat F, et al. A high-throughput protein refolding screen in 96-well format combined with design of experiments to optimize the refolding conditions. Protein Expres Purif, 2011, 75(2):192-203.
[23] García-Fruitós E, González-Montalbán N, Morell M, et al. Aggregation as bacterial inclusion bodies does not imply inactivation of enzymes and fluorescent proteins. Microb Cell Fact, 2005, 4(1):27.
[24] Jevsevar S, Gaberc-Porekar V, Fonda I, et al. Production of nonclassical inclusion bodies from which correctly folded protein can be extracted. Biotechnol Prog, 2005, 21(2):632-639.
[25] Peternel S, Grdadolnik J, Gaberc-Porekar V, et al. Engineering inclusion bodies for non denaturing extraction of functional proteins. Microb Cell Fact, 2008, 7(1):34-42.
[26] Lu SC, Lin SC. Recovery of active N-acetyl-d-glucosamine 2-epimerase from inclusion bodies by solubilization with non-denaturing buffers. Enzyme Microb Tech, 2012, 50(1):65-70.
[27] Sans C, García-Fruitós E, Ferraz RM, et al. Inclusion bodies of fuculose-1-phosphate aldolase as stable and reusable biocatalysts. Biotechnol Prog, 2012, 28(2):421-427.
10.3969/cmba.j.issn.1673-713X.2012.04.008
“重大新药创制”科技重大专项(2011ZX09506-007)
610052 成都,中国医药集团总公司四川抗菌素工业研究所(罗莉);610023 成都生物制品研究所有限责任公司(李坤、王保成、王明蓉)
王明蓉,Email:mignrongw2007@163.com
2012-05-03
