电子基因微芯片在医学研究中的应用
2012-04-13郭育奇赵春燕
郭育奇,赵春燕
·综述·
电子基因微芯片在医学研究中的应用
郭育奇,赵春燕
基因芯片(DNA microchip)也称 DNA 微阵列(DNA microarray),是把大量基因探针或基因片段按特定的排列方式固定在芯片载体上,形成致密有序的 DNA 分子点阵,按碱基互补配对原则与样品 DNA 杂交,然后通过计算机进行解读和分析获取大量信息,实现对生物样品高效、平行地检测或医学诊断。由于具有高通量、快捷、便宜等优点,基因芯片在各个领域起着越来越重要的作用。芯片可以根据探针的性质、固体表面的支撑物、探针寻址或目标检测方法的不同分为原位合成寡核苷酸微阵列(situ-synthesized oligonucleotide microarray)、高密度微珠阵列(high-density bead arrays)、电子微芯片(electronic microarrays)等。近年来,电子微芯片技术的发展越来越引起人们的关注,它是利用电场、热循环或化学方法等吸引带负电的 DNA 或其他生物分子和探针结合到芯片特定位点上从而进行主动杂交检测[1]。该种芯片在精确性、准确性和严格性控制方面优于传统芯片,可用于病原生物的检测和鉴定、微生物的分型、基因表达分析、基因突变及多态性分析、疾病诊断和预测等。本文就电子基因芯片的工作原理、优势和在医学方面的应用作简要综述。
1 电子基因芯片的工作原理和操作流程
电子基因芯片的工作原理如图 1 所示,它可借助电子场加快带电的生物分子结合到芯片上的速度。芯片上的每一个位点代表了一个电极,每一个电极都可被分别通电。通电后产生的电场可加速带负电的 DNA 探针或其他生物分子的结合。电子场一方面吸引特异性的寡核苷酸探针到特定位点,另一方面捕获目的 DNA 并特异性杂交到寡聚探针上。芯片载体上建有电极矩阵,其上的每一个位点都有一个独立的金属线接头。该矩阵通过标准的硅平版印刷术被组合在一起,并用 CMOS 片包埋,CMOS 片上的每个电极可被电压和电流单独控制。整个矩阵被包埋入一个一次性流体式的芯片中,这样便可提供一个样品或试剂的自动控制。电极矩阵上覆盖了一层 1 ~ 2 μm 厚的水凝胶渗透层,该层含有亲合素,亲合素促使探针或目的基因被直接捕获。在矩阵操作过程中,高电位促使水在阳极发生氧化反应,阴极发生还原反应。水的氧化反应产物是 H+和氧。这项技术就是利用阳极产生的 H+进行高效率的 DNA 杂交。反过来,如果阴极被激活产生 OH–,这样带负电的 DNA 分子就远离电极。因此,这种电子式的严格性可识别出单个碱基的不同,因而可用来分析单核苷酸多态性(single nucleotide polymorphisms,SNP)[2-3]。
Nanogen 公司生产的 Nanochip 检测系统包括 3 个部分:微电子芯片、上样系统和检测系统。微电子芯片是由 100 或 400 个位点组成,通过连接器来控制。通过对位点微电极的电位控制,在微电极的上方完成探针分子的选择性固定,靶核酸定向转移集中并与探针主动杂交。上样系统是通过电场作用将探针或其他分子锚定到微电子芯片上,锚定完成之后对非特异性结合的物质进行洗涤,减少干扰反应。检测系统用来检测杂交后荧光信号的强度。运用互补金属氧化半导体技术来进行核酸的电子寻址,如果探针和目的 DNA之间发生了特异性杂交反应,电子芯片被扫描后将呈现阳性结果[3-4]。
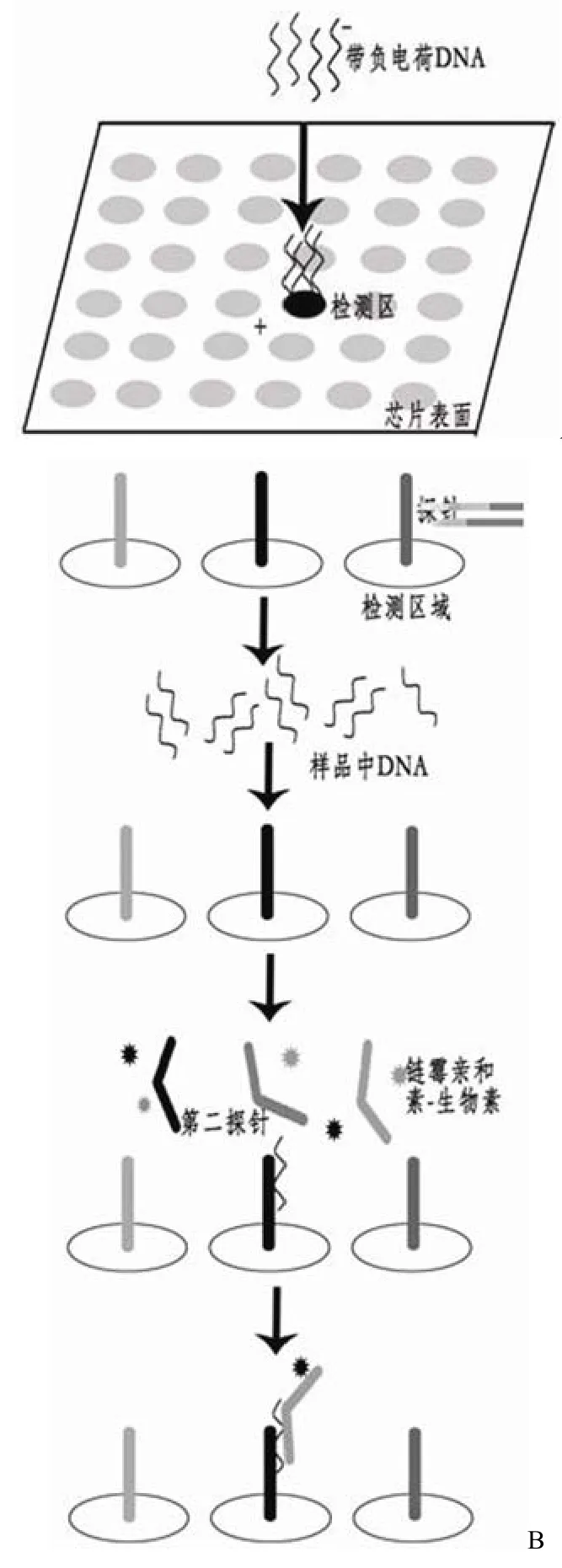
图1 电子基因芯片的工作原理图
2 电子基因芯片的优势
与传统的基因芯片相比,Nanochip 电子基因芯片具有如下几点独特性:①Nanochip 系统利用电子场和热循环技术,达到了精确性、准确性和严格性的控制。②在芯片上,通过对电位的控制来移动和浓缩 DNA,对生物分子的电子吸引力大大加速了分子结合到芯片上的速度,与传统的被动式芯片(如 Affymetrix,Genechip)相比,增加了 1000 倍以上。被动式芯片的点样需要花几个小时,而主动式电子芯片仅需要 30 ~ 60 s。因而,这种高效性可使 Nanochip 检测极低浓度的靶基因。③多重性分析:电子式芯片提供了一个灵活的平台。例如,在同一个芯片上可同时检测一个样品的多个基因,或一个基因的多个样品,或多个样品的多个基因。相反,传统 DNA 芯片上的位点不能被分别控制,因此,只能检测一个样品的多个基因。该系统还配有晶载内存,用于储存遗传分析和检测传染病病原体的关键信息,为多路的检测运转提供了更高的通量。以最小的消耗来检测多样的目标产物。④电子式严格性:可识别单一碱基的不同,并可确定单核苷酸多态性。⑤结构开放性:允许实验用户定义、选择和建立个性化的测试面板或从预先设定好的分析物特效试剂(analyte specific reagents,ASRs)中选择。⑥性价比高:该系统简化了工作流程,缩短了手动操作的时间。将开发和检测突变的功能集中在一张芯片上,比传统的芯片性价比高[2, 5-6]。
3 电子基因芯片在医学中的应用
3.1微生物的检测和鉴定
在临床微生物学研究中,通过中低密度的电子基因微芯片对微生物的目标遗传序列进行评价是非常重要的。检测中最常用的靶基因是细菌中的 16S rRNA、真菌中的 28S rRNA 和 rRNA 基因间的转录间隔区[7]。在芯片检测前需对样本用宽范围 PCR 或多重 PCR 处理,该措施可有效地提高灵敏度。在基因多样性的筛选中,微芯片的平行性也高于其他筛选方法。对于细菌中的分枝杆菌属可将 gyrB、rpoB 和 katG 基因作为目标基因。Sanguinetti 等[8]用 Nanochip电子微阵列来鉴定临床相关分枝杆菌的种属。先将临床菌株进行分离培养,然后用 PCR 方法扩增分枝杆菌的 rRNA基因,最后将产物和种属选择性的探针进行杂交。结果证明该方法比传统的鉴定方法更加快速、精确。
电子基因芯片结合宽范围的 PCR 技术还可用于检测和鉴定真菌、寄生虫和病毒病原体。Takahashi 等[9]对流感病毒 A 和 B,呼吸道合胞病毒,副流感病毒 1,2,3 型进行检测。比较了 Nanochip 400 系统和直接荧光抗体染色法(direct fluorescent-antibody staining,DFA)两种方法检测 122 份标本的结果,两者一致性达到 86.9%。若把 DFA作为检测的金标准,Nanochip 400 系统的灵敏度和特异度分别为 84.6% 和 100%。差异分析表明 Nanochip 400 系统灵敏度和特异度可达 98.6% 和 100%。同时还比较了Nanochip 400 系统和实时定量 PCR(real-time PCR)两种方法检测 130 份标本的结果,两者的一致性为 93.9%。在差异分析中,Nanochip 400 系统敏感性和特异性分别为98.6% 和 100%,而且 Nanochip 400 系统检测过程在 4 h内即可完成。由此可见 Nanochip 系统检测法是一种简便、快速、灵敏的检测常见呼吸道病毒的方法。
除此之外,该芯片还可以用于检测环境中的有害微生物,防止因有害微生物大量繁殖造成的疾病或生态灾难。Barlaan 等[10]对有害藻类大量繁殖过程(harmful algal blooms,HABs)中的细菌聚集物进行研究。通过变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)对 16S rRNA 基因进行分析。而后设计并合成特异的Nanochip微阵列的探针,通过杂交反应检测物种。该方法可检测 HABs 的发生和相对丰度,并可找出与 HABs 发生相关的核酸序列,该研究表明电子微阵列可检测和监控环境中的微生物。
3.2微生物的分型
利用电子基因芯片对不同种类基因组做检测可对微生物进行分型。该技术能克服传统分型方法的许多限制,与遗传学方法相结合后可补充或取代传统的血清型方法。该技术现已用于病原体及其繁殖,患者的再感染与复发,患者的院内感染以及抗药性菌株传播等方面的研究。
Saunders 等[11]通过膜蛋白基因 V1 区对人类免疫缺陷病毒 1 型(human immunodeficiency virus type 1,HIV-1)进行分型。通过对样品进行巢式反转录 PCR,将扩增产物与 Nanochip 微阵列相连后与寡核苷酸探针杂交,根据探针杂交的模式对病毒株分型。文中将该方法与测序方法或异源双链迁移率分析法(heteroduplex mobility assay,HMA)比较,不但准确性可与后两种方法相媲美,并且是一种方便和高效的 HIV-1 分型方法。
在雷特综合征患者中,女性 X 染色体相偶联的MeCP2 基因中的 8 个点突变占了正向突变总数的 70%。Thistlethwaite 等[12]以 Nanogen 系统为基础开发出了一种新的方法来检测这 8 个突变。然后将结果与变性高效液相色谱和测序方法结果进行比较。结果表明该分型技术表现出100% 的特异性和 3% 的多义性。在特异性和精确性方面,该方法与单引物扩增反应在同一水平,并且更快更经济。
3.3基因的差异表达分析
微芯片技术显著地增强了对基因差异表达的检测能力。与传统的微芯片相比,电子基因芯片能更快、更精确地分析不同组织中基因表达的变化。通过检测基因的差异表达,从而研究特殊基因与疾病之间关系。对疾病的诊断和治疗有重要意义。Corradi 等[13]应用该芯片对人白血病相关的融合基因进行研究。特定基因的表达量、基因多态性分布等生物学特征,在危险分层、靶向治疗或检测微量残存疾病的标记等方面有重要作用。作者将多重 RT-PCR、电杂交技术与Nanochip 系统相结合,对人普通的白血病转录融合基因进行筛选。实验证明该方法是高效的基因表达分析方法。
3.4基因突变检测和多态性分析
研究表明大多数疾病是由多个核苷酸突变引起的,因此研究这些核苷酸突变能获取有价值的医学信息。在临床诊断中,单核苷酸多态性可作为识别疾病基因的遗传标志,它既可用于分析病人与对照组之间的相关性又可解释同种微生物之间的接合作用。通过 SNP 分析可绘制和诊断疾病相关的等位基因。电子微阵列技术简单、快速、自动化程度高,可用来筛选和检测大量相关的 SNP,从而确定特殊微生物的 SNP 和应答病原体毒素的 SNP。
Evans 等[14]建立了一种以 Nanochip 系统为基础的新方法来检测凝血因子 V Leiden(factor V Leiden,FVL)中点突变,而后将该方法得出的结果与 Third Wave 分析法得出结果进行比较。Nanochip 系统在野生型,杂合子和纯合子特征化的精确度均为 100%,而 Third Wave 分析的精确度为 99.2%,90.2% 和 100%,前者显然优于后者。同样可对家族性地中海热(familial Mediterranean fever,FMF)中FMF(MEFV),factor V(F5)和 factor II(F2)基因型进行鉴定,并做突变和 SNP 分析。结果证明新方法是一种自动化、高通量的 SNP 检测方法[15]。
在儿童急性淋巴细胞白血病(acute lymphoblastic leukemia,ALL)人中,硫嘌呤甲基转移酶(thiopurine methyltransferase,TPMT)活性、基因型对 6-硫嘌呤(6-mercaptopurine,6-MP)化疗个体化有重要意义。Albayrak 等[16]以患 ALL 病的土耳其儿童为研究对象,应用 Nanochip 技术对患者的遗传多态性进行检测,探究多态性与个体低 TPMT 活性和耐受 6-MP 突变体之间的关系。检测结果表明患者 TPMT 多态性频率和分布与其他白种人相似,而 6-MP 毒性过大导致突变体的产生,所以对TPMT 基因的分型须在用 6-MP 治疗之前完成。
在慢性阻塞性肺疾病(chronic obstructive pulmonary disease,COPD)和弥散性支气管扩张(disseminated bronchiectasis,DB)患者中,Papatheodorou等[17]对 TNF-α基因启动子的多态性与基因的转录活性的关系进行研究,运用 Nanochip 系统对 5 个启动子的 SNP 进行分析。结果显示在 COPD 组、DB 组和健康吸烟者组三者之间 5 个SNP 的基因型频率没有显著性差异。仅 COPD 患者组的单倍体频率(haplotype frequencies)与普通人组之间出现了差异。在确定遗传风险度研究中,作者对 SERPINA1、2 和ADRB2 基因中的 5 个 SNP 进行分型。结果表明SERPINA1 中 p.V213A 的多态性与 DB 的风险有关。ADRB2 的 p.G16R 是严重 COPD 风险因素[18]。
3.5其他方面
除此之外,Weigum 等[19]开发出了一种新型的 BNC 传感技术(bio-nanochip sensor technique),并用此技术检测脱屑性细胞学(exfoliative cytology)样品中的口腔癌细胞和生物标记物(oral cancer biomarkers)。通过对 41 例牙科患者和 11 例正常人的样品的表皮细胞和表皮生长因子受体(EGFR)进行检测和分析,包括细胞核直径和面积、细胞核和细胞质(N/C)比例、生物标记的表达量。结果发现几乎所有细胞的这 4 项关键的参数都得到明显的提高。将检测的一系列特征结果进行综合分析,能特异性地提高对口腔癌及癌变前的状态的辨别力度。
在卵巢癌的早期诊断方面,Raamanathan 等[20]应用程序化的生物纳米芯片(programmable bio-nano-chip)平台对样品中的生物标记物进行检测。该技术可在 45 min内对血清中 CA125 做定量检测和分析。该系统在卵巢癌诊断过程中有非常好的应用前景。
4 电子基因微芯片前景展望
电子基因芯片发展到现在不足十年的时间,通过它,人类显著地提高了认识生命本质的能力,为研究生命这个复杂的网络系统打下基础[21]。现在各国的研究机构都在对此进行积极的研究,电子基因芯片在基因表达谱分析、病原生物鉴定、基因分型、突变检测及单核苷酸多态性分析等方面都已呈现出广阔的应用前景。虽然电子基因芯片技术还存在问题,但随着新技术和新思想的不断产生,电子基因芯片的发展与完善将对医学研究与应用产生深远的影响[22]。
参考文献
[1] Heller MJ, Forster AH, Tu E. Active microelectronic chip devices which utilize controlled electrophoretic fields for multiplex DNA hybridization and other genomic applications. Electrophoresis, 2000, 21(1):157-164.
[2] Keen-Kim D, Grody WW, Richards CS. Microelectronic array system for molecular diagnostic genotyping: Nanogen NanoChip 400 and molecular biology workstation. Expert Rev Mol Diagn, 2006, 6(3):287-294.
[3] Forster AH, Krihak M, Swanson PD, et al. A laminated, flex structure for electronic transport and hybridization of DNA. Biosens Bioelectron, 2001, 16(3):187-194.
[4] Cao NN, Huang HL, Zhao CY. Development of electric-field assisted isothemal on-chip amplication for detecting E.coli O157:H7. Chin Med Biotechnol, 2010, 5(6):455-457. (in Chinese)曹娜娜, 黄红兰, 赵春燕. 电子基因芯片DNA等温扩增技术检测大肠埃希菌O157:H7方法建立. 中国医药生物技术, 2010, 5(6): 455-457.
[5] Sosnowski R, Heller MJ, Tu E, et al. Active microelectronic array system for DNA hybridization, genotyping and pharmacogenomicapplications. Psychiatr Genet, 2002, 12(4):181-192.
[6] Melissa BM, Tang YW. Basic concepts of microarrays and potential applications in clinical microbiology. Clin Microbiol Rev, 2009, 22(4): 611-633.
[7] Kakinuma K, Fukushima M, Kawaguchi R. Detection and identification of Escherichia coli, Shigella, and Salmonella by microarrays using the gyrB gene. Biotechnol Bioeng, 2003, 83(6): 721-728.
[8] Sanguinetti M, Novarese L, Posteraro B, et al. Use of microelectronic array technology for rapid identification of clinically relevant mycobacteria. J Clin Microbiol, 2005, 43(12):6189-6193.
[9] Takahashi H, Norman SA, Mather EL, et al. Evaluation of the NanoChip 400 system for detection of influenza A and B, respiratory synctial, and parainfluenza viruses. J Clin Microbiol, 2008, 46(3): 1724-1727.
[10] Barlaan EA, Furukawa S, Takeuchi K. Detection of bacteria associated with harmful algal blooms from coastal and microcosm environments using electronic microarrays. Environ Microbiol, 2007, 9(3):690-702.
[11] Saunders NA, Alexander S, Tatt I. env Gene typing of human immunodeficiency virus type 1 strains on electronic microarrays. J Clin Microbiol, 2005, 43(4):1910-1916.
[12] Thistlethwaite WA, Moses LM, Hoffbuhr KC, et al. Rapid genotyping of common MeCP2 mutations with an electronic DNA microchip using serial differential hybridization. J Mol Diagn, 2003, 5(2): 121-126.
[13] Corradi B, Fazio G, Palmi C, et al. Efficient detection of leukemia-related fusion transcripts by multiplex PCR applied on a microelectronic platform. Leukemia, 2008, 22(2):294-302.
[14] Evans JG, Lee-Tataseo C. Determination of the factor V Leiden single-nucleotide polymorphism in a commercial clinical laboratory by use of NanoChip microelectronic array technology. Clin Chem, 2002, 48(9):1406-1411.
[15] Moutereau S, Narwa R, Matheron C, et al. An improved electronic microarray-based diagnostic assay for identification of MEFV mutations. Hum Mutat, 2004, 23(6):621-628.
[16] Albayrak M, Konyssova U, Kaya Z, et al. Thiopurine methyltransferase polymorphisms and mercaptopurine tolerance in Turkish children with acute lymphoblastic leukemia. Cancer Chemother Pharmacol, 2011, 68(5):1155-1159.
[17] Papatheodorou A, Latsi P, Vrettou C, et al. Development of a novel microarray methodology for the study of SNPs in the promoter region of the TNF-alpha gene: their association with obstructive pulmonary disease in Greek patients. Clin Biochem, 2007, 40(12):843-850.
[18] Papatheodorou A, Makrythanasis P, Kaliakatsos M, et al. Development of novel microarray methodology for the study of mutations in the SERPINA1 and ADRB2 genes--their association with Obstructive Pulmonary Disease and Disseminated Bronchiectasis in Greek patients. Clin Biochem, 2010, 43(1-2):43-50.
[19] Weigum SE, Floriano PN, Redding SW, et al. Nano-bio-chip sensor platform for examination of oral exfoliative cytology. Cancer Prev Res (Phila), 2010, 3(4):518-528.
[20] Raamanathan A, Simmons GW, Christodoulides N, et al. Programmable bio-nano-chip systems for serum CA125 quantification: toward ovarian cancer diagnostics at the point-of-care. Cancer Prev Res (Phila), 2012, 5(5):706-716.
[21] Trevors JT, Masson L. DNA technologies: what's next applied to microbiology research? Antonie Van Leeuwenhoek, 2010, 98(3): 249-262.
[22] Xiong W. The research status and application prospect of gene chip technology in life sciences research. Life Sci Instruments, 2010, 8(4):32-36. (in Chinese)熊伟. 基因芯片技术在生命科学研究中的应用进展及前景分析.生命科学仪器, 2010, 8(4):32-36.
DOI:10.3969/cmba.j.issn.1673-713X.2012.04.010
基金项目:吉林省卫生厅项目(2008Z001)
作者单位:130021 长春,吉林大学白求恩医学院病原生物学系
通讯作者:赵春燕,Email:chunyanzhao44@yahoo.com
收稿日期:2012-03-16
