基于SRAP标记的莲种质资源遗传多样性分析
2012-03-22欧阳冬梅刘凰徐金星刘春华邹东旺杨良波唐记平谢克强
欧阳冬梅,刘凰,徐金星,刘春华,邹东旺,杨良波,唐记平,谢克强
(1.江西广昌县白莲产业发展局,344900;2.广昌县白莲科学研究所)
基于SRAP标记的莲种质资源遗传多样性分析
欧阳冬梅1,刘凰1,徐金星2,刘春华2,邹东旺1,杨良波2,唐记平1,谢克强1
(1.江西广昌县白莲产业发展局,344900;2.广昌县白莲科学研究所)
利用SRAP标记对来自国内外莲主要产区在20世纪初期育出、并在莲育种中具有重要利用价值的40份莲属材料进行了分析。11对引物在40份材料中共扩增了184条条带,多态性条带127条,多态性频率为69.0%,表明莲属具有丰富的遗传多样性。聚类分析结果表明,莲属种质资源可分为3大类群,这与传统意义上的形态学分类相一致。且40份莲品种(材料)表现出很强的地域相似性,这说明来自同一个地域内的品种(材料)分化程度低,遗传多样性不丰富,极易造成品种单一化。
莲;SRAP;遗传多样性分析
我国是莲的原产地之一,莲在我国不仅栽培历史悠久,而且分布广泛,且是最具特色、栽培面积最大、品种资源最丰富的水生经济作物[1]。迄今为止,中国拥有花莲品种资源800多个[2],藕莲品种和子莲品种资源分别数十个。莲是常异花授粉植物,不同品种间的基因交流很容易通过昆虫的授粉来完成,导致品种间遗传资源的混合和品种内的遗传差异较大[3]。目前,我国莲同种异名现象时有发生,同一地区品种单一化现象严重,大大增加了莲栽培的风险性。为了避免这一现象,选取了40份代表性品种(材料)进行SRAP遗传多样性分析,以期明确它们的遗传背景,探讨全国各地主要传统型品种与广昌县现有栽培品种的关系,从而为莲品种选育提供理论依据。
1 材料与方法
1.1 试验材料
试验所用40份莲品种(材料)为国内外莲主要产区在20世纪初期广泛应用于生产的优异材料。材料取自广昌县白莲科学研究所博览园内。这40份材料包括野生莲、全国各地传统品种、广昌县现栽培品种和花莲品种,材料详细信息见表1。
1.2 样品制备
采集莲幼嫩叶片置于塑料袋中(自封袋),再加入变色硅胶,摇动使叶片与硅胶充分均匀接触。一般来说,硅胶与叶片质量比应为10∶1,以保证叶片在12 h内干燥,一旦发现硅胶从深蓝变为粉红,及时更换硅胶,干燥的叶片材料在室温下保存即可。
1.3 DNA的提取
选取上述硅胶干燥的叶片30 mg,用液氮磨成粉状,再用DNA提取试剂盒提取植物基因组DNA。DNA质量检测用1%的琼脂糖凝胶进行,电泳缓冲液为0.5×TBE,电压100 V,电泳30 min。
1.4 SRAP分析
PCR扩增反应总体积为15 μL,反应混合液中包括:50 ng/μL DNA模板1 μL,10×PCR buffer 1.5 μL,10 mmol/L dNTPs 0.6 μL,10 mmol/L上下游引物各0.5 μL,2 U/μL Taq DNA聚合酶0.5 μL,不足部分用无菌水补充。
PCR扩增反应程序为95℃5 min;95℃1 min,35℃ 1 min,72℃ 1 min,5个循环;95℃ 1 min,50℃1 min,72℃ 1 min,35个循环;72℃延伸 10 min,温度降到4℃即完成扩增。
扩增产物加上10 μL的上样缓冲液94℃预变性5 min,采用6%的变性聚丙烯酰胺凝胶,0.5× TBE缓冲液,65 W恒功率电泳1.5 h。银染检测电泳结果。
1.5 SRAP数据处理
对各莲材料各引物扩增情况作统计,有带记为1,无带记为0,缺失记为2。作矩阵图输入计算机,应用平均距离法进行聚类分析,并采用NTSYS-pc(Version 2.1)软件处理,建立聚类图。
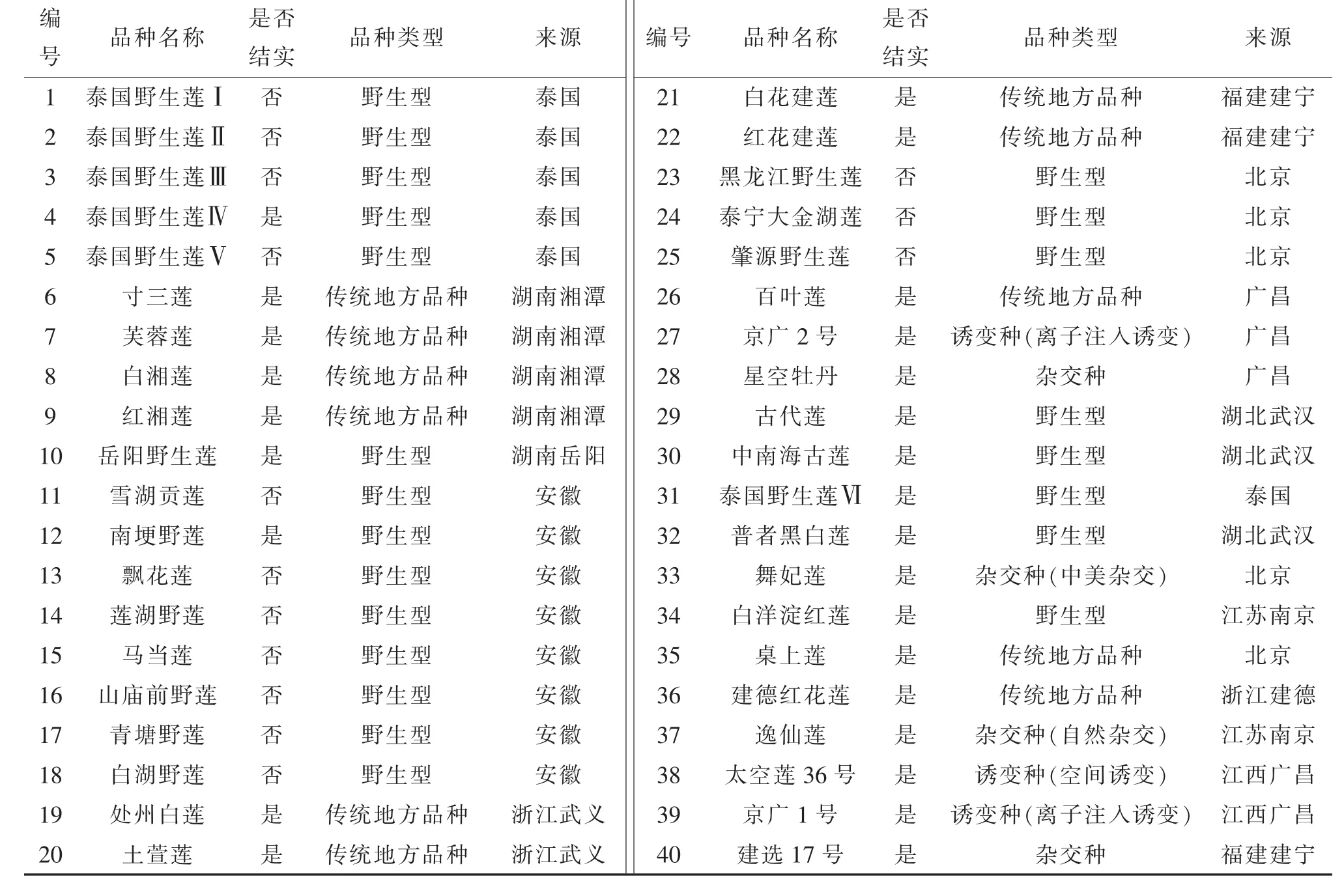
表1 试验材料信息
2 结果与分析
2.1 多态性分析
从40对SRAP引物中筛选出11对扩增稳定、多态性适中的引物对这40份材料进行扩增,11对引物共扩增产生184条带,平均每个引物对扩增出16.7条。其中多态性条带127条,多态性频率为69.0%。不同引物对多态性频率差异较大,多态性频率最高的为 EM14+ME10和 EM11+ME10,为100%,多态性频率最低的为EM9+ME10,为52.6%(表 2)。
2.2 指纹图谱的构建
以“0”“1”和“2”分别表示扩增的“有”、“无”和“缺失”,将11对多态性引物产生的127个多态性位点进行排序,形成一个数据距阵(表3),建立品种计算机化的DNA指纹,以区分供试品种(材料)。结果显示,每一份种质都有自己独特的指纹图谱。
2.3 聚类结果
根据这 11对多态性引物的扩增结果,经NTSYS-pc聚类分析,得出品种(材料)间的聚类图(图1)。结果表明,各品种 (材料)间相似系数在0.68~0.93。其中以雪湖贡莲和马当莲的相似系数最大,为0.93,说明它们之间的遗传差异很小。在相似系数为0.79的位置,可将参试品种分为三大类。第Ⅰ类包括大部分野生莲品种和一个花莲品种(桌上莲);第Ⅱ类包括泰国野生莲Ⅳ、Ⅵ,传统子莲品种和一个子莲花莲兼用品种(星空牡丹);第Ⅲ类为花莲品种。

表2 11对SRAP引物对的多态性比较
另外,从聚类图上还可以看出,各种质材料的聚类结果与地理位置有一定的联系,来自同一个地方的种质除个别外(如泰国野生莲Ⅳ和Ⅵ),大部分都聚在了一起。这反映出同一个地方的品种材料的亲本可能相同或者相近,表明同一地区品种单一化程度较高。

表3 部分材料对引物EM3+ME6和EM10+ME11的扩增结果
3 小结与讨论
SRAP (seq uence-related amplified polymorphism,特征序列扩增多态性),以全基因组为靶标,通过设计的引物对ORFs(open reading frames)进行扩增,上游引物对外显子进行特异性扩增,下游引物对内含子区域、启动子区域进行特异扩增。与其他分子标记相比,该标记具有简便、稳定、中等产率的特点,是一种较理想的分子标记[4,5]。本研究利用11对SRAP标记基本区分了40份莲品种材料,证明了SRAP标记应用于莲种质资源鉴定的可行性。雪湖贡莲和马当莲相似系数为0.93,可能由于供试品种之间很相似,或是因为供试品种较多,而可用的SRAP引物有限,两者的特异性条带较少,使这两个品种未能很好地分开。
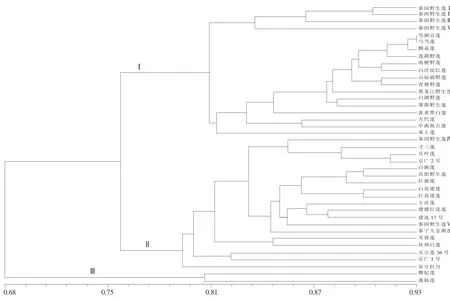
图1 40份莲品种材料的聚类树系分析
莲在我国栽培历史悠久,依据其利用目的已明显演化为三大类群:子莲、藕莲和花莲。在本研究中,40份莲品种材料也分为三大类,与园艺学分类系统相吻合[6],说明莲三大类型之间遗传分化十分明显。
本研究中来自全国各地的野莲材料表现为叶片厚实、花无或少量、不结实或瓣化,在农艺性状上与藕莲极其相似,生产上习惯将它们划分为藕莲品种,泰国野生莲Ⅰ、Ⅱ、Ⅲ和Ⅴ,在农艺性状上也与藕莲相似,在聚类图上得到了证实,与野生莲聚在了第Ⅰ类群。而泰国野生莲Ⅳ和Ⅵ在农艺性状上与子莲相似,表现为花多且结实,在聚类图中与传统子莲品种聚在第Ⅱ类群。这说明泰国野生莲品种之间遗传背景差异较大,在利用这些野生莲品种进行育种时,需根据育种目标的不同,选择不同的材料做亲本。同时也说明,分子鉴定应该与形态学分类相结合,两者互相佐证,才能在分类上更加准确可信。我们还发现本研究中的40份莲品种材料表现出很强的地域相似性,与前人的研究结果相似[7],这说明来自同一个地域内的品种分化程度低,遗传多样性不丰富,极易造成品种单一化,在生产上需多加注意。
在本研究中,古代莲和野生莲聚成一类,古代莲是考古出土的千年古莲子萌发繁育的后代,野生莲生长在自然湖泊中,很少有人工驯化积累的变异,而藕莲在长期的栽培过程中,只以产藕为驯化目的。野莲分布很广,前人一致认为野莲是现在栽培莲的共同祖先,根据聚类结果,野莲与藕莲的遗传差异相对较小,暗示藕莲可能是由野生莲直接驯化而来[8]。
[1]柯卫东,李峰,刘玉平,等.我国莲资源及育种研究综述(上)[J].长江蔬菜,2003(4):5-9.
[2]张行言.中国荷花新品种图志Ⅰ[M].北京:中国林业出版社,2011.
[3]瞿桢,魏英辉,李大威,等.莲品种资源的SRAP遗传多样性分析[J].氨基酸与生物资源,2008,30(3):21-25.
[4]Ferriol M,Picó B,Nuez F.Genetic diversity of some accession of Cucurbita maxima from Spain using RAPD and SRAP markers[J].Genetic Resources and Crop Evolution, 2003,50(3):227-238.
[5]Ferriol M,Picó B,Nuez F.Genetic diversity of germplasm collection of Cucurbita pepo using SRAP and AFLP markers[J].Theor Appl Genet,2003,107:271-282.
[6]倪学明.莲的品种分类研究[J].园艺学报,1983(3):207-210.
[7]郭宏波.莲属(Nelumbo)资源分类[D].武汉:华中农业大学,2003.
[8]向巧彦.莲种质资源遗传多样性研究及DNA指纹图谱构建[D].北京:北京林业大学,2008.
Genetic Diversity Analysis ofNelumboAccessions Based on SRAP Markers
OUYANG Dongmei1,LIU Huang1,XU Jinxing2,LIU Chunhua2,ZOU Dongwang1, YANG Liangbo2,TANG Jiping1,XIE Keqiang1
(1.Guangchang Industrial Development Bureau of White Lotus,Jiangxi 344900;2.Guangchang White Lotus Research Institute)
40Nelumboaccessions were analyzed with SRAP markers to estimate their genetic diversity.184 bands were obtained from 11 pairs of selected primers,and 127 bands were polymorphic,with the polymorphic rate of 69.0%,which indicated that the genetic diversity ofNelumbowas very rich.The results showed that all the materials could be divided into 3 groups,which was similar to the results of the traditional morphological classification by horticultural characteristics. Regional similarity was observed among the 40 materials,and the materials from the same region had low differentiation degree and poor genetic diversity,which easily resulted in the simplification of theNelumboaccessions.
Nelumbo;SRAP;Genetic diversity analysis
10.3865/j.issn.1001-3547.2012.16.010
国家公益性行业(水生蔬菜)科研专项子项目(200903017-05)资助
欧阳冬梅(1983-),女,研究生,助理农艺师,主要从事子莲育种
谢克强,通信作者,E-mail:xiekeqiang@126.com
2012-07-08
