重组大肠杆菌产番茄红素的研究进展
2012-02-02李燕飞徐志南
李燕飞,黄 磊,王 普,徐志南
(1.浙江工业大学药学院,浙江杭州310032;2.浙江大学化学工程与生物工程学系生物工程研究所,浙江杭州310027)
番茄红素是一种重要的天然植物色素,分子式为C40H56,是由11个共轭双键和2个非共轭双键组成的多不饱和脂溶性烃类化合物,在自然界中主要存在于西瓜、番茄等蔬菜水果中[1]。番茄红素具有很强的抗氧化能力,能够通过接受电子激发态的能量,使单线态氧的能量转移到番茄红素中,从而淬灭单线态氧和清除自由基,防止DNA受到氧化破坏,减轻细胞损伤和预防癌症的发生。此外,通过降低血清脂质过氧化和低密度脂蛋白的氧化,番茄红素还能减轻脂肪肝病变程度,防止或延缓动脉粥样硬化的形成[2,3],近年来其相关产品的研发已成为国际上新药研究的热点。到目前为止,番茄红素生产方法主要有天然提取法、化学合成法和发酵法三种。天然提取法主要是以西红柿为原料通过萃取的方法获得番茄红素,但由于原料含量太低导致生产成本居高不下,且生产受季节影响明显,无法满足市场需求。化学合成法虽然成本较低,但由于双键立体选择性难以控制致使产物不可控,且使用的化学试剂在产品中会有不同程度的残留。相比之下,微生物发酵法具有工艺简单、生产周期短、不受季节气候影响、成本低等优点,成为番茄红素最为理想的生产方法。迄今为止,能够用于生产番茄红素的微生物主要有红色细菌、三孢布拉氏霉菌、小球藻以及基因工程菌,特别是随着转基因技术的迅速发展,利用重组大肠杆菌生产番茄红素已成为研究的热点。
1 番茄红素的生物合成途径
1.1 番茄红素在植物、真菌和细菌中的生物合成途径 番茄红素是类胡萝卜素生物合成途径中的一个重要的中间产物,研究表明所有的类胡萝卜素都是由类异戊二烯化合物或萜类化合物合成的。在植物细胞液和真菌中,番茄红素通过甲羟戊酸途径(MVA途径)合成:最初3个乙酰辅酶A分子通过失去1个分子的羧基缩合成3-甲基-3,5-二甲基戊酸(MVA),MVA通过两步激酶反应生成甲羟戊酸-5-磷酸(MVAP)和甲羟戊酸-5-焦磷酸(MVAPP),MVAPP最后通过脱羧反应生成异戊烯焦磷酸(IPP),IPP在IPP异构酶作用下异构化形成3,3-二甲基丙烯焦磷酸酯(DMAPP),DMAPP再与3个分子的 IPP作用缩合依次生成牻牛儿焦磷酸(GPP)、法尼基焦磷酸(FPP)和牻牛儿基牻牛儿焦磷酸(GGPP),两个分子GGPP在八氢番茄红素合成酶(PSY)作用下聚合产生第一个无色类胡萝卜素—八氢番茄红素。八氢番茄红素经过连续的脱氢去饱和生成第一个有色类胡萝卜素—番茄红素。在细菌和植物质体中番茄红素是通过MEP途径合成的:由糖酵解途径合成的甘油醛-3-磷酸(G3P)和丙酮酸(Pyr)通过1-脱氧-D-木酮糖-5-磷酸合酶(DXS)催化缩合生成1-脱氧-D-木酮糖-5-磷酸(DXP),DXP由1-脱氧-D-木酮糖-5-磷酸还原异构酶(DXR)催化生成2-C-甲基-D-赤藓糖-4-磷酸(MEP),MEP再经过多步反应最后生成异戊烯焦磷酸(IPP)和二甲酰基焦磷酸(DMAPP)[4-6]。具体代谢途径如图1所示:
1.2 番茄红素在重组大肠杆菌中的生物合成途径 和其他微生物相比,大肠杆菌具有生长快、遗传背景清楚、基因操作方便等优点,已成为番茄红素生产的首选菌株。大肠杆菌本身不能合成番茄红素,但TCA循环的中间体如苹果酸、草酰乙酸等可以形成类胡萝卜素和脂类物质的骨架结构。大肠杆菌细胞内含有的成分如多萜醇、苯醌等化合物和番茄红素一样也都是由共同的前体IPP转化而来。野生型大肠杆菌只能将IPP转化成GGPP,而缺乏必要的酶将GGPP转化成番茄红素。研究表明只要将三个番茄红素合成酶的基因(crtE、crtB和crtI)导入大肠杆菌就可以最终建立番茄红素的生物合成途径(图2)[7~9]。其中crtE编码GGPP合酶,引入crtE可以增加FPP向GGPP的转化;crtB编码八氢番茄红素合成酶,可将GGPP转化成八氢番茄红素;而crtI编码八氢番茄红素脱氢酶,能促使八氢番茄红素向番茄红素转化。
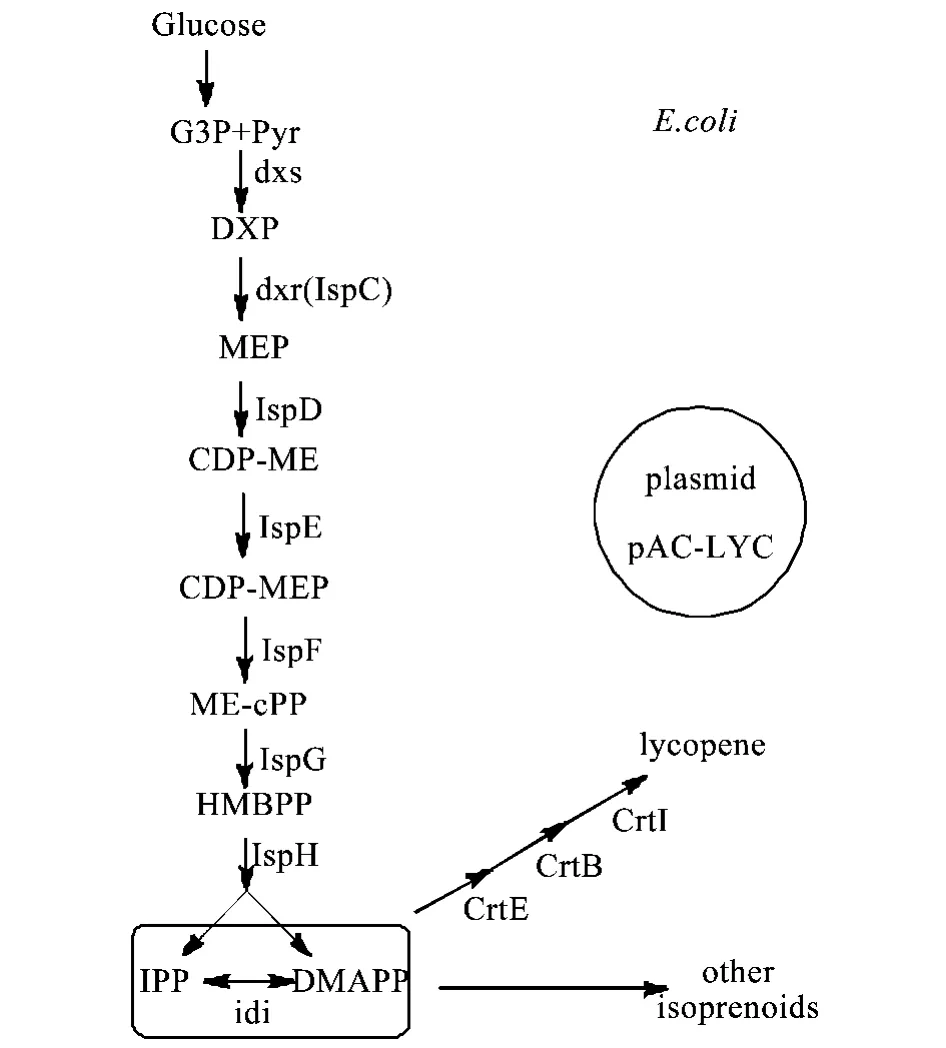
图2 番茄红素在大肠杆菌中的生物合成途径
CDP-ME:4-二磷酸-2C-甲基-D-赤藓糖;CDPMEP:4-二磷酸-2C-甲基-D-赤藓糖-2-磷酸;ME-cPP:2C-甲基-D-赤藓糖-2,4-环二磷酸;HMBPP:4-羟基-3-甲基-2-丁烯基-4-二磷酸;IspC-IspH:异戊烯焦磷酸(IPP)和二甲酰基焦磷酸(DMAPP)的合成酶;plasmid(pAC-LYC):含有crtE、crtB和crtI基因的质粒;other isoprenoids:其他类异戊二烯。
2 利用代谢工程构建重组大肠杆菌生产番茄红素
为达到利用大肠杆菌高效生产番茄红素的目的,需要对番茄红素的代谢途径进行改造和优化。一般先要找到影响目标代谢流的关键基因,利用基因工程的方法使其过量表达,对其进行抑制或敲除等,但由于番茄红素的合成涉及菌体内的多步酶催化反应,所以往往需要对多个基因同时进行操作。重组工程菌构建完成后,还可通过微生物培养工程的系统优化进一步提高番茄红素的生产能力。利用代谢工程和发酵工程的方法提高番茄红素产量一般有以下几种方法:①同源基因的原位取代;②代谢旁路基因的敲除;③代谢途径关键基因的过量表达;④多基因的协同调控;⑤工程菌培养条件的优化。
2.1 同源基因的原位取代 同源基因是来自同一物种或不同但相关物种的、在进化过程中源于共同祖先的基因,是在进化过程中特定基因在不同物种间的转移和重排所产生的结果,这种基因演化是微生物进化的强大推动力。选取高酶活同源基因对代谢途径中的关键基因进行原位取代是代谢工程研究中的常用手段。
大肠杆菌引入crtE、crtB和crtI三个关键基因后使得代谢流导向番茄红素的合成,而这三个基因来源的不同会对番茄红素的产量产生明显影响。Ju-Eun Kim等[10]发现当crt基因来源于Pantoea agglomerans时,番茄红素产量可以达到27 mg·L-1,而当crt基因来源于Pantoea ananatis时,番茄红素的产量只有12 mg·L-1。研究表明,番茄红素产量的差异主要是crtE基因引起的,crtE编码的GGPP合酶可以增加FPP向GGPP的转化,是番茄红素合成途径的限速酶。对比发现,Pantoea agglomerans来源的crtE基因具有更高的活性,有利于提高番茄红素的产量。IPP和它的同分异构体DMAPP是番茄红素合成的重要前体,idi编码的IPP异构酶有助于维持IPP和DMAPP之间的代谢平衡,在番茄红素合成途径中扮演着重要角色。Albermann等[11]用来源于地衣芽孢杆菌(Bacillus licheniformis)的idi基因替换大肠杆菌的idi基因,可以提高重组菌产番茄红素的能力。来源于大肠杆菌和地衣芽孢杆菌idi基因的大小分别为549 bp和1 050 bp,编码的氨基酸序列相似度只有6%。这说明了异种同源基因在进化过程中发生歧化从而导致他们在结构和功能上某种程度的差异,选用合适来源的酶对代谢网络的优化具有重要的意义。
2.2 代谢途径旁路基因的敲除 通过旁路代谢途径基因的敲除增加目的产物的代谢流量是代谢工程的常用方法之一。微生物体内代谢途径非常复杂,除目标产物的合成途径外,许多旁路代谢流对微生物的生长都有重要的作用。如果为了加强产物合成代谢流而任意敲除旁路代谢基因往往会影响菌体的正常生理功能导致产物产量的下降,所以找到合适的待敲除基因是整个旁路代谢网络优化的首要任务。
Stephanopoulos课题组[12]根据重组大肠杆菌整体代谢网络,建立了一个代谢通量计算模型,通过对全基因组范围的代谢通量平衡分析和基因敲除模拟计算,筛选出了6个目标基因靶点(gdhA、gpmA、gpmB、aceE、fdhF和talB)。经过单基因,双基因和三基因组合敲除实验,发现敲除gdhA、aceE和fdhF三个基因能使番茄红素的产量提高近40%。其中,gdhA编码谷氨酸脱氢酶,催化α-酮戊二酸和NADPH转化为谷氨酸,敲除gdhA可以增加前体NADPH的供应量。aceE编码丙酮酸脱氢酶,敲除aceE能阻止丙酮酸流向乙酰辅酶A的合成,从而增加前体物质丙酮酸的量。fdhF编码甲醛脱氢酶,催化甲醛转化为CO2和H2O,敲除fdhF可减少丙酮酸流向甲醛及其他副产物,结果同样是增加丙酮酸的量而促进番茄红素的合成。翁志明等[13]选择BW25113作为基因敲除的起始菌,利用Red重组技术分别对gdhA、fdhF和aceE这三个基因进行敲除,结果表明基因型为 ΔgdhAΔaceE和ΔgdhAΔfdhFΔaceE的两种菌株合成番茄红素的能力最强,番茄红素产量分别为3.8 mg·g DCW-1和3.6 mg·gDCW-1。ackA-pta可以催化乙酰辅酶A转化为乙酸,Vadali等[14]发现敲除ackA-pta基因可以将番茄红素产量提高约45%。ackA-pta基因的敲除一方面可以降低乙酰辅酶A的消耗,从而减弱了丙酮酸流向乙酰辅酶A的代谢流;另一方面还可以降低乙酸的积累可以减弱其对菌体生长的不利影响。在此基础上通过链霉菌甲羟戊酸途径的引入,提高了番茄红素前体IPP的含量,进一步将番茄红素的产量提高了1倍。
2.3 代谢途径关键基因的过量表达 番茄红素代谢途径中一些前体物质的积累有利于番茄红素的合成,这些物质可以通过相关酶的过量表达来增加其含量,以满足番茄红素高效合成的需求。idi基因编码代谢途径中的IPP异构化酶,能够促进IPP向DMAPP的转化,从而调节这两个代谢中间产物的平衡。而ispA是中间产物FPP合成酶基因,idi或ispA的过量表达或两者同时过量表达均可使番茄红素产量得到提高,其中同时过量表达idi和ispA时番茄红素产量比原始菌提高了69%[15]。Kang等[16]利用鸟枪法分析大肠杆菌基因组发现过量表达dxs、crl、rpoS和appY均可以促进重组大肠杆菌番茄红素的合成。其中dxs是MEP途径的关键酶基因,其过量表达使得流向IPP的代谢流增强,直接提高关键前体IPP的量;crl编码细胞表面纤维蛋白,增大外膜空间有利于番茄红素的储存;rpoS编码σs因子,σs因子为RNA聚合酶的亚基,起到调节基因转录的作用;appY编码能量代谢调控蛋白。研究发现,过量表达dxs或appY时番茄红素产量可达到2.8 mg·gDCW-1,而当dxs分别同其他三个基因一起过量表达时,均比单独过量表达 dxs效果好。此外,Jin和Stephanopoulos[17]通过基因组合分析又发现过量表达yjiD、ycgW、yhbL、purDH和yggT也可以提高番茄红素的产量。研究将旁路基因的敲除和限速基因的过量表达结合运用,筛选到一株基因型为WS140(T5p-dxs,T5p-idi,rrnBp-yjiD-ycgW,ΔgdhAΔaceEΔfdhF,pACLYC)的菌株,产番茄红素的量是原始菌的4倍,达到了16 mg·gDCW-1。用生物信息学的方法线性规划细胞生长速率和产物生成速率可显示能增强目标产物产量的代谢流,Choi等[18]在此基础上结合基因敲除和启动子替换等方法构建了一株重组菌WLGB-RPP (pTrcDx-idi-mdh,pLyc184)将番茄红素的产量提高到了283 mg·L-1。
2.4 代谢途径多基因协同调控 微生物的代谢网络十分复杂,目标产物的最终合成需要经过一系列的转化,而各步反应间的代谢流存在相互影响和制约的作用,因此只对若干个基因进行操作将难以达到全面发掘与提升产物合成能力的目标。Wang等[19]以番茄红素生物合成途径的20种关键基因(dxs、dxr、ispD、ispE、ispG、ispH、idi、ispA、appY、rpoS、crl、elbA、elbB、yjiD、purH、rnlA、yggT、ycgZ、ymgA、ariR)为研究目标,建立了一种能对大量基因的转录水平进行同时调控的方法(MAGE)。针对这20种基因的SD序列进行研究,首先化学合成了转录效率不等的SD序列基因库,通过电转化方法将基因库导入大肠杆菌感受态细胞,利用同源重组技术随机替换目标SD序列,实现了对全部基因转录水平的整体调控。该实验在3天之内就筛选到了一株番茄红素产量提升5倍的突变株,分析表明由于该突变株的dxs和idi基因的转录水平得到了增强使得番茄红素产量大幅提高。
2.5 通过培养条件的优化提高产量 不同的生长环境对微生物菌体的生长和代谢产物的生成都会产生影响。番茄红素为脂溶性物质,一般都积累在细胞膜上,这在一定程度上改变了细胞表面的形状,使细胞产生聚集现象。表面活性剂可以有效地减少表面张力,使细胞分散,从而改善菌体的生长状况。Yoon等[20]通过在培养基中添加表面活性剂吐温80将番茄红素的产量提高了27%。碳源在大肠杆菌中的运输方式主要有两种:磷酸烯醇式丙酮酸(PEP)依赖型和非磷酸烯醇式丙酮酸依赖型。Jongrae Kim等[21]研究了三种磷酸烯醇式丙酮酸(PEP)依赖型附加碳源(葡萄糖、甘露糖和麦芽糖)和三种非磷酸烯醇式丙酮酸依赖型碳源(甘油、半乳糖和乳糖)对番茄红素产量的影响,结果表明甘油为附加碳源时的产量最高,是葡萄糖做为附加碳源时的30倍。Yeong-Su Kim等[22]也对工程菌E.coli K12的培养基碳源进行优化,发现在含有甘油的培养基中加入10 mg·L-1葡萄糖和7.5 mg·L-1的L-阿拉伯糖发酵34 h时可使番茄红素的产量提高到1 350 mg·L-1,比未优化培养基提高了7.1倍。除了碳源,培养温度对番茄红素产量也会产生影响,有报道相对低的温度(20~30℃)培养更有利于番茄红素的积累[14,23,24],但是此时菌体的生长却相对缓慢,会因为发酵时间延长而影响经济效益。运用温度梯度培养可以有效地解决这一矛盾,Kim等[25]将工程菌E.coli DH5α先在37℃下培养,20 h后转移到25℃,在此期间补加甘油作为碳源和阿拉伯糖作为诱导剂,60 h后番茄红素产量可达到260 mg· L-1,比一直在25℃条件下培养时产量提高了20%。
3 总结与展望
利用基因工程菌发酵生产番茄红素已经成为研究的重要发展方向,通过对番茄红素代谢网络的全面研究,可以有效地指导生物合成途径的遗传修饰,从而提高番茄红素的生产能力。但和番茄红素合成相关的酶大多分布于脂膜上,在非膜环境下很容易失去活性,使得番茄红素代谢网络的酶学研究变得十分困难。不仅如此,番茄红素的最终合成涉及一系列步骤的反应,各步反应之间相互联系,互为影响,如何使之更好的协同作用来提高番茄红素的合成能力也是一个亟待解决的关键问题。因此,对基因工程菌的基因转录组和蛋白质组进行分析,测定番茄红素生产过程中多基因转录变化情况,研究关键基因群及其作用机理,在深入理解代谢网络构成和调控机制的基础上,通过有效协同抑制各代谢支流及增强目标代谢流,进一步提高番茄红素产量,将成为利用微生物发酵法生产番茄红素的新的研究方向之一。
[1] Qu MY,Zhou Z,Chen CH,et al.Lycopene protects against trimethyltin-induced neurotoxicity in primary cultured rat hippocampal neurons by inhibiting the mitochondrial apoptotic pathway[J].Neurochem Int,2011,59(8):1095- 1103.
[2] Lu R,Dan H,Wu R,et al.Lycopene:Features and potential significance in the oral cancer and precancerous lesions[J].J Oral Pathol Med,2011,40(5):361-368.
[3] Omoni AO,Aluko RE.The anticarcinogenic and antiatherogenic effects of lycopene:a review[J].Trends Food Sci Tech,2005,16(8):344-350.
[4] Hirschberg J.Carotenoid biosynthesis in flowering plants[J].Curr Opin Plant Boil,2001,4(3):210-218.
[5] Ronen G,Cohen M,Zamir D,et al.Regulation of carotenoid biosynthesis during tomato fruit development:expression of the gene for lycopene epsilon-cyclase is downregulated during ripening and is elevated in the mutant Delta[J].Plant J,1999,17(4):341-351.
[6] Rohmer M.The discovery of a mevalonate-independent pathway for isoprenoid biosynthesis in bacteria,algae and higher plants[J].Nat Prod Rep,1999,16(5):565-574.
[7] Rohdich F,Hecht S,Gärtner K,et al.Studies on the nonmevalonate terpene biosynthetic pathway:metabolic role of IspH(LytB)protein[J].P Natl Acad Sci,2002,99(3): 1158-1163.
[8] Cunningham FX,Gantt E.A portfolio of plasmids for identification and analysis of carotenoid pathway enzymes:A-donis aestivalis as a case study[J].Photosynth Res,2007,92(2):245-259.
[9] Harada H,Misawa N.Novel approaches and achievements in biosynthesis of functional isoprenoids in Escherichia coli[J].Appl Microbiol Biotechnol,2009,84(6):1021-1031.
[10]Yoon SH,Kim JE,Lee SH,et al.Engineering the lycopene synthetic pathway in E.coli by comparison of the carotenoid genes of Pantoea agglomerans and Pantoea ananatis[J].Appl Microbiol Biotechnol,2007,74(1):131-139.
[11]Rad SA,Zahiri HS,Noghabi KA,et al.Type 2 IDI performs better than type 1 for improving lycopene production in metabolically engineered E.coli strains[J].World J Microbiol Biotechnol,2012,28(1):313-321.
[12]Alper H,Jin YS,Moxley JF,et al.Identifying gene targets for the metabolic engineering of lycopene biosynthesis in Escherichia coli[J].Metab Eng,2005,7(3):155-164.
[13]Liu JZ,Weng ZM,Wang Y,et al.Metabolic engineering based on systems biology for chemicals production[J].Front Biol,2009,4(3):260-265.
[14]Vadali RV,Fu Y,Bennett GN,et al.Enhanced lycopene productivity by manipulation of carbon flow to isopentenyl diphosphate in Escherichia coli[J].Biotechnol Prog,2005,21(5):1558-1561.
[15]卢韦.大肠杆菌基因工程菌生产番茄红素研究[D].北京:首都师范大学,2007:47-52.
[16]Kang MJ,Lee YM,Yoon SH,et al.Identification of genes affecting lycopene accumulation in Escherichia coli using a shot-gun method[J].Biotechnol Bioeng,2005,91(5): 636-642.
[17]Jin YS,Stephanopoulos G.Multi-dimensional gene target search for improving lycopene biosynthesis in Escherichia coli[J].Metab Eng,2007,9(4):337-347.
[18]Choi HS,Lee SY,Kim TY,et al.In silico identification of gene amplification targets for improvement of lycopeneProduction[J].Appl Environ Microbiol,2010,76(10):3097-3105.
[19]Wang HH,Isaacs FJ,Carr PA,et al.Programming cells by multiplex genome engineering and accelerated evolution[J].Nat Lett,2009,460(7257):894-898.
[20]Yoon SH,Lee YM,Kim JE,et al.Enhanced lycopene production in Escherichia coli engineered to synthesize isopentenyl diphosphate and dimethylallyl diphosphate from mevalonate[J].Biotechnol Bioeng,2006,94(6):1025-1032.
[21]Kim J,Kong MK,Lee SY,et al.Carbon sources-dependent carotenoid production in metabolically engineered Escherichia coli[J].World J Microbiol Biotechnol,2010,26(12):2231-2239.
[22]Kim YS,Lee JH,Kim NH,et al.Increase of lycopene production by supplementing auxiliary carbon sources in metabolically engineered Escherichia coli[J].Appl Microbiol Biotechnol,2011,90(2):489-497.
[23]Lee PC,Mijts BN,Schmidt-Dannert C.Investigation of factors influencing production of the monocyclic carotenoid torulene in metabolically engineered Escherichia coli[J].Appl Microbiol Biotechnol,2004,65(5):538-546.
[24]Kim SW,Kim JB,Jung WH,et al.Over-production of beta-carotene from metabolically engineered Escherichia coli[J].Biotechnol Lett,2006,28(12):897-904.
[25]Kim SW,Kim JB,Ryu JM,et al.High-level production of lycopene in metabolically engineered E.coli[J].Process Biochem,2009,44(8):899-905.
