转座子PiggyBac在哺乳动物中的应用
2012-02-01马元武张连峰
马元武,张连峰
(中国医学科学院,北京协和医学院,医学实验动物研究所,卫生部人类疾病比较医学重点实验室,国家中医药管理局人类疾病动物模型三级实验室,北京 100021)
转座元件或转座子是指宿主基因组内发生基因座位置的改变的遗传元件。当对转座子进行基因工程改造,使之携带一段外源基因,当在转座过程中插入基因的内部或是邻近位置,会对被插入基因造成基因功能的突变或失活,用以研究基因的功能[1]。转座子一般由转座酶和转座酶所识别一段DNA序列 (转座识别序列)所组成,转座序列能够在转座酶的作用下完成转座反应。按转座过程是否经过RNA中间体而分为I型转座子和II型转座子。I型转座子(或称逆转座子),需要先被转录成RNA,然后在由转座子编码的转座酶的作用下逆转录成DNA插入宿主基因组内。II型转座子 (或称为DNA转座子),II型转座子转座过程中不经过RNA中间体,在由转座子编码转座酶的作用下,通过复制或直接剪切的方式将转座子DNA转座入宿主基因组内(图1)。
基因工程手段对DNA转座子进行改造,将转座子的转座识别序列和转座酶相分离,并在转座序列内插入一段外源感兴趣的基因,当在有转座酶存在的条件下识别两端的转座序列介导转座反应的发生,已 经 应 用 于 黑 腹 果 蝇 (Drosophila melanogaster),线虫 (Caenorhabditis elegans)等多种模式生物的基因功能研究。虽然人类基因组内存在着大量转座序列(约45%)[2-3],但由于进化过程中的累计突变造成其活性丧失,因此,有活性的转座子在哺乳动物中的应用一直受到限制。直到DNA转座子Sleeping Beauty(SB)才使得DNA转座子作为一种遗传工程工具应用于哺乳动物基因功能的研究。SB来源于鲑鱼 (salmanoid)基因组,是一种的Tc1/mariner样转座子,Zoltan Ivics等通过分子重建的方式使其重新获得了活性,并能在人和小鼠细胞内转座[4]。新型转座子的逐渐被发现和应用,目前发现在哺乳动物中有活性的转座子有:1) hAT样转座子;2)Tcl样转座子包括 Sleeping Beauty和Frog Prince;3)PiggyBac转座子家族。这三类转座子中,转座活性最高的为 Sleeping Beauty和PiggyBac,对这两种转座子的特性和转座活性比较[5],发现甘蓝蠖度尺蛾 (cabbage looper moth Trichoplusia ni)来源的转座子 PiggyBac相对具有更高的转座活性,转座过程中更高的携带外源基因插入宿主基因组的能力,并能够实现转座子及所携带外源基因的完全转座,不会在供体位置造成任何遗传物质的改变,这些特性使其在转基因动物、癌基因发现、iPS研究及基因治疗研究方面前景诱人。
1 PiggyBac转座子
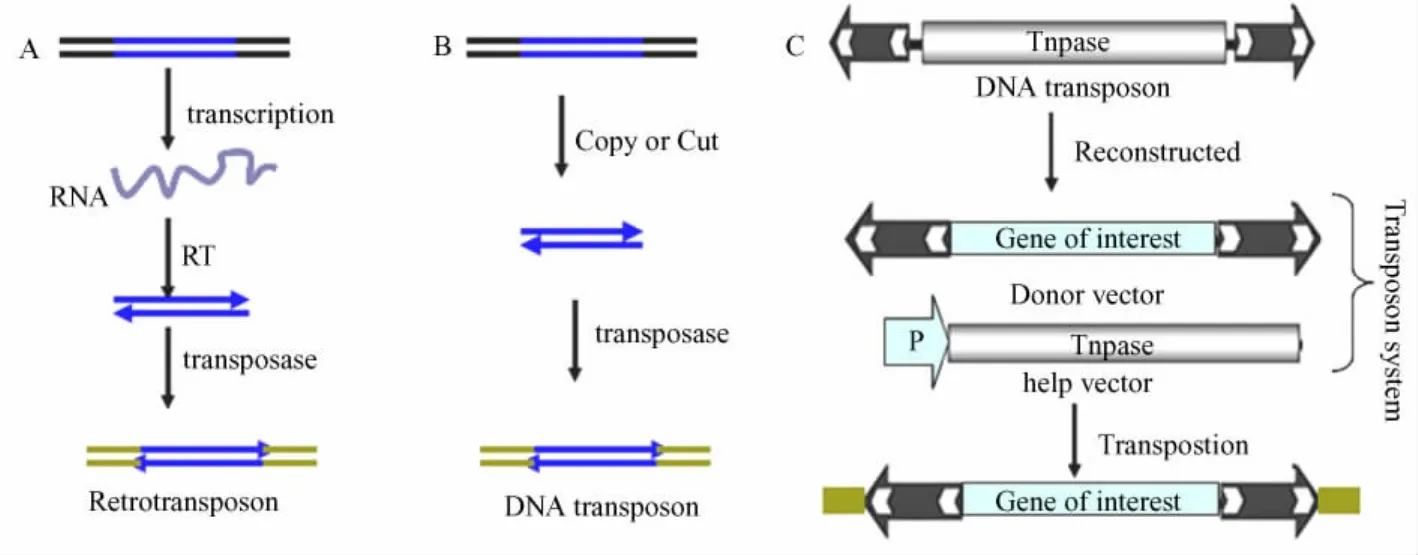
图1 转座的分子机制。DNA线性表示,RNA曲线表示Fig.1 Molecular mechanisms of transposition.DNA is represented by straight lines.RNA transcripts are wavy lines
PiggyBac分离于甘蓝蠖度尺蛾[6],最初能够在果蝇、红腋斑粉蝶 (red flour beetle Tribolium Castaneum)及昆虫中转座,明显不同于其它已发现的转座子类型,命名PiggyBac转座子家族,简称PB[7-8].PB元件是2472bp的转座子序列拥有13 bp的末端反向重复序列以及编码594个氨基酸的转座酶序列组成[9],识别TTAA序列采用剪切、黏贴的DNA转座模式(类似于Sleeping Beauty(SB)转座子)[10]。
PiggyBac样转座元件 (PiggyBac-like elements,PLE)普遍存在多物种内,很少具有转座活性以及完整的转座子结构,目前成功分离出了如 PBGD(1,3,5)、AyPLE和 AaPLE等多种PLE[11]。经过密码子优化过的mPB以及hyPB(mPB基础上的进一步优化获得具有更高的转座活性),是目前发现的在哺乳动物中转座活性最高的 DNA转座子[5,12-13]。转座识别序列和转座酶的分离可以使得转座子发展为二元系统,见图1 C。提高了转座安全性、携带外源基因的能力并增强了可操作性。转座反应需要转座子序列和转座酶同时存在。当对转座识别序列内部插入一段外源基因时,可实现目的基因向宿主基因组的靶向运输[1]。PB转座系统作为一种有效的基因工程操作工具由于具有的高容量[14]、无痕迹转座[9,13],安全性强等优势应用在大规模研究基因的插入突变及基因功能研究、癌基因/抑癌基因的发现、iPS研究以及基因治疗研究方面。
2 哺乳动物中的应用
在不影响转座效率的情况下,PB转座系统可高效携带十几kb外源DNA靶序列转座,用于靶基因的转基因研究[12]。此外,当所携带的外源基因含有基因捕获元件时,PB在转座过程中,可捕获基因组内基因造成基因的插入突变或失活,通过转座子的末端重复序列可非常容易定位插入基因的位点,用以研究基因组内大规模基因功能。由于转座造成基因的插入失活,研究基因与生长,基因与发育,以及基因与基因之间协调作用关系[3,15]。生物体任何功能的行使并不是单个基因所能完成的,他需要多个基因的协同调节表达共同来实现。PB的高转座效率可造成多个基因功能的丧失,可研究多基因与生长、发育以及基因之间的关系[3,15]。
2.1 基因突变
传统的基因突变方式主要是通过粒子射线、化学诱变剂以及病毒感染来实现的。虽然这些方法能够有效的产生突变,获得感兴趣的表型变化,但是后续突变位点的确定却是费事耗力[16]。几十年来,由于缺少有效插入突变工具,哺乳动物基因组的大规模遗传学分析受到了很大的限制[17]。
近年来,DNA转座子SB和PB的发现和功能重塑,能够在哺乳动物体内有效转座,使得这一局面得以改变。DNA转座子通常由末端重复序列以及位于中间的转座酶基因构成[1],转座反应需要转座酶识别序列和转座酶同时存在。转座酶缺失的转座元件在有转座酶瞬时表达的情况下就能够发生转座反应。转座序列和转座酶可以分离的特性,可构建转座识别序列和转座酶相分离的二元转座系统[15]。在转座识别序列之间插入我们感兴趣的基因 (如功能基因、基因捕获组件等),当在转座酶存在下,可以有效的将外源基因运输进入宿主基因组内。当外源基因为基因捕获组件时,可以在转座进入基因组时有效捕获基因组内基因,实现基因的插入突变或失活。只要存在转座酶即可以实现转座,不同于以往的基因打靶方式,这一方法可实现基因组的大规模基因功能遗传学分析[18]。
2.2 癌基因的发现
癌变的发生是受到遗传和环境等多种因素影响所致,体细胞基因的突变在肿瘤的发生和发展中起至关重要的作用。在过去的几十年里,已经应用插入突变的方式在小鼠,大鼠等动物模型中简单进行癌变的遗传学分析[19]。然而癌变的发生是一个复杂的过程,正常细胞的癌变可能需要多种突变基因的逐渐积累才能完成。有效基因突变工具的发现和应用对于研究癌变发生的遗传学因素具有重要的意义。PB转座子是目前发现的转座活性最高的DNA转座子,在转座时能够实现转座子序列的完全剪切和转座,在供体位置处不留任何转座痕迹,通过转座子的末端重复序列,可以通过接头连接的PCR非常方便的对转座子在基因组中的插入位点进行定位,基于这些特点,使得PB作为一种非常有潜力的遗传工程工具,可以应用于哺乳动物大规模筛选癌基因的研究[20],目前已经构建出多种用于癌基因发现的转座系统载体如 T2/Onc,T2/Onc2和T2/Onc3等[19,21],为癌基因的发现提供了基础。通过对插入位点的确定,可以研究基因组特定位点与肿瘤发生之间的关系[22-23]。
2.3 基因治疗与iPS
基因治疗,要求目的基因能够特异、有效的导入靶细胞内,并长时间表达,产生尽量少的毒副作用。目前大部分基因治疗都采用病毒载体。然而,病毒载体的构建和准备不仅费时、费力同时也存在许多其它问题,如逆病毒载体 (retroviruses)能够长效的实现目的基因的表达,但由于偏向于插入常染色体的转录起始位点和基因的内含子内,容易造成基因的插入失活,易至癌变[24];腺病毒载体(adenoviruses)可以将DNA运输到处于分裂或静止期的细胞内,由于不插入基因内部,不造成致癌突变,但不能保证目的基因的长效、稳定表达,且易引起宿主的免疫应答[25];另一类为腺联病毒载体(adeno-associated viruses),降低了宿主的免疫应答反应稳定的表达,但目的基因的大小却受到限制[26]。新型的非病毒载体的应用就显得非常重要。
SB和PB转座子的发现,使得转座子作为一种有用的基因工程工具应用于哺乳动物,SB和PB的“剪切-黏贴”的DNA转座模式,相对低的费用、低的免疫反应性、高的转座效率、高容量,安全性强等特点[27],使其在基因治疗方面具有非常大的应用潜力。自SB于1997年发现以来,人们一直致力于将其应用于基因治疗,并取得了许多可喜的成果,如可以用于小鼠遗传缺陷造成的基因病的治疗研究,如 I型酪氨酸血症[28],A型[29]和 B型[30]血友病,亨廷顿病[31]等遗传型疾病的治疗研究。随着研究的不断深入,通过对 SB转座系统引入嵌合体抗原受体来介导T细胞的重定向特异性,已经用于 I/II期临床试验[27]。然而,SB的相对较低的转座活性,以及明显的供体附近位置转座偏好性,限制了其在基因治疗方面的应用。PB转座系统的高转座活性和无痕迹转座,使得PB成为一种比SB转座系统具有更广阔应用前景的DNA转座子[32-34]。
SB和PB虽然在基因治疗方面潜力巨大,但它们的安全性仍是基因治疗的所考虑重点,非特异性整合会导致所不期望的后果[35]。这一不足正在通过在转座酶中嵌合入 DNA结合结构域 (DNA binding domain,DBD)进行弥补,前期的实验表明,这一改进能有效提高转座系统的转座效率和特异性[36-38]。此外,只要有转座酶的存在就会有转座的发生,也使得转座相对不稳定,Johann Urschitz等通过对转座系统进行改进,获得了一种不依赖于辅助载体的转座系统,转座发生后会造成转座酶的失活,解决这一问题[39]。
iPS的显著优势是具有类似于胚胎干细胞的多潜能性,并且可以用于诱导分化成为内胚层、中胚层和外胚层用以移植来治疗退行性或遗传性疾病[40]。通常情况下,鼠或人的成纤维细胞的重新编程为多潜能干细胞,需要四种转录因子 c-Myc,Klf4,Oct4 and Sox2[41]的辅助.以往应用于iPS研究辅助因子的运输工具主要为病毒载体,但是病毒载体存在着类似于基因治疗方面的缺陷,很难排除由于外源基因导入基因组所造成的改变对 iPS的影响。
PB转座系统在转座后不留任何足迹,在由 PB转座系统提供四种转录因子 c-Myc,K lf4,Oct4和Sox2完成 iPS后,可以通过提供 PB转座酶在基因组内消除载体。由于 PB的无痕迹转座,不会造成遗传物质的改变,可以用于探索体细胞的重新编程时,消除外源载体因素所可能造成的影响,为iPS的研究提供了有力的工具[39,42-43]。
3 小结
PB转座子是目前在哺乳动物中转座活性最高的转座子,并且可以携带高达十几Kb的外源基因转座而不影响其效率,使其在哺乳动物的转基因、癌基因的大规模筛选、基因治疗方面具有巨大的应用潜力。此外,PB的无痕迹转座对产生无转基因、无遗传物质改变的iPS研究也具有非常重要的意义和应用前景。然而,转座子的非特异性转座,也影响了其在哺乳动物中的应用。因此,DNA转座子虽然具有诱人的应用前景,但应用的道路却很漫长。
[1]马元武,张连峰.转座子Sleeping Beauty和PiggyBac[J].中国生物化学与分子生物学报,2010,26(9):783-787.
[2]Lander ES,Linton LM,Birren B,et al.Initial sequencing and analysis of the human genome[J].Nature,2001,409(6822): 860-921.
[3]Horie K,Kuroiwa A,Ikawa M,et al.Efficient chromosomal transposition of a Tc1/mariner-like transposon Sleeping Beauty in mice[J].Proc Natl Acad Sci U S A,2001,98(16):9191 -9196.
[4]Ivics Z, Hackett PB, Plasterk RH, et al. Molecular reconstruction of Sleeping Beauty,a Tc1-like transposon from fish,and its transposition in human cells[J].Cell,1997,91 (4):501-510.
[5]Liang Q,Kong J,Stalker J,et al.Chromosomalmobilization and reintegration of Sleeping Beauty and PiggyBac transposons[J].Genesis,2009,47(6):404-408.
[6]Fraser MJ,Ciszczon T,Elick T,et al.Precise excision of TTAA-specific lepidopteran transposons piggyBac(IFP2)and tagalong(TFP3)from the baculovirus genome in cell lines from two species of Lepidoptera[J].Insect Mol Biol,1996,5(2): 141-151.
[7]Lorenzen MD,Berghammer AJ,Brown SJ,et al.piggyBacmediated germ line transformation in the beetle Tribolium castaneum[J].Insect Mol Biol,2003,12(5):433-440.
[8]Bonin CPMann RS.A piggyBac transposon gene trap for the analysis of gene expression and function in Drosophila[J].Genetics,2004,167(4):1801-1811.
[9]Ding S,Wu X,Li G,et al.Efficient transposition of the piggyBac(PB)transposon in mammalian cells and mice[J].Cell,2005,122(3):473-483.
[10]Wu SC,Meir YJ,Coates CJ,et al.piggyBac is a flexible and highly active transposon as compared to sleeping beauty,Tol2,and Mos1 in mammalian cells[J].Proc Natl Acad Sci U S A,2006,103(41):15008-15013.
[11]Wu M,Sun Z,Luo G,et al.Cloning and characterization of piggyBac-like elements in lepidopteran insects[J].Genetica,2011,139(1):149-154.
[12]Yusa K,Zhou L,Li MA,et al.A hyperactive piggyBac transposase formammalian applications[J].Proc Natl Acad Sci U S A,2011,108(4):1531-1536.
[13]Wang W,Lin C,Lu D,et al.Chromosomal transposition of PiggyBac in mouse embryonic stem cells[J].Proc Natl Acad Sci U S A,2008,105(27):9290-9295.
[14]Lacoste A,Berenshteyn FBrivanlou AH.An efficient and reversible transposable system for gene delivery and lineagespecific differentiation in human embryonic stem cells[J].Cell Stem Cell,2009,5(3):332-342.
[15]Geurts AM,W ilber A,Carlson CM,et al.Conditional gene expression in the mouse using a Sleeping Beauty gene-trap transposon[J].BMC Biotechnol,2006,6(30):1-15.
[16]Kong J,Wang F,Brenton JD,et al.Slingshot:a PiggyBac based transposon system for tamoxifen-inducible ‘ selfinactivating’insertional mutagenesis[J].Nucleic Acids Res,2010,38(18):e173.
[17]Starr TKLargaespada DA.Cancer gene discovery using the Sleeping Beauty transposon[J].Cell Cycle,2005,4(12): 1744-1748.
[18]Largaespada DA.Transposon mutagenesis in mice[J].Methods Mol Biol,2009,530:379-390.
[19]Dupuy AJ.Transposon-based screens for cancer gene discovery in mouse models[J].Semin Cancer Biol,2010,20(4):261 -268.
[20]Rad R,Rad L, Wang W, et al. PiggyBac transposon mutagenesis:a tool for cancer gene discovery in mice[J].Science,2010,330(6007):1104-1107.
[21]Collier LS,Carlson CM,Ravimohan S,et al.Cancer gene discovery in solid tumours using transposon-based somatic mutagenesis in themouse[J].Nature,2005,436(7048):272 -276.
[22]Su Q,Prosser HM,Campos LS,et al.A DNA transposon-based approach to validate oncogenic mutations in themouse[J].Proc Natl Acad Sci U S A,2008,105(50):19904-19909.
[23]Mattison J,Kool J,Uren AG,et al.Novel candidate cancer genes identified by a large-scale cross-species comparative oncogenom ics approach[J].Cancer Res,2010,70(3):883 -895.
[24]Laufs S,Nagy KZ,Giordano FA,et al.Insertion of retroviral vectors in NOD/SCID repopulating human peripheral blood progenitor cells occurs preferentially in the vicinity of transcription start regions and in introns[J].Mol Ther,2004,10(5):874-881.
[25]McCaffrey AP,Fawcett P,Nakai H,et al.The host response to adenovirus,helper-dependent adenovirus,and adeno-associated virus in mouse liver[J].Mol Ther,2008,16(5):931-941.
[26]Claeys Bouuaert CChalmers RM.Gene therapy vectors:the prospects and potentials of the cut-and-paste transposons[J].Genetica,2010,138(5):473-484.
[27]Hackett PB,Largaespada DACooper LJ.A transposon and transposase system for human application[J].Mol Ther,2010,18(4):674-683.
[28]Montini E,Held PK,Noll M,et al.In vivo correction ofmurine tyrosinemia type I by DNA-mediated transposition[J].Mol Ther,2002,6(6):759-769.
[29]Liu L,Mah CFletcher BS.Sustained FVIII expression and phenotypic correction of hemophilia A in neonatalmice using an endothelial-targeted sleeping beauty transposon[J].Mol Ther,2006,13(5):1006-1015.
[30]Ohlfest JR,Frandsen JL,Fritz S,et al.Phenotypic correction and long-term expression of factor VIII in hemophilic mice by immunotolerization and nonviral gene transfer using the Sleeping Beauty transposon system[J].Blood,2005,105(7):2691-2698.
[31]Chen ZJ,Kren BT,Wong PY,et al.Sleeping Beauty-mediated down-regulation of huntingtin expression by RNA interference[J].Biochem Biophys Res Commun,2005,329(2):646 -652.
[32]Feschotte C.The piggyBac transposon holds promise for human gene therapy[J].Proc Natl Acad Sci U SA,2006,103(41): 14981-14982.
[33]Wilson MHGeorge AL,Jr.Designing and testing chimeric zinc finger transposases[J].Methods Mol Biol,2010,649 353 -363.
[34]Nakazawa Y,Huye LE,Dotti G,et al.Optimization of the PiggyBac transposon system for the sustained genetic modification of human T lymphocytes[J].J Immunother,2009,32(8): 826-836.
[35]Yant SR,Wu X,Huang Y,et al.High-resolution genome-wide mapping of transposon integration in mammals[J].Mol Cell Biol,2005,25(6):2085-2094.
[36]Wilson MH,Kaminski JMGeorge AL,Jr.Functional zinc finger/ sleeping beauty transposase chimeras exhibit attenuated overproduction inhibition[J].FEBS Lett,2005,579(27): 6205-6209.
[37]Ivics Z,Katzer A,Stuwe EE,et al.Targeted Sleeping Beauty transposition in human cells[J].Mol Ther,2007,15(6): 1137-1144.
[38]Maragathavally KJ,Kaminski JMCoates CJ.Chimeric Mos1 and piggyBac transposases result in site-directed integration[J].Faseb J,2006,20(11):1880-1882.
[39]Urschitz J,Kawasumi M,Owens J,et al.Helper-independent piggyBac plasmids for gene delivery approaches:strategies for avoiding potential genotoxic effects[J].Proc Natl Acad Sci U S A,2010,107(18):8117-8122.
[40]Hanna J,Wernig M,Markoulaki S,et al.Treatment of sickle cell anemiamousemodelwith iPS cells generated from autologous skin[J].Science,2007,318(5858):1920-1923.
[41]Takahashi KYamanaka S.Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors[J].Cell,2006,126(4):663-676.
[42]Desponts CDing S.Using small molecules to improve generation of induced pluripotent stem cells from somatic cells[J].Methods Mol Biol,2010,636 207-218.
[43]Vanden Driessche T,Ivics Z,Izsvak Z,et al.Emerging potential of transposons for gene therapy and generation of induced pluripotent stem cells[J].Blood,2009,114(8): 1461-1468.
