pBIFC-VN173-CXCR4 和pBIFC-VC155-NT21MP 真核表达质粒的构建及其在活细胞内的作用
2012-01-22高艳军杨清玲陈昌杰丁勇兴
高艳军,杨清玲,陈昌杰,丁勇兴
(1.蚌埠医学院临床检验诊断学实验中心;2.蚌埠医学院生化与分子生物学教研室;3.蚌埠市中心医院普外肿瘤科,安徽 蚌埠 233030)
肿瘤转移是影响癌症患者预后的重要因素之一,而转移是由多个因素参与和多个步骤发展的过程。研究发现,趋化因子在为肿瘤细胞提供迁移信号,促进其定向转移并准确定位中可能扮演着至关重要的角色。CXCR4(chemokine receptor 4、CXCR4 和CD184)属G蛋白偶联受体家族,与其配体基质细胞衍生因子1α(stromal cell-derived factor 1、SDF-1α 和CXCL12)构成的生物学轴,可诱导肿瘤细胞趋化、转移等诸多生物学效应[1-2]。CXCR4 抑制性多肽(N-terminal 21-mer peptide,NT21MP)是趋化因子巨噬细胞炎症蛋白Ⅱ(viral macrophage inflammatory protein-Ⅱ,vMIP-Ⅱ)N端部分氨基酸序列,可以选择性结合CXCR4 受体,通过竞争性结合CXCR4 活性位点降低CXCL12/CXCR4 信号轴的作用,进而抑制乳腺癌细胞的趋化和转移[3-4]。双分子荧光互补分析技术(bimolecular fluorescence complementation,BiFC)是近年来出现的一种新技术,该技术可直观地判断目的蛋白在活细胞内的定位及相互作用[5]。本研究通过构建NT21MP 和CXCR4 的BiFC 真核表达载体,求证二者在活细胞内是否可以相互结合从而启动NT21MP 的生物学抑制效应。
1 材料与方法
1.1 材料 乳腺癌细胞株SKBR3、非洲绿猴肾成纤维细胞株COS-7、大肠杆菌DH5α 菌及JM109 菌为本实验室保存;pBiFC-VC155 载体、pBiFC-VN173 以 及 pBIFC-VN173-JUN 和pBIFC-VC155-FOS 阳性对照载体由美国普渡大学胡长灯教授惠赠;TRIzol 试剂购自Gibco 公司;焦碳酸二乙酯(DEPC)购自Sigma 公司;cDNA 合成试剂盒购自Fermentas 公司;CXCR4引物、NT21MP 均由上海捷瑞公司合成;TaKaRa Ex Taq® PCR 扩增试剂盒、限制性内切酶、PMDTM-18T 载体、T4 DNA 连接酶及胶回收试剂盒均购自TaKaRa 公司;质粒抽提试剂盒购自北京天根生化科技有限公司;退火缓冲液购自碧云天生物制剂公司。
1.2 方法
1.2.1 vMIPⅡN 端21 肽(NT21MP)目的基因的获取 根据vMIP-ⅡN 端的氨基酸序列(Genbank:AAB62642)以及编码vMIP-ⅡN 的开放阅读框K4 基因(Genbank:U93872.2:21495 -21779)的核苷酸的序列确定了编码vMIP-ⅡN 末端21个氨基酸肽的核苷酸序列作为目的基因。目的基因的两端分别引入限制性内切核酸酶KpnⅠ和EcoRⅠ的靶位序列,将设计好的2 条核苷酸序列由捷瑞生物工程有限公司合成。其核苷酸序列:A:5'-AATTCAGAT GCTGGGAGCGTCCTGGCATAGACCGGACAAGT GCTGTCTCGGTTACCAGAAAAGACCATTACCAG GTAC-3';B:5'-CTGGTAATGGTCTTTTCTGGTA ACCGAGACAGCACTTGTCCGGTCTATGCCAGGA CGCTCCCAGCATCTG-3'。首先用消毒的双蒸水溶解2 条单链核苷酸至100μmol/L;然后,在PCR 反应管中分别加入双蒸水15μl、退火缓冲液5μl、寡核苷酸A 2.5μl 和寡核苷酸B 2.5μl,应用PCR 仪进行退火反应。PCR 反应条件:95℃变性2 min;每30 s 下降1℃,降至25℃;4℃保存。
1.2.2 pBiFC-VC155-NT21MP 的构建与鉴定将pBiFC-VC155 质粒载体用限制性核酸内切酶KpnⅠ和EcoRⅠ在37℃双酶切,电泳分离后纯化回收。将已纯化的酶切质粒与NT21MP的退火产物于16℃连接过夜,转化至DH5α 感受态细胞。使用含氨苄西林(100 mg/L)的LB琼脂平板筛选阳性克隆。抽提质粒,以KpnⅠ和EcoRⅠ酶切鉴定,酶切鉴定符合的克隆送生工生物工程(上海)有限公司进行测序,测序结果与GenBank 中的数据进行分析。
1.2.3 人类CXCR4 基因的PCR 扩增 根据Genbank 中收录的CXCR4 的mRNA 序列(NM_003467.2),设计PCR 上游引物:5'-GGAATT CAATGGAGGGGATCAGTATATACAC-3',设 有EcoRⅠ酶切位点;下游引物:5'-GGGGTACCA AGCTGGAGTGAAAACTTGAAGACTCAGAC-3',设有Kpn I 酶切位点。应用Trizol 法提取乳腺癌SKBR3 细胞株中总RNA,逆转录为cDNA 后以该cDNA 为模板应用上述引物进行PCR 扩增。PCR 反应条件:95℃预变性5 min,94℃变性45 s,66℃退火45 s,72℃延伸90 s,35个循环,72℃延伸10 min,将PCR 产物进行琼脂糖凝胶电泳,观察结果。
1.2.4 人类CXCR4 基因的T 载体连接和鉴定PCR 产物进行琼脂糖凝胶电泳鉴定后,割胶回收目的片段,用试剂盒纯化后与PMD18-T 载体连接(按试剂盒说明书进行)。CaCl2法转化至JM109 感受态细胞,筛选氨苄青霉素抗性克隆,抽提纯化质粒,重组质粒PMD-18-T-CXCR4经KpnⅠ和EcoRⅠ双酶切鉴定后,委托生工生物工程(上海)有限公司进行测序验证。
1.2.5 重组载体(pBiFC-VN173-CXCR4)的构建与鉴定 测序正确的CXCR4-T 载体和pBiFC-VN173 载体分别应用EcoRⅠ和KpnⅠ进行酶切。将酶切后的目的基因与载体经过割胶回收纯化后经T4 DNA 连接酶于16℃连接过夜,并转化感受态细胞DH5α 菌,使用含氨苄西林(100 mg/L)的LB 琼脂平板筛选阳性克隆,抽提纯化质粒,以KpnⅠ和EcoRⅠ酶切鉴定,酶切鉴定符合的克隆委托生工生物工程(上海)有限公司测序。
1.2.6 重组载体共转染COS-7 细胞 将COS-7 细胞接种于24 孔板,每孔105个细胞,于500μl 无抗生素DMEM 完全培养液(含10%胎牛血清)中37℃、5%CO2培养箱培养,待细胞融合度达80%至90%且细胞生长状态良好时进行转染。转染分组:①pBIFC-VN173 和pBIFCVC155 共转染组;②pBIFC-VN173-CXCR4 转染组;③pBIFC-VC155-NT21MP 转染组;④pBIFCVN173-CXCR4 和pBIFC-VC155-NT21MP 共转染组;⑤pBIFC-VN173-JUN 和pBIFC-VC155-FOS 共转染组(阳性对照)。取2 支EP 管,分别标记为A 和B,各加入50μl 减血清培养液(OPTI-MEM),A 管加入脂质体2μl,混匀后室温静置5 min,B 管加入质粒1μg(共转染组每种0.5μg),然后混匀A 和B 液,室温静置20 min,最后加入24 孔板中,37℃、5%CO2培养箱培养24 h,荧光显微镜下观察。
2 结果
2.1 BiFC 重组质粒pBiFC-VC155-NT21MP 测序鉴定 将NT21MP 的DNA 与双酶切后的pBiFC-VC155 质粒连接转化,挑取阳性单克隆摇菌,取菌液送上海生工进行基因测序。测序结果显示,在测序序列的343 至420 位碱基为目的插入片段,与目的DNA 比对完全正确(图1),其插入方向及阅读框架正确,证明BiFC 重组质粒pBiFC-VC155-NT21MP 构建成功。
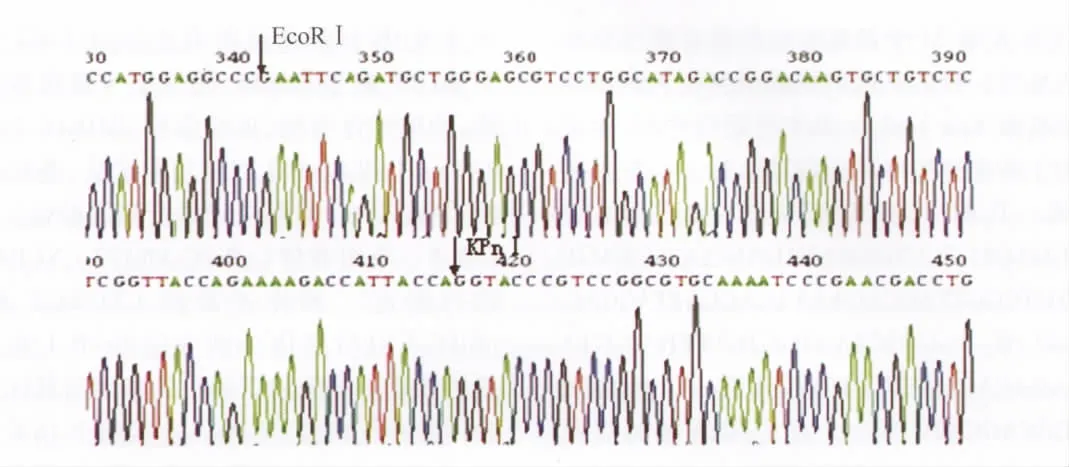
图1 pBiFC-VC155-NT21MP 重组质粒部分测序图Fig.1 Partial sequencing results of recombinant plasmid pBiFC-VC155-NT21MP
2.2 RT-PCR 对人类CXCR4 扩增 从乳腺癌SKBR3细胞中提取总RNA,逆转录为cDNA 后应用上述CXCR4 引物扩增后行琼脂糖凝胶电泳。在约1 074 bp处有一明显条带,与预期的CXCR4 基因片段相符(图2)。

图2 CXCR4 基因PCR 产物琼脂糖电泳Fig.2 Agarose gel electrophoresis analysis of CXCR4 gene PCR products
2.3 PMD18-T-CXCR4 载体和BiFC 重组载体pBiFC-VN173-CXCR4 构建与鉴定 CXCR4 的PCR 产物经胶回收纯化后与PMD18-T 载体连接转化后,挑取单克隆摇菌,抽提质粒。应用EcoRⅠ和KpnⅠ双酶切鉴定重组质粒,切出约1 074 bp 和2.8 kb 大小的片段,同预期的长度和CXCR4 的PCR 结果均一致(图3);同样方法双酶切鉴定重组质粒pBiFC-VN173-CXCR4,琼脂糖电泳显示约1 074 bp 和5.2 kb 的2 片段(图4)。两重组质粒测序结果与GenBank 中收录的人CXCR4 的编码序列除+621 bp(ATG为1)处单个碱基突变(但不影响氨基酸的编码)外,其余完全相同(图5),且插入方向及阅读框架正确,说明BiFC 重组真核表达载体pBiFC-VN173-CXCR4 构建成功。

图3 PMD-18T-CXCR4 重组质粒限制性酶切鉴定Fig.3 The restriction enzyme digestion analysis of PMD-18T-CXCR4 recombi-nant vector
2.4 CXCR4 和NT21MP 相互结合的BiFC 分析 用不同的质粒单独或共转染COS-7 细胞24 h后荧光显微镜观察BiFC 信号(绿色)。图6 示,在pBIFC-VN173 和pBIFC-VC155 共转染组、pBIFC-VN173-CXCR4 转染组及 pBIFCVC155-NT21MP 转染组中均检测不到BiFC 信号的表达,而在pBIFC-VN173-CXCR4/pBIFCVC155-NT21MP 共转染组B4和C4中,可以检测到BiFC 信号,且绿色荧光主要定位于胞质内。同样在阳性对照B5和C5中也可以观察到荧光。

图4 pBiFC-VN173-CXCR4 重组质粒限制性酶切Fig.4 The restriction enzyme digestion of pBiFC-VN173-CXCR4 recombinant vector
3 讨论
趋化因子受体CXCR4(CD184)由Feng等[6]于1996年发现的,其是一种结构高度保守的7次跨膜受体,是G 蛋白偶联受体超家族的成员。基质细胞来源因子1α(stromal cellderived factor 1,SDF-1α)又名CXCL12,其唯一受体为CXCR4[7]。研究表明CXCL12/CXCR4生物学轴对肿瘤的影响是多方面的,在多种肿瘤的发生、发展和转移过程中发挥重要作用[2,8-12]。
vMIP-ⅡN 端21 肽(NT21MP)是本室根据vMIP-ⅡN 端与CXCR4 结合的高度保守区,通过固相合成技术制备的活性多肽,能够与CXCR4 特异性结合,在活性测定和动物实验中均表现出高效的CXCR4 抑制活性[3-4]。前期研究发现,NT21MP 能够抑制乳腺癌细胞的趋化及增殖,并诱导乳腺癌细胞的凋亡,其可能作用机制是NT21MP 通过与CXCL12 竞争结合于CXCR4,进而抑制了CXCL12/CXCR4 的AKT、ERK1/2等多种胞内信号途径的激活[4,13]。但这些实验研究都是建立在体外实验和通过动物实验间接测定的基础之上,目前还没有在活细胞内直接观察NT21MP 与CXCR4 相互作用的实验报道。

图5 pBiFC-VN173-CXCR4 重组质粒部分测序图Fig.5 Partial sequencing results of recombinant plasmid pBiFC-VC155-NT21MP

图6 NT21MP 和CXCR4 在COS-7 细胞中的双分子荧光互补分析图Fig.6 BiFC analysis of NT21MP and CXCR4 in recombinant plasmid-transfected COS-7 cells
BiFC 技术是近年来由胡长灯教授首创的一种直观、快速地判断目的蛋白在细胞中定位和相互作用的新技术[14]。Geoffrey 等[15]研究发现,在GFP 的2个β 片层之间的环结构上至少有10个位点可以插入外源蛋白而不影响GFP 的荧光活性。BiFC 技术正是利用该荧光蛋白家族的这一特性,在环结构的特异位点将荧光蛋白切成N 端片段和C 端片段两部分后,再由一小段氨基酸序列linker 分别与目标蛋白连接。将重组好的基因构建入载体,共转染细胞,两段重组基因在细胞中融合表达。若目标蛋白间存在相互作用,通过linker 牵引荧光蛋白的N 端片段与C 端片段就能够相互靠近,形成荧光蛋白生色团重新发出荧光。该技术的关键是将拟观察的目的蛋白的基因定向插入至相应的真核表达载体。将构建好的质粒共转染细胞后,在荧光显微镜下检测是否有互补荧光产生,就可以推断出两目标蛋白是否发生了相互作用[16]。
本实验成功构建了vMIP-ⅡN 端21 肽(NT21MP)和CXCR4 基因的BIFC 真核表达载体。在构建的质粒中,NT21MP 和CXCR4 编码序列分别与Venus C 端155-238 位氨基酸残基和N 端1-172 位氨基酸残基的编码序列片段连接。共转染二者的重组质粒,若NT21MP 和CXCR4 可以结合,二者在胞内相互作用即可以使荧光蛋白重新恢复荧光活性。本实验应用空BiFC 载体以及重组载体单独和共转染至COS-7 细胞,转染24h 后通过荧光显微镜观察,在空载体共转染以及重组质粒单独转染组中无BiFC 信 号,在 pBIFC-VN173-CXCR4/pBIFCVC155-NT21MP 共转染组以及阳性对照组中则检测到绿色荧光信号(图6)。利用BiFC 技术观察到二者在活细胞中的相互作用,这充分说明二者在胞内可以相互结合;同时,为我们前期研究NT21MP 通过竞争性的结合受体CXCR4上的活性位点,CXCL12/CXCR4 生物轴的胞内信号无法进行有效激活,而进一步抑制乳腺癌细胞株的趋化、增殖以及促进凋亡的作用机制提供了一个有力的佐证。
肿瘤分子靶向治疗是一种研究发现的新兴肿瘤治疗方法之一。CXCR4 参与多种疾病发生发展,为多种疾病的重要治疗靶点,如艾滋病、肿瘤和风湿性关节炎等,尤其在肿瘤转移方面[17]。NT21MP 作为化学合成肽能够直接作用于CXCR4 靶点,在本实验中得到了充分的证明,为NT21MP 向生物活性制剂靶向治疗肿瘤转移的研究方向的设想提供了理论支持。传统的研究方法存在一定的缺陷:如免疫荧光的透化过程会对细胞造成损伤,而免疫共沉淀技术要求必须破碎细胞。它们都无法做到在活细胞生理条件下实时、直接、快速地对细胞内NT21MP 与CXCR4 间相互作用进行动态研究。BiFC 技术能弥补这一缺陷,并在研究转录因子间的相互作用及其亚细胞定位[14],寻找G 蛋白可能存在相互作用的β 和γ 亚基[18],利用多色荧光互补技术同时检测多种蛋白质间的相互作用[19]以及检测泛素化途径[20]等相关生命科学领域中均得到具体应用。
总之,本实验成功构建了应用于BiFC 技术的真核表达载体,并且在活细胞内检测到NT21MP 与CXCR4 的互相结合,为进一步研究NT21MP 通过CXCL12/CXCR4 轴发挥的受体拮抗作用奠定了良好的基础。
[1]GASSMANN P,HAIER J,SCHLUTER K,et al.CXCR4 regulates the early extravasation of metastatic tumor cells in vivo [J].Neoplasia,2009,11:651-661.
[2]KRYCZEK I,WEI S,KELLER E,et al.Stromaderived factor (SDF-1/CXCL12)and human tumor pathogenesis [J].Am J Physiol Cell Physiol,2007,292(3):C987-C995.
[3]YANG Qing-ling,LI Cheng-hua,DING Yong-xing(杨清玲,李成华,丁勇兴).Inhibitory effect of polypeptide on CXCR4 related metastasis of bresat cancer cell line [J].Cancerous Distortion Mutation(癌变 畸变 突变),2008,20(2):89-92.(in Chinese)
[4]YANG Q L,DING Y X,CHEN C J,et al.Suppression of murine breast cancer metastasis by selective inhibition of CXCR4 by synthetic polypeptide derived from viral macrophage inflammatory proteinⅡ[J].Huazhong Univ Sci Technol[Med Sci],2011,31(4):497-503.
[5]FAN Jin-yu,CUI Zong-qiang,ZHANG Xian-en(樊晋宇,崔宗强,张先恩).Bimolecular fluorescence complementation technique [J].China Biochemistry and Molecular Biology(中国生化学与分子生物学报),2008,24(8):767-774.(in Chinese)
[6]KLENERMAN P,ZINKERNAGEL RM.Original antigenic sin impairs cytotoxic T lymphocyte responses to viruses bearing variant epitopes[J].Nature,1998,394(6692):482-485.
[7]BARBERO S,BONAVIA R,BAJETTO A,et al.Stromal cell-derived factor 1alpha stimulates human glioblastoma cell growth through the activation of both extracellular signal-regulated kinases 1/2 and Akt[J].Cancer Res,2003,63(8):1969-1974.
[8]FISCHER L,KORFEL A,PFEIFFER S,et al.CXCL13 and CXCL12 in central nervous system lymphoma patients [J].Clin Cancer Res,2009,15:5968-5973.
[9]SUNG B,JHURANI S,AHN KS,et al.Zerumbone down-regulates chemokine receptor CXCR4 expression leading to inhibition of CXCL12-induced invasion of breast and pancreatic tumor cells[J].Cancer Res,2008,68(21):8938-8944.
[10]SAUVE K,LEPAGE J,SANCHEZ M,et al.Positive feed-back activation of estrogen receptors by the CXCL12-4 pathway [J].Cancer Res,2009,69(14):5793-5800.
[11]SINGH S,BOND V C,POWELL M,et al.CXCR4-120-IIIB interactions induce caspase-mediated apoptosis of prostate cancer cells and inhibit tumor growth[J].Mol Cancer Ther,2009,8(1):178-184.
[12]KWONG J,KULBE H,WONG D,et al.An antagonist of the chemokine receptor CXCR4 induces mitotic catastrophe in ovarian cancer cells[J].Mol Cancer Ther,2009,8(7):1893-1905.
[13]YANG Qing-ling,YANG Zhi-feng,GAO Yan-jun(杨清玲,杨志峰,高艳军).Expression and purification of vMIP-Ⅱ N-terminal GST fusion protein and identification of its bioactivity [J].China Biotechnology(中国生物工程杂志),2010,30(9):80-86.(in Chinese)
[14]HU C D,CHINENOV Y,KERPPOLA T K.Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation [J].Mol Cell,2002,9(4):789-798.
[15]GEOFFREY S B,DAVID A Z,ROGER Y.Circular permutation and receptor insertion within green fluorescent proteins [J].Proc Natl Acad Sci USA,1999,96(20):11241-11246.
[16]KERPPOLA T K.Visualization of molecular interactions using bimolecular fluorescence complementation analysis:characteristics of protein fragment complementation [J].Chem Soc Rev,2009,38(10):2876-2886.
[17]KHAN A,GREENMAN J,ARCHIBALD S J.Small molecule CXCR4 chemokine receptor antagonists:developing drug candidates [J].Curr Med Chem,2007,14(21):2257-2277.
[18]HYNES T R,TANG L N,MERVINE S M,et al.Visualization of G protein β γ dimers using bimolecular fluorescence complementation demonstrates roles for both β and γ in subcellular targeting [J].J Biol Chem,2004,279(29):30279-30286.
[19]HU C D,KERPPOLA K T.Simultaneous visualization of multipleprotein interactions in living cells using multicolor fluorescence complementation analysis [J].Nature Biotechnology,2003,21(5):539-545.
[20]FANG D Y,KERPPOLA K T.Ubiquitin-mediated fluorescence complementation reveals that Jun ubiquitinated by Itch/AIP4 islocalized to lysosomes[J].Proc Natl Acad Sci USA,2004,101(41):14782-14787.
