辣椒抗黄瓜花叶病毒QTL分析
2011-08-07王立浩毛胜利张正海云兴福张宝玺
赵 娟 王立浩 毛胜利 张正海 云兴福 张宝玺*
(1中国农业科学院蔬菜花卉研究所,北京 100081;2内蒙古农业大学农学院,内蒙古呼和浩特010019)
近年来,黄瓜花叶病毒(Cucumber mosaic virus,CMV)对辣椒(Capsicumm annuumL.)的危害日益严重,其主要通过蚜虫进行传播,可引起辣椒系统花叶、矮化、皱缩,导致果实畸形,严重减产(Xu & Barnett,1983)。培育抗黄瓜花叶病毒的辣椒新品种是辣椒育种的目标之一(张宝玺 等,2005)。分子标记技术的成熟为辅助辣椒抗病育种提供了条件,可通过基因定位构建遗传图谱,在此基础上分离克隆抗病基因,进一步了解病害的发生机制(张丽英 等,2008)。
辣椒分子标记和数量遗传的研究近年逐步发展起来,国外研究者采用RFLP、RAPD、AFLP等分子标记技术得到了一些辣椒抗黄瓜花叶病毒的QTL位点(Caranta et al.,1997;Ben Chaim et al.,2001;Caranta et al.,2002),国内研究者在辣椒抗疫病(安静 等,2007)、辣椒红素含量(张芳芳 等,2010)的QTL定位方面取得一定进展。但关于辣椒抗黄瓜花叶病毒QTL定位的研究鲜见报道,本试验采用SRAP和SSR分子标记技术相结合,构建辣椒分子遗传图谱,针对辣椒抗黄瓜花叶病毒基因进行QTL定位分析,以期为今后辣椒抗黄瓜花叶病毒育种提供参考。
1 材料与方法
1.1 试验材料
供试辣椒品种 perennial,抗黄瓜花叶病毒,果实指形,朝天,味辣,从法国农业科学院引进;甜椒品种茄门,感黄瓜花叶病毒,果实灯笼形,主要园艺性状优良,来自中国农业科学院蔬菜花卉研究所。
试验于 2008年 3~11月在本所进行。采用抗病材料 perennial和感病材料茄门杂交的 F2为作图群体,共146株。F2自交得到F3,共146个株系,每个株系3次重复,每处理10株,共4 380株。
2008年3月播种双亲以及F2群体的146个单株;9月播种双亲以及F3株系,每个苗盘种植6行,每行10株,F3每个株系种植30株,以75-3-1为感病对照,进行室内人工接种抗病性鉴定。
1.2 分子标记筛选
采用CTAB小量法(Fulton et al.,1995)提取双亲、F2的146个单株叶片DNA。SRAP分子标记引物参考 Li和 Quiros(2001)、Riaz等(2001)、Lin等(2003)、Budak等(2004)、王刚等(2004)、雷剑和柳俊(2006)等。SRAP扩增反应体系参考 Li和 Quiros(2001)的方法。SSR分子标记引物参照Sol genomics network上公布的引物(http://solgenomics.net/),共150对(上海生工生物工程技术服务有限公司合成),利用亲本进行引物的筛选。SSR扩增反应体系参照Tam等(2005)的方法,扩增产物在4%变性聚丙烯酰胺凝胶中电泳、银染。将银染后晾干的胶板放置于白光灯箱上,统计多态性条带,与母本茄门带型相同记为a,与父本perennial带型相同记为b,具有共显性的带型记为h,由于各种原因造成的数据不清晰或缺失者记为“-”。在SRAP标记中,F2出现与母本不同的记为c,与父本不同的记为d。
1.3 抗病性鉴定
本试验采用的黄瓜花叶病毒为重花叶株系,该病毒由本所病毒真菌课题组提供。在防虫温室中,待幼苗 3~4片真叶时采用人工摩擦接种法(孙秀东 等,2008)进行接种。接种前,要保证幼苗的生长整齐一致。以75-3-1为感病对照。取已感染黄瓜花叶病毒的三生烟(Nicotiana tobacumcv.Samsun NN)鲜叶,按照鲜叶与0.03 mol·L-1的磷酸缓冲液(pH=8.0)1M∶5V混合。在冰浴中研磨成匀浆,离心过滤得到接种液,接种液现配现用。接种前在辣椒真叶上洒少许过600目筛金刚砂,然后用手指蘸取接种液轻轻摩擦叶面,接种第1、2真叶。接种后用水冲掉叶面上多余的汁液。接种后温度控制在25~30 ℃。第1次接种3 d后进行第2次接种,保证所有单株均接有该病毒。接种20 d后调查发病症状。
病情分级标准参照国家“八五”辣椒抗病育种攻关组的分级标准(李树德,1995):0级,无任何症状;1级,心叶明脉或接种叶急性小枯斑;3级,系统花叶或茎上产生坏死斑点;5级,系统重花叶、畸形或茎上产生坏死条斑;7级,多数叶片畸形、蕨叶、植株矮化或茎、枝和叶脉系统坏死;9级,植株严重矮化,停止生长或严重系统坏死,至全株死亡。群体抗病类型划分标准参考孙秀东等(2008)的方法,免疫(I):病情指数=0;高抗(HR):病情指0.1~5.0;抗病(R):病情指数5.1~20.0;耐病(MR):病情指数20.1~40.0;感病(S):病情指数大于40.0。

1.4 数据分析
根据孔繁玲(2006)的世代对比法计算F2群体的广义遗传力。结合SRAP和SSR分子标记在 F2上的多态性,采用 JoinMap3.0软件构建连锁遗传图谱,取 LOD>3.0。参照 Ben Chaim等(2001)的方法,将遗传图谱的数据结合辣椒抗黄瓜花叶病毒的接种鉴定结果,运用MapQTL4.0软件中的Permutation Test功能,计算并确定连锁群的LOD值;利用Composite Interval Mapping功能即复合区间作图法进行QTL分析,并分析QTL的加性效应,解释表型变异的贡献率。
2 结果与分析
2.1 SRAP、SSR分子标记多态性
筛选出有差异的SRAP引物426对,选择扩增多态性谱带较清晰,且在父母本间表现差异明显的SRAP引物35对,在F2群体上进行DNA多态性分析,得到83个多态性标记,平均每对引物可以扩增出 2条明显的差异谱带。其中引物Bm8F×K2R在 F2群体上部分单株的多态性见图1。
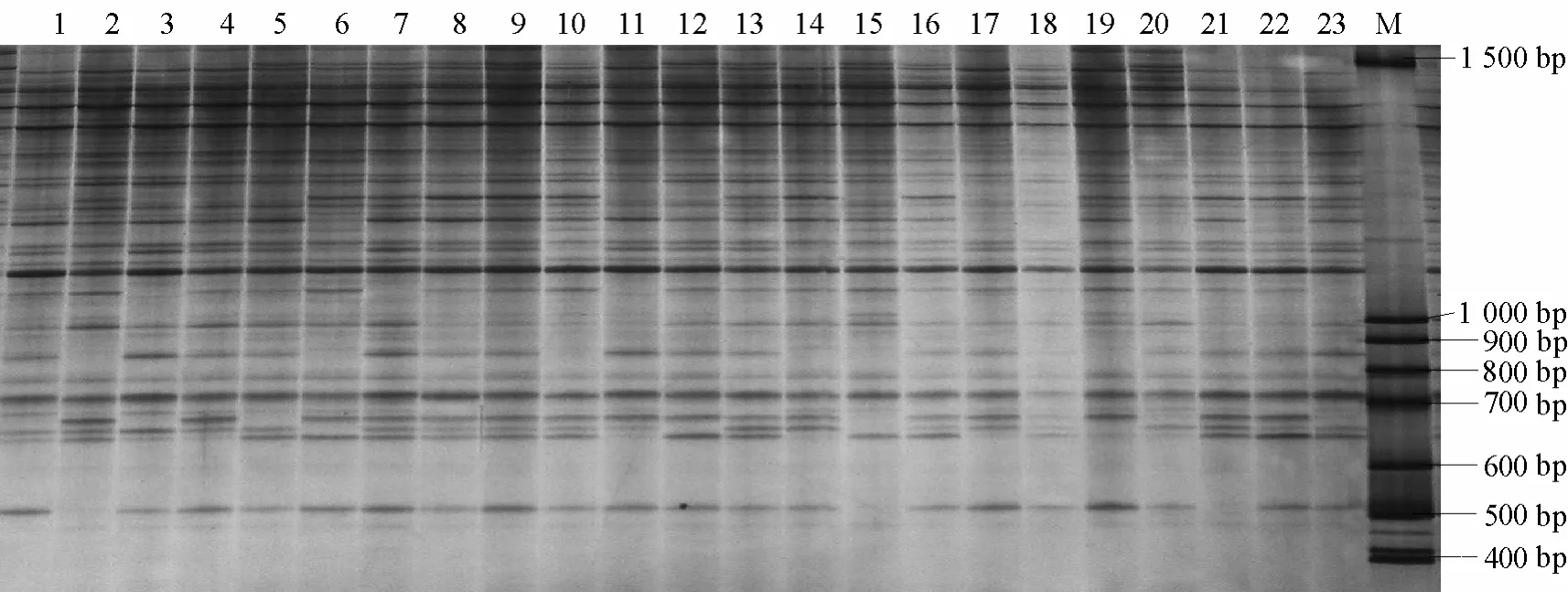
图1 引物Bm8F×K2R在F2群体上的多态性分析
筛选出有差异的SSR引物56对,选择扩增多态性谱带较清晰,且在父母本间表现差异明显的SSR引物36对,在F2群体上进行DNA多态性分析。其中引物SSR088在F2群体上部分单株的多态性见图2。
2.2 抗黄瓜花叶病毒鉴定结果
从 F3群体的表现来看,各株系间差异明显(图3)。父本perennial整体无明显感病症状,仅个别叶片上产生枯斑;母本茄门感病症状明显,出现花叶、矮化、条斑,部分单株甚至死亡;F1的感病症状介于亲本之间,有枯斑和花叶出现(图4)。
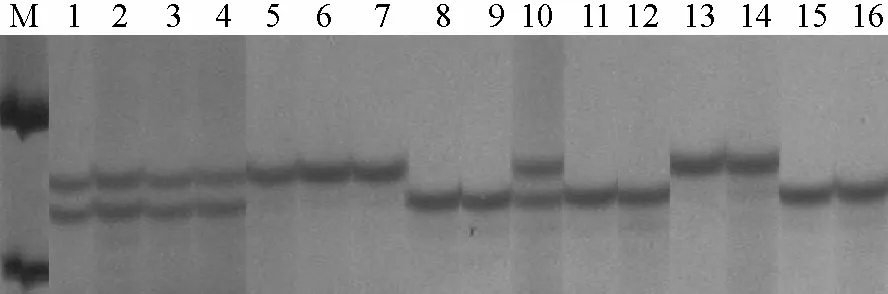
图2 引物SSR088在F2群体上的多态性分析

图3 F3群体接种黄瓜花叶病毒的发病情况

图4 父母本及F1接种黄瓜花叶病毒的发病情况
将F3群体抗病鉴定的病情指数取平均值来反映F2世代146个单株的抗病性(Ben Chaim et al.,2001),就是利用扩大接种鉴定的群体数量来提高鉴定F2抗性的准确性。经统计,F3群体中没有对黄瓜花叶病毒免疫的株系,病情指数在5.1~20.0之间的抗病(R)株系25个,病情指数在20.1~40.0之间的耐病(MR)株系109个,病情指数大于 40.0的感病(S)株系 12个。其中父本perennial的病情指数为4.44,是对CMV高抗的品种,而母本茄门和感病对照75-3-1的病情指数分别为51.11、59.84,均远大于40.0。
由图5可以看出,F3株系抗黄瓜花叶病毒病情指数分布呈正态分布,可用于数量性状的定位分析。
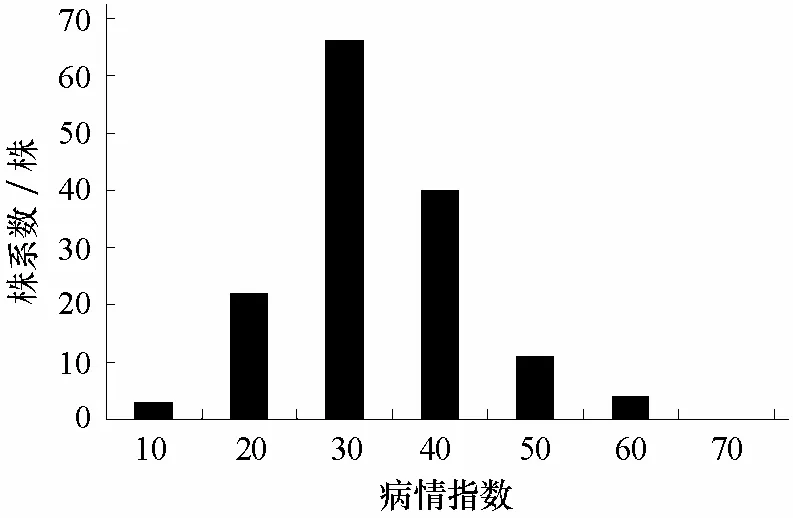
图5 F3株系抗黄瓜花叶病毒病情指数分布图
2.3 广义遗传力分析
如表1所示,辣椒抗黄瓜花叶病毒病的广义遗传力不高,仅为 45.47%,说明抗性可能受到多个基因控制,同时环境因素对黄瓜花叶病毒病的抗性表现有较大影响,在进行抗病育种时应放宽选择标准。

表1 F2的广义遗传力
2.4 分子遗传图谱和QTL定位
利用SRAP分子标记得到83个多态性标记,结合36个SSR多态性标记,应用JoinMap3.0软件进行连锁分析,取LOD值为3.0,得到的分子连锁图谱包括13个连锁群、76个标记位点,其中SRAP标记55个,SSR标记21个。图谱覆盖长度为830.4 cM,标记间平均图距为10.92 cM,连锁群上的标记数在2~16之间,长度处于0~152.2 cM范围内(图6)。经QTL分析,在第1、4、7连锁群上检测到3个QTL位点(表2)。第1连锁群上的QTL位点的LOD值为3.78,在连锁群上支持的区域为136.1~146.1 cM,与其距离最近的两个标记是C9C10-4、B11C16-2,可解释表型差异的贡献率为12.7%;在第4连锁群上的QTL位点LOD值为3.35,在连锁群上支持的区域为57.0~62.0 cM,与其距离最近的标记为B13C10、HpmsE072;在第7连锁群上检测到的QTL位点在HpmsE124和hpms2-2两个标记之间,其LOD值为3.73,支持区域为94.3~109.3 cM;后2个QTL的解释表型差异的贡献率分别为38.8%和11.0%。
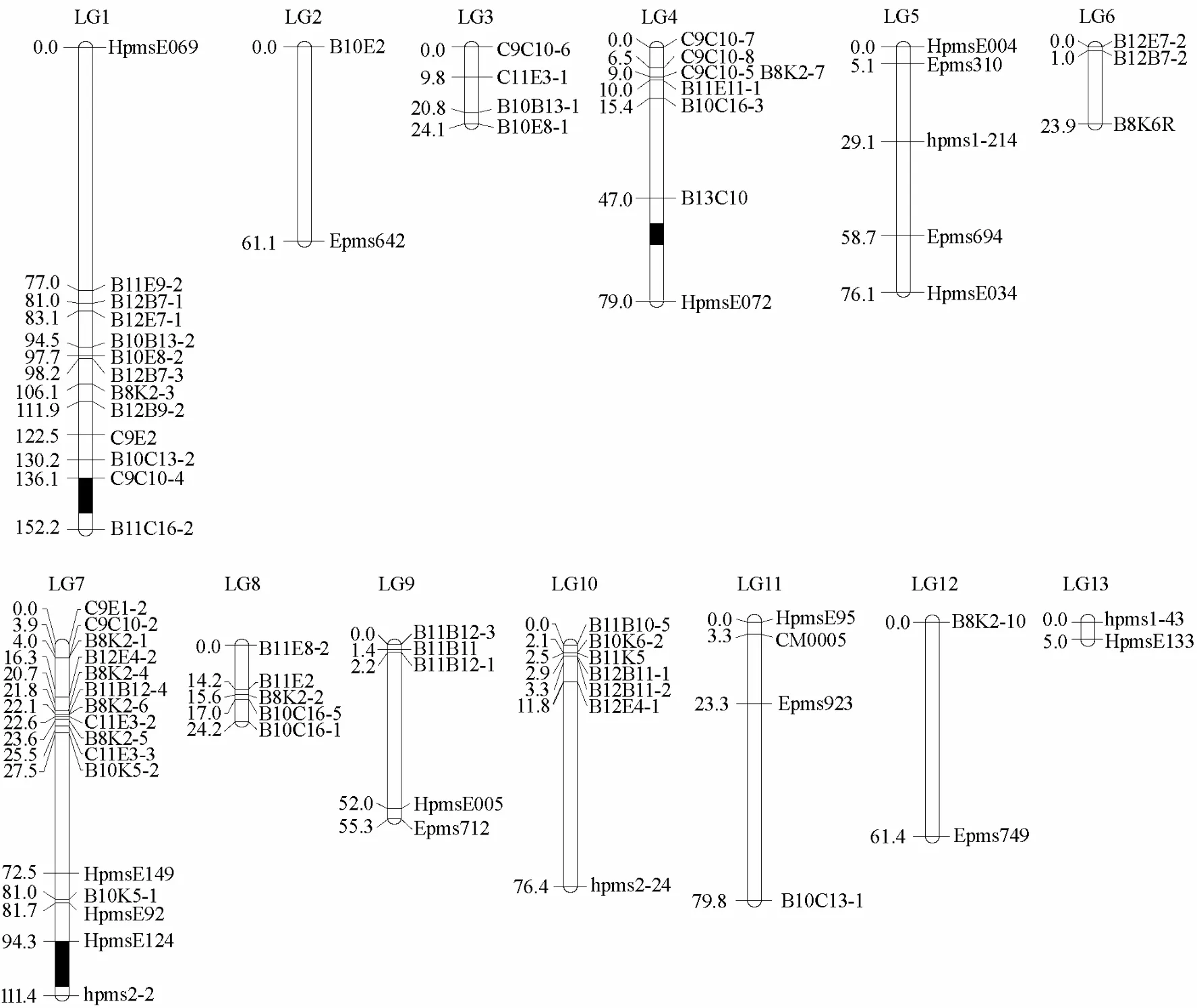
图6 辣椒抗黄瓜花叶病毒分子遗传连锁图谱
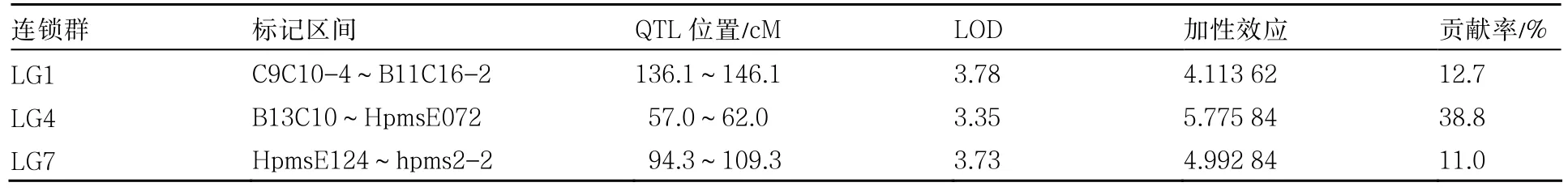
表2 辣椒抗黄瓜花叶病毒QTL分析
3 结论与讨论
国外对于辣椒抗黄瓜花叶病毒的QTL定位的研究取得了一些成果。Caranta等(1997)通过构建辣椒遗传图谱结合对F1抗病性鉴定,找到3个有关辣椒抗CMV的QTL位点,解释的表型变异在57%以上。之后又利用复合区间作图法在新的亲本组合中找到8个抗CMV的QTL位点,其中1个主效QTL解释的表型变异为45.0%~63.6%(Carahta et al.,2002)。Ben Chaim等(2001)在由180个F3世代单株为作图群体构建辣椒遗传图谱的基础上,采用区间作图法对辣椒抗CMV进行定位研究,得到4个QTL位点,其中1个主效QTL解释的表型变异为16%~33%。
本试验采用复合区间作图法得到3个QTL位点,分布在3个连锁群上,能解释表型变异率分别为 12.7%、38.8%和 11.0%。与第 1个 QTL位点距离较近的标记是 C9C10-4、B11C16-2,与第2个QTL位点距离较近的标记是B13C10、HpmsE072,与第3个QTL位点距离较近的标记是HpmsE124、hpms2-2,在这6个标记中,HpmsE072、HpmsE124和hpms2-2属于SSR标记。通过对本试验中构建的辣椒遗传图谱的第 7连锁群、Lee等(2004)构建的辣椒遗传图谱的第11连锁群、已公布(http://solgenomics.net/)的辣椒第10和第11条染色体DNA序列、Ben Chaim等(2001)构建的辣椒抗CMV遗传图谱的第11连锁群上的标记进行比较分析发现,可以通过其上的标记将本试验定位的第3个QTL位点与Ben Chaim等(2001)定位的主效QTL位点cmv11.1联系起来。说明本试验定位的QTL位点有可能与Ben Chaim等(2001)定位的QTL位点是同一位点,也可能是同一连锁群上的不同抗CMV位点。这需要进一步加密遗传图谱,寻找更多的抗CMV位点。
本试验中得到3个QTL位点的LOD值分别为3.78、3.35、3.73,QTL数量较少,可能与标记数量偏少和图谱密度不够有关,有待于进一步增加新的标记,丰富遗传图谱。
安静,胡勇胜,张宝玺,毛胜利,王立浩.2007.辣椒分子连锁遗传图谱的构建及抗疫病QTL定位.中国蔬菜,(10):9-12.
孔繁玲.2006.植物数量遗传学.北京:中国农业大学出版社.
雷剑,柳俊.2006.一个与马铃薯青枯病抗性连锁的SRAP标记筛选.中国马铃薯,20(3):150-153.
李树德.1995.中国主要蔬菜抗病育种进展.北京:科学出版社.
孙秀东,雷建军,周淑梅,陈国菊,曹必好,刘媛媛.2008.辣椒CMV广州分离物的鉴定及辣椒材料抗病性筛选.中国蔬菜,(3):11-14.
王刚,潘俊松,李效尊,何欢乐,吴爱忠,蔡润.2004.黄瓜SRAP遗传连锁图的构建及侧枝基因定位.中国科学C辑:生命科学,34(6):510-516.
张宝玺,王立浩,毛胜利,郭家珍.2005.我国辣椒育种研究进展.中国蔬菜,(10/11):4-7.
张芳芳,张正海,毛胜利,王立浩,胡鸿,张宝玺.2010.辣椒成熟果实中辣椒红素含量的QTL定位.中国蔬菜,(20):14-18.
张丽英,陈儒钢,张俊红,欧阳波,肖景华,李汉霞,叶志彪.2008.辣椒抗病基因同源序列的克隆与分析.中国农业科学,41(1):169-175.
Ben Chaim A,Grube R C,Lapidot M.2001.Identification of quantitative trait loci associated with resistance tocucumber mosaic virusinCapsicum annuum.Theor Appl Genet,102:1213-1220.
Budak H,Shearman R C,Parmaksiz I,Gaussoin R E,Riordan T P,Dweikat I.2004.Molecular characterization of buffalogross germplasm using sequence-related amplified polymorphism markers.Theor Appl Genet,108:328-334.
Caranta C,Palloix A,Lefebvre V,Daubèze A M.1997.QTLs for a component of partial resistance tocucumber mosaic virusin pepper:restriction of virus installation in host-cells.Theor Appl Genet,94:431-438.
Caranta C,Pflieger S,Lefebvre V,Daubèze A M,Thabuis A,Palloix A.2002.QTLs involved in the restriction ofcucumber mosaic virus(CMV)long-distance movement in pepper.Theor Appl Genet,104:586-591.
Fulton T M,Chunwongse H,Tanksley S D.1995.Microprep proteol for extraction of DNA from tomato and other herbaceous plant.Plant Mol Biol Rep,13(3):207-209.
Lee J M,Nahm S H,Kim Y M,Kim B D.2004.Characterization and molecular gengtic mapping of microsatellite loci in pepper.Theor Appl Genet,108:619-627.
Li G,Gao M,Yang B,Quiros C F.2003.Gene for gene alignment between theBrassica and Arabidopsisgenomes by direct transcriptome mapping.Theor Appl Genet,107:168-180.
Li G,Quiros C F.2001.Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging inBrassica.Theor App1 Genet,103:455-461.
Lin Z X,Zhang X L,Nie Y C,He D H,Wu M Q.2003.Construction of a genetic linkage map for cotton based on SRAP.Chinese Science Bulletin,8(19):2063-2067.
Riaz A,Li G,Quresh Z,Swati M S,Quiros C F.2001.Genetic diversity of oilseedBrassica napusinbred lines based on sequence-related amplified polymorphism and its relation to hybrid performance.Plant Breeding,120(5):411-415.
Tam S M,Mhiri C,Vogelaar A,Kerkveld M,Pearce S R,Grandbastien M A.2005.Comparative analyses of genetic diversities within tomato and pepper collections detected by retrotransposon-based SSAP,AFLP and SSR.Theor Appl Genet,110:819-831.
Xu Z,Barnett O W.1983.Identification of a cucumber mosaic virus strain from naturally infected peanut in China.Plant Disease,68:386-389.
