来源于角毛壳菌的木聚糖酶xyn24基因克隆及特性1)
2011-03-29王艳君杨谦
王艳君 杨谦
(福建师范大学福清分校,福清,350300)(哈尔滨工业大学)
木聚糖是大量存在于植物体内的植物纤维,在自然界的储量仅次于纤维素[1]。木聚糖酶(EC 3.2.1.8)是催化降解木聚糖的一类糖基水解酶,它可将由β-1,4-木糖苷键组成的木聚糖降解成低聚糖或木糖。木聚糖酶在饲料工业、功能性食品、纺织业、废水处理、生物燃料、造纸等方面具有广阔的应用前景而备受关注。国内外对木聚糖酶进行了较多的研究,尤其是丝状真菌可产生分泌到胞外的木聚糖酶,便于酶的分离纯化,加之丝状真菌的产酶水平高于细菌及酵母菌[2],因此,对丝状真菌尤其是木霉属(Trichoderma)和曲霉属(Aspergillus)的研究较多。角毛壳菌(Chaetomium cupreum)是一种重要的生物防治真菌,其生防作用除产生毒素及抗生素外,还可以产生大量的细胞壁降解酶,许多木聚糖酶已从微生物中克隆得到,但在角毛壳菌中尚未见报道。本研究从角毛壳菌中克隆得到木聚糖酶基因(xyn24),为研究该菌的生防机制及其生防能力的改造奠定基础。利用生物信息学手段进行分析,为进一步表达、纯化及大量制备重组xyn24蛋白及相关功能研究提供理论依据。
1 材料与方法
1.1 材料
角毛壳菌菌株为哈尔滨工业大学微生物实验室保存;Ex-Taq DNA聚合酶、T4连接酶、限制性内切酶、IPTG等为宝生物工程(大连)有限公司产品;引物由上海英俊生物技术有限公司合成;胶回收试剂盒购自上海华舜公司;其他生化试剂均为国产分析纯。
1.2 方法
1.2.1 木聚糖酶xyn24基因全长cDNA的获得
角毛壳菌菌丝体培养:角毛壳菌孢子接入PD液体培养基中,在27℃、220 r/min条件下培养48 h。4 000 r/min离心10 min收集菌丝,液氮速冻后于冰箱中-80℃保存备用。
角毛壳菌木聚糖酶基因ESTs序列的获得:哈尔滨工业大学生命科学与工程实验室构建了角毛壳菌菌丝体时期的cDNA文库,利用BlastX软件对角毛壳菌菌丝体的3 066条ESTs序列进行相似性搜索[3]。从中获得角毛壳菌木聚糖酶基因的EST序列,登录号为DV547992。
1.2.2 木聚糖酶xyn24基因全长DNA的获得
角毛壳菌基因组的提取:采用改良的CTAB法[4],取1.0 g的菌丝于液氮中研成粉末后,加600 μL预热的2×CTAB使之充分混匀,65℃水浴10 min,不时混匀;10 000 r/min离心,弃去上清,加600 μL预热的2×CTAB使之充分混匀,65℃水浴10 min,不时混匀;加等体积氯仿/异戊醇(V(氯仿)∶V(异戊醇)=24∶1)进行抽提,缓慢颠倒混匀10 min,10 000 r/min下高速离心10 min,上清转入新的离心管中,弃去沉淀(重复2~3次);上清液加入等体积的异丙醇,于室温沉淀DNA 15 min,10 000 r/min离心10 min,弃去上清;75%乙醇洗涤沉淀2次;加入适量TE,65℃溶解10 min;纯化后的角毛壳菌菌丝基因组DNA于-20℃保存备用。
角毛壳菌木聚糖酶基因的扩增:以角毛壳菌基因组为模板,PCR扩增xyn24基因的DNA序列所使用的引物为:上游引物,5'-GACGATGGTCTCCTTCAAGGCTCT-3';下游引物,5'-CGGTTAGACAGTGATGCTGGAAGAAC-3'在上下游引物中分别引入EcoRⅠ和XhoⅠ酶切位点,(加框处为酶切位点)。扩增程序为94℃、5 min;94℃、30 s,56℃、30 s,72℃、30 s,35个循环;72℃、7 min。PCR产物回收纯化,与pMD18-T载体连接,转化大肠杆菌DH5α感受态,涂布在含有氨苄青霉素的LB平板上。用IPTG和X-gal进行蓝白斑选择,从白色菌落(氨苄抗性转化子)提取质粒并酶切鉴定,将阳性克隆命名为pMD-T/Xyn,送上海博亚公司测序。
1.2.3 序列分析
用ORF软件(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)寻找开放读码框;Pfam(http://www.sanger.ac.uk/Software/Pfam/)进行蛋白家族预测以及用ProtParam软件(http://au.expasy.org/tools/protparam.html)计算蛋白的相对分子质量及等电点。
用BlastP进行相似性搜索,选择了11条与角毛壳菌xyn24基因氨基酸序列相似性高的真菌木聚糖酶氨基酸序列;用MEGA4.0软件对上述12种真菌的木聚糖酶基因的氨基酸序列进行多序列比对,构建进化树;BlastX(http://www.ncbi.nlm.nih.gov/BLAST/)进行序列同源性搜索;BlastP(http://www.ncbi.nlm.nih.gov/BLAST/)进行保守区预测;Prosite(http://www.expasy.org/cgi-bin/prosite)分析蛋白质结构域。SignalP 3.0 Server(ExPASy Proteomics tools)进行信号肽预测。
2 结果与分析
2.1 角毛壳菌xyn24基因全长DNA及cDNA序列的获得
提取角毛壳菌基因组作为模板,PCR扩增后的产物片段如图1所示,琼脂糖凝胶电泳纯化回收后,送交上海博亚公司测序。获得全长DNA序列,并提交GenBank注册,登录号为EF026977。

图1 xyn24基因的PCR扩增结果
2.2 角毛壳菌xyn24基因核苷酸序列分析
开放读码框由起始密码子ATG至终止密码子TAA(图2),共有690个碱基组成,编码229个氨基酸,由1个内含子和2个外显子组成,cDNA登录号为DQ886937,DNA登录号为EF026978。ProtParam分析表明该蛋白质的相对分子质量为24700,理论等电点为7.83,分子式为C1105H1620N306O340S3,不稳定系数为17.47,属于稳定的蛋白质。

图2 xyn24的ORF序列及推断的氨基酸序列
2.3 角毛壳菌xyn24基因进化树结构
xyn24基因推断的蛋白质序列与其相似性较高的11种真菌木聚糖酶蛋白质序列进行BlastP比对,结果表明,角毛壳菌xyn24基因推断的氨基酸序列与嗜热革节孢菌(Scytalidium thermophilum AB114442)、细丽毛壳菌(Chaetomium gracile D49850)、柰恩端梗霉(Acrophialophora nainiana DQ641721)、球毛壳菌(Chaetomium globosum XM001221294)、粗糙链孢霉(Neurospora crassa XM959052)、嗜热毛壳菌(Chaetomium thermophilum AJ508932)、费希尔曲霉(Neosartorya fischeri XM001258637)、构巢曲霉(Aspergillus nidulans XM656125)、土曲霉(Aspergillus terreus XM001216082)、禾旋孢腔菌(Cochliobolus sativus AJ004802)、玉米大斑病菌(Setosphaeria turcica AJ548879)木聚糖酶基因序列相似性分别为78%、75%、74%、72%、72%、66%、64%、64%、64%、63%、61%。采用邻接法分析12种真菌的木聚糖酶蛋白的进化顺序,结果如图3所示,xyn24蛋白与来源于嗜热革节孢菌的的木聚糖酶处在同一组中,两者的亲缘关系最近,与球毛壳菌、嗜热毛壳菌、细丽毛壳菌的亲缘关系相对较近。
2.4 角毛壳菌xyn24蛋白结构域及合成后的化学修饰位点预测
通过BlastP进行保守区预测(图4),该木聚糖酶基因属于糖基水解酶11家族,具有糖基水解酶家族11的保守区结构域,预测结果表明,糖基水解酶家族11结构域上具有2个活性位点,分别在第123~133位及第214~225位的氨基酸位置上。分析表明,该木聚糖酶蛋白共有N-豆蔻酰化位点10个;蛋白激酶C磷酸化位点4个;N-糖基化位点2个。表1列出了各位点在229个氨基酸序列中的位置及氨基酸组成。
2.5 角毛壳菌xyn24蛋白信号肽预测
木聚糖酶大多是胞外酶,都具有信号肽序列,经SignalP 3.0-NN预测结果(图5)表明,xyn24在16(A)~17(L)位为信号肽切割位点。比较其他11种不同来源的同源性较高的木聚糖酶蛋白的信号肽切割位点见表2所示,经选择12种不同真菌的木聚糖酶蛋白的信号肽切割位点符合下列通式:AX1A~X2P(其中X1为L,A,F;X2为L,F,A,M,S,T出现的频率较高)。
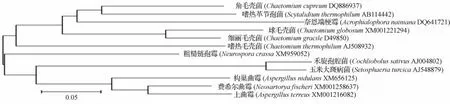
图3 12种真菌木聚糖酶蛋白进化树
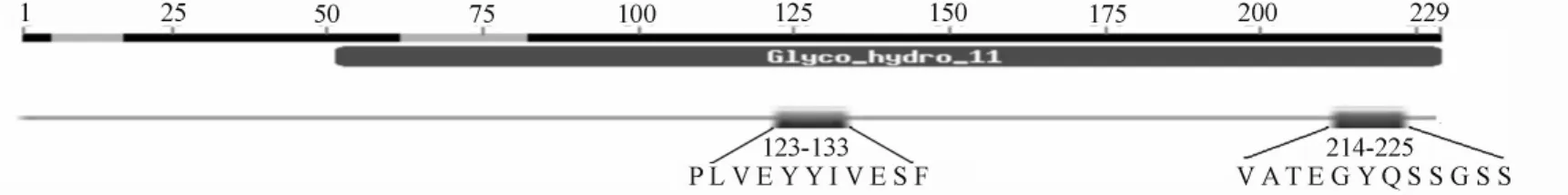
图4 木聚糖酶xyn24保守区预测结果
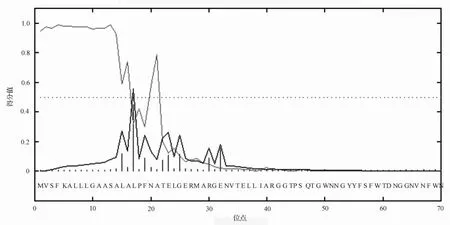
图5 角毛壳菌xyn24信号肽预测
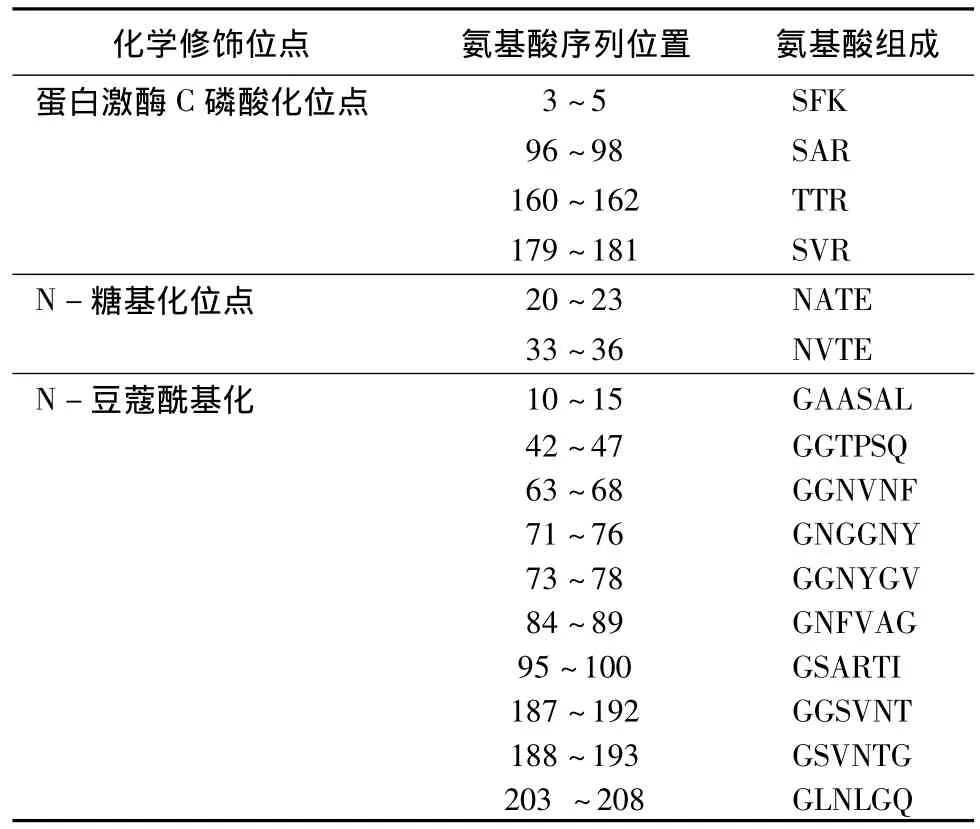
表1 木聚糖酶xyn24合成后的化学修饰位点

表2 不同来源的真菌木聚糖酶基因的信号肽序列
2.6 角毛壳菌xyn24蛋白三级结构预测
通过SWISS-MODEL(http://swissmodel.expasy.org/repository/)对其三级结构进行预测,结果如图6所示,xyn24含有一个α-螺旋并在外侧,β折叠扭曲成一个空穴,可作为酶作用的活性位点。这种单一的β折叠结构符合糖基水解酶家族11的结构特征。
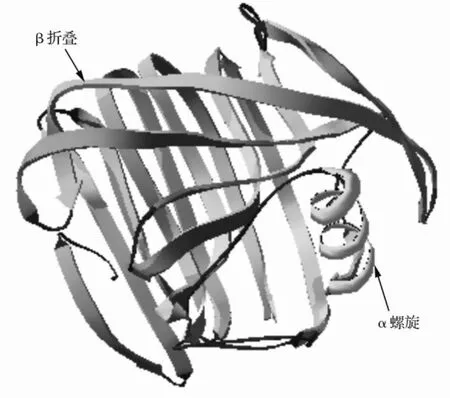
图6 预测木聚糖酶xyn24三级结构
3 结论与讨论
角毛壳菌基因组的提取过程中,真菌细胞壁的破碎是不可忽视的一个重要环节,本研究采用液氮研磨的方法,快速、低温、研磨充分,高质量的基因组模板提高了后续实验成功的可能性。
木聚糖酶根据其催化区氨基酸序列的组成不同,可分为糖基水解酶10和11家族[5],此外还有一些木聚糖酶属于第5、7、8、43糖基水解酶家族。糖基水解酶10家族的木聚糖酶的催化区是圆柱型(β/α)8结构[6],而糖基水解酶11家族的催化区结构是2个反相平行的β片状褶折和一个α螺旋组成,仅含有唯一的α-螺旋并在外侧[7],本研究克隆得到的xyn24具有糖基水解酶11家族催化区结构域的明显结构特征。
xyn24基因具有一个很强的信号肽区,含有信号肽的蛋白质一般能够被分泌到细胞外,可能作为重要的细胞因子起作用,从而具有潜在的应用价值。从本研究克隆得到的木聚糖酶基因xyn24及其相似性比对结果发现,其信号肽切割位点处的氨基酸A-A-P是进行信号肽识别和切割的关键部位。
蛋白质生物合成后活性调节的另外一种形式是化学修饰,包括蛋白质的糖基化和磷酸化。木聚糖酶的重要翻译后加工位点是N-位和O-位糖基化。木聚糖酶蛋白经过糖基化修饰以后可以使整个酶蛋白在体内避免蛋白水解酶的作用,保持其稳定性,对于正常发挥木聚糖酶的功能具有重要作用。磷酸化是蛋白质合成后广泛存在的一种化学修饰,是控制酶活性的重要步骤。许多情况下,磷酸化的蛋白质其酶活性大大提高;也有些情况下,磷酸化的蛋白质其酶活性下降甚至消失,从而对蛋白质的功能调节起重要作用。通过对xyn24蛋白的化学修饰位点的预测推断,该蛋白的体外表达载体的选择十分重要,如选择比较优秀的巴斯德毕赤酵母表达系统,是木聚糖酶蛋白产物在体外具有酶活性的关键。因此,通过生物信息学的分析论证为后续实验的成功奠定基础。
[1]Prade R A.Xylanases:from biology to biotechnology[J].Biotech Genet Eng Rev,1996,13:101-131.
[2]Polizeli M L T,Rizzatti A C S,Monti R,et al.Xylanase from fungi:properties and industrial applications[J].Appl Microbiol Biotechnol,2005,67(5):577-591.
[3]Zhang Haiyan,Yang Qian.Expressed sequence tags-based identification of genes in the biocontrol agent Chaetomium cupreum[J].Appl Microbiol Biotechnol,2007,74:650-658.
[4]萨姆布鲁克J,弗里奇E F,曼尼阿蒂斯T.分子克隆实验指南[M].2版.金冬雁,黎孟枫,候云德,等,译.北京:科学出版社,2002:880-887.
[5]Henrissat B,Bairoch A.New families in the classification of glycosyl hydrolases based on amino acid sequence similarities[J].Biochemical Journal,1993,293(3):781-788.
[6]Fujimoto Z,Kuno A,Kaneko S,et al.Crystal structure of Streptomyces olivaceoviridis E-86 β-xylanase containing xylan-binding domain[J].Journal of Molecular Biology,2000,300(3):75-585.
[7]Kumar P R,Eswaramoorthy S,Vithayathil P J,et al.The tertiary structure at 1.59Å resolution and the proposed amino acid sequence of a family-11 xylanase from the thermophilic fungus Paecilomyces varioti Bainier[J].Journal of Molecular Biology,2000,295(3):581-593.
