水产养殖环境常见四环素类抗生素抗性基因tet(M)的演化及传播的初步分析
2011-03-14陈琳琳刘维青张高生任宗明
陈琳琳,刘维青,刘 静,张高生,任宗明
(1.中国科学院 烟台海岸带研究所,山东 烟台 264003; 2.黄岛出入境检验检疫局,山东 青岛 266555;3.中国科学院 海洋研究所,山东 青岛 266071)
自20世纪中后期,发达国家即开始对环境中抗生素的含量及可能的生态效应高度关注,并相继建立了不同环境介质中的抗生素浓度标准[1]。而中国相关方面的研究起步较晚,但随着临床医学中抗生素使用剂量的不断增大及耐药性的出现,国内学者也开始越来越关注抗生素的环境污染与生态毒性效应[2-5]。抗生素的长期使用会造成生态环境中耐药性细菌及抗生素抗性基因(Antibiotic Resistance Genes,ARGs)的产生[6]。ARGs在生物体中可能长久而持续地传播,即使携带ARGs的细胞被杀灭或消亡,它释放到环境中的DNA仍可能通过垂直传播或水平转移转染给同属或不同属的其他细菌,进而在环境中传播、扩散,从而对公共健康和食品卫生、饮用水安全等构成威胁[7-8]。众多国内外学者认为ARGs在环境中的持续性残留、菌群间的迁移、转化和传播,比抗生素残留本身对生态环境的危害更大[9-10]。针对这些问题,Pruden等[11]2006年首次提出将抗生素抗性基因作为一类新型环境污染物。
四环素类抗生素由于其广谱性和低毒性,广泛应用于医药卫生、畜牧业、农业以及养殖业。而近几年,四环素耐药细菌及抗性基因的不断发现成为多个领域面临的严重问题[12]。细菌对四环素类的抗性主要由两种机制介导:一种是能量依赖性排出泵(energy dependent efflux pump),另一种是核糖体保护蛋白(ribosomal protection proteins,RPPs)[13]。目前已经发现了40多种四环素抗性基因,两种机制的抗性基因大多可以通过可转移的质粒或转座子传播,进而渗透到各种需氧或厌氧的革兰氏阳性和阴性细菌中[14]。其中RPPs基因广泛分布于地球多种环境介质中,并且它们与生物蛋白质合成过程中起重要作用的延长因子,如EF-G(原核生物蛋白质合成过程中催化 tRNA的移位和多肽延伸的每个循环后期mRNA从核糖体上掉下来)/EF-2(参与真核生物蛋白质合成,功能相当于原核生物的EF-G)以及EF-Tu(介导氨酰-tRNA进入核糖体的空位)/EF-1α(真核生物EF-1的α亚基,相当于原核生物中的EF-Tu亚基)具有近缘关系[15-17]。通常抗性基因作为“附件”基因单位(“accessory” genes),一般位于质粒、转座子等可动遗传因子上,他们与细菌自然环境下的成长并没有必然的联系,而延长因子是细菌生长所必需的基因,并且来自具有很长进化历史的参与蛋白合成的GTPase家族[18]。鉴于 RPPs基因与延长因子的特殊关系,对RPPs基因本质的研究是了解该类抗性基因的抗药性机制和传播途径的非常有效且直接的方法。
本研究关注的tet(M)基因就是一类重要的 RPPs基因,它通常和接合性转座子Tn1545-Tn916相结合,而Tn1545-Tn916是一个广宿主转座子,它可以在多种细菌之间转移[14,19-20]。这使得tet(M)基因可以通过染色体垂直传播或质粒、转座子水平传播进而在环境中广泛传播。研究显示,tet(M)基因在水产养殖环境中分布广泛,很有可能迅速在环境中造成抗性基因污染[21]。国外学者对不同环境介质不同细菌寄主的tet(M) 基因进行了研究,试图从分子水平分析tet(M)基因的起源[22-25]。根据我们掌握的文献,目前国内尚无单独对tet(M)基因的研究报道,只有从不同环境介质检测到tet(M)基因的存在[26]。为了研究不同来源的tet(M)基因与其环境介质、细菌宿主的相关性,从DNA序列这一最本质的层面揭示抗生素抗性基因的分化及潜在的传播机制,本文采用分子系统学的方法针对水产养殖环境中常见的四环素类抗生素抗性基因tet(M)的序列特点及系统发育进行分析,旨在为今后分析抗生素抗性基因来源、污染机理及控制对策奠定基础。
1 材料与方法
1.1 养殖环境tet(M)基因数据获取
从GenBank下载来自不同水产养殖生物和养殖环境的常见四环素类抗性基因tet(M)序列,基本信息包括抗性基因种类、细菌种属、环境介质及序列接受号Accession number列于表1。
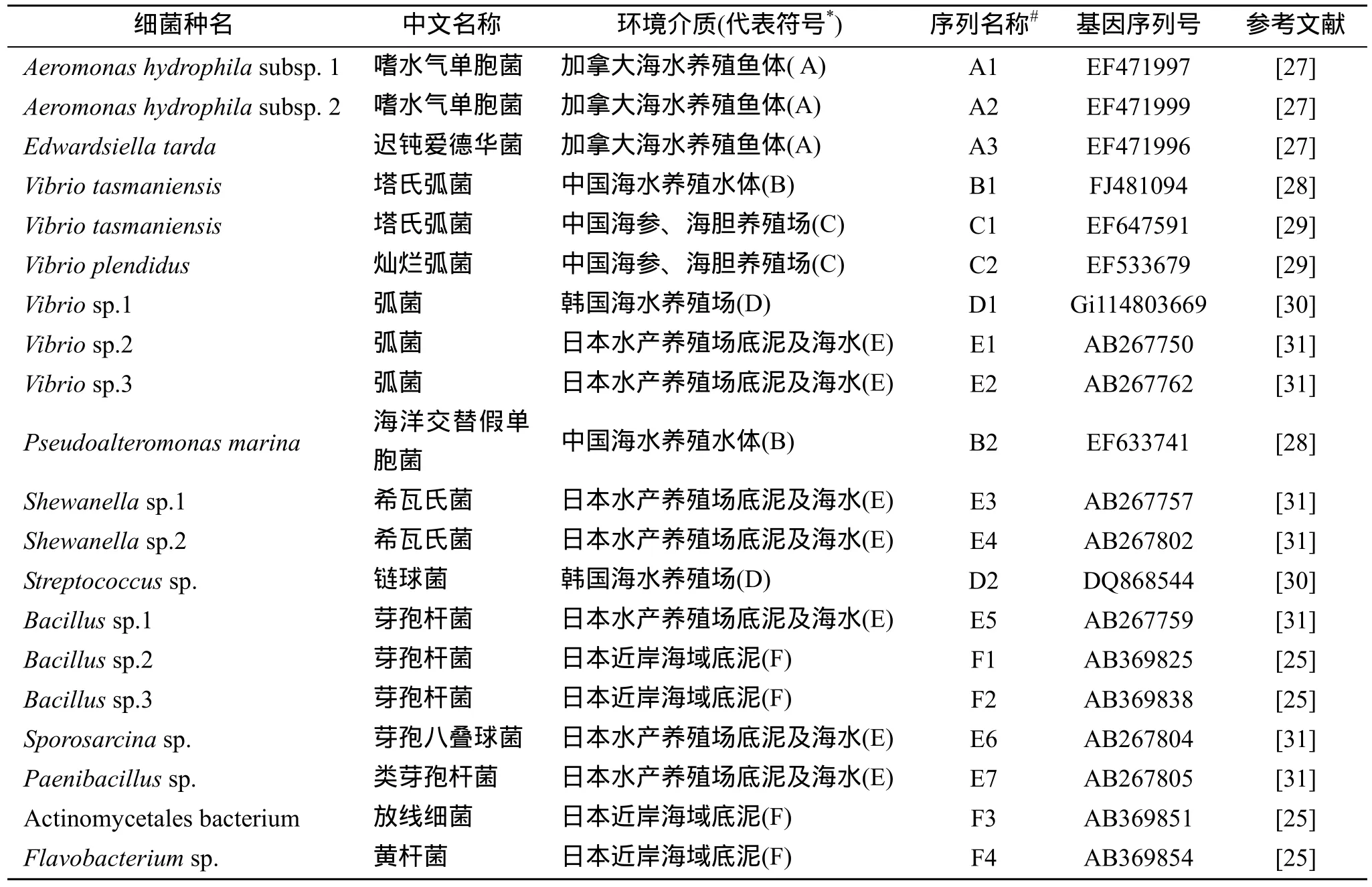
表1 水产养殖环境检出的tet(M)基因Tab.1 Detection of tet(M) genes in aquaculture environment
1.2 序列比对与系统学分析
将 20条序列用 Clustal W 软件[32]比对,DnaSP 4.0[33](DNA sequence Polymorphism)软件进行单倍型及多态性分析。采用MEGA 4.0[34](molecular evolution genetic analysis)软件,用最大简约信息(Most Parsimony,MP)法和邻近(Neibourgh Joining)法构建系统发育树。再应用自展法(bootstrap)检验树的可靠性(500次重复)。空位始终作为缺失状态,大肠杆菌延长因子EF-Tu基因(Gi| 296507628:5254-6438)作为外源基因,并结合寄主细菌分类及环境介质与tet(M)基因的系统发育作相关性分析。
2 结果与分析
2.1 序列多态性分析
序列比对得到662 bp同源片段,55个多态位点。20条基因序列有 4种基因型(表 2),其中基因型tet(M)-a存在于来自澳大利亚养殖鱼体内分离的3种细菌,包括嗜水气单胞菌两个亚种及迟钝爱德华菌Edwardsiella tarda; b基因型广泛分布于多种细菌中,包括 B1、C1、C2、D1、E2、E4、E6、E7、F2、F3、F4 、B2共5种环境介质12种细菌,其中B1、C1、C2、D1、E2等5种细菌均来自弧菌属(Vibrio); c基因型只存在于1种链球菌中,且与d基因型具有很近的亲缘关系,仅在405bp位点上有一个单碱基的A/G转换; d基因型存在于4种细菌,包括2种芽孢杆菌F1、E5,一种弧菌E1,以及一种希瓦氏菌E3。4种基因型的平均核苷酸差异度为4.2%,最大值8.3%出现在b和c两种基因型之间,最小值在c和d之间,为0.2%。
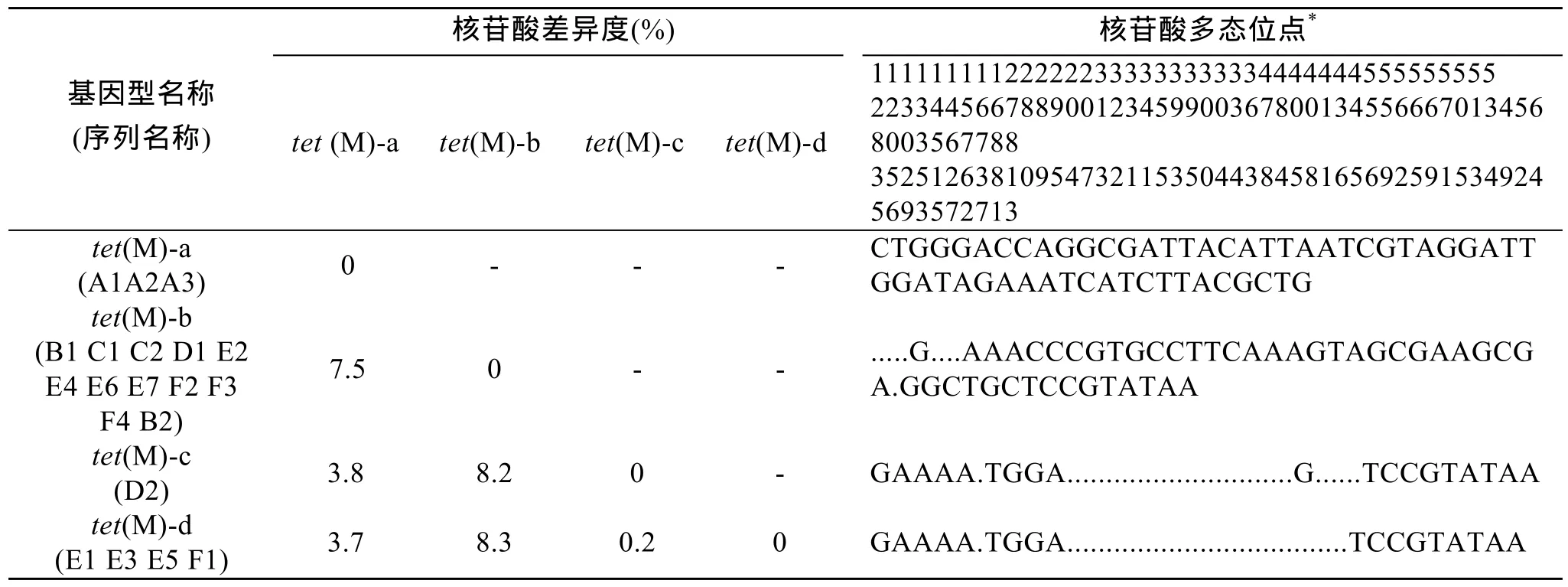
表2 tet(M)基因型核苷酸差异度及多态位点Tab.2 Nucleotide divergence and polymorphic sites in different tet(M) genotypes
2.2 系统发育分析
最大简约树显示20条tet(M)序列清晰分成三大分支(图 1),其中 Clade1对应于tet(M)-a基因型,Clade2对应于tet(M)-c和 d基因型,Clade3对应于tet(M)-b,500次重复检验,枝上置信度均为100%。邻近法构建的系统树与此树形结构相同,文中未列出。
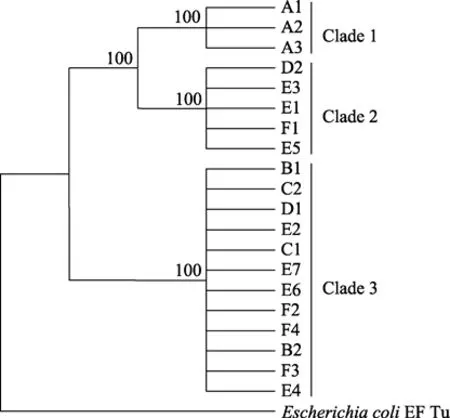
图1 水产养殖环境tet(M)基因分子系统发育树(分支上数字为自展支持率)Fig.1 Molecular phylogenetic tree of tet(M) genes in Aquaculture environment (The numbers above the branches indicate bootstrap support of MP)
3 讨论
从表1可以看出,水产养殖环境中tet(M)基因存在于多种细菌中,从海水环境中常见的病原菌到一些环境细菌。有研究表明,日本东京海域 21%~96%四环素抗性细菌携带有tet(M)基因,越南湄公河养殖区及中国海洋生物养殖场也检测到大量的tet(M)基因,暗示tet(M)基因是养殖环境细菌四环素抗性的主要机制[25,29,35]。tet(M)基因存在于细菌的可动遗传元件如质粒、转座子、接合转座子以及整合子[14,22,36],这使得tet(M)基因能更容易从一个物种传播到另一个物种[37],进而导致其在环境中大量细菌内的广泛分布[15]。在中国,长期以来四环素类抗生素因其广谱及低毒性,一直作为水产养殖的常用药,并作为添加剂低剂量加入饵料以促进养殖生物的生长,使得养殖环境成为四环素类抗生素的重要潴留池。因此,tet(M)基因的环境毒理效应,应作为中国养殖环境重要的关注对象。
基因序列多态性分析显示(表2),20条水产养殖环境中tet(M)基因共有4种基因型,其中,tet(M)-a基因型仅存于从澳大利亚养殖鱼体分离的3种细菌中,其他3种基因型在各种介质中均有出现,暗示着a基因型具有一定的环境介质特异性。相同的环境介质代表着相同或近似的抗生素使用背景。研究发现抗生素抗性基因与抗生素的使用背景有着直接的关系,抗生素对耐药菌株抗性基因的诱导具有专一性,因此,理论上抗生素本身在环境中的迁移、转化及归趋等环境行为与其所诱导的抗生素抗性基因,在环境中的传播应该具有很大的相似性和一致性,抗生素的使用历史与其抗性基因之间存在相关性[38-39]。日本科学家对太西洋远洋海域的抗性基因研究发现,在大洋深处未被抗生素污染的区域,仍然检测到了tet(M)抗性基因的存在[40]; Neela等[41]研究发现,有些革兰氏阳性菌虽然携带tet(M)基因,但仍对四环素敏感,说明tet(M)抗性基因的检出频率并不代表着抗性水平,还需要从基因表达的角度去分析。本研究中,大多数tet(M)的基因型与环境介质并无直接关系,相同或相近的环境介质内,tet(M)基因型可能相同或近缘(如A1、A2与A3),但也可能远缘(如D1与D2,F1与F2),说明环境介质与tet(M)基因型并无严格的相关性,暗示着tet(M)基因在环境中的迅速传播及分化。根据前人报道,很多抗生素抗性基因其实在抗生素使用之前就已存在,Kobayashi等[42]的分子系统学研究结果更加证实了RPPs基因包括tet(M),早在地球上生物总界分化之前已经存在,也就是说,RPPs在原核与真核生物分化之前就已经开始进化。从寄主细菌的分类地位角度看,水产养殖环境中 4种基因型在细菌中分布没有严格的种属特异性(表2)。相同或相近的tet(M)基因型可以存在于近缘或相同的细菌中(如弧菌属的5种细菌均携带tet(M)-b基因型),也可以存在于远缘的物种(如tet(M)-d同时存在于芽孢杆菌F1、E5,弧菌E1以及希瓦氏菌E33类细菌中)。同种或近缘的细菌应该具有相同或类似的遗传背景,而本研究中相同的遗传背景包含不同的tet(M)等位基因或者相同的tet(M)在远源的细菌株系中发现,进一步显示了tet(M)基因活跃的运动和进化[23]。
系统发育树上 20条基因序列分成了三大分支(图 1),其中 Clade1对应于tet(M)-a基因型,Clade2对应于tet(M)-c和d基因型,Clade3对应于tet(M)-b基因型。Rahman等[25]对日本近岸海域细菌抗性的研究,将海洋环境的tet(M)基因分成两种类型:tet(M)-A和tet(M)-B,其中前者代表起源于前抗生素时代即存在的自然基因储备(natural gene pool); 后者则代表通过选择或水平基因转移获得的基因。三大支系中,Clade2包含的tet(M)-d基因型与tet(M)-A相同,clade3对应的tet(M)-b则与tetM-B型相同,二者的平均核苷酸差异度达到8%; 这两种基因型与前人研究结果相比,分别对应于Agerso等[43]2006年所分的tet(M)-II和tet(M)-III,Gevers等[24]2003年所分的tet(M)-2和tet(M)-1,以及Huys等[44]2004年所分的type-I和type-V。Clade1对应的tet(M)-a及Clade2中的tet(M)-c与tet(M)-d近缘,但已有一定的分化,核苷酸差异度分别为3.7%和0.2%。Oggioni研究显示,tet(M)的基因变异是由于两种远缘的等位基因的同源重组产生的,这两种远缘等位基因类型分别发现于Tn1545和Staphylococcus aureus[45]。新的tet(M)基因型可能是由于前两种等位基因重组形成的镶嵌结构(mosaic structure),进而形成了介于两类远缘等位基因之间的基因型[23,24,46]。由此本研究中,tet(M)-a与tet(M)-c应为tet(M)-b和tet(M)-d两种远缘基因型的中间类型。
综上所述,tet(M)基因的分型与细菌的分类及环境介质的来源并无严格的相关性,因此今后在研究此类基因的环境毒理效应时,不应只分析基因的检出率及抗生素背景,还应从分子水平研究基因的变异及表达在不同细菌寄主及环境介质中的变化,了解该类基因的抗性分化机制,以期从分子手段入手降低甚至消除其抗药性或阻断其传播途径。中国作为水产养殖大国,在生产过程中抗生素特别是四环素类抗生素滥用严重,抗药细菌及抗生素抗性基因的增多将大大增加养殖业及人体耐药病原菌病害发生和流行的可能性及危害程度。tet(M)基因是水产养殖环境中的优势抗性基因,传播及进化活跃,广泛分布于多种耐药性病原菌及环境细菌中,因此,在今后的科研及环境保护过程中应予以一定的重视。
[1]Boxall A B A,Fogg L A,Blackwell P A,et al.Veteri-nary medicines in the environment [J].Reviews of Environmental Contamination and Toxicology,2004,180:1-91.
[2]胡莹莹,王菊英,马德毅.近岸养殖区抗生素的海洋环境效应研究进展[J].海洋环境科学,2004,23(4):76-80.
[3]张浩,罗义,周启星.四环素类抗生素生态毒性研究进展 [J].农业环境科学学报,2008,27(2):407-413.
[4]朱力军.动物大肠杆菌的抗药变化趋势[J].中国兽药杂志,2001,35(2):16-18.
[5]邹世春,朱春敬,贺竹梅,等.北江河水中抗生素抗性基因污染初步研究 [J].生态毒理学报,2009,4(5):655-660.
[6]Kerry J,Coyne R,Gilroy D,et al.Spatial distribution of oxytetracycline and elevated frequencies of oxytetracycline resistance in sediments beneath a marine salmon farm following oxytetracycline therapy [J].Aquaculture,1996,145(1-4):31-39.
[7]Crecchio C,Ruggiero P,Curci M,et al.Binding of DNA from Bacillus subtilis on montmorillonite-humic acids-aluminum or iron hydroxypolymers:Effects on transformation and protection against DNase [J].Soil Science Society of American Journal,2005,69(3):834-841.
[8]Hill K E,Top E M.Gene transfer in soil systems using microcosms [J].FEMS Microbiology Ecology,1998,25(4):319-329.
[9]Kummerer,K.Resistance in the environment [J].Journal of Antimicrobial Chemotherapy,2004,54(2):311-320.
[10]罗义,周启星.抗生素抗性基因(ARGs)——一种新型环境污染物 [J].环境科学学报,2008,28(8):1499-1505.
[11]Pruden A,Pei R,Storteboom H,et al.Antibiotic resistance genes as emerging contaminants:studies in northern Colorado [J].Environmental Science & Technology,2006,40(23):7445-7450.
[12]Schmidt A S,Bruun M S,Dalsgaard I,et al.Occurrence of antimicrobial resistance in fish-pathogenic and environmental bacteria associated with four danish rainbow trout farms [J].Applied and Environmental Microbiology,2000,66(11):4908-4915.crobiology,2000,66(11):4908-4915.
[13]Roberts M C.Tetracycline resistance determinants:Mechanisms of action,regulation of expression,genetic mobility,and distribution [J].FEMS Microbiology Reviews,1996,19(1):1-24.
[14]Chopra I,Roberts M.Tetracycline antibiotics:mode of action,applications,molecular biology,and epidemiology of bacterial resistance [J].Microbiology and Molecular Biology Reviews,2001,65(2):232-260.
[15]Aminov R I,Garrigues-Jeanjean N,Mackie R I.Molecular ecology of tetracycline resistance:development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J].Applied and Environmental Microbiology,2001,67(1):22-32.
[16]Miranda C D,Kehrenberg C,Ulep C,et al.Diversity of tetracycline resistance genes in bacteria from Chilean salmon farms [J].Antimicrobial Agents and Chemotherapy,2003,47(3):883-888.
[17]Sanchez-Pescador R,Brown J T,Roberts M,et al.Homology of the TetM with translational elongation factors:implications for potential modes of tetM-conferred tetracycline resistance [J].Nucleic Acids Research,1988,16(3):1218.
[18]Connell S R,Tracz D M,Nierhaus K H,et al.Ribosomal protection proteins and their mechanism of tetracycline resistance [J].Antimicrobial Agents and Chemotherapy,2003,47(12):3675-3681.
[19]Salyers A A,Shoemaker N B,Stevens A M,et al.Conjugative transposons - an unusual and diverse set of integrated gene-transfer elements [J].Microbiological Reviews,1995,59(4):579-590.
[20]Clewell D B,Flannagan S E,Jaworski D D.Unconstrained bacterial promiscuity - the Tn916-Tn1545 family of conjugative transposons [J].Trends in Microbiology,1995,3(6):229-236.
[21]高盼盼,罗义,周启星,等.水产养殖环境中抗生素抗性基因(ARGs)的研究及进展 [J].生态毒理学报,2009,4(6):770-779.
[22]Agerso Y,Jensen L B,Givskov M,et al.The identification of a tetracycline resistance gene tet(M),on a Tn916-like transposon,in the Bacillus cereus group [J].FEMS Microbiol Letter,2002,214(2):251-256.
[23]Doherty N,Trzcinski K,Pickerill P,et al.Genetic diversity of the tet(M) gene in tetracycline-resistant clonal lineages of Streptococcus pneumoniae [J].Antimicrobial Agents and Chemotherapy,2000,44(11):2979-2984.
[24]Gevers D,Danielsen M,Huys G,et al.Molecular characterization of tet(M) genes in Lactobacillus isolates from different types of fermented dry sausage [J].Applied and Environmental Microbiology,2003,69(2):1270-1275.
[25]Rahman M H,Nonaka L,Tago R,et al.Occurrence of two genotypes of tetracycline (TC) resistance gene tet(M) in the TC-resistant bacteria in marine sediments of Japan[J].Environmental Science & Technology,2008,42(14):5055-5061.
[26]陈明娜,党宏月.海参、海胆养殖水体中多抗性细菌抗性基因的初步研究 [J].海洋科学,2008,32(3):13-18.
[27]Akinbowale O L,Peng H,Barton M D.Diversity of tetracycline resistance genes in bacteria from aquaculture sources in Australia [J].Journal of Applied Microbiology,2007,103(5):2016-2025.
[28]Dang H Y,Song L S,Chen M N,et al.Concurrence of cat and tet genes in multiple antibiotic-resistant bacteria isolated from a sea cucumber and sea urchin mariculture farm in China [J].Microbial Ecology,2006,52(4):634-643.
[29]Dang H,Zhang X,Song L,et al.Molecular characterizations of oxytetracycline resistant bacteria and their resistance genes from mariculture waters of China [J].Marine Pollution Bulletin,2006,52(11):1494-1503.
[30]Kim S R,Nonaka L,Suzuki S.Occurrence of tetracycline resistance genes tet(M) and tet(S) in bacteria from marine aquaculture sites [J].FEMS Microbiology Letter,2004,237(1):147-156.
[31]Nonaka L,Suzuki S.New Mg2+-dependent oxytetracycline resistance determinant tet 34 inVibrio isolatesfrom marine fish intestinal contents [J].Antimicrobial Agents Chemother,2002,46(5):1550-1552.
[32]Thompson J D,Higgins D G,Gibson T J.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice [J].Nucleic Acids Research,1994,22(22):4673-4680.
[33]Rozas J,Sanchez-DelBarrio J C,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods [J].Bioinformatics,2003,19(18):2496-2497.
[34]Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular evolutionary genetics analysis (MEGA) software version 4.0 [J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[35]Kobayashi T,Suehiro F,Cach Tuyen B,et al.Distribution and diversity of tetracycline resistance genes encoding ribosomal protection proteins in Mekong river sediments in Vietnam [J].FEMS Microbiology Ecology,2007,59(3):729-737.
[36]Roberts M C.Plasmid-mediated Tet M in Haemophilus ducreyi [J].Antimicrobial Agents and Chemotherapy,1989,33(9):1611-1613.
[37]Licht T R,Christensen B B,Krogfelt K A,et al.Plasmid transfer in the animal intestine and other dynamic bacterial populations:the role of community structure and environment [J].Microbiology,1999,145(Pt 9):2615-2622.
[38]Aarestrup F M,Agerso Y,Gerner-Smidt P,et al.Comparison of antimicrobial resistance phenotypes and resistance genes in Enterococcus faecalis and Enterococcus faecium from humans in the community,broilers,and pigs in Denmark [J].Diagnostic Microbiology and Infectious Disease,2000,37(2):127-137.
[39]Pei R,Kim S C,Carlson K H,et al.Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J].Water Research,2006,40(12):2427-2435.
[40]Rahman M H,Sakamoto K Q,Nonaka L,et al.Occurrence and diversity of the tetracycline resistance gene tet(M) in enteric bacteria of Antarctic Adelie penguins[J].Journal of Antimicrobial Chemotherapy,2008,62(3):627-628.
[41]Neela F A,Nonaka L,Suzuki S.The diversity of multi-drug resistance profiles in tetracycline-resistant Vibrio species isolated from coastal sediments and seawater[J].Journal of Microbiology,2007,45(1):64-68.
[42]Kobayashi T L.Nonaka M,Rahman H,et al.Molecular evidence for the ancient origin of the ribosomal protection protein that mediates tetracycline resistance in bacteria[J].Journal of Molecular Evolution,2009,66:228-235.
[43]Agerso Y,Pedersen A G,Aarestrup F M.Identification of Tn5397-like and Tn916-like transposons and diversity of the tetracycline resistance gene tet(M) in enterococci from humans,pigs and poultry [J].Journal of Antimicrobial Chemotherapy,2006,57(5):832-839.
[44]Huys G,D'Haene K,Collard J M,et al.Prevalence and molecular characterization of tetracycline resistance in Enterococcus isolates from food [J].Applied and Environmental Microbiology,2004,70(3):1555-1562.
[45]Oggioni M R,Dowson C G,Smith J M,et al.The tetracycline resistance gene tet(M) exhibits mosaic structure [J].Plasmid,1996,35(3):156-163.
[46]Stanton T B,Humphrey S B.Isolation of tetracycline-resistant Megasphaera elsdenii strains with novel mosaic gene combinations of tet(O) and tet(W) from swine [J].Applied and Environmental Microbiology,2003,69(7):3874-3882.
