碱性pH条件下白念珠菌钙离子通道CCH1和MID1基因对钙内流的影响及Crz1p转录因子对其的调控作用
2011-02-10王慧徐宁邢来君李明春魏东盛
王慧,徐宁,邢来君,李明春,魏东盛
南开大学微生物学系 分子微生物学与技术教育部重点实验室,天津 300071
钙离子是微生物细胞生长必须的营养元素之一,并且在细胞内充当着重要的第二信使分子的角色[1-2]。真核生物细胞对外界环境变化的适应能力对其存活至关重要,酵母细胞在暴露于多数环境压力时,如:离子饥饿[3]、高等渗压力[4]、碱性 pH[5]以及其他信号分子 (如α-因子[6]和药物胺碘酮[7]) 等,都会引起胞内钙离子浓度的瞬间变化。细胞内钙离子浓度的变化机制极其复杂,可能是外钙内流的结果,也可能是胞内钙库,如液泡或高尔基体释放钙离子的结果。Brand等发现钙离子通道 (VGCC) 电压门控膜蛋白Cch1p和Mid1p,与白念珠菌钙内流有关[8]。胞外钙离子通过Cch1p和Mid1p进入细胞内,引起细胞内钙浓度的瞬间变化进而激活钙调神经磷酸酶 (CaN),使得转录因子 Crz1p去磷酸化,与CDRE (CaN依赖性的应答元件) 结合来进一步激活下游的应答基因,随后将胞内的钙离子浓度降低到变化前的水平[5]。
微生物体必须在环境变化的适应过程中存活,这对于人体条件致病性真菌白念珠菌尤为重要。pH是一种重要的生理环境压力条件,对白念珠菌的形态有显著影响,细胞对环境pH的适应能力对其致病性至关重要[9]。酵母细胞内钙离子的动态平衡与pH密切相关,在酿酒酵母中对碱性pH转录应答的研究首次给出钙离子信号转导途径介导 pH压力应答的证据。DNA微阵列数据[10]表明在碱性pH条件下,共有150个pH依赖性的基因在mRNA水平上表达上调至少2倍。碱性pH条件诱导的许多基因的表达在CNA1/CNB1或CRZ1基因缺失的菌株中下调,甚至在添加CaN的免疫抑制剂药物FK506时完全受到抑制。对 ENA1启动子元件分析的结果表明,在其中有2个pH应答区,包含着最小的CDRE元件,来激活碱性pH条件引发的转录应答。可见,以钙离子信号作为信使,转录因子 Crz1p介导的钙信号途径是pH压力应答中的重要途径。
为了研究钙信号途径在碱性 pH条件应答中的重要作用,本文构建了钙通道基因 CCH1和 MID1的缺失突变株,研究其在碱性pH条件下对钙内流的影响。微生物细胞胞内钙浓度变化的测定方法很多,其中钙发光蛋白指示法[11]有过多次报道,然而很少使用流式细胞术来检测酵母细胞中钙离子的变化。本文利用流式细胞术在全细胞水平上测定碱性 pH条件下胞内钙波变化的动力学,分析CCH1和MID1缺失对钙内流的影响,通过 β-半乳糖苷酶体系和实时定量PCR方法进一步追踪Crz1p转录因子的调控作用。总之,对钙信号途径的研究将有助于加深对白念珠菌碱性pH应答机制的了解。
1 材料与方法
1.1 材料
1.1.1 主要试剂
Taq、Taq plus、long Taq DNA聚合酶、常用限制性内切酶购于 TaKaRa公司;限制性内切酶BspQⅠ、NogM Ⅳ和 AseⅠ购于 NEB公司;pGEM-T Easy Vector、T4 DNA连接酶及反转录酶购于 Promega北京生物技术有限公司;DNA快速纯化/回收试剂盒、M199培养基、ONPG和dNTPs等购于北京鼎国生物技术有限公司;转化使用试剂PEG3350、LiAc以及钙离子螯合剂EGTA和钙探针Fluo-3AM购于Sigma公司;荧光定量PCR使用天根公司的 SYBR-Green试剂盒。其余试剂均为国产分析纯;所有引物均由北京奥科公司合成。
1.1.2 菌株、质粒和引物
实验使用的菌株和质粒见表1,引物见表2。
1.1.3 培养基
LB培养基:1%胰蛋白胨,0.5%酵母提取物,1% NaCl。白念珠菌培养使用的所有培养基(SC-ura除外) 均添加终浓度80 µg/mL尿苷。YPD培养基:1%酵母浸出粉,2%蛋白胨,2%葡萄糖。SC培养基:0.67%无氨基酵母氮源,0.2%省却混合物粉末,2%葡萄糖。M199培养基:M199粉末一袋,150 mmol/L HEPES,80 µg/mL尿苷,调溶液的pH至 4或 8,抽滤除菌。钙离子螯合剂 EGTA配成100 mmol/L贮液,过滤后添加到培养基中使终浓度达到10 mmol/L,形成钙离子缺陷的培养条件,CaN的免疫抑制剂FK506配成1 mg/mL贮液,添加到培养基的终浓度为1 µg/mL,CaCl2配成1mol/L贮液,添加时终浓度为200 mmol/L。

表1 本研究使用的菌株和质粒Table 1 Strains and plasmids used in this study

表2 本研究所用引物Table 2 Primers used in this study
1.2 白念珠菌基因的敲除
使用二步法PCR介导的基因敲除的方法[12]。分别以质粒 pRS-Arg∆SpeI和 pDDB57为模板,MID1/CCH1-5DR和3DR为引物,通过PCR获得含有ARG4或URA3选择性标记的PCR产物。将PCR产物直接转化野生型菌株 DAY1。白念珠菌细胞的转化使用醋酸锂法,具体方法参见文献[13]。使用MID1/CCH1-5detect和3detect引物通过PCR的方法进行转化子的鉴定。
1.3 Fluo-3荧光染料负载及流式细胞术测钙
离心收获对数生长期的细胞,用 HBSS缓冲液(氯化钾0.4 g/L、氯化钠8.77 g/L、六水合氯化镁0.2 g/L、氯化钙 0.22 g/L、HEPES 2.38 g/L、葡萄糖1.98 g/L,pH 7.0) 洗涤细胞,荧光染料Fluo-3AM贮液用20% Pluronic F-127 DMSO溶解至浓度为1 mg/mL,使用时终浓度为5 µg/mL。加染料的细胞置于37 ℃避光孵育50 min,负载后用HBSS洗涤细胞3次。使用FACSCalibur流式细胞仪测定胞内钙离子浓度,在加入刺激物质前,钙离子的荧光值平稳,显示为基线,30 s后加入KOH或HCl,混匀流式管中的细胞后跟踪细胞内钙离子的动态变化482 s,总时间为512 s。数据使用Winmdi 2.9软件进行分析。
1.4 pPHO89-LacZ-His重组质粒的构建
使用PHO89-Pdistal和PHO89-ATG引物扩增得到PHO89启动子,终质粒pPHO89-LacZ-His的构建方法参见文献[14]。
1.5 β-半乳糖苷酶活性测定
转化不同菌株的 pPHO89-LacZ-His质粒,在SC-His缺陷培养基上筛选正确转化子,将转化子接种于30 mL不同条件的M199培养基中,30 ℃振荡培养至OD600≈0.8~1.0时收集菌体,β-半乳糖苷酶活性的测定具体步骤见文献[13],β-半乳糖苷酶的活性计算公式,OD420×1 000/[OD600×t (min)×3(稀释因子)],单位为Miller,每组数据的获得均来自至少3次独立平行实验。
1.6 白念珠菌总RNA提取
将白念珠菌接种至3 mL M199 (pH 7) 培养基中,30 ℃过夜振荡培养。稀释过夜培养的菌液,转接至50 mL M199 (pH 4或pH 8)培养基中,起始OD600≈0.1,30 ℃振荡培养至 OD600≈0.6~0.8,离心收集菌体,洗涤后液氮冻存。使用玻璃珠破壁的方法提取RNA[15]。提取的RNA样品保存于−80 ℃超低温冰箱中备用。
1.7 cDNA的合成和荧光定量PCR
使用Promega公司的Oligo(dT) 引导cDNA第一链的合成,反应程序为:37 ℃,30 min使引物结合;42 ℃、1 h进行反转录;70 ℃、15 min灭活反转录酶。反转录得到的cDNA样品在−80 ℃保存,荧光定量PCR使用Bio-rad IQ5仪器,ACT1-5RT和ACT1-3RT引物用于ACT1的扩增,MID1-5RT/3RT、CCH1-5RT/3RT引物分别来扩增MID1和CCH1基因,每个反应设4个平行。PCR扩增条件为:94 ℃预变性3 min;94 ℃ 20 s,58 ℃ 20 s,68 ℃ 30 s,40个循环。PCR完成后,温度从58 ℃升至94 ℃,仪器自动收集荧光信号,得到融解曲线。进行 3次独立平行实验后,使用Bio-radIQ5软件分析实验结果,计算MID1和CCH1的Threshold cycle (Ct值),使用内参基因ACT1的Ct值矫正目的基因的Ct值得到 ∆Ct值,然后进一步比较野生型菌株和缺失突变株中同一基因的 Ct值得到 ∆∆Ct值,基因相对表达量使用2−∆∆Ct方法计算[16]。
2 结果
2.1 白念珠菌CCH1和MID1基因缺失突变株的构建
CCH1和MID1基因杂合缺失突变株比较容易获得,挑取 SC-Arg选择性平板上的转化子,分别提取基因组DNA,进行转化子的PCR鉴定。结果显示:对于CCH1,筛选的14个转化子中,有11个出现3.8 kb的CCH1基因野生型条带和2.3 kb的ARG4条带 (图1A,泳道3),获得杂合子的阳性率为78.6%。对于MID1,筛选的21个转化子中,有17个出现1.7 kb的MID1基因野生型条带和2.4 kb的ARG4条带 (图1B,泳道2),获得杂合子的阳性率为 80.9%。但是纯合突变株不易获得,第 2次重组后大部分转化子是由 URA3替换了 ARG4造成的杂合突变株,从SC-Ura平板分别筛选了75个CCH1和80个MID1的转化子,仅获得1株纯合突变株cch1::ARG4/cch1::URA3 (图1A,泳道1) 和mid1::ARG4/mid1::URA3 (图1B,泳道3),阳性率分别为1.33%和1.25%。
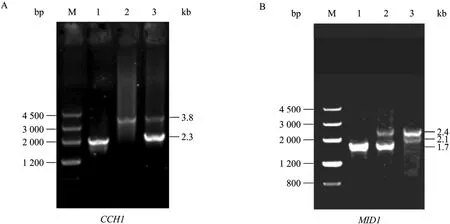
图1 CCH1和MID1基因缺失突变株的PCR检测Fig. 1 PCR confirmation of CCH1 and MID1 mutants. (A) 1: NKC73 (cch1::ARG4/cch1::URA3); 2: wild type; 3: NKC72 (cch1::ARG4/CCH1). (B): 1: wild type; 2: NKC67 (mid1::ARG4/MID1); 3: NKC68 (mid1::ARG4/mid1::URA3); M: marker Ш 4.5 kb, 3.0 kb, 2.0 kb, 1.2 kb, 800 bp from top to bottom.
2.2 碱性 pH 条件引起胞内钙离子浓度的瞬间变化以及CCH1和MID1基因缺失对钙波变化的影响分析
经Fluo-3AM负载的细胞上流式管,图2A为白念珠菌DAY1细胞FSC/SSC散点图,图中细胞最为集中的R1区域设定为研究范围。野生型白念珠菌细胞受到3 mol/L KOH刺激后,由于胞内的pH由中性变为碱性,200 s内即可看到明显的钙峰出现,而在250 s后胞内钙离子浓度又回到刺激前的基线水平(图2B)。使用6 mmol/L的HCl刺激同样的细胞,使胞内 pH由中性变为酸性,却没有明显的钙峰出现(图 2C)。为了进一步研究 pH对胞内钙波变化的影响,使用不同浓度的KOH和HCl刺激DAY1细胞,比较每一种 pH条件下钙波变化区域的平均荧光强度值与最大变化区域值的比值来确定能引起钙波变化最为显著的pH条件范围。从图2D可以看出,当胞外pH达到7.5时,比值大于50%,证明开始出现了较为明显的钙波变化并且在pH 8.2时达到最大,在pH 8.5后,这种变化又逐渐减弱,表明不同pH引起的胞内钙离子浓度的瞬间变化是剂量依赖性的。我们使用同样浓度的 KOH刺激 cch1∆/∆和mid1∆/∆细胞 (图2E、2F),使胞内的pH达到8.2,但并没有看到明显的钙峰出现,表明CCH1和MID1基因缺失影响了碱性pH条件下钙离子的内流。
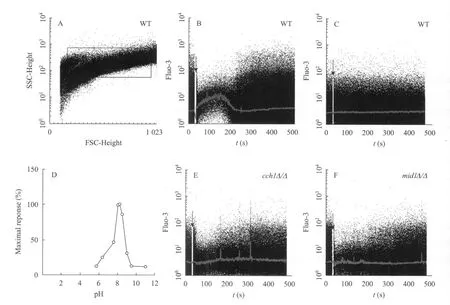
图2 不同pH条件下钙波变化的动力学及cch1∆/∆和mid1∆/∆对碱性pH条件下钙内流的影响Fig. 2 Kinetics of the calcium fluctuation in response to different pH and effect of cch1∆/∆ and mid1∆/∆ mutants on calcium fluctuation in response to alkaline stress.
2.3 CCH1和MID1基因缺失对PHO89-LacZ活性的影响分析
PHO89是一个碱性pH应答基因,且它的激活要通过钙调神经磷酸酶途径[11]。如果碱性 pH能引起胞内钙浓度的瞬间升高,我们推测许多钙离子或CaN应答的启动子将会被激活,而在钙离子浓度没有发生变化的情况下,启动子无应答。为证实这种假设,我们构建了PHO89-LacZ-His报告基因并将其分别转化DAY1、cch1∆/∆和mid1∆/∆,利用β-半乳糖苷酶报告体系,通过比较不同pH以及不同钙浓度条件下启动子活性的强弱来分析CCH1和MID1基因缺失对PHO89-LacZ活性的影响。结果发现:1) 在pH 4条件下,PHO89启动子几乎无应答,只有较低的 LacZ活性,但是添加 Ca2+后,LacZ活性在野生型菌株中上升了10倍,在突变株中也上升了2~5倍。2) 在pH 8条件下,cch1∆/∆和mid1∆/∆中 LacZ活性比野生型菌株中的活性下降近 2倍(图3),说明碱性pH条件下缺失突变株对钙离子浓度变化的影响直接影响到了PHO89启动子的活性,而在pH8+EGTA条件下LacZ活性的明显下降进一步说明PHO89-LacZ活性是钙离子依赖性的。
2.4 Crz1p转录因子对钙离子依赖性的 PHO89-LacZ活性的调控作用
Crz1p是钙信号转导途径的核心,为了研究这种碱性pH诱导的钙离子依赖性的PHO89-LacZ活性是否受到转录因子Crz1的调控作用,我们将含报告基因的质粒进一步转化了crz1∆/∆菌株,并且在pH 4和pH 8两种背景环境下,同时添加Ca2+和EGTA,以及CaN的免疫抑制剂FK506,通过测定12种条件下 LacZ的活性,比较野生型和突变型菌株中 β-半乳糖苷酶的活性差异,来分析 Crz1p转录因子对钙离子依赖性的 PHO89-LacZ活性的调控作用。从图4可以看出,1) 在crz1∆/∆菌株中,两种pH条件时无论是否添加 Ca2+或药物 FK506,都没有明显的 LacZ活性,证明PHO89启动子的活性是受到 Crz1p正向调控的;2) 野生型菌株中,pH 4和pH 4+FK506这两种条件下,只能检测到较低的LacZ活性,补加Ca2+后,LacZ活性上升10倍,但由于FK506的添加,LacZ活性又下降了2倍;相比较酸性条件 (pH 4),碱性条件下 (pH 8) 无论是否添加FK506都有较高LacZ活性,但在添加Ca2+螯合剂 EGTA后,LacZ活性明显下降,而且 FK506的添加使得LacZ活性又下降了2倍。综上可以得出,碱性pH压力下钙离子诱导的PHO89启动子的激活是CaN/Crz1p依赖的。
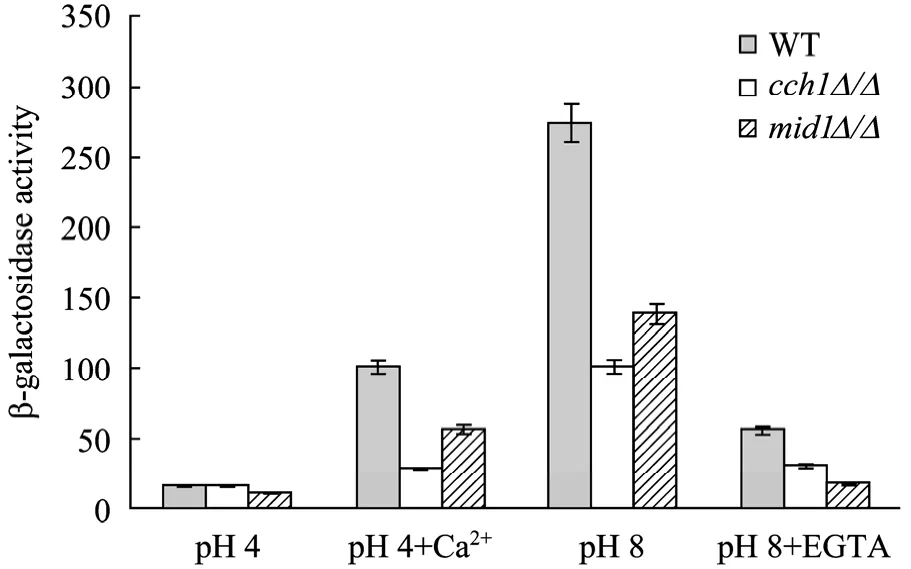
图3 含 PHO89-LacZ片段的野生型和缺失突变株在不同培养条件下的β-半乳糖苷酶活性分析Fig. 3 β-Galactosidase activity analysis of PHO89-LacZ band containing wild type and mutant strains.
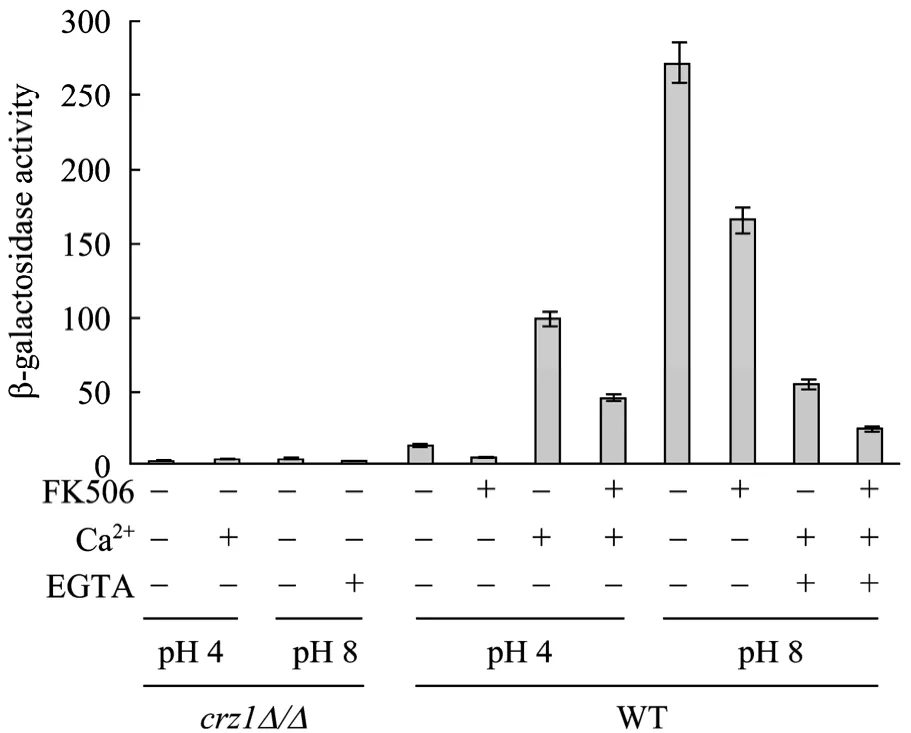
图4 Crz1p对碱性pH压力下钙离子诱导的PHO89启动子的调控作用Fig. 4 Regulating role of Crz1p in activation of calciumresponsive PHO89 promoter under alkaline stress.
2.5 Crz1p转录因子对钙通道基因CCH1和MID1的调控作用
对碱性pH的应答需要Rim101p、Crz1p和CaN,并且依赖于许多Na+-ATP酶转运体基因的表达。例如,在白念珠菌中,ENA2受到Rim101的严格调控,但是 Crz1在碱性 pH应答中似乎需要更多的除Na+-ATP酶转运体基因以外的靶点[10]。CCH1和MID1基因作为钙离子内流的通道,是CaN激活必需的,而cch1∆/∆和mid1∆/∆在碱性pH条件下对钙内流以及对 PHO89-LacZ活性的影响,很大程度是由于CaN没有被激活。因此,我们推测为了应答碱性pH,Crz1p对CCH1和MID1基因也具有一定的调控作用。
通过荧光定量PCR,我们比较了pH 4和pH 8两种条件下 CCH1和 MID1基因在野生型菌株和crz1∆/∆缺失菌株中的表达差异,从图5中的结果可以看出,在 pH 8条件下,CCH1和 MID1基因在crz1∆/∆缺失菌株中的表达量都有显著下降,是其在野生型菌株中的 40%~60%,添加药物 FK506对CCH1和MID1基因的表达没有明显影响,证明碱性pH条件下CCH1和MID1基因的表达受到Crz1p转录因子的正向调控作用,但非 CaN依赖。而在pH 4条件下,CCH1和MID1基因在crz1∆/∆缺失菌株中的表达量与其在野生型菌株中没有太大的差异,但是药物 FK506的添加,使其表达量下降了将近50%,说明CaN对酸性pH条件诱导的CCH1和MID1基因的表达是必需的。

图5 CCH1和MID1基因在野生型菌株和crz1∆/∆缺失菌株中的表达差异Fig. 5 Differential expression of Candida albicans MID1 and CCH1 in wild type and crz1∆/∆ mutant strains.
3 讨论
真核生物细胞能够通过许多信号转导机制来对各种各样的环境压力变化作出应答,钙调神经磷酸酶CaN/Crz1途径在pH变化过程中调控离子的动态平衡[17]。本文重点研究了以Cch1p/Mid1p通道为起点影响碱性 pH压力条件下的钙内流,以及 Crz1p为核心对相关基因进行调控的钙离子信号转导途径在碱性pH压力应答中的重要作用。实验中首先需要解决的问题是寻找合适的方法测定碱性 pH条件对白念珠菌细胞胞内钙波变化的影响,而报道较多的钙发光蛋白法、Fura-2荧光指示剂负载法,或是使用单细胞钙离子成像分析系统测定胞内钙离子浓度变化的方法,都只能反映单个细胞在环境变化下胞内钙波变化的情况,却不能代表整体细胞的平均水平。本文结合 Fluo-3AM荧光染料负载与流式细胞术,对碱性pH压力下胞内钙波变化动力学的检测,是微生物细胞胞内钙离子测定方法的重要改进,实现了全细胞水平,使实验结果更为科学、可信。
实验中,我们发现碱性pH条件能引起胞内钙离子浓度的瞬间变化并且这种变化在 cch1∆/∆和mid1∆/∆突变株中消失,Cch1p和Mid1p作为酵母细胞膜上高亲和力的钙离子转运系统通道,能够将胞外的钙运送至胞内,CCH1和MID1基因的缺失必然会导致这种钙内流受阻。我们的实验结果显示了CCH1和MID1基因在碱性pH诱导的胞内钙离子浓度的瞬间升高过程中发挥着重要的作用,但这并不排除胞内钙库可能向细胞中释放钙离子,从而引起胞内钙浓度升高的可能性,高的等渗压力就能够诱导液泡通过Yvc1p向胞内释放钙离子[18]。在酿酒酵母中,P-型ATP酶Pmr1p转运体能够将Ca2+和Mn2+运送回高尔基体,PMR1基因的缺失也可能会导致胞内钙离子浓度的积累[19]。所以本实验也使用同样的方法分析了保存的 yvc1∆/∆和 pmr1∆/∆突变株在碱性pH条件下胞内的钙波变化,结果发现突变菌株与野生型菌株中的钙波变化基本一致,再次证明CCH1和MID1基因在钙离子动态平衡和碱性pH条件下外钙内流的调控过程中的重要作用。
Crz1p是酵母细胞钙信号转导过程中的核心转录因子,能够特异性地与一个24 bp的CDRE序列结合,这对于钙离子诱导的、CaN依赖的基因表达是必不可少的[20]。在碱性pH条件下,PHO89基因的表达上调,在其启动子序列中很可能含有CDRE,我们的实验证实了这一点,β-半乳糖苷酶体系的建立一方面说明了pPHO89-LacZ的表达是钙离子依赖性的,另一方面在 crz1∆/∆ 突变菌株中启动子应答的消失以及在野生型菌株中添加FK506后LacZ活性的减弱,证明PHO89基因的表达是受到Crz1p严格调控的,并且是CaN依赖性的。既然Crz1p对碱性pH应答基因具有调控作用,鉴于其在钙信号转导途径中的核心角色,而CCH1和MID1基因作为压力变化时钙离子信号传递的重要通道因子,我们推测Crz1p对CCH1和MID1基因也具有一定的调控作用,所以最后利用荧光定量PCR证实了这一点,但是Crz1p对CCH1和MID1基因的调控作用不是CaN依赖性的。
总之,白念珠菌对环境pH的应答与其致病性密切相关,对钙信号途径在碱性pH压力应答中作用的研究将有助于我们进一步了解这种在医学上极为重要的病原菌是如何在宿主中存活的,对临床上念珠菌感染的治疗和新药物靶点导向具有重要的理论和实际意义。
致谢:本实验中的部分菌株和质粒由美国明尼苏达大学Dr. Dana Davis教授惠赠,在此表示衷心感谢!
[1] Kader MA, Lindberg S. Cytosolic calcium and pH signaling in plants under salinity stress. Plant Signal Behav, 2010, 5(3): 1559−2324.
[2] Bootman MD, Lipp P, Berridge MJ. The organization and functions of local Ca2+signals. J Cell Sci, 2001, 114(12): 2213−2222.
[3] Wiesenberger G, Steinleitner K, Malli R, et al. Mg2+deprivation elicits rapid Ca2+uptake and activates Ca2+/calcineurin signaling in Saccharomyces cerevisiae. Euk Cell, 2007, 6(4): 592−599.
[4] Gennemark P, Nordlander B, Hohmann S, et al. A simple mathematical model of adaptation to high osmolarity in yeast. In Silico Biol, 2006, 6: 193−214.
[5] Viladevall L, Serrano R, Ruiz A, et al. Characterization of the calcium-mediated response to alkaline stress in Saccharomyces cerevisiae. J Biol Chem, 2004, 279(42): 43614−43624.
[6] Muller EM, Locke EG, Cunningham KW. Differential regulation of two Ca(2+) influx systems by pheromone signaling in Saccharomyces cerevisiae. Genetics, 2001, 159(4): 1527−1538.
[7] Muend S, Rao R. Fungicidal activity of amiodarone is tightly coupled to calcium influx. FEMS Yeast Res, 2008, 8(3): 425−431.
[8] Brand A, Shanks S, Duncan VMS, et al. Hyphal orientation of Candida albicans is regulated by a calcium dependent mechanism. Curr Biol, 2007, 17(4): 347−352.
[9] Kullas AL, Martin SJ, Davis D. Adaptation to environmental pH: integrating the Rim101 and calcineurin signal transduction pathways. Mol Microbiol, 2007, 66(4): 858−871.
[10] Serrano R, Ruiz A, Bernal D, et al. The transcriptional response to alkaline pH in Saccharomyces cerevisiae: evidence for calcium-mediated signalling. Mol Microbiol, 2002, 46(5): 1319−1333.
[11] Gupta SS, Ton VK, Beaudry V, et al. Antifungal activity of amiodarone is mediated by disruption of calcium homeostasis. J Biol Chem, 2003, 278(31): 28831−28839. [12] Wilson RB, Davis D, Mitchell AP, et al. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J Bacteriol, 1999, 181(6): 1868−1874.
[13] Yong-Un B, Samuel JM, Dana AD. Evidence for novel pH-dependent regulation of Candida albicans Rim101, a direct transcriptional repressor of the cell wallβ-Glycosidase Phr2. Eukaryotic Cell, 2006, 5(9): 1550−1559.
[14] Gui L, Liang Y, Wei DS, et al. Regulating promoter element of iron-dependent gene FRP1 in Candida albicans by site-directed mutation. Chin J Biotech, 2008, 24(8): 1348−1353.桂磊, 梁勇, 魏东盛, 等. 利用定点突变分析白念珠菌依赖铁基因 FRP1启动子元件. 生物工程学报, 2008, 24(8): 1348−1353.
[15] Zhang YF, Jiao LX, He D, et al. Isolation method of total RNA from Candida Albicans. J Jilin Univ: Med Ed, 2007, 33(2): 359−3611.张云峰, 焦立新, 贺丹, 等. 白色念珠菌总RNA的提取方法. 吉林大学学报: 医学版, 2007, 33(2): 359−3611.
[16] Schmittgen TD, Livak KJ. Analyzing real-time PCR data by the comparative CT method. Nat Protoc, 2008, 3(6): 1101−1108.
[17] Bensen ES, Martin SJ, Li M, et al. Transcriptional profiling in Candida albicans reveals new adaptive responses to extracellular pH and functions for Rim101p. Mol Microbiol, 2004, 54(5): 1335−1351.
[18] Denis V, Cyert MS. Internal Ca2+release in yeast is triggered by hypertonic shock and mediated by a TRP channel homologue. J Cell Biol, 2002, 156(1): 29−34.
[19] Bates S, MacCallum D M, et al. Candida albicans Pmr1p, a secretory pathway P-type Ca2+/Mn2+-ATPase, is required for glycosylation and virulence. J Biol Chem, 2005, 280(24): 23408−23415.
[20] Stathopoulos AM, Cyert MS. Calcineurin acts through the CRZ1/TCN1-encoded transcription factor to regulate gene expression in yeast. Genes Dev, 1997, 11(24): 3432−3444.
