山葡萄核心种质初步构建1)
2011-01-17温景辉申海林邹利人刘洪章
温景辉 申海林 邹利人 陈 蕾 刘洪章
(吉林农业大学,长春,130118) (吉林省农业科学院) (吉林农业大学)
山葡萄核心种质初步构建1)
温景辉 申海林 邹利人 陈 蕾 刘洪章
(吉林农业大学,长春,130118) (吉林省农业科学院) (吉林农业大学)
采用扩增稳定且谱带清晰的18对SSR引物,对360份山葡萄资源进行遗传多样性分析。用逐级压缩法选择核心种质,比较了样本数不同的5个核心样本群的等位基因数、等位基因频率、有效等位基因数、Nei’s遗传多样性和Shannon’s信息指数等参数,最终选择了79个样本组成的样本群作为核心种质。用DPS软件对初始种质和核心种质群体的有效等位基因数、Nei’s遗传多样性和Shannon’s信息指数分别作t测验,核心种质保留了初始种质21.9%的样品,等位基因数、有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数的保留率分别为94.1%、106.3%、101.9%、102.2%,核心种质能很好地代表初始种质。
山葡萄(Vitis amurensis Rupr.);种质资源;遗传多样性;SSR;核心种质
植物遗传资源是新品种选育和种质创新的基础,种质资源的收集、保存、评价和利用非常重要[1-2]。Hintum 等[3]认为核心种质库能以最少的种质材料代表一个种及野生近缘种的形态特征、地理分布、基因与基因型最大范围的遗传多样性,可作为有利基因挖掘、新技术应用和资源深入研究的优先样品集,能够提高种质资源的利用效率。然而,数量庞大的种质资源不便于优良种质的保存、评价和利用,尤其是多年生木本果树植物,个体占用土地面积较大、管理费用较高,不便于种质资源的有效管理和深入研究。
DNA分子标记数据能够直接反映出DNA序列水平上的变化,具有多态性高、不受环境影响和基因表达与否的限制等特点,近年来逐渐成为检测植物种质遗传多样性、构建高质量核心种质的有效特征数据。目前已在芝麻[4-5]、水稻[6]、大豆[7]、豌豆[8]、桃[9]、果梅[10]等植物上建立了核心种质,但利用分子标记技术对我国山葡萄资源进行核心种质构建鲜有报道。笔者以中国农科院左家特产研究所的国家山葡萄资源圃现存的360份山葡萄资源为材料,采用SSR分子标记技术,初步构建了山葡萄种质资源核心种质,旨在为今后山葡萄种质资源的开发利用和遗传育种提供科学依据。
1 材料与方法
1.1 材料
供试山葡萄资源360份由中国农业科学院特产研究所国家果树种质山葡萄资源圃提供。
1.2 分子标记方法
采用SSR方法进行遗传多样性分析。SSR标记技术见文献[11]。引物序列由东北林业大学提供,上海生工合成。采用聚丙烯酰胺凝胶电泳。聚类分析采用Ntsys统计软件,数据处理采用EXCEL电子表格。每个标记位点,以1和0记录等位基因的有和无。
1.3 核心种质的遗传多样性评价参数
选择等位基因数、等位基因频率(pi)、有效等位基因数(A)、Nei’s遗传多样性(H)、Shannon’s多样性信息指数(I)等指标评价核心种质的遗传多样性[11]。
1.4 核心种质构建方法
本试验采用多次聚类结合优先取样法,其具体原理如下:首先判断处于最低分类水平的每组两个遗传材料的性状是否具有极端表现,并优先选取具有极端表现的个体。如果两个体性状都具有极端表现,则两个个体都被优先选入核心库,如果都不具有极端的表现则采用随机取样的方法剔除其中一个。对资源多次聚类,采用逐级压缩方法,建立样本群,直到代表性或核心种质达到要求,组成核心群体。
用上述方法分别抽取292、204、134、79、53个样品组成核心样本群进行分析比较,样品群代号分别为1、2、3、4、5。
1.5 核心种质数据分析
将从SSR标记中得到的原始数据输入计算机,通过EXCEL电子表格计算各样本群的等位基因数、等位基因频率、有效等位基因数、Nei’s遗传多样性和 Shannon’s信息指数,并用DPS(v7.05版)软件对所得数据进行t测验。
2 结果与分析
2.1 各样本群的遗传多样性比较
根据评价遗传多样性公式,采用EXCEL电子表格计算得出了各样本群的等位基因数、等位基因保留率、等位基因频率、有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数,结果见表1。比较不同取样数目所获得的各种遗传参数,可知样本群4代表的79份样品除等位基因频率低于样本群5外,有效等位基因数、Nei’s遗传多样性和 Shannon’s信息指数都是最高的,应把79份样本作为核心种质。
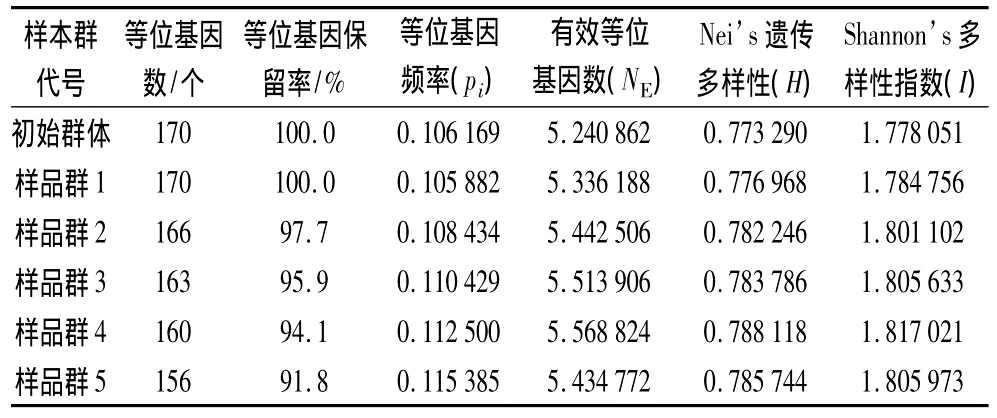
表1 各样本群的遗传多样性比较
2.2 初始种质与核心种质比较
初始种质与核心种质的等位基因数、有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数,结果见表2。从表2可看出核心种质保留了初始种质21.9%的样品,等位基因数、有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数的保留率分别为94.1%、106.3%、101.9%、102.2%。

表2 核心种质与初始种质遗传多样性比较
用DPS软件对2个群体的有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数分别作t测验,结果见表3。可以看出核心种质的有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数在概率0.05水平上与初始种质差异不显著,可见核心种质能很好的代表初始种质群体基因型值的多样性。

表3 初始种质与核心种质t测验结果
2.3 保留种质与核心种质比较
保留种质即原始种质除去核心种质后保留下来的种质资源,这部分种质作为后备资源,当在核心种质中无法找到所需性状时,就可从保留种质中寻找。核心种质与保留种质的等位基因数、有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数,结果见表4。

表4 核心种质与保留种质遗传多样性对比
用DPS软件对核心种质和保留种质的有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数分别做t测验,结果如表5。可以看出,在概率0.05水平上核心种质的有效等位基因数、Nei’s遗传多样性和Shannon’s信息指数与保留种质差异不显著。但从表4可知核心种质的有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数均高于保留种质,应该优先使用。
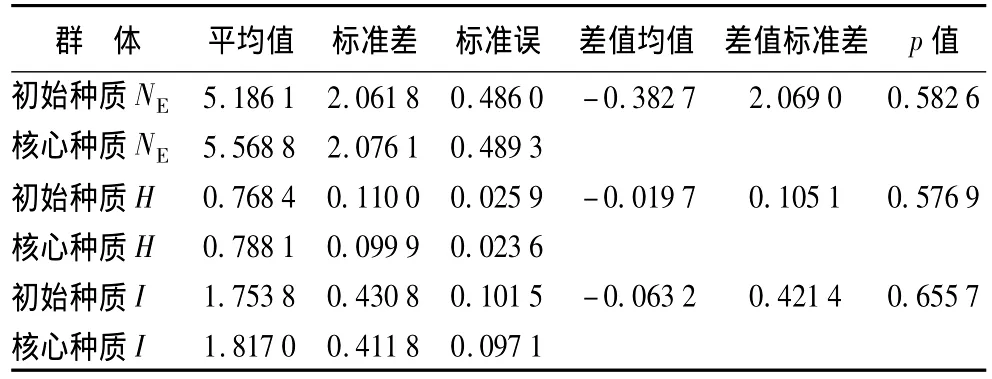
表5 核心种质与保留种质t测验结果
2.4 山葡萄资源核心种质主要性状
79份核心种质中包含两性花、雌能花和雄株3个类群。其中两性花31份,占两性花样本的21.7%;雌能花46份,占雌能花样本的21.7%;雄株2份,占雄株样本的40%。
3 讨论
3.1 核心种质的代表性、异质性和多样性
构建核心种质时要选择有代表性、异质性和多样性、类型齐全的样本作为核心种质。本研究采用SSR标记方法构建的核心种质中,不仅包含野生资源、人工培育品种(系)2大类,而且包含某些特殊性状的种质,如除两性花外,还有雌能花和雄株等。初始种质与核心种质相比,有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数分别经t测验差异不显著;同时,核心种质的有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数也均高于保留种质。这说明所构建的79份核心种质是山葡萄资源遗传多样性很高的种群,具有很好的代表性。
3.2 核心种质的构建方法
以往核心种质的构建多采用形态学方法[4-8]构建,形态农艺性状数据作为研究核心样品的指标,有简便和研究费用低的优点,但易受环境和基因显隐性的影响。自分子标记广泛应用于植物遗传资源研究以来,RFLP、RAPD、AFLP、SSR等标记已经逐渐成为评价植物资源多样性、构建核心种质的有效方法。本研究运用SSR标记,成功地初步构建了360份山葡萄资源的核心种质,具有一定应用价值。分子标记能从DNA水平检测材料的遗传差异,但分子标记不是直接控制形态一农艺性状变异的基因,仅用分子标记研究核心种质也存在着不足。如何整合这两类数据构建核心种质是核心种质研究中仍需解决的问题。
3.3 核心种质的取样比例
在国内外不同植物核心种质库的构建中总体取样比例为该物种总资源的5%~30%,或总量不超过3 000份[12]。由于生物进化及人工选择对作物的干预,产生了各个物种独特的特性,对整体取样比例的确定不能格式化、简单化,应视研究作物的遗传结构及遗传多样性而定。目前,在已构建的多种果树资源核心种质的取样比例均较高,取样比例为10%~30%。其中,张春雨[13]利用SSR标记构建的新疆野苹果核心资源取样比例为22.9%,刘勇[14]等构建的柚类核心资源取样比例为22.73%,程丽莉[15]构建的燕山地区实生板栗核心种质取样比例为27.2%,高志红[16]构建的果梅核心资源取样比例为10%,马玉敏[17]构建的野生板栗核心资源取样比例为20%。在本研究中,通过采用SSR分子标记,经过遗传多样性评价和显著性t测验,确定了我国山葡萄资源核心种质合适的取样比例为初始种质的21.9%。
4 结论
采用SSR分子标记,选择组内聚类逐步压缩取样方法,对360份山葡萄资源进行核心种质构建,获得了包含79份资源的核心种质。核心种质保留了初始种质21.9%的样品,等位基因数、有效等位基因数、Nei’s遗传多样性、Shannon’s信息指数的保留率分别为94.1%、106.3%、101.9%、102.2%,核心种质能很好的代表初始种质群。
致谢:感谢中国农科院特产研究所提供试验材料及农艺性状数据,感谢中国农业大学王军教授提供引物序列,特此致谢。
[1] 章秋平,刘威生,刘宁,等.普通杏(Prunus armeniaca)初级核心种质资源的构建及评价[J].果树学报,2009,26(6):819-825.
[2] 李保印,张启翔.我国园艺作物核心种质研究进展[J].果树学报,2007,24(2):204-209.
[3] van Hintum T J L,Brown A H D,Spillane C,et al.Core collections of plant genetic resources[M].Rome:Wiley Publication,1995.
[4] Zhang X,Zhao Y,Cheng Y,et al.Establishment of sesame germplasm core collection in China[J].Genetic Resources and Crop E-volution,2000,47:273-279.
[5] Kang C W,Kim S Y,Lee S W,et al.Selection of a core collection of Korean sesame germplasm by a stepwise clustering method[J].Breeding Science,2006,56:85-91.
[6] Hu J,Zhu J,Xu H M.Methods of constructing core collections by stepwise clustering with three sampling strategies based on the genotypic values of crops[J].Theoretical and Applied Genetics,2000,101(1):264-268.
[7] Wang J C,Hu J,Xu H M,et al.A strategy on constructing core collections by least distance stepwise sampling[J].Theoretical and Applied Genetics,2007,115(1):1-8.
[8] Upadhaya H D,Ortiz R.A mini core subset for capturing diversity and promoting utilization of chickpea genetic resources in crop improvement[J].Theoretical and Applied Genetics,2001,102(8):1292-1298.
[9] 李银霞,安丽君,姜全,等.桃(Prunus persica(L.)Batsch.)品种核心种质的构建与评价[J].中国农业大学学报,2007,12(5):22-28.
[10] 高志红,章镇,韩振海,等.中国果梅核心种质的构建与检测[J].中国农业科学,2005,38(2):363-368.
[11] 徐海明.种质资源核心库构建方法的研究及其应用[D].杭州:浙江大学,2005.
[12] Frankel O H,Brown A H D.Plant genetic resources today:a critical appraisal[M]//Holden J H W.Crop genetic resources:conservation and evaluation.London:George Allen and Unwin Press,1984:249-257.
[13] 张春雨.新疆野苹果(Malus sieversii)群体遗传结构与核心种质构建方法[D].泰安:山东农业大学,2008.
[14] 刘勇,孙中海,刘德春,等.利用分子标记技术选择柚类核心种质资源[J].果树学报,2006,23(3):339-345.
[15] 程丽莉.燕山板栗实生居群遗传多样性研究与核心种质初选[D].北京:北京林业大学,2005.
[16] 高志红.果梅核心种质的构建与分子标记的研究[D].北京:中国农业大学,2003.
[17] 马玉敏.中国野生板栗(Castanea mollissim Blume)群体遗传结构和核心种质构建方法[D].泰安:山东农业大学,2009.
Primary Establishment of Core Collection of Vitis amurensis in Northeast China
/Wen Jinghui(School of Life Sciences,Jilin Agricultural University,Changchun 130118,P.R.China);Shen Hailin,Zou Liren,Chen Lei(Jilin Academy of Agricultural Sciences);Liu Hongzhang(Jilin Agricultural University)//Journal of Northeast Forestry University.-2011,39(6).-35~37
Vitis amurensis;Germplasm resources;Genetic diversities;SSR;Core collections
S663.1
1)现代农业产业技术体系建设专项资金资助(nycytx-30)。
温景辉,男,1963年10月生,吉林农业大学生命科学学院,博士研究生,工作于吉林省农业科学院,研究员。
刘洪章,吉林农业大学生命科学学院,教授。E-mail:lhz999@126.com。
2010年12月17日。
责任编辑:戴芳天。
Eighteen pairs of SSR primers with steady amplification and clear spectrum were used to analyze the genetic diversity of 360 accessions of Vitis amurensis Rupr.in northeast China.Stepwise condensation method was used to screen the core collection.A total of 79 samples were chosen as core collection by comparing the parameters of 6 sample groups,including number of alleles,allele frequency,effective number of alleles,Nei’s genetic diversity,and Shannon information index.T-test for initial collection and core collection was performed by using DPS software.Results showed that the core collection hold 21.9 percent of samples of initial collection,and the number of alleles,the effective number of alleles,Nei’s genetic diversity,and Shannon information index reached 94.1%,106.3%,101.9%and 102.2%respectively.It is demonstrated that the core collection can express initial collection well.
