汾酒大曲和酒醅样品DNA提取方法的优化*
2011-01-13闫亮珍李晓然全哲学马恩波
闫亮珍,李晓然,全哲学,马恩波
汾酒历史悠久、风味独特,是清香型白酒的典型代表,产于山西省杏花村,有着几千年的传统工艺酿造历史。汾酒酿造工艺流程中所含有的微生物种群是构成其独特风味的主要因素之一[1]。
PCR技术现已成为功能基因组学研究广泛应用的技术[2]。在 PCR扩增过程中,模板 DNA的提取质量及纯度往往是决定 PCR能否扩增出目标条带的限制因素[3]。DNA提取是分子生物学研究中的关键技术,其结构的完整性和纯度直接决定后续实验能否顺利进行[4]。提取汾酒酒曲及酒醅微生物总 DNA,对其中细菌特异 DNA序列 16S rDNA基因及真菌的ITS序列进行特异性扩增,通过扩增分析 PCR产物,可鉴定汾酒酒曲和酒醅样品的微生物种类及主要优势菌。
目前用于微生物 DNA的提取有很多种方法,如Martin法、高盐改进法及试剂盒法等[5],虽然试剂盒法操作简单,能获得较高质量的 DNA,但据文献报道,此种方法 DNA得率较低且成本昂贵,因此选择一种可靠的适合汾酒样品的DNA提取方法显得尤为重要。常用的DNA提取方法主要有以下 3种:(1)物理方法,即通过外力作用破坏细胞结构,主要有液氮冻融法、玻璃珠振荡法、液氮研磨法、超声振荡法和微波热休克法等。理论上,物理方法是最有可能得到微生物群落完整 DNA的方法,但由于物理方法较为剧烈,得到的 DNA片段往往较小。(2)化学法,最常用的化学裂解试剂为十二烷基硫酸钠 (SDS),它可以溶解细胞膜上的疏水材料,其它试剂如洗涤剂等通常在热处理时与螯合剂 (如 EDTA)共同使用。(3)酶法,常用的为溶菌酶,主要用于细菌 DNA的提取,加入其它酶如溶壁酶可用于提取真菌 DNA,蛋白酶 K用于处理DNA中的蛋白质污染等[6]。本文综合分析了上述各种方法的优越性,采用酶物理化学法的 DNA提取方法,不仅获得了汾酒样品中高质量的微生物DNA,而且成本较试剂盒提取法为低。
1 材料与方法
1.1 材料与主要试剂
样品:汾酒酒曲、汾酒酒醅和环境样品均取自山西杏花村汾酒股份有限公司。
主要实验试剂:Lysozyme(溶菌酶 )、15μL 15 mg/mL Lyticase(溶壁酶 )、蛋白酶 K、DH5α感受态大肠杆菌,均购于 TI ANGEN公司;pMD18-T,购于 TaKa-Ra公司;玻璃珠,购于 Sigma公司。
Lysing buffer(配制):0.1 mol/L Tris-HCl,0.1 mol/L EDTA(pH值 8.0),0.75 mol/L蔗糖。
Extraction Buffer(配制):0.1 mol/L磷酸盐,0.1 mol/L EDTA(pH值8.0),0.1 mol/L Tris-HCl(pH 7.0),1.5 mol/L NaCl,1%CTAB。
1.2 样品总 DNA提取方法
1.2.1 酶法 +液氮冻融法 +化学法 (即酶物理化学法[7])
1.2.1.1 样品预处理
(1)酒醅样品预处理
酒醅处理方法 1:取约 250μL(约 0.25 g)样品置于 2 mL离心管中,加 1 mL无菌水,振荡器振荡 1 min,10 000 r/min离心 5 min,去掉液体,重复 3次。
酒醅处理方法 2:取约 250μL(约 0.25 g)酒醅置于 2 mL离心管中,加入 1 mL无菌水,振荡器振荡 1 min,振荡完后直接取振荡浑浊液 (不含酒醅中的颗粒物质)于一新的离心管中,10 000 r/min离心 5 min,去掉上清液,重复 3次。
(2)酒曲样品预处理
酒曲曲块质地比较坚硬,在取样之前需先将曲样粉碎成均匀的粉末。用已灭菌处理过的药勺取约250μL(约 0.1 g)已粉碎好的样品置于 2 mL离心管中,加 1 mL无菌水,于振荡器振荡 1 min,10 000 r/min离心 5 min,去掉液体,重复 1~2次。
(3)环境样品预处理
环境样品的采样方法是将环境微生物吸附于滤纸或棉球上。在样品处理步骤中用已灭菌的剪刀和镊子将滤纸和棉球剪碎,加入 1 mL无菌水,振荡器振荡 1 min,10 000 r/min离心 5 min,去掉上清液。
1.2.1.2 DNA的提取
(1)加 450μL Lysing buffer,10μL 50 mg/mL Lysozyme(溶菌酶 )和 15μL 15 mg/mL Lyticase(溶壁酶),37℃水浴 30 min。(2)加入 25μL 20%SDS和5μL 20 mg/mL蛋白酶 K。(3)55℃水浴 2 h,加 0.1 g(约 100μL)玻璃珠,剧烈振荡 5 min。(4)液氮冻融3次。 (5)加入 80μL 5 mol/L NaCl和 60μL 10%CTAB/NaCl,混匀,65℃水浴 20 min。(6)加入等体积的酚 /氯仿 /异戊醇 (25∶24∶1),混匀。 (7)12 000 r/min离心 10 min,把上清液转移到新的 1.5 mL离心管中,若上层水相很混浊或颜色深,需重复步骤 7~8。(8)加等体积的三氯甲烷 /异戊醇 (24∶1),混匀。(9)12 000 r/min离心 10 min,把上清液转移到新的1.5 mL离心管中,若上层水相很混浊或颜色深,需重复步骤 9~10。(10)加 0.6倍体积的异丙醇,-20℃过夜。(11)12 000 r/min离心 15 min,尽量倒掉液体。(12)用 500μL预冷 70%乙醇洗涤,12 000 r/min离心 15 min。(13)弃上清液,65℃烘干 30 min,至烘干为止,最后加 50μL TE溶解,保存。
1.2.2 液氮研磨法 +化学法 (即物理化学法[8])
(1)称取 2 g样品和 1 g玻璃珠置于已灭菌的研钵中。(2)加液氮覆盖样品并于解冻前用力研磨,重复本步骤 3~4次,把样品转移至 50 mL离心管。(3)加 9 mL Extraction Buffer,小心混匀。(4)60℃水浴 3 min。 (5)加 1 mL 20%SDS,小心混匀。(6)60℃水浴 15 min,每 5 min摇匀 1次。(7)4 400 r/min离心 10 min。(8)将上清转移至新的 50 mL离心管中,沉淀重复步骤 3~7。(9)合并上清液,4400 r/min离心 10 min。(10)将 CTAB覆盖下的液体转移到一个新的 50 mL离心管中,加等体积的氯仿 /异戊醇 (24∶1),4 400 r/min离心 20 min。(11)收集上清水相,加 0.6体积的异丙醇小心混匀,室温沉淀 30 min。(12)16 000 r/min离心 20 min。(13)小心倒掉液体,65℃烘干 30 min,至烘干为止。(14)加 120μL TE溶解。
1.3 细菌 16S rDNA及真菌 ITS DNA片段的 PCR扩增
采用对细菌 16S rDNA具有特异性的通用引物(Muyzer et al.,1993)。正向引物 27f:5’-AGA GTT TGA TCM TGG CTC AG-3’,反向引物 1512r:5’-ACG GHTACC TTG TTA CGA CTT-3’。真菌 ITS区特异性正向引物 NAS3:5’-AAA CTC TGT CGT GCT GGG GATA-3’,反向引物 NLC2:5’-GAG CTG CAT TCC CAA ACA ACT C-3’。引物由北京 Invitrogen公司合成,Master Mix购于天根生化科技 (北京)有限公司。在 0.2 mL的离心管中配制 PCR反应体系 (表1)。PCR反应条件为 95℃预变性 5 min,94℃变性 5 min,56℃退火 1 min,72℃延伸 1 min 30 s,反应 30个循环,最后 72℃延伸 15 min,4℃结束。

表1 PCR反应体系
1.4 1%琼脂糖凝胶电泳检测
1.5 克隆文库构建及测序分析
胶回收产物连入载体 pMD18-T并转入 DH5α感受态大肠杆菌中,转化培养建立克隆文库,对每个克隆文库随机挑取克隆进行测序。
1.6 克隆文库的微生物多样性分析
使用 DOTUR软件对相似度大于 97%(即 cut off值 <0.03)的基因序列进行操作分类单元 (OTU)归类。采用 Chao丰富度指数,ACE丰富度指数以及rarefaction指数值进行微生物物种的多样性分析。
1.7 实验结论的验证
为了验证酶物理化学法是否对其它样品DNA也有很好的提取效果,我们选择了其它的酒醅和酒曲样品进行DNA提取和 PCR扩增。
2 结果与分析
2.1 酒醅样品两种不同预处理方法结果
在酒醅 2种不同预处理方法比较中,DNA提取方法采用的是酶化学物理法。图1中 1、2、6、7为酒醅处理方法 2得到的结果,3、4、8、9为酒醅处理方法1得到的结果,5、10是空白对照,从图中可以看出处理方法 1较处理方法 2有更好的DNA得率。
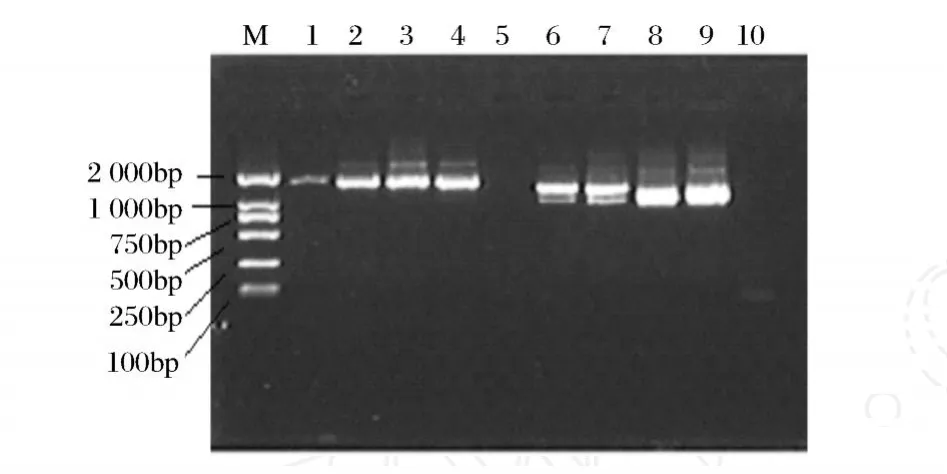
图1 酒醅样品不同预处理方法 PCR产物的电泳检测结果
2.2 酶物理化学法与物理化学法比较
本文将物理化学法和酶化学物理法 2种不同的DNA提取方法进行了比较,样品预处理采用较好的样品处理方法 1(见图1),对得到的细菌和真菌的克隆文库进行了群落组成分析 (见表2和表3)。结果表明,通过酶物理化学法可以得到更多的汾酒微生物分类信息,同时由于酶在适宜条件下高效专一的特点,使得酶物理化学法操作起来简便、省力。
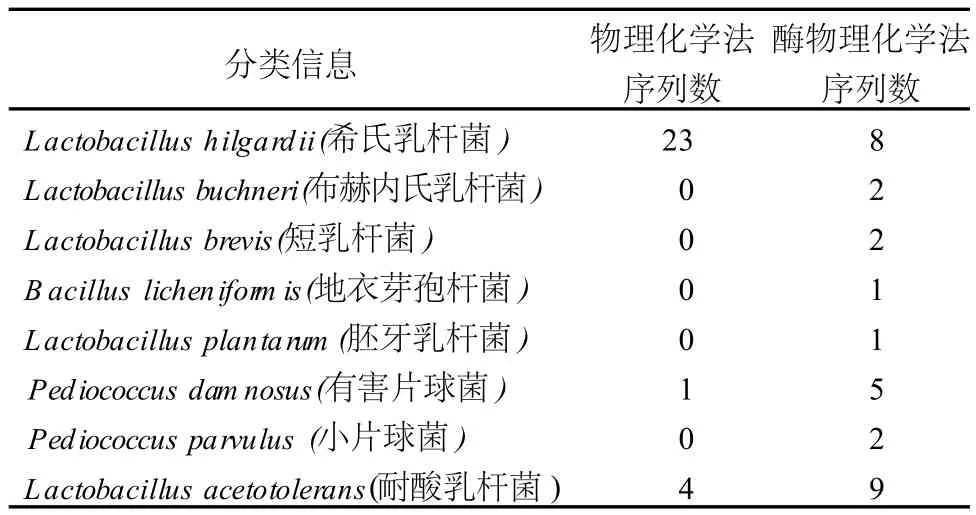
表2 两种 DNA提取方法得到的细菌克隆文库比较
2.3 两种不同 DNA提取方法得到的微生物多样性比较分析
多样性计算结果见表4。从表4可以看出,酶物理化学法得到的真菌和细菌的 Chao丰富度指数明显高于物理化学法得到的种类,同样,酶物理化学法得到的微生物 ACE丰富度指数也明显高于物理化学法,rarefaction值的差异虽然没有 Chao和 ACE明显,但同样也说明了酶物理化学法的优越性。由此可见酶物理化学法提取DNA更能体现酒曲样品中微生物种类的多样性。
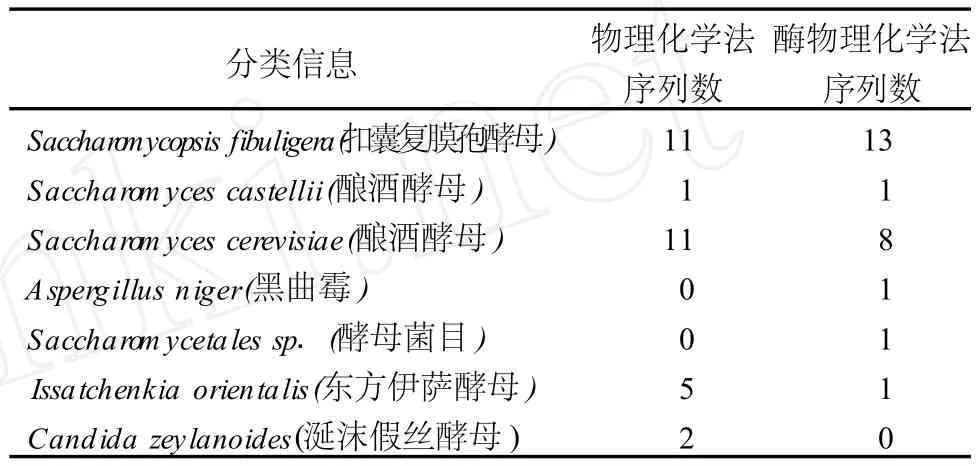
表3 两种 DNA提取方法得到的真菌克隆文库比较
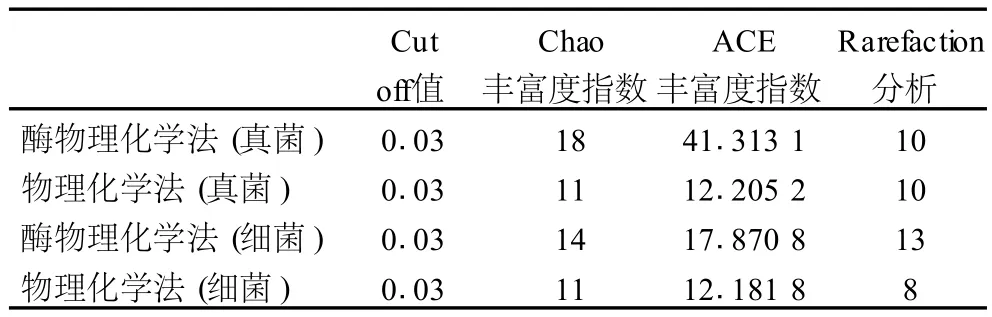
表4 不同DNA提取方法得到的微生物多样性比较分析
2.4 酶物理化学法提取的总 DNA及 PCR产物检测结果

图2 汾酒样品总DNA的检测结果
为了进一步验证酶物理化学法 (酒醅样品预处理采用方法 1,酒曲样品预处理见 b)的可行性,我们对其它酒醅和酒曲样品进行了DNA提取。从图2中可以看出采用酶物理化学法,获得了清晰可见的基因组 DNA条带,这为 16S r DNA和 ITS特异片段的 PCR扩增、特异片段克隆培养及测序提供了重要条件。图3为 PCR产物的电泳结果,得到了清晰的扩增条带,也证实了DNA的成功提取。

图3 酒醅不同时期 PCR产物的琼脂糖凝胶电泳检测结果
3 讨论
常规的微生物DNA提取多根据不同微生物的特点或具体实验要求选择合适的提取方法。汾酒酒醅和酒曲样品及环境样品中微生物含量丰富,种类繁多,从中提取的 DNA可用于分析在常规培养条件下难以培养出的微生物,跟踪其中某些主要菌株的微生物生长情况,亦可用来分析酒曲样品中微生物种类的多样性及其随时间和环境条件的变化情况[9]。因此,从汾酒不同样品中提取高质量的 DNA就成为最关键的技术之一。张宁等对各种DNA提取方法进行研究总结[9],虽然不同生物的 DNA提取方法不尽相同,但各种方法亦可相互借鉴。
汾酒酒醅中含有发酵过程所产生的酒精等微生物次级代谢产物,而微生物次级代谢产物无疑对后续DNA提取时所加入的酶,如溶菌酶 (Lysosome)、溶壁酶 (Lyticase)、蛋白酶 K(Proteinase K)等活性产生抑制,所以在进行酒醅 DNA的提取时,我们通过加入无菌蒸馏水反复洗涤离心的方法达到去除残留酒精的目的;对于酒曲样品 DNA的提取,无菌蒸馏水反复洗涤离心这一步骤也很重要,原因是不同时期酒曲样品中水分含量很少,曲块硬,导致其吸水性强,如果不进行蒸馏水的反复洗涤,直接加入 lysing buffer以及溶菌酶及溶壁酶,酒曲样品会将 lysing buffer全部吸收,从而使得溶菌酶及溶壁酶起不到酶解的作用;环境样品的取样具有一定的特殊性,一般是通过滤纸和棉球吸附的方式取样,DNA提取前的样品处理尤为重要,处理的方法是在加入无菌蒸馏水的情况下,将吸附有微生物的滤纸和棉球用剪刀剪尽量剪碎。这样在离心后滤纸和棉球能够沉淀于离心管底部,而不是贴在离心管壁上,保证 DNA的顺利提取。对于不同汾酒样品 DNA的提取,样品前处理是一个相对耗时的过程,但此步骤很关键,一旦样品处理好,之后就可以进行快速、大量的 DNA提取。另外,细胞破碎对真菌DNA的提取也是很重要的步骤。本文采用酶裂解法以及液氮冻融法处理真菌细胞壁,即物理破壁法,保证了真菌DNA的顺利提取。
酒醅样品的不同处理方法结果表明,汾酒酒醅样品微生物大多富集在酒醅中的谷物上,方法 2通过涡旋振荡可以将其洗涤下来,但由于洗涤不完全,导致实验不能将酒醅样品中的微生物DNA尽量完整地提取出来,这无疑为酒醅样品中微生物群落多样性的分析带来影响。而方法 1不需要细胞提取,且提取的DNA更能代表样品的微生物群落,它的缺点是提取DNA的同时也提取了样品中的其他成分,有些成分会干扰后续实验[10]。方法 2是在裂解释放 DNA之前,先将微生物细胞从它们的环境基质中分离出来,这一方法的局限主要是很多细菌会吸附在样品颗粒上不能被分离出来,结果将会导致所反映的样品中微生物群落具有片面性,本文结果也说明了这一点 (见图1)。综上所述,方法 1与方法 2各具优缺点,实际操作中要根据实验的具体情况选择合适的方法,本实验中由于汾酒酒醅本身所具有的特殊性,即不同发酵阶段的酒醅样品对微生物的吸附程度有所不同,所以采用了酒醅处理方法 1,该方法操作简单易行,同时也消除了方法 2中洗涤不完全因素的影响,保证了酒醅样品中微生物 DNA的完全提取。因此,方法 1较方法 2为好。
酒醅样品不同 DNA提取方法结果表明,利用酶物理化学方法可从汾酒样品中将真菌及细菌的基因组 DNA完整地提取出来,后续克隆测序结果表明,通过酶化学物理法提取获得的高质量DNA能全面反映汾酒制备过程中微生物群落结构的多样性。
综上所述,对于酒醅样品预处理来说,方法 1较好;对于样品不同的 DNA提取方法,酶物理化学法较好,因此采用酶物理化学法结合酒醅样品处理方法 1进行DNA的提取。
[1] 王佳丽,王丽,李凯,等.汾酒大曲中红曲霉的分离和鉴定[J].山西大学学报:自然科学版,2009,32(2):289-293.
[2] Ray Chaudhuri S,Pattanayak AK,Thakur AR.Microbial DNA extraction from samples of varied origin[J].Current Science,2006,91(12):1 697-1 700.
[3] Manuel Porcar,Silvia Ramos A,Mparo Latorre.A si mple DNA extractionmethod suitable for PCR detection of geneticallymodified maize[J].Journal of the Science of Food and Agriculture,2007,87:2 728-2 731.
[4] 张瑞福,曹慧,李顺鹏,等.土壤微生物总 DNA的提取和纯化[J].微生物学报,2003,43(2):276-282.
[5] 王丽娜,许修宏,宛煜嵩,等.三种土壤微生物总 DNA提取方法的比较 [J].基因组学与应用生物学,2009,28(2):331-334.
[6] Robe P,Nalin R,Capellano C,et al.Extraction of DNA from soil[J].European Journal of Soil Biology,2003,39:183-190.
[7] Schmidt TM,DeLong E F,Pace N R.Analysis of a marine picoplankton community by 16S rRNA gene cloning and sequencing[J].Journal of Bacteriology,1991,173(14):4 371-4 378.
[8] Zhou J,BrunsMA,Tiedje JM.DNA recovery from soils of diverse composition[J].Applied EnvironmentalMicrobiology,1996,62(2):316-322.
[9] 张宁,王凤山.DNA提取方法进展[J].中国海洋药物杂志,2004,98(2):40-46.
[10] Ogram A,G S Sayler,TBarkay.The extraction and purification ofmicrobialDNA from sediment samples[J].Journal ofMicrobiologicalMethods,1987,2-3(7):57-66.
