3个花鳗鲡地理种群的AFLP分析
2011-01-11朱友芳林凤钦王艺磊谢芳靖
朱友芳, 林凤钦, 王艺磊, 谢芳靖
(1. 福建省莆田市水产科学研究所, 福建 莆田 351100; 2. 集美大学 水产学院 福建省高校水产科学技术与食品安全重点实验室, 福建 厦门 361021)
3个花鳗鲡地理种群的AFLP分析
朱友芳1, 林凤钦2, 王艺磊2, 谢芳靖2
(1. 福建省莆田市水产科学研究所, 福建 莆田 351100; 2. 集美大学 水产学院 福建省高校水产科学技术与食品安全重点实验室, 福建 厦门 361021)
应用AFLP分子标记技术首次对澳大利亚、海南岛和菲律宾3个种群的共60尾花鳗鲡(Anguilla marmorata)幼鱼样品进行了遗传多样性分析。4对选择性扩增引物共扩增得到180个位点, 平均每对引物扩增出45个位点。澳大利亚、海南岛和菲律宾3个种群的多态位点分别为78.33%、79.44%和84.44%,Nei′s遗传多样性指数分别为 0.330 2±0.188 9、0.337 2±0.194 6 和 0.357 1±0.176 7, Shannon′s多样性指数分别为0.477 3±0.264 7、0.484 4±0.270 2和0.514 6±0.243 3; 根据基因分化系数和AMOVA分析估算,3个花鳗鲡种群遗传变异分别有89.25%和96.07%存在于种群内, 10.75%和3.93%存在于种群间。基因分化系数、AMOVA分析、遗传距离、基因流、系统树和显性基因型频率分析结果表明: 3个花鳗鲡种群间出现了遗传分化, 地理距离越大遗传分化程度越高, 三者之间出现一定的基因交流。研究结果对花鳗鲡养殖具有指导意义。
花鳗鲡(Anguilla marmorata); 种群; AFLP; 遗传变异
花鳗鲡(Anguilla marmorata)隶属鳗鲡目(Anguilliforme)、鳗鲡科(Anguillidae)、鳗鲡属(Anguilla), 属热带和亚热带江海洄游鱼类, 分布范围广, 东达太平洋中部诸岛, 西达非洲东部, 南达澳大利亚南部, 北达朝鲜、日本南部, 中国产于长江以南至海南岛各江河水域[1-2]。
有关不同种群花鳗鲡的骨骼结构差异及遗传多样性已有研究报道, Watanabe等[3]通过对花鳗鲡脊椎骨的数目研究, 得出了密克罗西尼亚岛(Micronesia)的花鳗鲡脊椎骨的数目与印度洋及太平洋其他12个花鳗鲡种群存在明显差异的结论; Minegishi等[4]采用线粒体DNA和微卫星分析的方法, 得出了可以将花鳗鲡划分为南太平洋、北太平洋、东印度洋和西印度洋4个种群的结论; Gong等[5]采用微卫星分析的方法, 得出了花鳗鲡的 Aus(澳大利亚)种群和中国种群的多样性指数相似、所有位点均符合哈迪-温伯格平衡的结论; 齐兴柱等[6]通过对线粒体细胞色素b基因的测序, 得出了花鳗鲡的日本和夏威夷种群的地理差异小于Hai(中国海南岛)种群与它们之间的地理差异的结论。
扩增片段长度多态性(Amplified Fragment Length Polymorphism,简称 AFLP)具有多态性强, 谱带丰富且清晰可辨, 实验结果稳定性、重复性好的特点, 可以在一次实验中同时观察到大量的限制性片段。因此, AFLP技术广泛应用于种质鉴定、基因克隆和定位、遗传图谱构建和遗传差异分析等研究[7-12]。目前中国花鳗鲡养殖的苗种主要来源于澳大利亚、海南岛和菲律宾三地,三者在水温、驯食和抗病力等方面均存在差异。为了分析产生这些差异的原因, 本研究首次采用 AFLP技术对花鳗鲡的 Aus种群、Hai种群和 Phi(菲律宾)种群进行遗传多样性分析, 研究结果对花鳗鲡养殖具有一定的指导意义。
1 材料和方法
1.1 实验鱼
收集 Aus墨尔本附近海域、Hai东海岸和 Phi棉兰老岛东部的花鳗鲡幼鱼各 20尾, 取肌肉固定于95%的酒精中, 4℃保存备用。
1.2 基因组DNA提取
DNA提取和纯化参照 Sambrook等[13], 采用苯酚/氯仿法提取总DNA。DU640紫外分光光度仪测定DNA浓度, 用无菌双蒸水将模板的一部分稀释到20 mg/L, 置于4℃中保存, 其余的未稀释的模板母液放于-20℃冰箱中备用, 通过 1%琼脂糖凝胶电泳检测DNA的完整性以及纯度, 凝胶电泳采用GDST500紫外凝胶成像分析系统。
1.3 AFLP指纹图谱构建
参照 Vos等[14]和 Wang等[15]方法构建 AFLP指纹图谱。AFLP接头和预扩增引物序列见表1。2种内切酶分别为EcoR I和Mse I。经过预实验从8种EcoR I引物(E-AA、E-AC、E-AG、E-AT、E-TA、E-TC、E-TG和 E-TT)和 8种 Mse I引物(M-CAA、M-CAC、M-CAG、M-CAT、M-CTA、M-CTC、M-CTG和 M-CTT)共 64对引物组合中, 筛选出 4对选择性扩增引物组合(4对引物组合分别为: E- TG/M-CAC,E-TG/M-CAT, E-AA/M-CAC, E-AA/M-CAA)进行AFLP分析。AFLP试剂盒购自invitrogen公司。
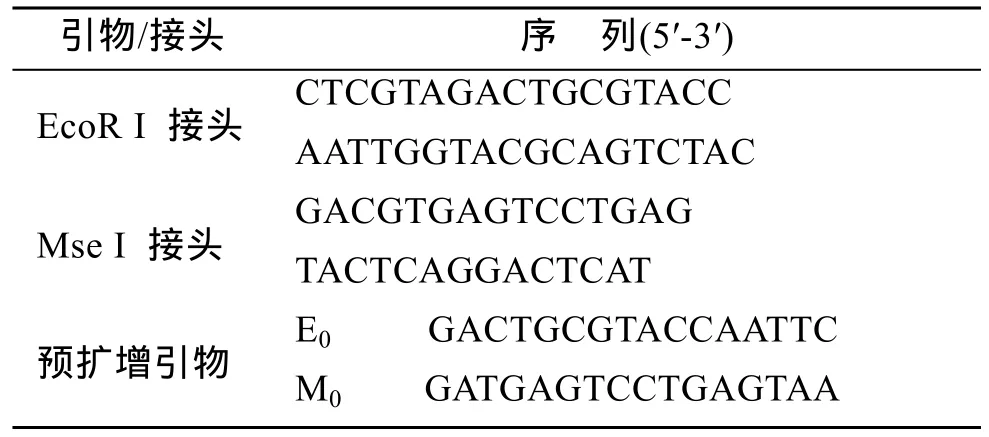
表1 AFLP接头及预扩增引物序列Tab. 1 Adaptors and pre-amplification primer sequences in AFLP analysis
1.3.1 酶切反应
每个样品的酶切混合液包括: 160 ng的 DNA样品, 0.2 µL的Mse I(Mse I 10 mol/L), 4 µL的10×Tango反应缓冲液, 再加双蒸水至反应总体积18 µL。将反应混合液置于 MJ-PTC200温度梯度 PCR仪中进行Mse I酶切, Mse I酶切反应条件: 65℃ 3 h, 80℃ 15 min, 4℃ 5 min。反应结束后, 再加入0.2 µL的 EcoR I(EcoR I 10 mol/L), 加双蒸水至总反应体积20 µL,进行EcoR I酶切, EcoR I酶切反应条件: 37℃ 3 h,80℃ 15 min, 4℃ 5 min。1%的琼脂糖凝胶电泳检测酶切效果, 4℃下保存备用。
1.3.2 连接反应
每个连接反应混合液包括: 酶切样品10 µL, 10 nmol/L EcoR I/Mse I接头 0.8 µL, 10×T4反应缓冲液2 µL, 1 mol/L T4连接酶 0.5 µL, 加双蒸水至总体积20 µL, 22℃恒温过夜。-20℃下保存备用。
1.3.3 预扩增反应
反应混合液包括: 2 µL 的连接产物, 2 µL的10×Taq buffer, 1.6 µL 的 25 nmol/L MgCl2, 0.5 µL 的dNTPs(10 mmol/L), 0.17 µL 的 E0(33 mol/L), 0.17 µL的M0(33 mol/L), 13.46 µL的无菌双蒸水, 0.1 µL的Taq DNA 聚合酶(5 U /µL), 共 20 µL。PCR 反应条件:先 94℃ 1 min; 再 94℃ 30 s, 56℃ 60 s, 72℃ 60 s,共20个循环; 4℃ 5 min。预扩增完成后, 将产物在1%琼脂糖凝胶中检测预扩增产物的效果。将预扩增产物取出5 µL加入45 µL双蒸水, 混合均匀作为选择性扩增模板。
1.3.4 选择性扩增反应
取5.0 µL的选择性扩增模板, 各加入15.0 µL选择性扩增反应混合液: 2.0 µL的10×Taq buffer, 1.2 µL的 25 nmol/L MgCl2, 0.4 µL 的 dNTPs(10 mmol/L),10.2 µL的无菌双蒸水, 0.5 µL的 EcoR I引物(10µmoL/L), 0.5 µL 的 Mse I引物(10 µmoL/L)。0.2 µL的Taq DNA聚合酶(5 U /µL)。PCR反应条件: 首先94℃ 1 min; 再 94℃ 30 s, 65℃ 30 s, 72℃ 60 s, 循环 13次(每一循环退火温度减少 0.7℃), 然后 94℃30 s, 65℃ 30 s, 72℃ 60 s, 23 个循环; 72℃ 10 min;4℃ 5 min。最后4℃保存。
1.3.5 变性聚丙烯酰胺凝胶电泳
AFLP扩增产物(共 20 µL)在电泳前加入 10 µL上样缓冲液, 95℃变性5 min, 立即放入冰盒中冷却(防止复性)。采用 6%的变性聚丙烯酰胺凝胶, 电泳缓冲液为 1×TBE, 在恒电压 2 000 V 下使用美国Bio-Rad聚丙烯酰胺凝胶电泳仪电泳, 先预电泳30~45 min, 直至温度达到 55℃。然后每个样品取5 µL上样, 恒电压2000V电泳2 h左右, 用银染法显示电泳结果。
1.4 数据统计分析
对所有扩增清晰的条带进行记录, 采用“0-1”系统记录谱带位置, 观察扩增条带的有无, 有带记为“1”, 无带记为“0”。将上述获得的 AFLP指纹图谱转换成 1和 0构成的数字矩阵, 结果填入Microsoft Excel 表格中。在种群处于Hardy-Weinberg平衡的条件下, 利用POPGENE VERSION 1.31[16]软件统计位点总数、多态位点数, 计算多态位点频率、显性基因型频率、Nei′s基因多样性指数、Shannon′s多样性指数、种群总基因多样性、种群内基因多样性、种群间的Gst(基因分化系数)、种群间的Nm(基因流系数)、遗传相似度和遗传距离等。利用 Arlequin 3.5进行 AMOVA分析, 采用 MEGA 3软件以UPGMA法对3个种群进行聚类分析, 构建群体间亲缘演化系统树图谱。
2 结果
2.1 3个种群AFLP扩增结果及遗传多样性
用 4对选择性引物(即引物组合 E-TG/M-CAC,E-TG/M-CAT, E-AA/M-CAC, E-AA/M-CAA)对花鳗鲡3个种群共60个样本的DNA进行AFLP分析, 共扩增出180条清晰的条带, 其中多态性条带为160条,Aus、Hai和Phi 3个种群的多态位点分别为78.33%、79.44%和84.44%, 总多态位点比率为88.89%(表2)。将每个扩增片段视为一个基因位点, 每对引物检出的位点数从34~50个不等, 平均每对引物检出45个位点。在3个种群中, Phi种群的遗传多样性最高, 而Aus种群的遗传多样性最低。
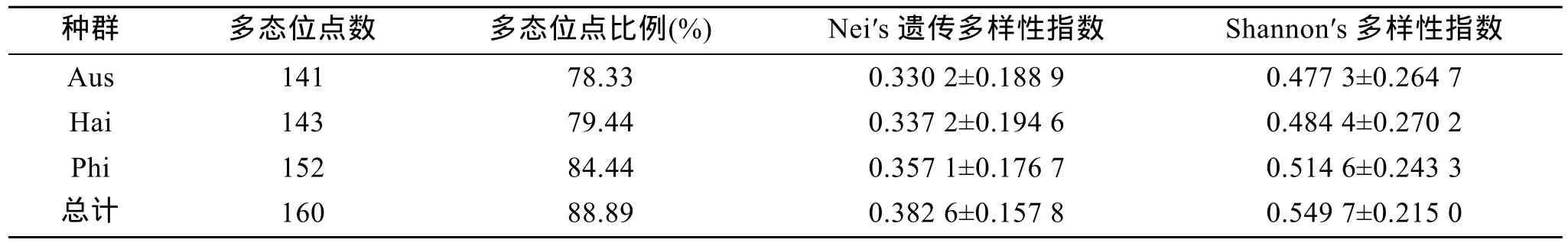
表2 3个种群扩增结果及遗传学参数Tab. 2 Amplification results and parameters of genetic diversity in the three Anguilla marmorata populations
2.2 3个种群的遗传分化及聚类分析
根据Gst(表3)和AMOVA分析估算(表4), 3个花鳗鲡种群遗传变异分别有89.25%和96.07%存在于种群内, 10.75%和3.93%存在于种群间。
根据 Wright[17]的标准,Gst值介于 0~0.05, 表明种群间遗传分化较弱; 介于 0.05~0.15, 表明种群间遗传分化中等; 0.15~0.25表明种群间遗传分化较大;当Gst值>0.25表明分化极大。由表3得出Aus种群~Hai种群、Aus种群~Phi种群和 Hai种群~Phi种群均出现中等遗传分化。
地理距离: Aus~Hai>Aus~Phi>Hai~Phi;Gst(表3): Aus种群~Hai种群>Aus种群~Phi种群>Hai种群~Phi种群; 遗传距离(表 5): Aus 种群~Hai种群>Aus种群~Phi种群>Hai种群~Phi种群, 可见地理距离越大遗传分化程度越高。
通常认为种群间的Nm<1, 种群间基因交流是有限的, 若Nm>1, 说明种群间通过某种渠道进行基因交流[18]。花鳗鲡两两种群间的Nm和 3个种群的总Nm均超过1, 说明种群间有基因交流。3个花鳗鲡种群间的Nm以Hai种群~Phi种群最高。
将 180个扩增位点的显性基因型频率以 10%为单位划分区间, 0和1分别设为一个单独的区间, 统计显性基因型频率位于各区间内的位点数。通过对不同种群的显性基因型频率在各区间内的位点数的走势比较, 可以分析出各种群遗传结构是否存在差异。由图1可以看出3个种群的曲线走势呈现出规律性, 总体趋势基本相似, 但有差异: Aus种群与Phi种群在0~9%和40%~69%区间走势不同; Aus种群与Hai种群在 0~9%和 50%~69%区间走势不同; Hai种群和Phi种群在40%~59%区间走势不同。说明3个种群在遗传结构方面存在一定的差异。通过计算花鳗鲡3个不同地理种群之间的Nei′s遗传相似度和遗传距离(表5), 根据遗传距离构建系统树(图2), 由图2中看出Hai种群和Phi种群亲缘关系较近, 首先聚在一起, 它们与Aus种群亲缘关系较远, 最后聚在一起。
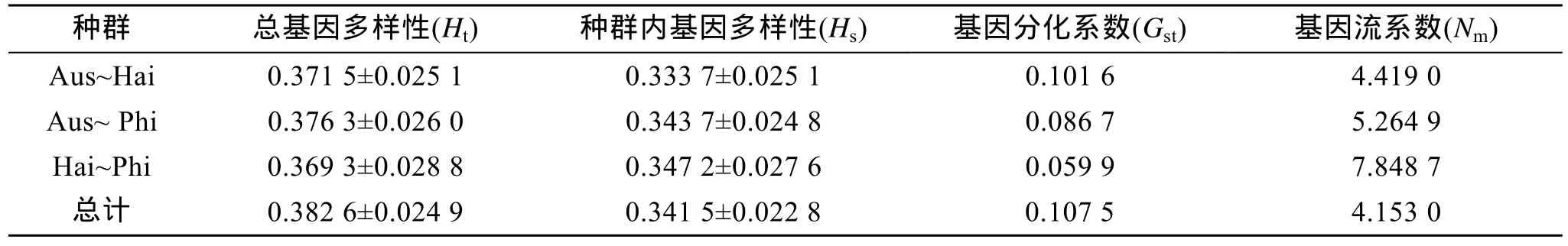
表3 3个花鳗鲡种群遗传分化Tab. 3 Genetic differentiation for the three populations of Anguilla marmorata
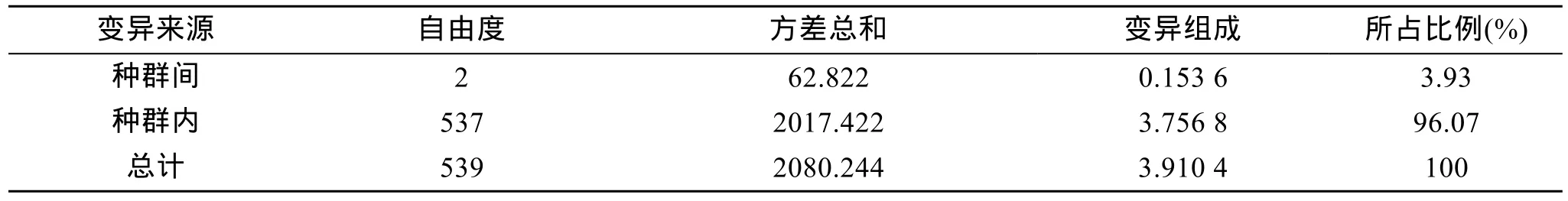
表4 花鳗鲡种群的AMOVA分析数据Tab. 4 Data derived from AMOVA of Anguilla marmorata
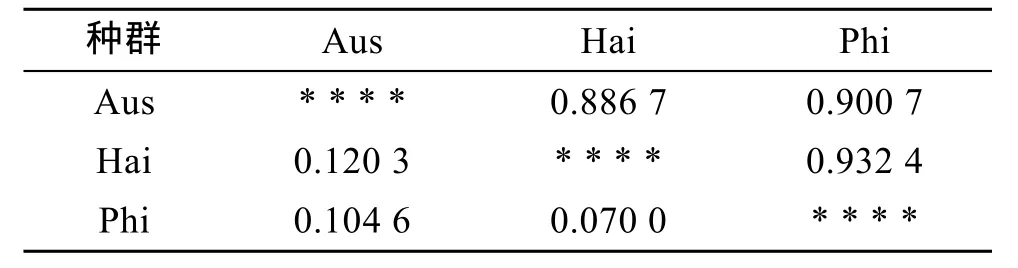
表5 种群间遗传相似度(对角线*上)及遗传距离(对角线*下)Tab. 5 Inter-population genetic identity (above diagonal)and genetic distance (below diagonal)
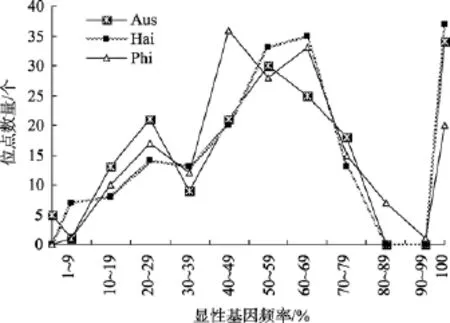
图1 扩增位点数在不同显性基因频率区间内分布Fig. 1 Distributions of amplified loci in different frequency intervals
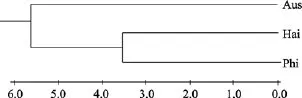
图2 UPGMA方法构建3个花鳗鲡种群的系统树Fig. 2 UPGMA dendrogram by AFLP in the three Anguilla marmorata populations
3 讨论
Aus种群和Hai种群的Nei’s遗传多样性指数分别为 0.330 2±0.188 9 和 0.337 2±0.194 6、Shannon′s多样性指数分别为0.477 3±0.264 7和0.484 4±0.270 2,Aus和Hai两种群多样性指数相似, 这与采用微卫星分析的方法得出的花鳗鲡 Aus种群和中国种群多样性指数相似的结论一致[5]。
有关研究人员采用 AFLP技术对日本鳗鲡(Anguilla japonica)[9]、蓝圆鲹(Decapterus maruadsi)[10]和鱼(Miichthys miiuy)[11]进行遗传多样性分析, 得出了这三种鱼的多态位点比例分别为62.81%~74.10%、64.65%和68.86%~72.51%, 并根据多态位点比例认为日本鳗鲡、蓝圆鲹和鱼遗传多样性较高。3个花鳗鲡种群多态位点比例为 78.33%~84.44%, 高于日本鳗鲡、蓝圆鲹和鱼, 可见3个花鳗鲡种群遗传多样性目前还处在较高的水平。我们认为与花鳗鲡自然产卵、产卵后亲鱼死亡、自然种群分布广、人为干扰因素较少等相关。
Minegishi等[4]采用线粒体DNA和微卫星分析的方法, 将花鳗鲡划分为南太平洋、北太平洋、东印度洋和西印度洋4个种群, 据此分析, 属于南太平洋的Aus种群与属于北太平洋的Hai种群和Phi种群出现了遗传分化, 这与本次研究所得的结论一致。齐兴柱等[6]在利用细胞色素 b基因对不同地域花鳗鲡种群进行分析时得出了同位于北太平洋花鳗鲡的日本种群和 Hai种群两者之间存在地理差异, 本次研究同样也得出了同位于北太平洋Hai种群和Phi种群间出现了遗传分化的结论。
研究结果表明, 3个花鳗鲡种群间出现了中等程度的遗传分化, 我们认为原因可能为: 花鳗鲡于成年时降河洄游到江河口附近性腺才开始发育, 而后入深海进行繁殖。初孵出仔鱼为白色薄软的叶状体,叶状体尚不能自主游动, 依靠海流将其带到陆地沿岸后发生变态, 变成短的圆线条状的幼鳗, 进入淡水河湖内索食生长。由于 3个花鳗鲡种群性腺成熟的时间及到达产卵场的时间不同, 不同种群亲鱼所产的仔鱼在不同的海流的作用下, 于特定的时间到达特定的地域, 因此3个种群难以融合在一起形成1个随机交配的大种群, 3个种群间出现中等程度的遗传分化。
花鳗鲡两两种群间的Nm和3个种群的总Nm均超过 1, 种群间有基因交流, 遗传分化与地理距离成正相关, 原因可能是: 虽然不同种群花鳗鲡的亲鱼因产卵时间的差异而无法交配, 但花鳗鲡鱼卵和仔鱼随海流漂浮和迁移, 不同年份海流的变化可能使少数花鳗鲡的鱼卵和仔鱼的漂浮和迁移路线发生变化, 混合到其他种群中, 种群内鱼卵和仔鱼的迁入迁出减弱了种群间的遗传分化, 产生了一定的基因交流, 地理距离越远, 携带不同种群花鳗鲡鱼卵和仔鱼的海流混合的机会越少, 因此遗传分化与地理距离成正相关。
中国花鳗鲡养殖苗种主要来源于Aus、Hai和Phi三地, 三者在水温、驯食和抗病力方面均表现出差异,如与Hai和Phi的苗种相比, Aus的苗种可适应较低的水温, 在精养池中比较容易驯食, 但容易发生单鳃病等病害, 而 Hai的苗种则为三者中最难驯食的,由水丝蚓(Limnodrilus hoffmeisteri)转为配合饲料往往无法转换成功, 究其原因可能与 3个种群间的遗传分化有关。因此, 在今后的养殖生产中必须加强花鳗鲡的基础生物学研究, 对不同产地的花鳗鲡采用不同的养殖模式、驯食方式和诱食剂等, 以满足各自生长的需要, 达到高产稳产的目的。
[1]Miller M J, Mochioka N, Otake T, et al. Evidence of a spawning area ofAnguilla marmoratain the western North Pacific [J]. Marine Biology, 2002, 140: 809-814.
[2]陈锤. 花鳗鲡的生态学特征与开发利用[J]. 北京水产, 2005, 1: 54.
[3]Watanabe S, Aoyama J, Miller M J, et al. Evidence of population structure in the giant mottled eel, Anguilla marmorata, using total number of vertebrae [J]. Copeia,2008, 3: 680-688.
[4]Minegishi Y, Aoyama J, Tsukamoto K. Multiple population structure of the giant mottled eel,Anguilla marmorata[J]. Molecular Ecology, 2008, 7(3): 3109-3122.
[5]Gong X L, Ren S J, Chen S Q. Highly polymorphic microsatellite loci from the giant mottled eel (Anguilla marmorata) [J]. Molecular Ecology Resources, 2009,9(6): 1544-1547.
[6]齐兴柱, 尹绍武, 娄甜甜, 等. 海南产花鳗鲡细胞色素 b基因的克隆及序列分析[J]. 海南大学学报自然科学版, 2007, 25(4): 397-401.
[7]王伟继, 岳志芹, 孔杰, 等. AFLP分子标记技术的发展及其在海洋生物中的应用[J]. 海洋水产研究, 2005,26 (1): 82-87.
[8]林斌彬, 张子平, 王艺磊, 等. AFLP技术发展及其在水产生物学研究中的应用[J]. 集美大学学报(自然科学版), 2008, 13(1): 45-51.
[9]张辛, 许璞, 许广平, 等. 中国东海日本鳗鲡种群遗传结构的 AFLP分析[J]. 水生态学杂志, 2010, 3(1):82-86.
[10]张丽艳, 苏永全, 丁少雄, 等. 福建近海蓝圆鲹种群遗传多样性的AFLP分析[J]. 水产学报, 2010, 34(5):680-687.
[11]彭志兰, 柳敏海, 傅荣兵, 等. 舟山鮸鱼群体遗传多样性的 AFLP研究[J]. 上海水产大学学报, 2010,19(2): 172-177.
[12]邵艳卿, 陆荣茂, 董迎辉. 三种蛏的遗传多样性分析[J]. 海洋科学, 2009, 33(10): 26-30.
[13]Sambrook J, Russell D. 分子克隆实验指南(第三版)[M]//北京: 科学出版社, 2002: 463-471.
[14]Vos P, Hogers R, Bleeker M, et al. AFLP: a new technique for DNA fingerprinting [J]. Nucleic Acids Research, 1995, 23: 4407-4414.
[15]Wang Z Y, Tsoi K H, Chu K H. Applications of AFLP technology in genetic and phylogenetic analysis of penaeid shrimp [J]. Biochemical Systematics and Ecology, 2004, 32(4): 399-407.
[16]Yeh F C, Yang R C, Boyle T.POPGEN(version1. 3.1).Microsoft Windows-based Freeware for Population Genetic Analysis [M].//Edmonton: University of Alberta, 1999: 1-28.
[17]Wright S. The genetical structure of population [J].Ann Eugen-ics, 1951, 15: 323-334.
[18]曲若竹, 侯林, 吕红丽, 等. 种群遗传结构中的基因流[J]. 遗传, 2004, 26(3): 377-382.
AFLP analysis of threeAnguilla marmoratageographic populations
ZHU You-fang1, LIN Feng-qin2, WANG Yi-lei2, XIE Fang-jing2
(1. Putian Municipal Institute of Fishery Science, Putian 351100, China; 2. The Key Laboratory of Science and Technology for Aquaculture and Food Safety, Fisheries College, Jimei University, Xiamen 361021, China)
Dec., 08, 2010
Anguilla marmorata; population; AFLP; genetic variation
This study is the first of its kind to document genetic diversity of giant mottled eel (Anguilla marmorata) among three geographic populations of Australia, Hainan Island, and Philippines. This study was performed on 60 young eels by using AFLP molecular marker technique. A total of 180 AFLP loci, of which 160 loci were polymorphic, were detected by four primer combinations. The percentage of polymorphic loci from Australia, Hainan Island, and Philippines was 78.33%, 79.44%, and 84.44%, respectively; Shannon's diversity index was 0.477 3±0.264 7, 0.484 4±0.270 2, and 0.514 6±0.243 3, respectively. TheGstand AMOVA analysis showed that there were 89.25% and 96.07% genetic variation within the three populations, and 10.75% and 3.93% genetic variation among populations. Our results indicate that genetic differentiation has occurred and has a positive correlation with geographic distant. Meanwhile, gene flows exist among all three populations. These data will be important for the farming management ofA. marmorata.
Q347
A
1000-3096(2011)08-0083-06
2010-12-08;
2011-06-13
福建省高校水产科学技术与食品安全重点实验室基金资助项目(2008J201)
朱友芳(1964-), 男, 福建仙游人, 副研究员, 硕士研究生,主要从事水产养殖及病害防治的研究, 电话: 0594-6298821, E-mail:e365cn@163.com
谭雪静)
