水稻苯丙氨酸解氨酶的生物信息学分析
2010-11-26康晓慧
康晓慧, 雷 桅, 张 梅
(1.西南科技大学生命科学与工程学院, 中国 绵阳 621010;2.中山大学生命科学学院, 国家教育部食品与健康工程研究中心, 中国 广州 510650)
抗病性是植物与其病原生物在长期的协同进化过程中相互适应、相互选择所形成的一种可遗传特性.植物被病原物侵染后,会发生一系列生理变化和防卫反应,包括植保素和木质素等抗病物质的合成和积累,其中苯丙烷类代谢与植物的抗病性密切相关,苯丙氨酸解氨酶(phenylalanine ammonia-lyase, PAL)是该途径中的第一个关键酶,也是限速酶.它催化L-苯丙氨酸非氧化性脱氨生成反式肉桂酸(cinnamic acid,CA),即是植保素、木质素、类黄酮和某些酚类等生物合成的通用前体.因此,该酶在植物病理学研究中有极重要的地位.PAL为多基因家族所编码,但遗传特性相当保守,目前已从马铃薯(Solanumtuberosum)、水稻(Oryzasativa)、烟草(Nicotianatabacum)和黄瓜(Cucumissativus)等多种植物中克隆了pal基因,并将其作为植物抗病机制研究的重要内容.
水稻是世界上最重要的粮食作物之一,而病害给水稻的产量和质量造成了巨大的损失,因此水稻抗病的分子机理和遗传改良成为一项重要的课题,而作为关键防卫基因的pal则有望成为理想的调控靶点.粳稻品种日本晴(Nipponbare)是单子叶植物基因组学研究的模式植物,测序发现其pal基因家族包含9个成员.本文利用生物信息学的方法,选取其中一个成员基因,对其核苷酸及相应氨基酸序列的理化性质、结构特征、生化功能和系统演化等进行了预测和分析,为今后深入研究PAL蛋白的酶学特性和植物病害胁迫的分子机理提供一定的理论参考.
1 材料与方法
粳稻品种日本晴的苯丙氨酸解氨酶(PAL)核苷酸与氨基酸序列均来自于GenBank中已登录的数据信息:水稻Oryzasativa(japonica cultivar-group)(Accession:AY224546).
利用Vector NTI Suit 8等序列分析软件和www.ncbi.nlm.nih.gov、www.expasy.org等网站提供的各种在线生物信息学工具进行预测和分析.核苷酸和氨基酸序列的分子结构和理化性质分析使用ProtParam工具和 Vector NTI Suit 8软件完成[1];序列同源性比对使用Vector NTI Suit 8完成;分子系统发生树使用Clustal X软件比对后在MEGA3的Phylogeny中以Neighbor-Joining方法构建[2];亚细胞定位情况的考察使用TargetP 1.1 Server在线完成[3];跨膜结构域和疏水性分析分别使用TMHMM[4]和ProtScale[5]完成;motif和蛋白质功能分别用Scanprosite[6]和Protfun[7]分析;蛋白质二级结构预测和三级建模分别使用GOR[6]和SWISS-MODEL[8-9]完成,3D模型在WebLab ViewerLite 4.2软件中编辑.
2 结果与分析
2.1 水稻pal基因的核酸及相应氨基酸序列的分子结构和理化性质分析

图1 水稻PAL的核苷酸及其编码氨基酸序列
水稻pal基因的全长mRNA序列及其推导的氨基酸序列如图1所示,利用Vector NTI Suit 8软件分析得到的Ospal基因的核苷酸序列及其相应氨基酸序列的结构和性质数据,发现Ospal基因全长为2 136 bp,其中开放阅读框(ORF)长为2 133 bp,起始密码子为ATG,终止密码子为TGA,3′和5′非编码区(UTR)均不存在.水稻Ospal基因编码蛋白的分子式为C3188H5129N891O973S24,相对分子质量为72 273,等电点为6.12,摩尔消光系数为37 590,肽链长为671 aa,其中含带电氨基酸27.72%,极性氨基酸23.85%,疏水性氨基酸38.15%,酸性和碱性氨基酸比例为11.33%和10.13%,而含量最丰富的4种氨基酸占比为Ala:10.88%;Leu:10.13%;Val:8.20%;Gly:7.90%.
2.2 水稻pal基因的氨基酸序列的比对分析

图2 水稻PAL和其他植物PAL的氨基酸序列多重比对注:一致位点显示为黑底白字,保守位点显示为灰底白字,其他显示为白底黑字.活性位点标注*,MIO区用方框表示.Salvia miltiorrhiza(GenPept accession No.:ABR14606); Trifolium pretense(GenPept accession No.: BAE71252); Daucus carota(GenPept accession No.:BAA23367); Ipomoea batatas(GenPept accession No.:BAA11459); Nicotiana tabacum(GenPept accession No.:BAA22947); Oryza sativa (GenPept accession No.: AAO72666).
选取水稻(japonica cultivar-group)等6条植物PAL氨基酸序列,在Vector NTI 8软件中进行多重比对分析.分析结果用不同颜色显示(图2),颜色越深的表示同源性越高,最深的区域则表示可能存在的重要功能域.分析结果显示所选植物PAL具有较高的同源性,一致性达到57.4%,可见PAL编码区高度保守,但是在N端也表现出较明显的长度和组成上的变异性.推测这可能是由于苯丙烷类途径是植物初生代谢转向次生代谢的重要通路,广泛存在于植物体中,而作为苯丙烷类化合物生物合成途径中的第一个关键酶PAL必须保持足够的遗传稳定性和演化趋同性.此外,在保守的PAL比对图谱中,发现水稻PAL具有典型的苯丙氨酸和组氨酸解氨酶的标志性模式,该保守基序(GTITASGDLVPLSYIAG)被包埋在酶活性中心;此外,也存在已查明的MIO域,它通过Ala-Ser-Gly三肽的自动环化合脱水生成,毗邻底物结合位点,并负责产物E-肉桂酸酯/氨的催化形成.
2.3 水稻pal基因的氨基酸序列的分子系统进化树分析
将9条来自不同植物的PAL氨基酸序列在Clustal X软件中进行完全比对后,在MEGA3软件中按照Neighbor-Joining方案进行分子进化树分析,获得PAL氨基酸序列的分子进化树分析结果(如图3).进化树用MEGA3软件基于Neighbor-Joining方法(重复1 000次)构建.遗传距离显示在分支上.9种植物的PAL氨基酸序列聚成3支:甘薯、番薯和烟草聚成第1支,它们的多酚类物质含量较高;粘毛黄芩、丹参、豌豆和胡萝卜聚成第2支,前二者是主产黄酮活性成分的草药,后二者是含有丰富的香豆素和花青素等的蔬菜;马蹄竹和水稻聚成第3支,这两种植物的木质素合成能力较强,且同属禾本科.该进化树除反映了这9种植物间的亲缘关系外,也反映了不同植物类群所含苯丙素类化合物的种类和含量差异,即说明该指标与植物的系统演化进程密切相关.苯丙烷类代谢作为植物次生代谢的起始途径,广泛存在于所有植物,其衍生途径随着生命进化而分化出黄酮类、木质素类、植保素类等某些植物的主流次生代谢通路,而PAL作为苯丙烷类途径的第一个关键酶,显然在植物的功能分化和系统进化过程发挥着重要作用.因此,以PAL序列分析作为分子指标进行的遗传聚类,对于系统分类和自然进化研究具有一定的参考价值,同时也反映出植物的抗病性与植物本身的种质特性和进化水平密切相关.

图3 水稻PAL家庭和其他植物PAL的分子进化分析
2.4 水稻pal基因编码蛋白的亚细胞定位和功能结构域分析
TargetP 1.1 Server在线工具分析结果显示,水稻PAL定位于细胞质基质,且无转运肽,预测可靠性等级均为2.可见水稻PAL在游离核糖体上起始合成后,不进行蛋白转运,而是继续保留在细胞质基质中,直接催化苯丙素类化合物的生成.
PAL的多个重要保守结构域在OsPAL蛋白中也被找到,包括S213、Y120、L148、N269、Q357、Y360、R363、F409、Q497和4-methylideneimidazole-5-one(MIO),这些区域对于PAL的底物结合、催化活性和MIO形成都至关重要.这与欧芹的实验结果一致,表明水稻PAL也属于PAL蛋白家族.
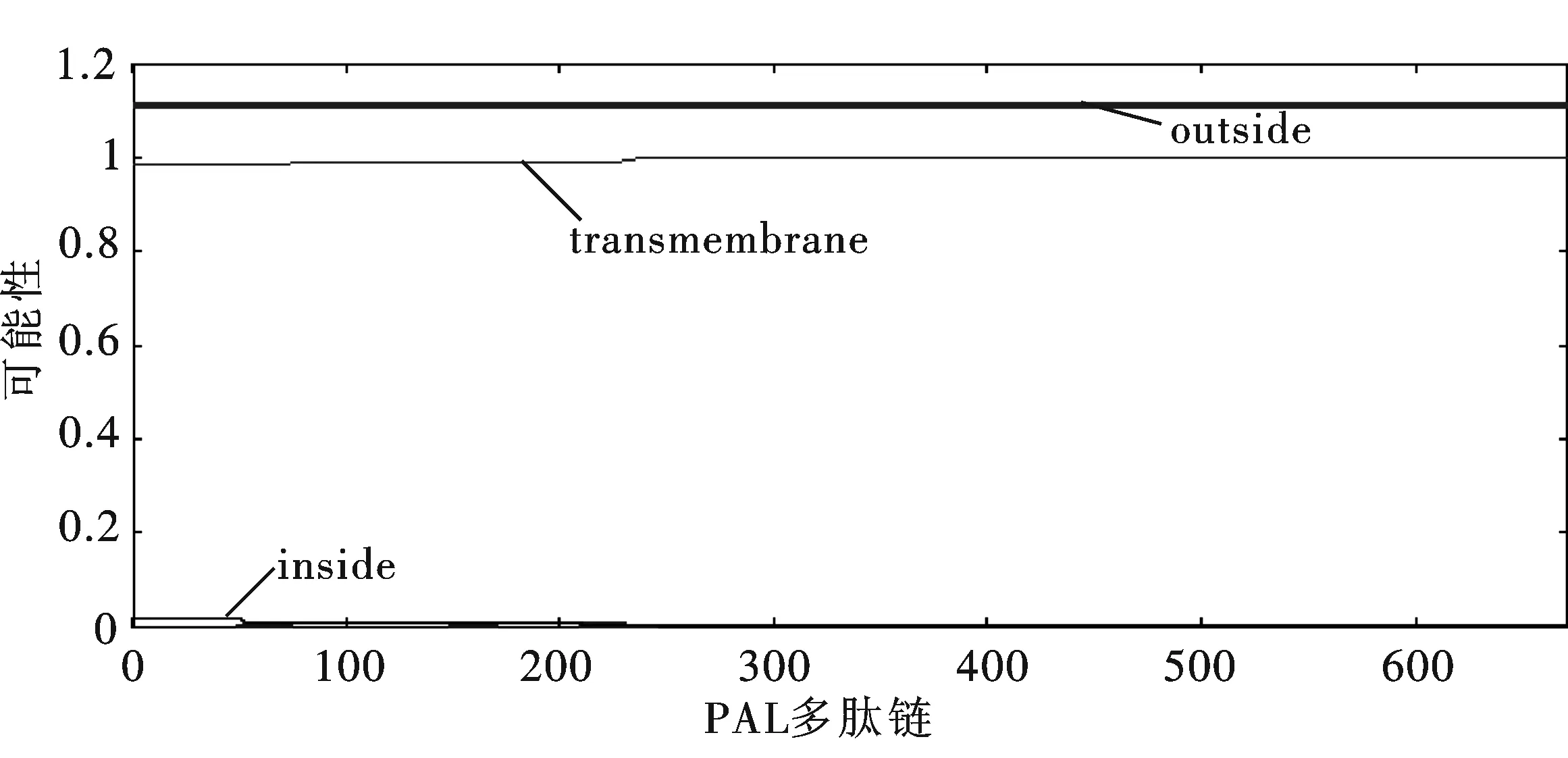
图4 水稻PAL蛋白的跨膜结构域预测
2.5 水稻pal基因编码蛋白的跨膜结构域和疏水性预测和分析
用TMHMM Server v. 2.0对水稻PAL氨基酸序列的跨膜结构域进行预测,结果如图4所示,水稻PAL整条肽链都位于细胞膜内,也即是水稻PAL不存在跨膜结构域,结合上述转运肽的预测,可以推断,水稻PAL在细胞质基质中合成后,不经蛋白转运,直接锚定于细胞质基质中的特定部位行使催化功能.这与上述亚细胞定位分析结果相一致.
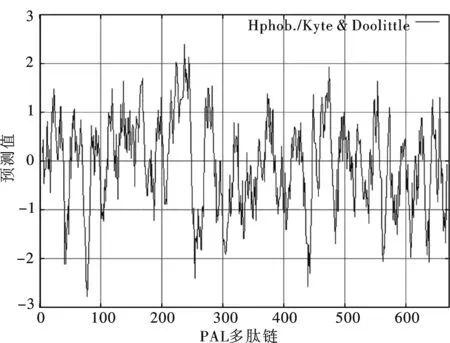
图5 水稻PAL蛋白的疏水性预测
由ProtScale进行水稻pal基因编码蛋白的氨基酸序列的疏水性预测结果表明(图5),多肽链具有最低分值-2.778,亲水性最强;而最高分值为2.389疏水性最强.整条多肽链表现为亲水性,但没有明显的亲水区域.结合跨膜结构域的预测结果,可以推断,水稻PAL不存在明显的疏水区域,与OsPAL不存在跨膜结构域的特征相吻合.
2.6 水稻pal基因的氨基酸序列二级和三级结构预测和分析
GOR4在线工具预测水稻PAL氨基酸序列的二级结构,结果如图6所示,水稻PAL多肽链中,随机卷曲占44.02%,α-螺旋占43.88%,延伸链占12.10%,即随机卷曲和α-螺旋是OsPAL多肽链中的主要结构元件,延伸链散布于整个蛋白质中.
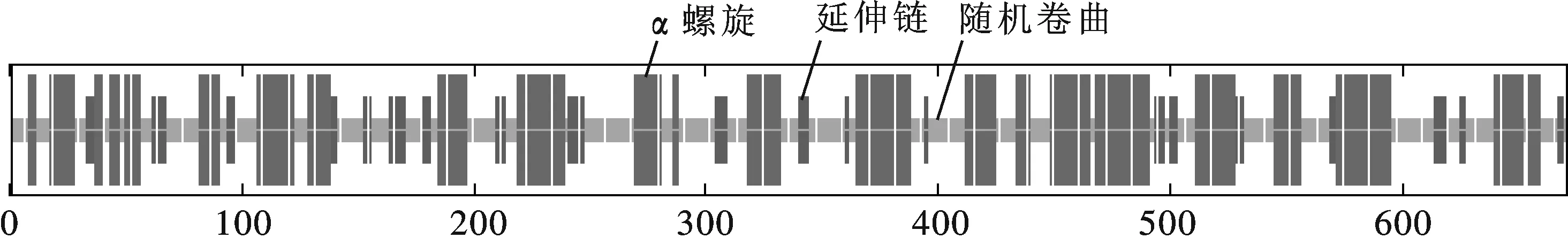
图6 水稻PAL蛋白的二级结构预测
在PROSTIE中通过与已知物种PAL功能位点的比对和计算,成功预测了水稻PAL的Motif,发现OsPAL含有5个重要的基序,分别是苯丙氨酸与组氨酸标记(Phenylalanine and histidine ammonia-lyases signature)(153~168 GTITASGDLVPLSYIA, 153~169 GTITASGDLVPLSYIAG),氨基化位点(183~186 DGKK),蛋白激酶C磷酸化位点(Protein kinase C phosphorylation site)(16~18 SLR, 74~76 SHR, 172~174 TGR, 260~262 THK, 452~454 SAR, 513~515 SAR, 517~519 SEK, 648~650 SER),酪氨酸激酶II磷酸化位点(Casein kinase II phosphorylation site)(335~338 SVND, 523~526 TAID, 532~535 SYAD, 575~578 TAFE, 635~638 SPGE),N-糖基化基序(N-glycosylation motif)(99~102 NGSD)和N-肉豆蔻酰化位点(N-myristoylation site)(20~25 GQVAAV, 65~70 GVTTGF, 95~100 GIFGNG).再将水稻PAL氨基酸序列上传到SWISS-MODEL的建模服务器中进行PAL结构的三维建模,通过同源搜索和比对,以殴芹(Petroselinum crispu)为模板初步构建了OsPAL的三维模型,然后在ViewerLite 4.2软件中进行序列编辑和位点设置,获得OsPAL的三级结构模型,相关功能位点见标示于图7.
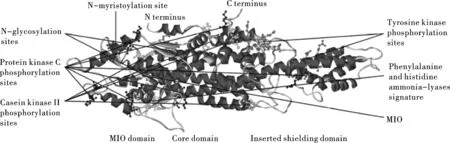
图7 水稻PAL的三维建模及重要基序
3 结论与讨论
苯丙氨酸解氨酶在木质素、植保素等植物抗病物质的代谢途径中起着重要作用,在水稻的抗病机制研究中也被公认为是重要的生理指标.生物信息学是后基因组时代,以计算机科学技术为平台,分析并挖掘生物学数据,从而为实验研究提供理论依据的新兴学科.本文利用生物信息学软件和方法,对日本晴水稻防卫基因pal的结构性质、生化功能和系统进化等进行了预测和分析,发现Ospal基因的核苷酸序列只包含ORF,缺少两端UTR,无跨膜结构域和质体转运肽,定位于细胞质基质.近年的研究也发现,细胞间质部分PAL活性最高,电镜技术显示PAL分散在细胞的基质中.这些实验结果也间接地验证了本定位预测的可能性.本项研究明确了PAL蛋白的二级结构以α-螺旋和随机卷曲为主,三维建模成功,并明确了多个功能基序.这些重要生物学参数的获得为进一步研究PAL的酶学性质和水稻的抗病机制奠定了理论基础.
PAL作为抗病性指标蛋白的水稻生物学研究已有诸多报道,但大多是以PAL酶活变化表征抗病能力强弱的生化研究.本文解析了OsPAL蛋白的分子结构,并描述了其细胞学特性,将有助于深入阐述水稻病理生理与抗性获得机制;而对Ospal基因的序列和系统学演化分析结果,将为基于Ospal的基因操作和遗传工程提供重要的调控靶点.因此,本项研究对于水稻抗病性分子育种也有重要参考价值.
参考文献:
[1] LEI W, LUO K M, SHUI X R,etal. Compute simulation to characterize structure and function of chalcone synthase from scutellaria baicalensis georgi[J]. Molecular Biology, 2009, 43(6): 1 012-1 017.
[2] KUMAR S K, TMAMURA K, JAKOBSENI I B,etal. MEGA2: Molecular evolutionary genetics analysis software[J]. Bioinformatics, 2001, 17: 1 244-1 245.
[3] OLOF E, HENRIK N, SØREN B,etal. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J]. J Mol Biol, 2000, 300:1 005-1 016.
[4] IKEDA M, ARAI M, LAO D M. Transmembrane topology prediction methods: A reassessment and improvement by a consensus method using a dataset of experimentally characterized transmembrance topologies[J]. In Silico Biol, 2002, 2: 19-33.
[5] GASTEIGER E, HOOGLAND C, GATTIKER A,etal. Protein Identification and analysis tools on the ExPASy Server[C]//John M Walker. The Proteomics Protocols Handbook. New York: Humana Press, 2005.
[6] COMBET C, BLANCHET C, GEOURJON C,etal. NPS@: Network protein sequence analysis[J]. TIBS, 2000, 25: 147-150.
[7] JENSEN J L, ST☞RFELDT H H, BRUNAK S. Prediction of human protein function according to Gene Ontology categories[J]. Bioinformatics, 2003, 19: 635-642.
[8] ARNOLD K, BORDOLI L, KOPP J,etal. The SWISS-MODEL Workspace: A web-based environment for protein structure homology modelling[J]. Bioinformatics, 2006, 22: 195-201.
[9] SCHWEDE T K J, GUEX N, PEITSCH M C. SWISS-MODEL: an automated protein homology-modeling server[J]. Nucleic Acids Res, 2003, 31:3 381-3 385.
