基于表位预测的花生过敏原Arah6免疫交叉反应性研究
2010-10-19陈红兵胡纯秋罗春萍
朱 盼,陈红兵,*,胡纯秋,李 欣,罗春萍,3
(1.南昌大学 食品科学与技术国家重点实验室,江西 南昌 330047;2.南昌大学中德联合研究院,江西 南昌 330047;3.南昌大学环境与化学工程学院,江西 南昌 330047 )
基于表位预测的花生过敏原Arah6免疫交叉反应性研究
朱 盼1,2,陈红兵1,2,*,胡纯秋1,2,李 欣1,2,罗春萍1,2,3
(1.南昌大学 食品科学与技术国家重点实验室,江西 南昌 330047;2.南昌大学中德联合研究院,江西 南昌 330047;3.南昌大学环境与化学工程学院,江西 南昌 330047 )
为了探讨花生过敏原Arah6与其他同源蛋白之间的交叉反应,通过各种生物信息数据库(Genbank,DiscoTope)以及网络工具及软件(BLAST,NPS@和Protean,Clustal x,PyMOL),开展基于表位预测的Arah6交叉免疫反应性研究。结果表明:肽段AA10~15、45~48、53~60、116~118区域可能是Arah6的线性表位优势区域;Arah6的构象型表位优势区有4个,它们存在的区域为AA1~13、35~51、53~59、89~105;基于线性表位预测的15种蛋白质与Arah6可能具有交叉反应性,其中4种(Arai6,conglutin,Arad6和conglutin 8)发生交叉反应的概率为100%;基于构象型表位预测的17种蛋白质与Arah6可能具有交叉反应性,其中4种(Ara i 6,conglutin,Arad6和conglutin 8)发生交叉反应的概率为100%。本结果对进一步了解Arah6的过敏性以及指导开展食品中Arah6的免疫学检测具有重要作用。
Arah6;花生过敏;表位;交叉反应;预测
Abstract:In order to explain cross-reactivity between peanut allergen Arah6 and other homologous proteins, various kinds of bioinformationcal databases, including Genbank and DiscoTope, and net tools/software, including BLAST, NPS@, Protean,Clustal x and PyMOL, were used for the epitope analysis between these proteins. It was indicated that the linear epitopes on Arah6 existed with high probability in the regions of AA10-15, 45-48, 53-60 and 116-118, and the conformational epitopes in the regions of AA1-13, 35-51, 53-59 and 89-105. According to the prediction of linear epitopes, there were fifteen proteins might cross-react with Arah6, and the possibility of cross-reactivity of four of them (Ara i 6, conglutin, Ara d 6 and conglutin 8) was 100%. However, according to the prediction of conformational epitopes, the number of proteins could cross-react with Arah6 with high probability was 17, and the possibility of cross-reactivity of four of them (Ara i 6, conglutin,Ara d 6 and conglutin 8) was 100%. These investigations will be beneficial for understanding peanut Arah6 allergy and help to develop an approach for the immunologic detection of Arah6 in foods in the future.
Key words:Arah6;peanut allergy;epitopes;cross-reactivity;prediction
花生广泛存在于各类食品中,花生过敏是一类较普遍的食物过敏反应[1],食品中极少量的花生过敏原便会产生严重的变态反应,有时可引起过敏性休克并危及生命,严重影响了部分过敏人群的生活质量[2]。目前国际免疫联合会命名小组委员会认可的花生过敏原有11种[3],其中,Arah6是一种主要的过敏原,属于2S白蛋白家族,与花生中另一主要过敏原Arah 2的氨基酸序列有59%的同源性[4]。另外,抗原表位是免疫交叉反应的物质基础[5],可分为线性表位和构象型表位。线性表位由肽链上连续的5~7个氨基酸组成[6],主要识别过敏原中特定的氨基酸序列。目前公认,在氨基酸数目超过80的蛋白质中,若序列同源性大于30%或有6个连续相同的氨基酸序列,则认为蛋白之间可能存在交叉反应[7]。构象型表位则是由蛋白中不连续氨基酸序列通过折叠等方式形成特定的三维空间结构而发生作用,占抗原表位中的90%[8-9]。通过蛋白质序列同源性和软件分析,利用已知蛋白三维结构模拟未知蛋白三维结构[8],经对比分析,可以预测具有相同/相似构象结构蛋白的交叉反应性。
迄今为止,蛋白质的结构包括表位的预测使用了很多生物信息库和软件。如,GenBank数据库是世界上著名的生物信息数据库,是由美国国立生物技术信息中心(NCBI)维护的一级核酸序列数据库[10],其中的BLAST工具主要用于序列对比和同源性分析。蛋白质结构数据库(PDB)是目前最主要的蛋白质分子结构数据库之一,其包含了大量的抗原抗体复合物晶体结构及相关信息[6]。而SWISS- MODEL是一个有注解的基于同源建模的蛋白质结构服务器[11],可提供蛋白质三维建模服务,任秀艳等[12]用此软件成功完成了HrpNEa蛋白的结构和功能的预测。另外,DNAStar软件是一种用于DNA和蛋白质分析的生物软件,其中Protean主要用于蛋白质结构分析,黎明等[13]采用此软件成功预测了猪传染性胃肠炎病毒的二级结构和B细胞抗原表位。ABCpred则采用人工神经网络来预测线性表位,预测敏感性约为67%,特异性约为64%[6,14],而且李明才等[15]采用此方案对与预测所得到的人白细胞介素-33蛋白的B细胞抗原表位结果进行最终验证。值得关注的是,DiscoTope是通过蛋白质三级结构数据来预测构象表位的一种新功能软件[16],纪滨等[17]采用此软件成功预测了H5N1亚型禽流感HA蛋白抗原的非线性B细胞表位,并通过了实验鉴定。
本研究利用生物信息学技术,将花生过敏原Arah6进行氨基酸序列分析,预测其二级结构、线性表位、构象型表位,从而预测交叉反应,对进一步了解Arah6的过敏性以及指导开展食品中Arah6的免疫学检测具有重要作用。
1 材料与方法
1.1 花生过敏原Arah6DNA序列来源
通过GenBank检索花生过敏原Arah6的基因序列,该基因在GenBank的登录号为AAD56337。应用DNAStar软件推导出该基因的氨基酸序列。
1.2 Arah6氨基酸序列同源性分析
利用BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)分析Arah6氨基酸序列,预测同源性蛋白。
1.3 基于Arah6线性表位的免疫交叉反应预测方案
1.3.1 Arah6蛋白的线性表位预测
利用NPS@网络服务器(http://npsa-phil.ibcp.fr/ cgibin/npsa_automat.pl? page=/NPSA/npsa_server.html)和DNAStar生物分析软件(Gamier-Robson方法)中的protean软件对Arah6蛋白质二级结构进行预测。Protean软件采用Kyte-Doolittle等方案预测Arah6的亲水性(hydrophilicity)[18],Emini法预测表面可及性(accessibility)[19],Jameson-Wolf法预测抗原性(antigenicity)[20],Karplus-Schulz法预测蛋白柔韧性(flexibility)[21],综合各因素,预测Arah6可能的线性表位区。
1.3.2 基于线性表位的免疫交叉反应预测
根据已知的Arah6表位区,在同源蛋白中,利用软件Clustalx进行多序列对比分析,查找相同/相似表位区域,预测可能与目的蛋白发生交叉反应的蛋白质。
1.4 基于Arah6构象型表位的免疫交叉反应预测方案
1.4.1 Arah6三维结构模拟预测构象型表位
利用网络数据库SWISS-MODEL(http://swissmodel.expasy.org/) 模拟Arah6的PDB构象图,并在网络数据库DiscoTope[22](http://www.cbs.dtu.dk/services/DiscoTope)中预测其空间构象型表位。
1.4.2 基于构象型表位的交叉反应预测
结合各蛋白的序列,利用网络数据库S W I S SMODEL模拟其PDB构象图,并在DiscoTope中预测构象型表位,与目的蛋白对比,预测免疫交叉反应。在预测中,通过网络数据库DisoTope是实现蛋白质三维结构预测构象型表位的一种新方法[6],本工作中选定特异性表位默认阈值-7.7,此时预测的特异性为75%,敏感性为47%。
2 结果与分析
2.1 花生过敏原Arah6的氨基酸序列
根据GenBank中登录号位AAD56337(Arah6)的DNA序列推导出的氨基酸序列如下:AHASAMRRERGR QGDSSSCERQVDGVNLKPCEQHIMQRIMGEQEQYDSYNFGS TRSSDQQQRCCDELNEMENTQRCMCEALQQIMENQCD GLQDRQMVQHFKRELMNLPQQCNFGAPQRCDLDVSGGRC。
2.2 Arah6氨基酸序列同源性分析
利用GenBank网络数据库对Arah6进行BLAST分析,具体结果见表1。

表1 Arah6的同源序列BALST分析结果Table 1 Homology sequences comparison of Arah6 using BALST
通过BLAST分析得知:能与Arah6发生交叉反应的蛋白主要来自于不同花生种属以及大豆、羽扇豆、大麦、黑麦、蓖麻等,共34种,见表1。表中1~3号蛋白与目的蛋白的相似度高达90%以上,且4~17号蛋白的相似度均在40%以上,它们均来自于花生,而花生是羽扇豆制品中的主要组成成分,这就大大增加了花生过敏的严重性和普遍性。
2.3 基于Arah6线性表位的免疫交叉反应预测结果
2.3.1 Arah6蛋白二级结构和特性分析及其线性表位的预测结果
2.3.1.1 蛋白二级结构和特性分析
用NPS@网络服务器对该基因的二级结构进行预测,结果如图1所示,对应相关数据见表2。
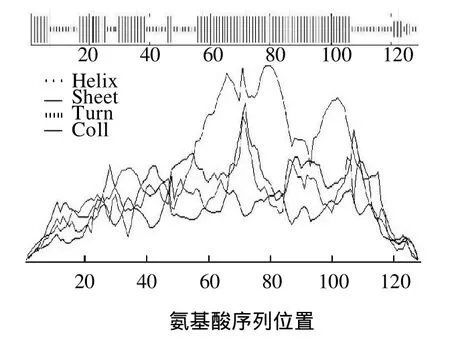
图1 NPS@预测Arah6二级结构Fig.1 Prediction analysis for secondary structure of Arah6 using NPS@

表2 NPS@预测Arah6二级结构结果Table 2 Location of four secondary structural classes of Arah6 predicted using NPS@
利用Protean软件对该蛋白的二级结构及蛋白特性再次进行预测,结果如图2所示,对应相关数据见表3。
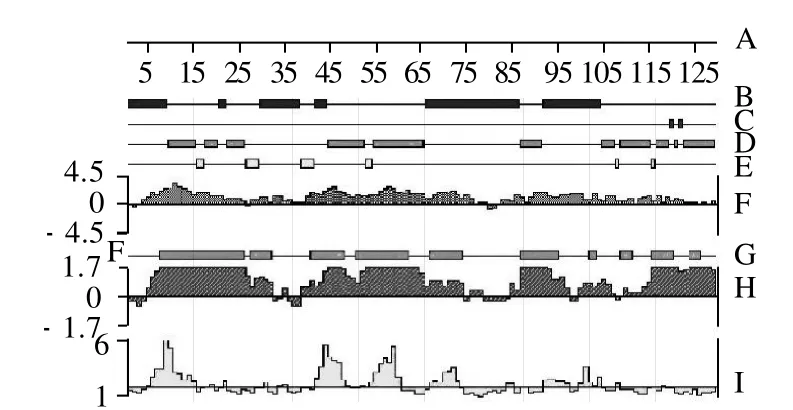
图2 Protean法预测的Arah6二级结构参数及蛋白特性Fig.2 Prediction analysis for secondary structure, antigenicity, hydrophilicity,surface prodability and flexibility of Arah6 using Protean appraoch

表3 Protean法预测的Arah6二级结构及相关预测指数的区域定位Table 3 Predicted location of four secondary structural classes and regions corresponding to 4 specific indexes of Arah6 using Protean approach
2.3.1.2 预测的Arah6线性表位
通过比较,用网络服务器NPS@预测得到的结果与Protean软件预测得到的结果基本相似,具有一定的可行性。当肽段位于β-转角和无规则卷曲区域[23-24],且亲水性>0,抗原指数>0,表面可及性>1时,即预测为优势B细胞抗原表位[25];且线性表位至少应由3~5个氨基酸组成。综合各参数可推导出Arah6的线性抗原表位优势区域为AA10~15,45~48,53~60,116~118(图3深色方框区域)。
2.3.2 基于线性表位对比的交叉反应预测
利用Clustal X对BLAST分析的结果进行多序列对比分析。结果显示:与Arah6具有完全相同的线性表位的蛋白质有4种,分别为Ara i 6、落花生conglutin、Ara d 6、conglutin 8(图3,其余图略),它们与目的过敏原蛋白质发生交叉反应的概率为100%;蔓花生conglutin和2S protein 1拥有2个与目的蛋白完全相同的线性表位;另外,拥有1个完全相同的线性表位的蛋白有9种,分别为表1中序号为7~15的蛋白质,它们多为不同花生亚种、羽扇豆科和大豆中的部分蛋白质,它们在很大程度上与目的蛋白可能发生交叉反应。

图3 线性表位(方框标示区域)对比分析Fig.3 Comparisons of linear epitopes between different proteins
2.4 基于Arah6构象型表位的免疫交叉反应预测结果
2.4.1 预测的Arah6构象型表位区
利用DiscoTope网络数据库预测得Arah6的构象型表位区有6个,用PyMOL软件绘制出来,如图4(彩色区域)所示,这6个表位区域在氨基酸序列中的位置分别为 AA1~13(红色)、16~17(绿色)、35~51(蓝色)、53~59(黄色)、86(紫色)、89~105(青色)。由于 AA16~17、86区域所含的氨基酸数目太少,无法构成构象型表位,故应剔除。综合考虑,Arah6的构象型表位区只有4个。

图4 Arah6的空间构象(灰色区域)和构象性表位区域(深色区域)Fig.4 Conformational epitopes on Arah6
2.4.2 基于构象型表位对比预测交叉反应
相同方法分别预测各蛋白质的空间构象型表位所在区域,并用PyMOL绘图,与目的蛋白进行对比分析。具有相同或相似的构象,即可发生交叉反应。

图5 不同蛋白相同/相似构象型表位对比图Fig.5 Conformational epitope comparison among different proteins
结果显示:与Arah6(图5R)具有相同的构象型表位(青色区域)的蛋白只有4种,分别为Ara i 6、落花生 conglutin、Ara d 6、conglutin 8(图 5A~D),它们与目的过敏原蛋白质发生交叉免疫反应的概率为100%;与目的蛋白具有相似构象型表位区域的蛋白有13种,分别为表1中序号分别为5~15(图5E~O)、17(图5P)和21(图5Q)的蛋白,它们在一定程度上也可能与Arah6发生交叉反应。
3 讨 论
近年来,随着生物信息学的迅速发展,已有许多分子生物学软件用于蛋白质的分析,线性表位和构象型表位的预测则是其中主要部分。关于线性表位的预测,目前被认可有效的方法主要包括二级结构方案、亲水性方案、可及性方案、可塑性方案、抗原性方案[23,26-27],但一般采用多种参数综合分析的方法。研究表明,在进行抗原线性表位预测时,运用生物信息软件和互联网服务器,采用多种方案综合分析可提高实验的成功率,增强实验的目的性[26,28]。本研究采用Protean软件和NPS@服务器预测了Arah6的线性表位,并用ABCpred对结果进行了验证,此法预测所得的B细胞线性表位区准确率达到73.9%。
由于已知晶体结构的抗原-抗体复合物数量较少、抗原-抗体结合机理研究不够清楚以及预测算法设计的困难性等方面原因,构象型表位预测的研究一直比较缓慢,远远落后于线性表位预测的研究[29]。但生物信息学软件DiscTope是目前新开发出的一种用于预测B细胞构象型表位的Web软件,它在95%特异度条件下,可正确识别构象型表位中的15.5%的残基,而普通线性表位预测方法在同等特异度条件下,只能识别约11%的残基[22]。本研究中采用DiscTope软件对目的蛋白进行了预测,得到了它的构象型表位分布优势区域。并通过绘图软件PyMOL对各相关蛋白的相关区域进行对比分析,结果表明拥有相同/相似的构象型表位优势区域即可与Arah6发生交叉反应。尽管如此,该结论最好通过抗体筛选噬菌体肽库等工作来进行实验验证[22]。
本研究基于生物信息学和网络数据库,分别从序列同源性分析、线性表位预测对比分析、构象型表位预测对比分析三个不同层次上对Arah6的交叉反应进行了预测。结果显示:Ara i 6、落花生conglutin、Ara d 6和落花生conglutin 8在序列同源性、线性表位和构象型表位上都与Arah6有很高的相似性,它们与目的蛋白发生交叉反应的概率为100%;构象型表位相似且线性表位相同的蛋白质,如Ara d 2.01、Arah 2.01、seed storage protein SSP2、白羽扇豆conglutin delta seed storage protein等,它们在理论上也能与目的蛋白发生交叉反应。另外,线性表位相同而构象型表位不同的蛋白也可能与目的蛋白发生交叉反应,如羽扇豆科conglutin delta、大豆napin-type 2S albumin 1、大豆2S albumin pre-propeptide等。
总之,本实验利用生物信息学方法,结合网络数据库,对花生蛋白质过敏原Arah6的交叉反应性进行了预测,从理论上阐述了Arah6与其他同源蛋白的免疫交叉反应性。
[1] 罗春萍, 高金燕, 胡纯秋, 等. 花生交叉过敏反应的研究进展[J]. 食品科学, 2009, 30(21):384-388.
[2] PARK C W, KIM G I, LEE C H. A comparison study on allergen components between Korean (Arachis fastigiataShinpung) and American peanut (Arachis hypogaeaRunner)[J]. KoreanMed Sci, 2000, 15(4):387-392.
[3] JIN Tengchuan, GUO Feng, CHEN Yuwei et al. Crystal structure of Arah 3, a major allergen in peanut[J]. Mol Immunol, 2009, 46(8/9):1796-1804.
[4] KLEBER J K, CRAMERI R, APPENZELLER U, et al. Selective cloning of peanut allergens including profilin and 2S albumins by phage disply technology[J]. Allergy Immunology, 1999, 119(4):265-274.
[5] 吴玉章, 朱锡华. 研究蛋白质抗原表位的方法学评述[J]. 国际免疫学杂志, 1991(5):245-249.
[6] 梁瑾. 一种B细胞构象表位预测方法的建立及初步应用[D]. 北京:中国农业科学院, 2009:13-16.
[7] GODMAN R. Practical and predictive bioinformatics methods for the identification of potentially cross-reactive protein matches[J]. Molecular Nutrition and Food Research, 2006, 50:655-660.
[8]HUANG Jian, HONDA W. “CED:a conformational epitope database”[DB]. BMC Immunol, 2006, 7:7. [2010-05-01](http://www.biomed central.com/1471-2172/717)
[9] SHARMA S, KUMAR P, BETZEL C, et al. Structure and function of proteins involved in milk allergies[J]. J Chromatogr B Biomed Sci Appl, 2001, 756(1/2):183-187.
[10] 李轶. GenBank数据库检索及其应用-Entrez检索功能[J]. 中华医学图书情报杂志, 2008,17(5):49-51.
[11] 谌容, 陈敏, 杨春贤, 等. 基于SWISS-MODEL的蛋白质三维结构建模[J]. 生命的化学, 2006, 26(1):54-56.
[12] 任秀艳, 高永闯, 李美茹, 等. HrpNEa蛋白的结构与功能分析[J]. 安徽农业科学, 2010(9):2254-2256.
[13] 黎明, 于天飞. DNAStar软件在动物病毒研究中的应用实例[J]. 高师理科学刊, 2010, 30(3):61-63.
[14] SAHA S, RAGHAVA G P S. Bcepred:prediction of continuous B-cell epitopes in antigenic sequences using physico-chemical properties[J].Lect Notes Comput Sci, 2004, 3239:197-2045.
[15] 李明才, 柳晓金, 苏绍波. 人白细胞介素-33蛋白的二级结构及B细胞抗原表位预测[J]. 汕头大学医学院学报, 2008, 21(1):1-3.
[16] ANDERSEN P H, NIELSEN M, LUND O. Prediction of residues in discontinuous B-cell epitopes using protein 3D structures[J]. Protein Sci, 2006, 15(11):2558-2567.
[17] 纪滨. H5V1亚型禽流感病HA蛋白抗原表位预测与筛选[D]. 长春:吉林大学, 2009.
[18] KVTE J, DOOLITTLE R F. A simple method for displaying the hydropathic character of a protein[J]. Mol Biol, 1982, 157:105-132.
[19] EMINI E A, HUGHES J V, PERLOW D S, et al. Induction of hepatitis a virus -neutralizing antibody by a virus - specific synthetic peptide[J]. J Viorol, 1985, 55:836-839.
[20] JAMESON B A, WOLF H. The antigenic index:a novel algorithmfor predicing antigenic determinants[J]. Comput Appl Biosci, 1988, 4(1):181-186.
[21] KARPLUS P A, SCHULZ G E. Prediction of chain flexibility in proteins [J]. Naturwissenschaften, 1985, 72(4):212-213.
[22] HASTA A P, NIELSEN M, LUND O. Prediction of residues in discontinuous B cell epitopes using protein 3D structures[J]. Protein Science,2006, 15:2558-2567.
[23] 吕燕波, 万瑛, 吴玉章, 等. SARS病毒基因组所编码的E蛋白的二级结构和B细胞表位预测[J]. 免疫学杂志, 2003, 19(6):407-410.
[24] 李海侠, 毛旭虎. 蛋白质抗原表位研究进展[J]. 微生物学免疫学进展, 2007, 35(1):54-58.
[25] 肖治军, 张改平, 乔松林. Ⅰ型与Ⅱ型IBDV前体多聚蛋白B细胞抗原表位的预测与比较[J]. 中国家禽, 2009, 31(21):22-25.
[26] MAEDA K, MIZUKOSHI F, HAMANO M, et al. Development of an equine heipesvirus type 4-specific enzyme-linked immunosorbent assay using a B-cell epitope as an antigen[J]. J Clin Microbiol, 2004, 42(3):1095-1098.
[27] MAHLER M, BLUTHNER M, POLLARD K M. Advances in B-cell epitope analysis of autoantigens in connective tissue diseases[J]. Clin Immunol, 2003, 107(2):65-79.
[28] ANANDARAO R, SWAMINATHAN S, KHANNA N. The identification of immunodominant linear epitopes of dengue type 2 virus capsid and NS4 a proteins using pin-bound peptides[J]. Vinus Res, 2005, 112(1/2):60-68.
[29] 黄艳新, 饱永利, 李玉新. 抗原表位预测的免疫信息学方法研究进展[J]. 中国免疫学杂志, 2008, 24(9):857-861.
Immunologic Cross-reactivity of Peanut Allergen Arah6 on the Basis of Epitope Prediction
ZHU Pan1,2,CHEN Hong-bing1,2,*,HU Chun-qiu1,2,LI Xin1,2,LUO Chun-ping1,2,3
(1. State Key Laboratory of Food Science and Technology, Nanchang University, Nanchang 330047, China;2. Sino-German Joint Research Institute, Nanchang University, Nanchang 330047, China;3. School of Environmental and Chemical Engineering,Nanchang University, Nanchang 330047, China)
Q517
A
1002-6630(2010)17-0318-05
2010-06-10
南昌大学食品科学与技术国家重点实验室目标导向项目(SKLF-MB-200807);江西省主要学科学术和技术带头人培养项目([2004]234号);教育部新世纪优秀人才支持计划项目(NCET-08-07-04)
朱盼(1987—),女,硕士研究生,研究方向为食品质量与安全。E-mail:xingmeng903@163.com
*通信作者:陈红兵(1967—),男,教授,博士,研究方向为食品营养与安全。E-mail:chbgjy@hotmail.com
