利用SPT3的定向进化提高工业酿酒酵母乙醇耐受性
2010-10-16赵心清姜如娇李宁杨晴白凤武
赵心清,姜如娇,李宁,杨晴,白凤武
大连理工大学生物科学与工程系,大连 116024
利用SPT3的定向进化提高工业酿酒酵母乙醇耐受性
赵心清,姜如娇,李宁,杨晴,白凤武
大连理工大学生物科学与工程系,大连 116024
利用对转录因子的定向进化可对多基因控制的性状进行有效的代谢工程改造。本研究对酿酒酵母负责胁迫相关基因转录的SAGA复合体成分SPT3编码基因进行易错PCR随机突变,并研究了SPT3的定向进化对酿酒酵母乙醇耐性的影响。将SPT3的易错PCR产物连接改造的pYES2.0表达载体并转化酿酒酵母Saccharomyces cerevisiae 4126,构建了突变体文库。通过筛选在高浓度乙醇中耐受性提高的突变株,获得了一株在10%(V/V)乙醇中生长较好的突变株M25。该突变株利用125 g/L的葡萄糖进行乙醇发酵时,终点乙醇产量比对照菌株提高了11.7%。由此表明,SPT3是对酿酒酵母乙醇耐性进行代谢工程改造的一个重要的转录因子。
定向进化,乙醇耐性,SPT3,酿酒酵母,易错PCR
Abstract:Directed evolution of transcription factors can be employed to effectively improve the phenotypes which are controlled by multiple genetic loci.In this study, we used error-prone PCR for the directed evolution of SPT3, which is the component of yeast Spt-Ada-Gcn5-acetyltransferase(SAGA)complex responsible for the transcription of stress-related genes,and studied its effect on the improvement of ethanol tolerance.Mutant library was constructed by ligating the error-prone PCR products with a modified pYES2.0 plasmid, and the expression plasmids were subsequently transformed to yeast industrial strain Saccharomyces cerevisiae 4126.One mutant strain M25 showing superior growth in presence of 10% ethanol was selected.M25 produced 11.7% more ethanol than the control strain harboring the empty vector when 125 g/L glucose was used as substrate.This study revealed that SPT3 is an important transcription factor for the metabolic engineering of yeast ethanol tolerance.
Keywords:directed evolution, ethanol tolerance, SPT3, Saccharomyces cerevisiae, error-prone PCR
利用易错PCR(Error prone PCR)或DNA 改组(DNA shuffling)对酶分子编码基因进行的定向进化,可获得催化效率提高的酶蛋白,并改善酶的一系列性质,如稳定性[1-2]、底物特异性[3]等,是蛋白质工程的重要研究工具。近年来,定向进化技术也成功应用于转录因子等控制细胞全局转录的调节蛋白的突变,并在酵母菌[4]、乳杆菌[5]等微生物的代谢工程改造中得到成功应用。酵母菌的乙醇耐受性与多个基因有关[6],因而通过传统的单基因敲除或过量表达很难达到提高酵母菌乙醇耐性的目的。2006年Alper等报道了通过全局转录工程(Global transcriptional machinery engineering,gTME)的方法提高酵母菌乙醇耐性的开创性研究,为酵母菌乙醇耐性的代谢工程操作提供了新的思路[4]。对乙醇耐性提高的突变菌株中SPT15蛋白的突变分析表明,其编码蛋白有3个氨基酸残基的突变,而对这 3个氨基酸残基位点的单独定点突变并没有起到同样的效果,因此作者提出,这 3个氨基酸同时突变的结果可能影响了SPT15与SPT3蛋白的结合,因为这3个突变在SPT3基因敲除突变体中没有起到同样的突变效果[4]。
SPT3是酵母菌转录起始共激活因子复合体SAGA(Spt-Ada-Gcn5-acetyltransferase)的成分之一,负责引导TATA结合蛋白TBP(TATA-binding protein)结合到特定的启动子区[7],近年的研究发现,SAGA复合体与多种环境胁迫因素相关基因的转录有关,这些胁迫因素包括高温、营养缺乏、DNA损伤等[8]。而SPT3的功能还不仅限于SAGA复合体成分,研究表明SPT3还与Ccr4-Not 复合体的功能有关[9],而这个复合体可影响酵母细胞内多种生理代谢过程,包括影响乙醇脱氢酶ADH2基因[10]等可能与乙醇发酵相关的基因的表达。此外,SPT3还参与调控酵母菌 NADP-特异性谷氨酸脱氢酶基因GDH1的调控,从而影响细胞内的辅酶平衡[11]。因此,SPT3基因在酵母细胞生长代谢以及乙醇合成等多方面生理过程中具有重要的调节作用,而且这种调节作用不局限于已报道的与SPT3与SPT15的结合,SPT3的其他功能也可能对细胞的转录机制产生重要影响。
国内学者利用gTME技术,对SPT15基因进行定向进化,获得了高效利用木糖并共代谢木糖和葡萄糖的酿酒酵母菌株[12],但对 SPT3在乙醇耐性中的研究还没有报道。在利用酵母单基因缺失突变体研究与乙醇耐性相关基因的报道中,发现SPT3缺失突变体对乙醇的敏感性提高,暗示了SPT3在酿酒酵母乙醇耐性中的重要作用[13]。本研究通过对 SPT3编码基因进行易错PCR,构建了突变体文库,并研究了SPT3的定向进化对酿酒酵母乙醇耐性的影响,证明了SPT3的突变可提高酿酒酵母的乙醇耐受性。
1 材料和方法
1.1 微生物菌种和培养基
大肠杆菌E.coli DH5α、工业酿酒酵母S.cerevisiae 4126[14]为本实验室保存。酿酒酵母菌株S288C由瑞典 Chalmers大学 Jens Nielson教授惠赠。质粒pFA6a-kanMX4由清华大学陈国强教授惠赠。大肠杆菌-酵母穿梭载体pYES2.0由大连理工大学李文利副教授惠赠。
大肠杆菌用 LB 培养基(g/L:酵母粉 5,胰蛋白胨10,NaCl 10)培养,转化子培养添加50 µg/mL氨苄青霉素。酿酒酵母转化子用 YPD培养基培养(g/L:酵母粉 4,蛋白胨 3,葡萄糖 20),液体培养时添加 100 µg/mL G418,固体培养时添加 300 µg/mL G418。乙醇发酵培养基(g/L:酵母粉5,蛋白胨4,葡萄糖125)。PCR相关试剂、T4 DNA连接酶、DNA快速连接试剂盒、限制性内切酶EcoR I、BamH I、pMD 20-T Vector均为宝生物工程(大连)有限公司的产品。限制性内切酶Nde I、Nhe I购自NEB公司。DNA纯化试剂盒为GE公司产品。凝胶回收试剂盒、抗生素购自Sigma公司。
1.2 实验方法
1.2.1 酿酒酵母基因组的提取
酿酒酵母S288C基因组DNA的提取参考文献[15]的方法进行,作为扩增SPT3基因的模板。
1.2.2 SPT3表达载体pRJ04及易错PCR突变文库的构建
本研究中所用的引物序列见表 1。引物 Kan-F/Kan-R用于扩增G418抗性标记基因,在正向引物中加入Nhe I酶切位点,反向引物中加入Nde I酶切位点。以质粒pFA6a-kanMX4为模板,扩增产物连接到pMD 20-T Vector,构建pMD20-T Kan。再分别对pYES2.0和pMD20-T Kan进行Nde I/Nhe I双酶切,凝胶回收5.7 kb的pYES2.0载体骨架片段和1.5 kb的pMD20-T 的Kan片段并连接,构建pYES2.0骨架上引入G418抗性基因的pRJ01。
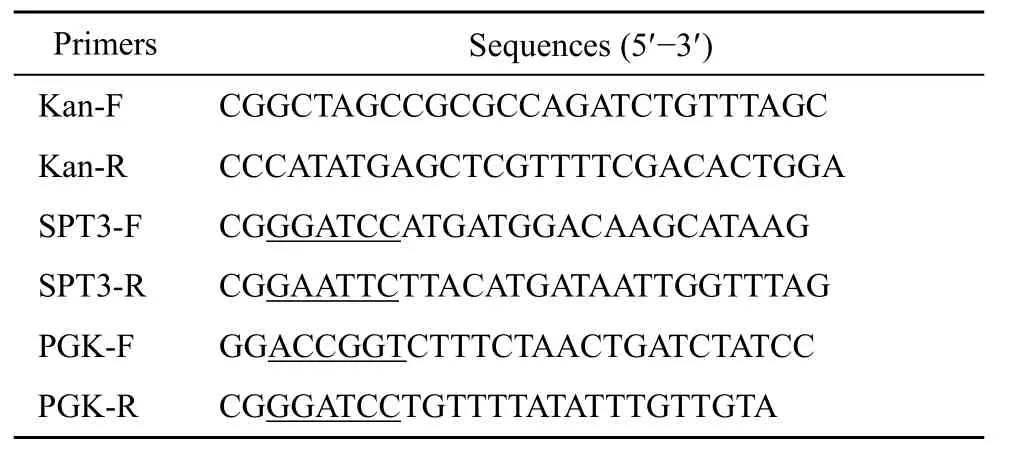
表1 本研究所用的引物Table 1 Primers used in this study
采用引物SPT3-F/SPT3-R(分别带有BamH I和EcoR I酶切位点)扩增得到SPT3基因,并连接到pMD 20-T Vector,得到pRJ02质粒。再利用EcoR I/BamH I酶切 pRJ01和 pRJ02,构建 pYES 2.0骨架上含有SPT3和G418抗性基因的pRJ03。利用引物PGK-F/PGK-R(分别带有 Age I和 BamH I酶切位点)扩增PGK启动子,将启动子连接入pRJ03,获得SPT3表达载体pRJ04。
易错 PCR突变反应体系如下:SPT3-F/SPT3-R(10 pmol/L 各 1 µL,10× buffer(Mg2+free)2.5 µL,Taq 酶 0.1 µL,dNTP Mixture(2.5 mmol/L)2 µL,pRJ02 2 µL,MnCl2(5 mmol/L)1 µL,MgCl2(25 mmol/L)7 µL,ddH2O 补充至 25 µL。PCR 扩增条件为 94℃ 30 s;94℃ 30 s,54℃ 30 s,72℃ 1.5 min,30个循环;72 ℃ 1 min 。将获得的载体混合物经BamH I和EcoR I酶切,连接入经同样酶切处理的pRJ04,构建SPT3表达载体的易错PCR文库,并转化感受态E.coli DH5α,构建突变体基因文库。提取突变基因文库的质粒,将混合质粒用电击转化法转化酿酒酵母S.cerevisiae 4126,获得的酵母转化子构成酵母突变体文库,共获得429个酵母菌落。将pRJ01空载体转化4126酵母菌株,获得酵母对照菌株。
1.2.3 SPT3突变体酵母文库的筛选
利用在高浓度乙醇中培养的方法富集乙醇耐性提高的突变体。将酵母突变体文库平板上的克隆用适量YPD液体培养基洗入摇瓶,于YPD液体培养基中30℃、150 r/min培养16 h。以2%接种量接种于含10%乙醇的YPD中,30℃、150 r/min培养24 h,然后取 1 mL培养物转接于 50 mL含 12%乙醇的YPD中,培养24 h后稀释涂平板。2 d后生长出来的菌落进行进一步的筛选,挑选较大的单菌落在试管中用3 mL YPD液体培养基,30℃、150 r/min培养16 h,测量其OD600,并按照相同起始OD600值接种于含10%乙醇的YPD液体培养基,48 h后测量其OD600吸收值。取在10%乙醇中生长好于对照的突变体,按2%的接种量接种于250 mL摇瓶(内有50 mL含10%乙醇的YPD)中,控制起始OD值一致,48 h后测量OD600,比较其生长情况,与含有空载体的对照菌株相比在10%乙醇中生长最好的为乙醇耐性提高的突变体。
1.2.4 突变体乙醇耐性和发酵性能的研究
将在YPD斜面上活化培养后的菌株接种于乙醇浓度为10%的YPD液体培养基中,150 r/min、30℃条件下培养,定期测量菌体OD600,绘制生长曲线,以菌体 OD值表征乙醇耐性,同时检测在不含有乙醇的YPD液体培养基中各菌株的生长情况。
将菌株在YPD斜面上活化培养后接种于发酵培养基,150 r/min、30℃条件下培养。定时取样测量发酵液的乙醇浓度。乙醇浓度的检测用HPLC方法,参考文献[16]。
菌株的生长测定和乙醇发酵实验均重复至少 2次,每次实验至少有2个平行,结果取测试的平均值,所有结果都具有可重复性。
1.2.5 突变体的遗传稳定性研究
突变体在含有YPD的斜面传代,每48 h转接1次,共传代 9次,用传代后的菌种进行生长和发酵实验,并和传代初的菌种进行比较,研究其遗传稳定性。
2 结果与讨论
2.1 SPT3对酵母菌乙醇耐性的影响
虽然 SPT3的缺失导致酿酒酵母在乙醇中的生长受到抑制[13],但对其过量表达后乙醇耐性的变化尚未见报道。为进一步确定SPT3与酿酒酵母乙醇耐性的关系,首先比较了SPT3过量表达菌株的生长和对高浓度乙醇的反应。SPT3过量表达菌株的生长与含有空载体的对照菌株基本一致,在10%乙醇中的生长也与对照无明显差别(结果未显示),说明过量积累SPT3对酵母菌的生长和代谢没有明显影响。进一步研究了 SPT3突变基因对酿酒酵母菌株S.cerevisiae 4126乙醇耐性的影响,在易错PCR突变体文库中随机挑取2个突变体M1和M2进行SPT3突变基因测序,并研究了2个突变体在10%乙醇中的生长情况。突变体在YPD培养基中的生长与对照类似,但在10%的乙醇中,突变体的生长明显滞后于对照菌株,体现在延迟期的延长和最终生物量的明显减少,测序结果表明,M1突变体中SPT3基因有6个氨基酸突变,其中 253位的Lys突变为终止密码,M2突变株同样具有6个氨基酸突变位点,分别分散在SPT3的N端和C端不同区域。上述结果表明,SPT3在酵母菌乙醇耐性中具有重要作用,其随机突变后有可能筛选到乙醇耐性提高的突变体。
2.2 SPT3易错PCR突变体文库的筛选和突变体乙醇耐性和温度耐性的研究
对SPT3的易错PCR突变体文库中的单菌落进行乙醇耐性的富集筛选,在初筛中获得了11株乙醇耐性高于对照的突变体,在摇瓶复筛中获得了乙醇耐性提高最明显的突变体M25。该突变体在YPD液体培养基中的生长与对照相似,但在10%乙醇中的生长明显优于对照株(图1),尤其在对数期差异最明显,36 h的OD600值是对照的2.62倍。
由于文献报道 SAGA复合体还与温度胁迫有关[8],本实验在不同培养温度下进行了突变体文库的筛选,但突变体文库在37℃、40℃温度下未筛选到耐高温的突变体,将耐乙醇的突变体在不同高温条件下(37℃、40℃)培养,也未发现耐温性的明显变化,说明本实验条件下未得到温度耐性改变的突变体,SPT3的突变能否引起酵母菌温度耐性的提高有待进一步研究。
2.3 突变株M25的乙醇发酵性能
对突变株M25和对照菌株的乙醇发酵实验表明,32 h后,突变株和野生型均将培养基中的糖耗尽(数据未显示),细胞生长无明显差别(图2A),但突变株M25的发酵终点乙醇产量略高于对照株(图2B),对照菌株36 h发酵产生的乙醇为48.0 g/L,而突变株为53.6 g/L,乙醇体积产率达到1.675 g/(L·h),比对照株提高了11.7%。突变体在多次传代后检测遗传稳定性,证明发酵性能比较稳定。
2.4 突变株的SPT3基因突变位点
对突变株 M25中的 SPT3基因进行测序结果表明,SPT3蛋白的337个氨基酸中,共8处氨基酸残基发生了突变,分别为:Ser18Pro、Ile59Thr、Thr104Ser、Lys135Arg、Arg177Gly、Lys179Glu、Cys199Ser、Asp214Gly。其中个别残基的突变,如18、59、177、214位氨基酸的突变改变了侧链的极性,可能造成蛋白质空间结构的变化,从而影响SPT3与其他蛋白复合体成分的结合,从而影响多个基因的转录。对突变位点的氨基酸进行定点突变,并研究单独或组合定点突变对乙醇耐性的影响,可获得SPT3蛋白中对乙醇耐性起关键作用的氨基酸残基的信息。
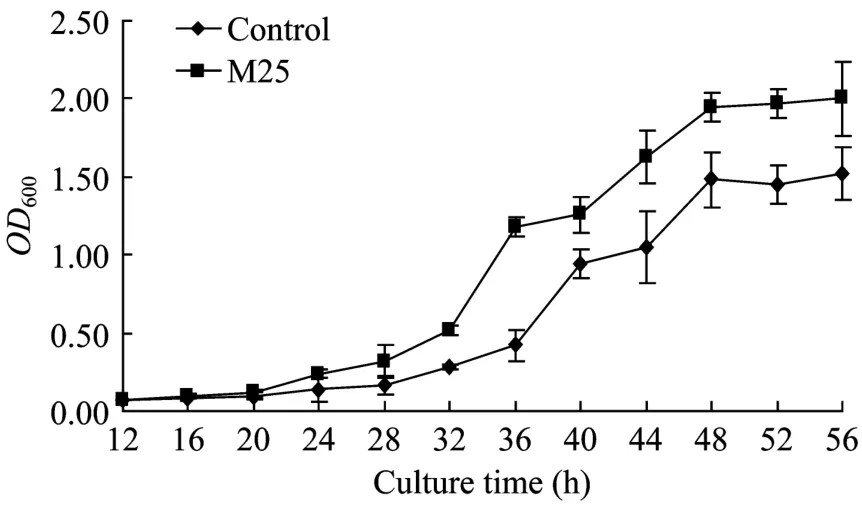
图1 突变体M25与对照菌株在10%乙醇中生长的差别Fig.1 Growth difference of mutant strain M25 in 10% ethanol comparing with that of control.
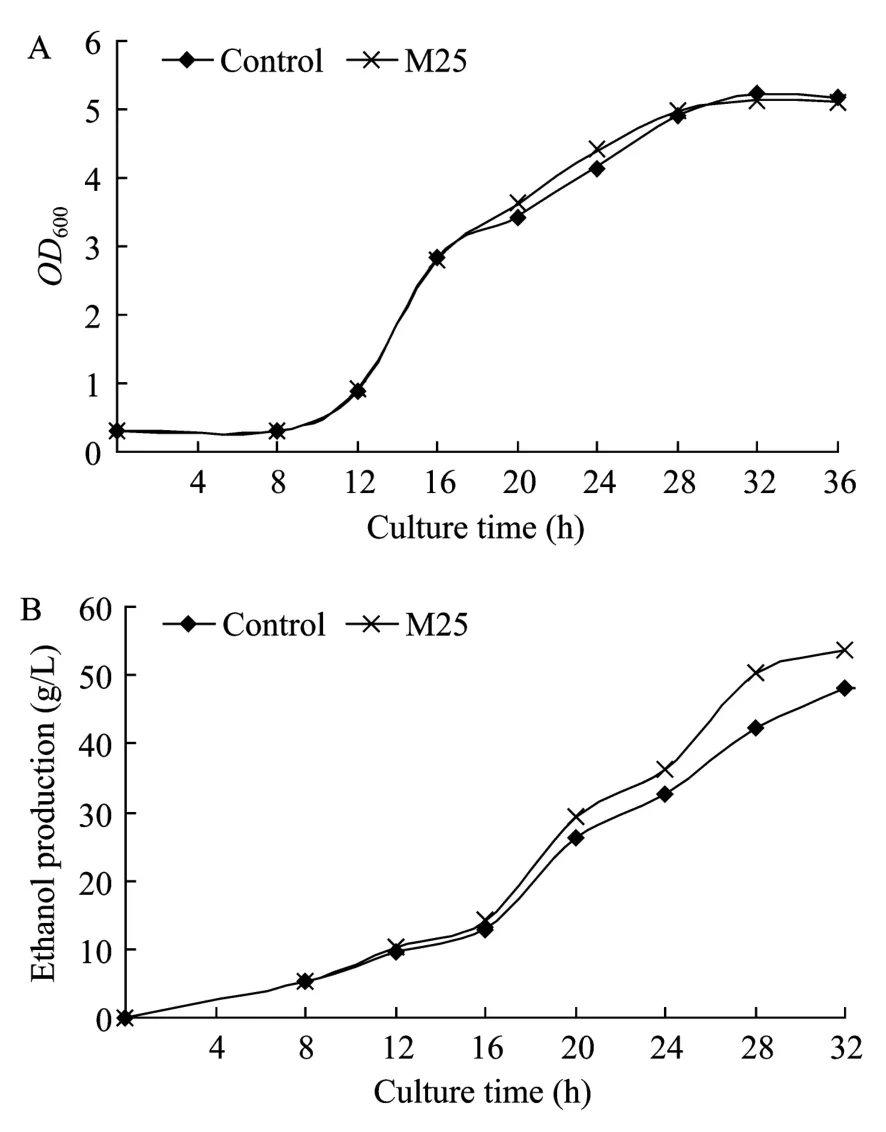
图2 突变体M25和对照菌发酵过程中细胞生长(A)和乙醇产量(B)比较Fig.2 Cell growth(A)and ethanol production(B)of mutant M25 comparing with that of control.
2006年Stephanopoulos研究小组首次报道通过全局转录工程(gTME)提高酵母菌的乙醇耐性,发现了酵母 RNA聚合酶 II转录复合体 TFIID成分SPT15中3个氨基酸的突变提高了酵母菌的乙醇耐性和乙醇产量[4]。他们采用的是低拷贝载体和弱启动子,目前还不清楚突变基因拷贝数的多少和启动子的强弱对突变体生理特性的影响,以及SPT3的突变频率对乙醇耐性的影响。进一步的研究将利用多种表达载体和启动子活性研究对突变文库特性的影响,已经证明提高易错PCR体系的模板量可降低突变频率,未来的研究将深入揭示SPT3的突变在酿酒酵母乙醇耐性中的作用。
Madison和 Winsto研究了乳酸克鲁维酵母Kluyveromyces lactis、裂殖酵母Schizosaccharomyces pombe和Clavispora opuntiae中的SPT3,证明了在不同的酵母中 SPT3的功能是保守的[17]。目前还不清楚导致酿酒酵母乙醇耐性提高的 SPT3的突变是否也可以提高其他酵母的乙醇耐受性和胁迫耐受性。BLAST搜索显示,多种低等真核生物都有SPT3同源的蛋白,在人类基因组中也有与SPT3部分同源的蛋白[18],对这些蛋白与各种胁迫因素的深入研究将进一步揭示SPT3对不同生物细胞代谢的深刻影响。
在本文的准备过程中,Hou等报道了在工业酵母 S.cerevisiae TH-AADY中过量表达 SPT3和SPT15突变基因提高酵母菌乙醇耐性的结果[18],在他们的研究中,过量表达SPT3提高了酵母菌的乙醇耐受性,与本实验的结果不一致,推测SPT3的作用可能和酵母菌的遗传背景有关。而本研究首次报道了利用SPT3的定向进化提高酵母菌的乙醇耐受性,为构建乙醇耐性提高的工业酵母菌株奠定了基础。本实验中 SPT3的定向进化得到了多株乙醇耐性提高的突变体,但多数突变体乙醇发酵性能没有明显变化。进一步优化突变频率,调节载体拷贝数和调节启动子活性,可能获得乙醇耐性、渗透耐性、高温耐性和乙醇产量均提高的新一代工业乙醇酵母。
3 结论
利用酿酒酵母 SPT3基因的易错 PCR,建立突变体文库,获得了乙醇耐性提高的酿酒酵母突变体,证明 SPT3基因可用于酿酒酵母乙醇耐性的代谢工程改造,本研究提供了利用全局转录工程(gTME)技术提高酵母菌乙醇耐性的又一个靶点基因。进一步的研究将揭示突变株乙醇耐性提高的分子机制,为构建乙醇耐受性和其他胁迫耐性提高的工业酵母,提高乙醇发酵的经济技术水平奠定了基础。
REFERENCES
[1]Eijsink VG, Gaseidnes S, Borchert TV, et al.Directed evolution of enzyme stability.Biomol Eng, 2005, 22:21–30.
[2]Frances HA, Patrick LW, Kentaro M, et al.How enzymes adapt: lessons from directed evolution.Trends Biochem Sci, 2001, 26(2): 100–106.
[3]Nicholas JT.Directed evolution of enzymes for applied biocatalysis.Trends Biotechnol, 2003, 21(11): 474–478.
[4]Alper H, Moxley J, Nevoigt E, et al.Engineering yeast transcription machinery for improved ethanol tolerance and production.Science, 2006, 314(5805): 1565–1568.
[5]Klein MD, Stephanopoulos G.Assessing the potential of mutational strategies to elicit new phenotypes in industrial strains.Proc Natl Acad Sci USA, 2008, 105(7): 2319–2324.
[6]Fujita K, Matsuyama A, Kobayashi Y, et al.The genome-wide screening of yeast deletion mutants to identify the genes required for tolerance to ethanol and other alcohols.FEMS Yeast Res, 2006, 6: 744–750.
[7]Mohibullah N, Hahn S.Site-specific cross-linking of TBP in vivo and in vitro reveals a direct functional interaction with the SAGA subunit Spt3.Gens Dev, 2008, 22:2994–3006.
[8]Huisinga KL, Pugh BF.A Genome-wide Housekeeping Role for TFIID and a highly regulated stress-related role for SAGA in Saccharomyces cerevisiae.Molecular Cell,2004, 13: 573–585.
[9]James N, Landrieux E.A SAGA-independent function of SPT3 mediates transcriptional deregulation in a mutant of the Ccr4-Not complex in Saccharomyces cerevisiae.Genetics, 2007, 177: 123–135.
[10]Malvar T, Biron RW.The CCR4 proten from Saccharomyces cerevisiae contains a leucine-rich repeatregion which is required for its control of ADH2 gene expression.Genetics, 1992, 132: 951–962.
[11]Riego L, Avendano A, DeLuna A, et al.GDH1 expression is regulated by GLN3, GCN4, and HAP4 under respiratory growth.Biochem Biophys Res Commun, 2002, 293: 79–85.
[12]Liu HM, Xu L, Yan M, et al.gTME for construction of recombinant yeast co-fermenting xylose and glucose.Chin J Biotech, 2008, 24(6): 1010–1015.刘红梅, 许琳, 严明, 等.gTME构建共发酵木糖和葡萄糖的重组酿酒酵母.生物工程学报, 2008, 24(6):1010–1015.
[13]van Voorst F, Houghton-Larsen J, Jønson L, et al.Genome-wide identification of genes required for growth of Saccharomyces cerevisiae under ethanol stress.Yeast,2006, 23(5): 351–359.
[14]Choi EY, Park JN, Kim HO, et al.Construction of an industrial polyploid strain of Saccharomyces cerevisiae containing Saprolegnia ferax β-amylase gene and secreting β-amylase.Biotechnol Lett, 2001, 24: 1785–1790.
[15]Burke D, Dawson D, Stearns T.Methods in Yeast Genetics.New York: Cold Spring Harbor Laboratory Press, 2000.
[16]Lei JJ, Zhao XQ, Xue C, et al.Influence of floc size distribution on the ethanol tolerance of a self-flocculating yeast strain SPSC01.Chin J Biotech, 2008, 24(2):309–314.雷娟娟, 赵心清, 薛闯, 等.絮凝颗粒粒度分布对自絮凝酵母 SPSC01乙醇耐受能力的影响.生物工程学报,2008, 24(2): 309–314.
[17]Madison JM, Winston F.Identification and analysis of homologues of Saccharomyces cerevisiae Spt3 suggest conserved functional domains.Yeast, 1998, 14(5):409–417.
[18]Hou L, Cao X, Wang C, et al.Effect of overexpression of transcription factors on the fermentation properties of Saccharomyces cerevisiae industrial strains.Lett Appl Microbiol, 2009, 49(1): 14–19.
Improving ethanol tolerance of Saccharomyces cerevisiae industrial strain by directed evolution of SPT3
Xinqing Zhao, Rujiao Jiang, Ning Li, Qing Yang, and Fengwu Bai
Department of Bioscience and Bioengineering, Dalian University of Technology, Dalian 116024, China
Received:October 16, 2009;Accepted:December 15, 2009
Supported by:Natural Science Foundation of China(No.30500011), National High Technology Research and Development Program of China(863 Program)(No.2007AA10Z358).
Corresponding author:Xinqing Zhao.Tel: +86-411-84706308; Fax: +86-411-84706329; E-mail: xqzhao@dlut.edu.cn国家自然科学基金项目(No.30500011),国家高科技研究发展计划(863计划)(No.2007AA10Z358)资助。
