运用基因芯片技术检测牛、山羊、猪和鸡源性成分
2010-10-11石丰运缪建锟张利平陶虹吕建强阮周曦宗卉
石丰运,缪建锟,张利平,陶虹,吕建强,阮周曦,宗卉
1 甘肃农业大学动物科学技术学院,兰州 730070
2 深圳出入境检验检疫局,深圳 518010
3 中国农业大学农业生物技术国家重点实验室,北京 100193
生物技术与方法
运用基因芯片技术检测牛、山羊、猪和鸡源性成分
石丰运1,2,缪建锟3,张利平1,陶虹2,吕建强2,阮周曦2,宗卉2
1 甘肃农业大学动物科学技术学院,兰州 730070
2 深圳出入境检验检疫局,深圳 518010
3 中国农业大学农业生物技术国家重点实验室,北京 100193
本研究通过对脊椎动物分子标记基因进行序列分析,最终选择线粒体DNA (mtDNA) 16S rRNA基因为目标基因,利用一对通用引物,在该引物扩增区间设计了4条特异性基因芯片检测探针及2条质控探针用于对牛、山羊、猪、鸡等4种动物源性成分进行检测。通过对PCR扩增体系及杂交体系的优化,该检测方法能实现对上述4种动物源性成分同时进行快速、准确地检测,具有很好的特异性,灵敏度均达到1 pg,最终建立了这4种动物源性基因芯片检测方法。该基因芯片检测技术将为我国进出口饲料中的动物源性成分的鉴别提供新的检测方法和技术支持。
线粒体16S rRNA基因,DNA芯片,动物源性
Abstract:We analyzed the sequence of vertebrate molecular marker genes, then we selected the mitochondrial DNA (mtDNA)16S rRNA gene as marker gene. In order to detect four kinds of animal-derived ingredients, which including bovine, goat, pig and chicken. We utilized a pair of universal primers, designed four sets of species-specific microarray probes and two pairs of quality control probes. We optimized the PCR amplifications and hybridization conditions, therefore these four kinds of animal-derived ingredients could be rapid and accurate detected by this approach. The detection limits were all reaches 1 pg. We established the detection platform of these four kinds of animal-derived ingredients. This universal PCR-microarray assay provides a new method for the identification of animal-derived ingredients in the import-export field.
Keywords:mitochondrion 16S rRNA gene, DNA chip, animal-derived ingredients
随着国际贸易的飞速发展,防御国际间动物疫病传播的难度也在不断加大。为了防止疯牛病、痒病、禽流感、甲流等经饲料、畜产品传播,世界各国政府和组织在规范对动物源性饲料使用的同时,积极开展动物源性成分检测技术方面的研究。目前国内外对于动物源性成分的检测方法的报道主要有显微镜检法、红外光谱、ELISA、普通 PCR、荧光定量PCR等方法。但上述的分子生物学方法尚不能很好满足快速、大规模、高通量检测的需要。因此,需要建立一种能快速准确鉴别、检测动物源性成分的方法,提高检验检疫通关速度,对保护我国畜牧业的安全生产具有重要的意义。
基因芯片基本原理是将核酸片段作为识别分子,按预先设置的排列固定于特定的固相支持载体的表面形成微点阵,利用反向固相杂交技术,将标记的样品分子与微点阵上的核酸探针杂交,以实现多到数万个分子之间的杂交反应,通过特殊的检测系统进行高通量、大规模地分析检测样品中多个基因的表达状况或者特定基因分子的存在[1]。基因芯片因具有高通量、高度并行性、高灵敏度等优点而得到了广泛的应用[2-3]。基因芯片目前主要产品包括基因表达谱芯片,转基因农产品检测芯片,新生儿基因检测芯片,肝炎病毒、艾滋病毒基因检测芯片,其中肿瘤检测、肝病检测、自身免疫疾病诊断芯片即将或已经进入临床应用和商业化运作。物种鉴定检测芯片研制仍处于起步阶段,如应用于临床致病真菌鉴定[4]和水产食品中常见病原微生物鉴定[5]等。但迄今为止,基因芯片技术应用于动物源性成分鉴定检测还未见报道。因此,本研究尝试建立用基因芯片技术同时对牛、山羊、猪、鸡 4种动物成分的快速检测方法,较好地弥补了常规分子检测方法的缺陷,能实现一次反应完成对多种动物源性成分的种类初筛和鉴定,为我国进出口饲料及畜产品快速筛查和种类鉴定提供有效的技术支持。
1 材料与方法
1.1 样品
牛肉、山羊肉、猪肉、鸡肉、绵羊肉、狗肉、兔肉、鸭肉、鹅肉、鹌鹑肉、火鸡肉、鹧鸪肉和鱼肉均购自广东深圳超市,驴肉和鹿肉均购自内蒙古呼和浩特超市。这些样品均经过品种鉴定并测序验证,结果证明均为其名称所述之物种。
1.2 试剂和仪器
细胞/组织-基因组提取试剂盒购自北京天根生物公司。ExTaqDNA聚合酶、dNTPs、DNA Marker DL2000购自宝生物工程 (大连) 有限公司。醛基化DNA芯片基片及盖片、4孔围栏购自北京博奥生物公司。
NANO-PLOTTER2芯片点样仪、GenePix4200A芯片扫描仪、ABI Verti PCR仪、凝胶成像系统(Bio-Rad公司)、烘箱 (BINDER)、核酸蛋白分析仪DU800 (Beckman) 均由深圳出入境检验检疫局动植中心提供。
1.3 引物和探针的设计
根据相关检测文献及对牛、山羊、猪、鸡、鱼等动物的DNA分子标记基因序列进行系统分析,最终选择mtDNA 16S rRNA基因为目标基因。根据文献选用了一对可扩增该基因的通用引物 Universal-Forward/Reverse[6],在引物的扩增区间设计出一系列探针,用于检测牛、山羊、猪、鸡源性成分。将候选探针序列用 GenBank的 Blastn软件进行比对分析,并进行实验验证,经过筛选选择了特异性较好的序列作为固相芯片检测探针,质控探针包括阳性定位点探针及Cy5标记互补链、阴性质控探针。质控探针均与牛、山羊、猪、鸡、鱼以及马等动物mtDNA的16S rRNA基因序列无同源性。引物和探针的合成、修饰由上海超世生物技术有限公司完成,具体序列见表1。
1.4 芯片的制备
固相探针用1× TE溶解至终浓度为40 µmol/L,与 50% DMSO等体积混匀后作为探针点样混合液,加入384孔板中,空白对照为50% DMSO,按预先设计的探针点阵排布顺序,点样至基片上。点样后基片在湿盒中37℃烘箱中水合12 h,用 0.2%SDS洗液漂洗2 min,去离子水漂洗3次,每次2 min。再放入0.2% NaBH4封闭液中封闭15 min,中间静置5 min;去离子水漂洗3次,每次2 min,2 000 r/min离心2 min干燥,4℃避光保存备用。
1.5 基因芯片的阵列设计
每张醛基化玻片上固定着4个矩阵,探针点阵排布如图1所示,每个阵列共4行11列,第1列和第1行均为阳性定位点探针,其他探针各设5个重复。
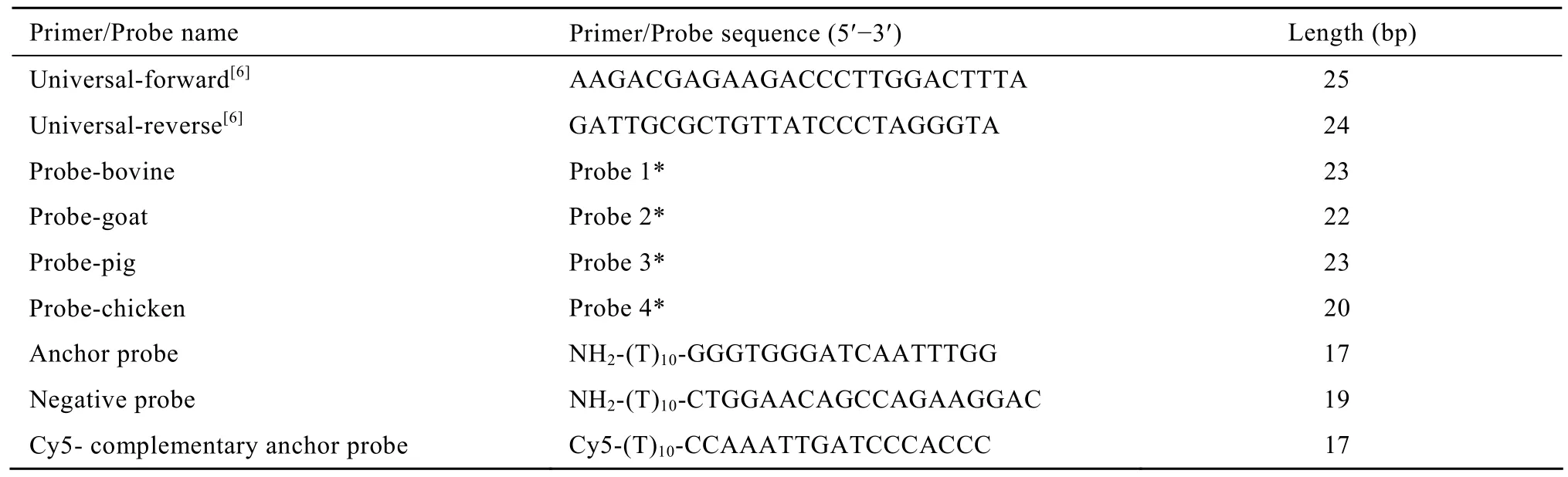
表1 动物源性基因芯片通用引物、检测探针和质控探针Table 1 Universal primers, species- specific probes and quality control probes used in this study

图1 基因芯片探针排布图Fig.1 Layout of microarray probes. A: anchor probe; N: negative probe; B: blank control.
1.6 荧光掺入法PCR扩增与标记
荧光掺入法PCR反应体系为:10× PCR buffer 2.5 µL、Mg2+2 µL、正向引物和反向引物各 0.5 µL(10 µmol/L)、Taq酶 0.2 µL (5 U/µL)、Cy5-dNTPs 0.4 µL、DNA 模板 (10 ng/µL) 1 µL、ddH2O 补足总体积到25 µL。空白对照为ddH2O代替DNA。PCR扩增条件为:94℃预变性 5 min;94℃变性30s,55℃退火30s,72℃延伸30s,共35个循环;72℃延伸 5 min。Cy5-dNTPs 配方为 ddH2O 2.85 µL、dATP(10 mmol/L) 1 µL、dCTP (10 mmol/L) 1 µL、dGTP(10 mmol/L) 1 µL、dTTP (10 mmol/L) 0.65 µL、Cy5-dCTP (1 mmol/L) 3.5 µL。
1.7 杂交及扫描
分别取 7 µL 芯片杂交液 (5× SSC,0.1% SDS),6 µL PCR标记产物、Cy5标记阳性定位探针互补链1 µL,混匀后95℃变性5 min,立即冰浴5 min。将其按预计排布顺序加入芯片点样区,盖上盖玻片置于杂交盒中52℃烘箱中避光杂交2 h。杂交后的芯片依次放入42℃预热洗液I (0.3× SSC,0.2%SDS)和洗液 II (0.06× SSC) 磁力搅拌清洗,各 3 min,2 000 r/min离心2 min干燥。激光共聚焦扫描仪设置为在635 nm的激发波长扫描芯片,激光功率为60%,PMT设置为60%进行扫描。图像用GenePixs V5.0软件分析,以JPEG和TIFF的格式保存。
1.8 特异性实验
为了更好地评价本检测方法的特异性,共选取15种不同种的动物组织 (见 1.1),用通用引物Universal-Forward/Reverse进行荧光掺入法 PCR扩增,扩增后产物进行1.5%琼脂糖凝胶电泳检测,杂交、扫描过程见1.7。
1.9 灵敏度实验
将提取的牛、山羊、猪、鸡的DNA模板原液测量浓度后母液均定量至 10 ng/µL,分别进行 10×梯度稀释,即 10−1~10−6共 6个梯度。用通用引物Universal-Forward/Reverse进行荧光掺入法 PCR扩增,扩增后产物进行1.5%琼脂糖凝胶电泳检测,杂交、扫描过程见1.7。
2 结果与分析
2.1 靶基因片段荧光掺入法PCR扩增结果
分别用牛、山羊、猪、鸡、绵羊、鹿、驴、狗、兔、鸭、鹅、鹌鹑、火鸡、鹧鸪、鱼等15种动物源性DNA模板和空白对照进行荧光掺入法PCR扩增,电泳检测结果见图 2。通用引物 Universal-Forward/
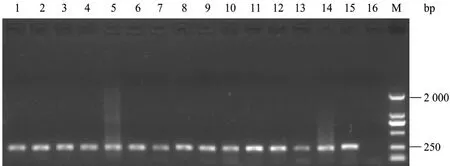
图2 通用引物动物源性PCR扩增电泳图Fig.2 Agarose electrophoresis analysis of PCR products by primers Universal-Forward/Reverse. M: DNA marker DL 2 000; 1−16:bovine, goat, pig, chicken, sheep, donkey, deer, dog, rabbit, duck, goose, quail, turkey, artridge, fish, blank control.
Reverse能有效扩增出以上15种动物源性的DNA模板,PCR产物经电泳分离,回收纯化并测序,结果表明15种动物源性PCR产物序列长度分别在234~249 bp之间,与预期扩增产物大小相同,空白对照无扩增条带。同时,用该通用引物对牛源性成分进行灵敏度检测,电泳结果见图 3,牛 DNA浓度为10−5稀释度时,5 µL PCR产物通过电泳已经看不到电泳条带。
2.2 基因芯片特异性检测结果

图3 灵敏度实验PCR电泳图Fig.3 Agarose electrophoresis analysis of sensitivity test of bovine by Universal-Forward/Reverse. M: DNA marker DL 2 000; 1−6: 10−1, 10−2, 10−3, 10−4, 10−5, blank control.

图4 特异性检测杂交结果Fig.4 Specificity test result of microarray. A−O: bovine, goat, pig, chicken, donkey, deer, dog, rabbit, duck, goose, artridge, quail,turkey, sheep, fish, respectively.
用通用引物进行荧光掺入法标记PCR扩增,按1.7方法进行杂交、扫描,结果如图4所示。为了能有效地对杂交信号进行评判,在根据文献[7]得出本系统的阳性判定标准为通过Gene Pix 4200A扫描识别的信噪比大于3的信号为阳性信号。阳性质控探针及阴性质控探针杂交信号正常,4条特异性探针与目的产物有很好的杂交信号,与其余11种供试材料之间无杂交信号,不存在交叉反应,这4条特异性探针有很好的特异性。
2.3 基因芯片灵敏度检测结果
用通用引物进行荧光掺入法标记PCR扩增,按1.7方法进行杂交、扫描,牛、山羊、猪、鸡源性成分的基因芯片灵敏度检测结果分别见图 5~8,最低检测限都达到了10−5稀释度,即浓度为1 pg。牛源性成分灵敏度检测电泳结果表明 (图3),牛DNA浓度为10−5稀释度时,5 µL PCR产物通过电泳已经看不到电泳条带 (山羊、猪、鸡电泳结果与牛基本一致),但是通过基因芯片杂交后仍然可以看到较为明显的杂交信号 (图5)。
2.4 基因芯片检测方法可重复性验证
从2批点制的检测芯片中各随机抽取4张,针对bovine和goat探针位点的靶基因做2次杂交,每次再从2批次芯片中随机抽取2张杂交,所用靶基因的量 (10 ng/µL) 及杂交条件都是相同的,用以验证该方法的重复性和稳定性。第 1次杂交各芯片标记为 1A、1B、2A、2B;第 2次杂交各芯片标记为1C、1D、2C、2D。从杂交扫描分析后的图像上可以看出,无论是批间还是批次杂交试验都得到了明显的信号结果 (图9),而且各个探针间也没有交叉反应。

图5 基因芯片对牛成分的灵敏度检测结果Fig.5 Sensitivity test result of bovine by microarray. (A) 10−1. (B) 10−2. (C) 10−3. (D) 10−4. (E) 10−5. (F) 10−6.

图6 基因芯片对山羊成分的灵敏度检测结果Fig.6 Sensitivity test result of goat by microarray. (A) 10−1. (B) 10−2. (C) 10−3. (D) 10−4. (E) 10−5. (F) 10−6.

图7 基因芯片对猪成分的灵敏度检测结果Fig.7 Sensitivity test result of pig by microarray. (A) 10−1. (B) 10−2. (C) 10−3. (D) 10−4. (E) 10−5. (F) 10−6.
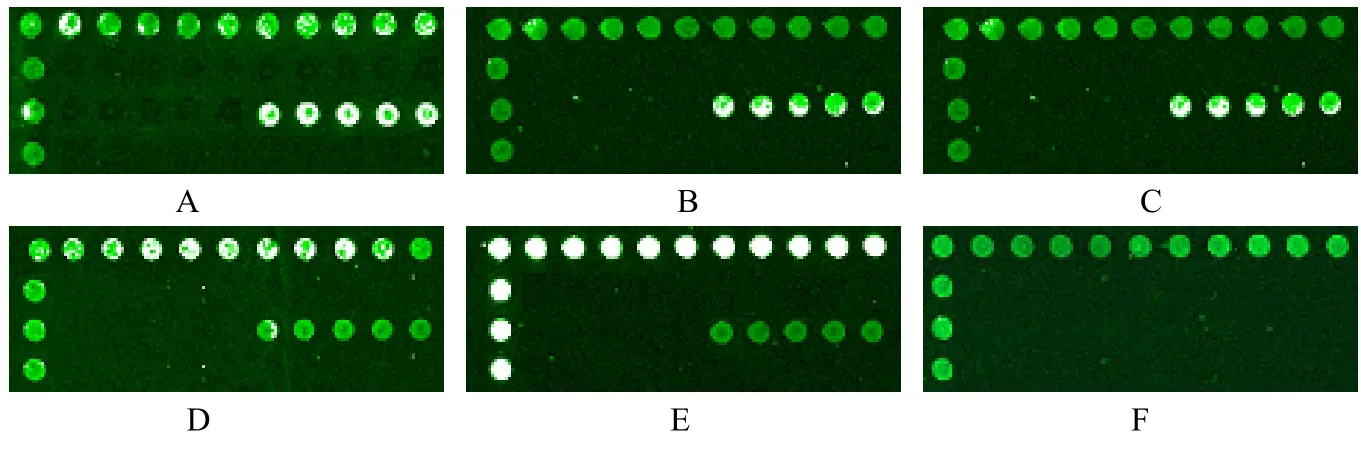
图8 基因芯片对鸡成分的灵敏度检测结果Fig.8 Sensitivity test result of chicken by microarray. (A) 10−1. (B) 10−2. (C) 10−3. (D) 10−4. (E) 10−5. (F) 10−6.

图9 可重复性杂交试验结果Fig.9 Repeatability results of hybrization. 1A, 1B, 1C, 1D: bovine; 2A, 2B, 2C, 2D: goat.
3 讨论
mtDNA是高等动物唯一的核外遗传物质,所有组织细胞中均含有大量的线粒体[8],mtDNA主要以编码序列构成,种内的异质基因很少,而在不同的物种间具有高度的变异性[9]。在高等植物中没有发现类似结构的基因,在植物mtDNA中也没有相似的编码同源蛋白的基因[10-11],因此,高等生物mtDNA 16S rRNA是一个比较理想的、保守性较好的分子标记基因。本研究通过该区段的一对通用引物Universal-Forward/Reverse可以对 15种脊椎动物mtDNA 16S rRNA基因进行有效扩增,结合特异性探针,构建固相基因芯片,可同时进行多种动物源性成分的鉴别检测,不仅可以降低检测步骤及时间,还可降低检测成本。
获得具有很好特异性和高灵敏度的探针是成功建立基因芯片检测技术的关键[12-14],因此如何进行探针的设计与筛选来消除探针杂交的不利影响,提高检测的特异性将是基因芯片应用技术研究的重点之一[15-16]。在探针筛选过程中,本实验发现探针的Tm值及杂交温度[17]和探针在 PCR产物序列上的位置对杂交信号的强弱有着较为明显的影响。杂交温度降低将增加探针的信号值,但探针的特异性将降低;杂交温度提高将降低探针的信号值,但探针的特异性将提高,而最佳的杂交温度一般低于探针的理论Tm值8℃~10℃,通过对杂交温度进行系统的梯度性优化试验获得最佳的杂交温度。由于探针的位置对特异性也有影响,在靠近PCR产物5′端的位置时,反向探针的杂交信号要好于正向的探针。当所设计的探针的位置在靠近PCR产物中间的位置时,正向探针的杂交信号与反向探针杂交信号相近[18],所以在设计探针时要考虑探针的选择位点及方向性。
本研究在进行基因芯片灵敏度检测方法过程中,发现 10−5梯度稀释的牛 DNA通过琼脂糖凝胶电泳无目的产物,但是通过基因芯片杂交后有杂交信号,说明基因芯片检测方法的灵敏度要高于常规PCR,这一研究结果与翟俊辉等[19]的研究结果相符。
本研究选取通用引物只通过一次PCR扩增就可以得到 15种动物的目的片段,结合PCR-基因芯片技术可同时鉴别检测多种动物源性成分,并且具有很好的特异性和灵敏度。通过对牛、山羊、猪和鸡动物源性成分检测方法的初步研究,获得了该体系PCR扩增及芯片杂交等步骤的参数,为进一步对其余的动物源性成分的检测奠定了基础。本方法大大地缩短了检测周期,为口岸检验检疫提供高效、快速、准确的检测手段,为我国进口食品、畜产品快速筛查和种类鉴定提供有效的技术平台,具有非常广阔的推广应用前景。
REFERENCES
[1] Bai DT, Qi ZB. The application of genechip.Prog Microbiol Immunol, 2002, 30(4): 100–103.
白东亭, 祁自柏. 基因芯片应用前景. 微生物学免疫学进展, 2002, 30(4): 100–103.
[2] Schena M. Zhang L,et al.Translated Microarray Analysis. Beijing: Science Press, 2004: 19–20.
谢纳 M, 张亮, 等译. 生物芯片分析. 北京: 科学出版社, 2004: 19–20.
[3] Liu YL, Zheng Y, Zhao H. Preliminary study of human papilloma virus detection and genotyping DNA microarray.Chin J Pract Gynecol Obst, 2005, 21(7):411–412.
刘玉玲, 郑英, 赵虎. 人乳头瘤病毒检测及分型基因芯片制备的初步研究. 中国实用妇科与产科杂志, 2005,21(7): 411– 412.
[4] Huang AH, Li JW, Shen ZQ,et al. Detection and identification of fungal pathogens by oligonuleotide microarray hybridization.Chin J Microbiol Immunol,2007, 27(2): 180–185.
黄爱华, 李君文, 谌志强, 等. 基因芯片技术检测鉴定临床常见致病真菌的初步研究. 中华微生物学和免疫学杂志, 2007, 27(2): 180–185.
[5] Gao S, Xie MJ, Jin DZ,et al. Detection and identification for foodborne pathogenic bacteria in fishery products using DNA chip.Lett Biotechnol, 2007, 18(1): 72–76.
高爽, 谢明杰, 金大智, 等. 运用基因芯片技术建立检测水产食品中常见病原微生物方法的研究. 生物技术通讯, 2007, 18(1): 72–76.
[6] Bottero MT, Civera T, Nucera T,et al. Design of universal primers for the detection of animal tissues in feedstuff.ProQuest Agric J, 2003, 27: 667–669.
[7] Prins TW, Van Dijk JP, Beenen HG,et al. Optimised padlock probe ligation and microarray detection of multiple (non-authorised) GMOs in a single reaction.BMC Genomics, 2008, 9: 584.
[8] Momcilovic D, Rasooly A. Detection and analysis of animal materials in food and feed.J Food Protect, 2000,63: 1602–1609.
[9] Tartaglia M, Saulle E, Pestalozza S,et al. Detection of bovine mitochondrial DNA in ruminant feeds: amolecular approach to test for the presence of bovine-derived materials.J Food Protect, 1998, 61: 513–518.
[10] Wang RF, Myers MJ, Campbell W,et al.A rapid method for PCR detection of bovine materials in animal feedstuffs.Mol Cell Probes, 2000, 14: 1–5.
[11] Montiel-Sosa J F, Ruiz-Pesini E, Montoya J,et al. Direct and highly species-specific detection of pork meat and fat in meat products by PCR amplification of mitochondrial DNA.J Agr Food Chem, 2000, 48: 2829–2832.
[12] Dorris DR, Nguyen A,et al. Oligo deoxyribose nucleotide probe accessibility on a three-dimensional DNA microarray surface and the effect of hybridization timeon the accuracy of expression ratios.BMC Biotechnol, 2003,11: 3–6.
[13] Chou CC, Chen CH, Lee TT,et al. Optimization of probe length and the number of probes per gene for optimal microarray analysis of gene expression.Nucleic Acids Res,2004, 32(12): e99.
[14] Hans B, Stephan P. Specific and nonspecific hybridization of oligonucleotide probes on microarrays.Biophys J,2005, 89: 337–352.
[15] Matveeva OV, Shabalina SA, Nemtsov VA,et al.Thermodynamic calculations and statistical correlations for oligo-probes design.Nucleic Acids Res,2003, 31:4211–4217.
[16] Naef F, Magnasco MO. Solving the riddle of the bright mismatches. Hybridization in oligonucleotide arrays.Phys Rev E, 2003, 68: 11906–11910.
[17] Lu CY, Shi CL, Zhang CX,et al.Development of single base extension-tags microarray for the detection of food-borne pathogens.Chin J Biotech, 2009, 25(4):554–559.
陆长勇, 施春雷, 张春秀, 等. 基于单碱基延伸标签反应的常见食源性致病菌基因芯片检测方法的建立. 生物工程学报, 2009, 25(4): 554–559.
[18] Zhang GM, Zhang Z. The Establishment of Approach of Detection Platform for Plant Pathogenic Fungi. Beijing:China Agriculture Press, 2005: 111–112.
章桂明, 章正. 植物病原真菌检测平台方法的建立. 北京: 中国农业出版社, 2005: 111–112.
[19] Zhai JH, Song YJ, Du ZM,et al. Study on universal microarray systemfor detection of common clinical infectious bacteria.Chin J Public Health, 2003, 19(4):430–431.
翟俊辉, 宋亚军, 杜宗敏, 等. 通用基因芯片检测感染性细菌方法的研究. 中国公共卫生, 2003, 19(4):430–431.
Detection of bovine, goat, pig and chicken derived ingredients in animal products with universal PCR-microarray method
Fengyun Shi1,2, Jiankun Miao3, Liping Zhang1, Hong Tao2, Jianqiang Lü2, Zhouxi Ruan2,and Hui Zong2
1Faculty of Animal Science and Technology,Gansu Agricultural University,Lanzhou730070,China
2Shenzhen Entry-exit Inspection and Quarantine Bureau,Shenzhen518010,China
3State Key Laboratory of Agro-biotechnology,China Agricultural University,Beijing100193,China
Received:January 14, 2010;Accepted:April 9, 2010
Supported by:Project of Shenzhen Entry-exit Inspection and Quarantine Bureau (No. SZ2008015).
Corresponding author:Hui Zong. Tel: +86-755-25588685; E-mail: zonghui32@yahoo.com.cn
深圳出入境检验检疫局科研项目 (No. SZ2008015) 资助。
