丙型肝炎病毒核心基因置换对病毒复制和感染影响的初步研究
2010-01-26黎刚曹明媚王文博任浩戚中田
黎刚,曹明媚,王文博,任浩,戚中田
第二军医大学微生物学教研室,上海市医学生物防护重点实验室,上海200433
丙型肝炎病毒(hepatitis C virus,HCV)是导致输血后肝炎的主要病原体之一,与肝硬化和肝细胞癌高度相关[1]。HCV核心(core)蛋白是HCV的重要结构蛋白,是一个具有膜蛋白特征的二聚化α螺旋蛋白[2],由191个氨基酸组成,序列十分保守,各分离株的同源性超过95%。核心蛋白是从基因水平治疗HCV感染和进行疫苗研究的重要区域,它在病毒增殖、慢性致病、肝细胞癌变、免疫功能障碍中起重要作用[3],也是病毒衣壳包装和产生感染性病毒颗粒的必要因素之一[4,5]。
核心蛋白在体外可非特异性结合核酸[6,7]。Duvignaud等[8]认为,核心蛋白可能通过N端区域包装基因组RNA。N端82个氨基酸缺乏二级结构,在体外和酵母中能有效诱导并形成核衣壳样颗粒(nucleocapsid-like particle,NLP),而Majeau等[9]发现,仅需N端79个氨基酸就足够在体外形成核衣壳蛋白。核心蛋白能与包膜糖蛋白E1和E2作用,与包膜区蛋白共同形成HCV的核衣壳[10,11]。
目前,核心蛋白在HCV复制中的作用还不明确。Goh等[12]用酵母双杂交系统研究发现,核心蛋白能与NS5A的N端部分共沉淀,两者相互作用所需的NS5A最小序列包括干扰素敏感决定区(interferon sensitivity-determining region,ISDR)、蛋白激酶R(protein kinase R,PKR)结合区和富含Pro的区域。核心蛋白和NS5A通过与其他蛋白共同作用形成一个复合体结构,但对病毒复制和感染性病毒颗粒组装的具体作用还不清楚。Fan等[13]发现,核心蛋白可特异性结合HCV基因组的5′非编码区,抑制病毒的翻译过程。核心蛋白是与病毒RNA结合的核衣壳蛋白,而NS5A在HCV的复制中起重要作用,可以推测核心蛋白通过与其他蛋白直接或间接作用参与病毒的复制和组装。
为探讨HCV型间差异对病毒复制和感染的影响,本研究采用我国高流行性的HCV1b型HC-J4的核心区平行置换2a型FL-J6JFH1的相应基因,构建FL-J6JFH/J4 core复制子,转染Huh7.5.1,分析核心基因置换后在病毒复制、蛋白翻译和感染性病毒颗粒产生能力方面的变化。
1 材料和方法
1.1 材料
1.1.1质粒、菌株、细胞株和培养基含有HCV2a J6JFH1株全长基因组的质粒pFL-J6JFH1由美国洛克菲勒大学HCV研究中心Charles M. Rice教授惠赠。含有HCV1b型J4全长cDNA的质粒pGEM-J4和大肠埃希菌(Escherichiacoli,E.coli)DH5α由本实验室保存。Huh7.5.1细胞由中国科学研究院巴斯德研究所钟劲研究员提供。
1.1.2试剂QuikChange®LightningSite-Directed Mutagenesis Kit为Stratagene公司(美国)产品。异丙基硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG)、5-溴-4-氯-3-吲哚-β-D-半乳糖苷(5-bromo-4-chloro-3-indolyl β-D-galactopyranoside,X-gal)、NZ胺(酪蛋白的酶促水解物)、酵母提取物等购自北京鼎国生物技术有限责任公司。14 ml BD Falcon聚丙烯管购自上海雅怡科学实验设备有限公司。异硫氰酸荧光素(fluorescein isothiocyanate,FITC)标记的羊抗人IgG购自Jackson免疫研究所(美国)。M-MLV反转录酶及T7体外转录试剂盒(RiboMAXTMLarge Scale RNA Production System-T7)购自Promega公司(美国)。LipofectamineTM2000购自Invitrogen公司(美国)。RNA抽提试剂购自上海华舜生物工程公司。
1.2 方法
1.2.1HCVJ4核心区的聚合酶链反应采用重 叠延伸聚合酶链反应(polymerase chain reaction,PCR)的方法,以pGEM-J4为模板,扩增出J4 核心基因片段(该片段两端含有J6核心基因两端配对序列),所用上游引物为J4-C-F: 5′-AAGCTTATGCATCACCATCACCATCACAGC-ACGAATCCTAAACC -3′,下游引物为J4-C-R:5′-GGATCCGAAGCGGAAGCTGGGATGGTC-3′(下划线:引入的互补配对序列)。
1.2.2HCVJ4核心置换的pFL-J6JFH/J4core复制子的构建回收J4核心片段后作为大引物(megaprimer),以pFL-J6JFH为模板进行热循环反应,扩增出pFL-J6JFH/J4 core质粒,DpnⅠ消化原始的pFL-J6JFH模板后,转化、鉴定,得到pFL-J6JFH/J4 core复制子,具体方法参照Stratagene公司的QuikChange®Lightning Site-Directed Mutagenesis Kit操作说明。
1.2.3HCVRNA转录体的制备和Huh7.5.1细胞转染将FL-J6JFH1、FL-J6JFH/J4 core经XbaⅠ线性化后,用T7体外转录试剂盒制备RNA体外转录体,RNase-free DNaseⅠ处理完全去除DNA模板后,用Tris饱和的苯酚∶氯仿∶异戊醇(25∶24∶1)抽提RNA,2%琼脂糖凝胶电泳检测其完整性和纯度。体外转录成RNA后用脂质体转染Huh7.5.1细胞,转染方法和步骤参照Invitrogen公司的LipofectamineTM2000试剂盒要求。
1.2.4转染细胞内HCVRNA水平检测设计HCV 5′非编码区的特异性引物(上游引物为F-HCVN:5′-GCGTTAGTATGAGTGTCGTG-3′;下游引物为R-HCVN:5′-TCGCAAGCACCCTATCAG-3′),预期产物213 bp。设计内参照人GAPDH扩增引物(上游引物:5′-TGGGCTACACTGAGCACCAG-3′;下游引物:5′-AAGTGGTCGTTGAGGGCAAT-3′,预期产物100 bp)。
转染后第5天,抽提转染细胞内RNA,转录成cDNA后以双标准曲线法[14]进行定量。HCV检测的标准品是含有全长HCV cDNA的质粒FL-J6JFH1 PCR后的纯化产物(稀释梯度为10-3~10-9),GAPDH 的标准品是纯化后的PCR产物(稀释梯度为10-1~10-6)。将两者梯度稀释,分别绘制标准曲线。各实验组样品的Ct值与标准曲线对比,计算检测样品的浓度。经内参基因GAPDH均一化后,即可算出转染细胞内HCV RNA的水平。
1.2.5转染细胞内HCV蛋白表达细胞的检测RNA转染后第8天,以丙型肝炎患者血清(预实验结果表明,以HCV单克隆抗体检测的荧光结果弱于患者血清,故本文选取患者血清作为一抗进行免疫荧光检测)为一抗,羊抗人-FITC为二抗,免疫荧光法(immunofluorescence assay, IFA)检测转染细胞中HCV蛋白的表达。Olympus倒置显微镜(CK40)观察并拍照记录。
1.2.6转染细胞上清液HCV感染性检测收集转染后第8天的细胞上清液,1 467g离心10 min,去除可能存在的细胞碎片,分装后保存在-80 ℃。naïve Huh-7.5.1 按每孔3×104个细胞接种到96孔细胞培养板,置含5% CO2的37 ℃培养箱孵育至40%~50%融合。加入收集的细胞上清液,继续培养72 h。以丙型肝炎患者血清为一抗,IFA检测转染细胞中HCV蛋白阳性细胞。Olympus倒置显微镜(CK40)观察并拍照记录。
2 结果
2.1 HCV J4核心置换的pFL-J6JFH/J4 core复制子的构建与体外转录
PCR扩增出HC-J4的核心基因片段(图1),克隆到pMD18-T载体上后进行测序验证,用基因置换方法构建了pFL-J6JFH/J4 core复制子,经测序验证,J6核心基因被J4核心基因成功置换。将FL-J6JFH1和FL-J6JFH/J4 core经XbaⅠ线性化体外转录成RNA,2%琼脂糖凝胶电泳结果显示,大小为6 000~10 000 bp(图2)。
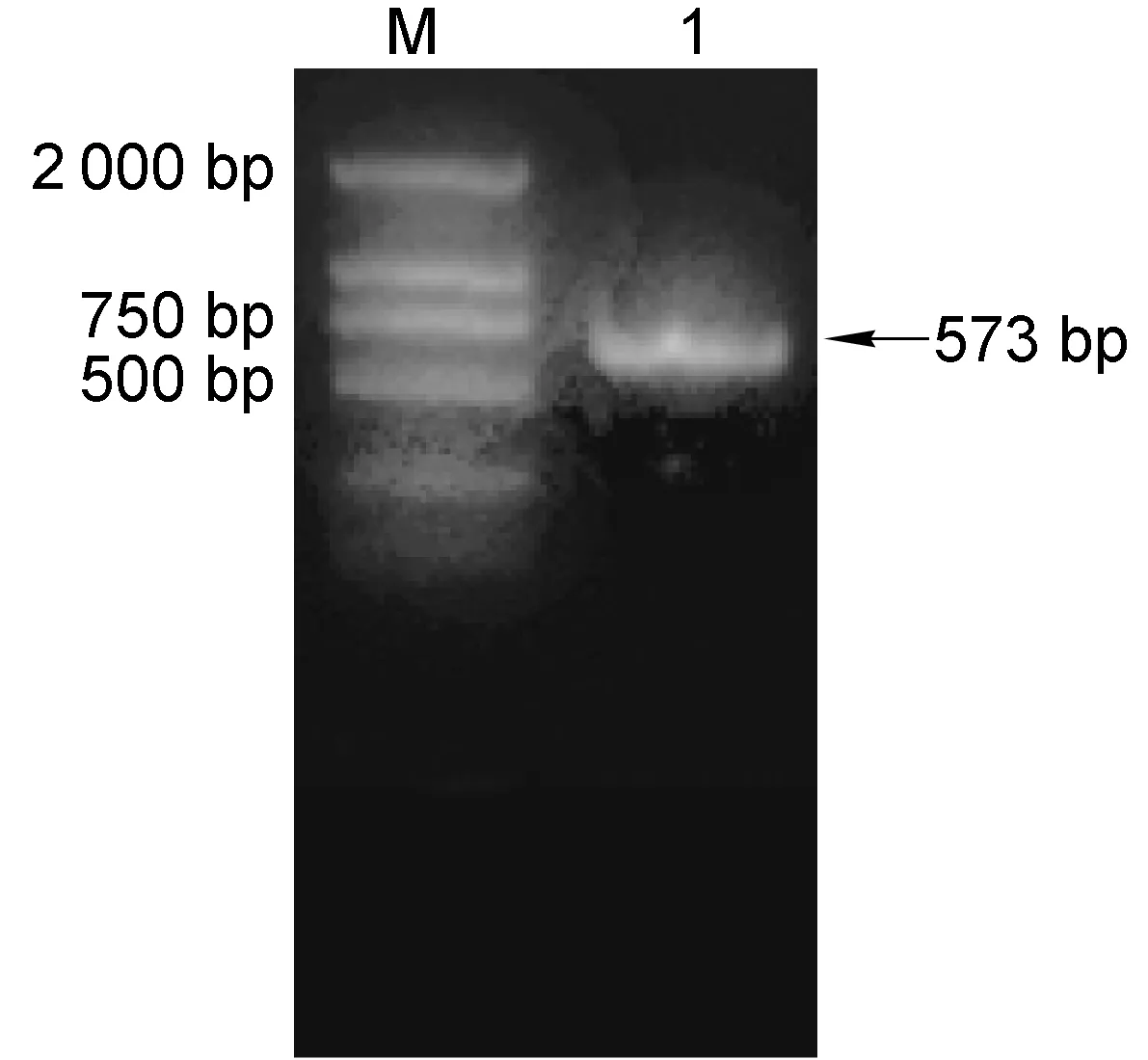
M, DNA marker 2000; 1, J4 core fragment.
图1HCVJ4核心的PCR扩增结果
Fig.1AmplificationofJ4corebyPCR

1, FL-J6JFH1; 2, FL-J6JFH/J4 core; M, RNA marker 10000.
图2FL-J6JFH1和FL-J6JFH/J4core转录体电泳
Fig.2GelelectrophoresisofsyntheticRNAtranscriptsofFL-J6JFH1andFL-J6JFH/J4core
2.2 转染细胞内HCV RNA水平的变化
将FL-J6JFH1和FL-J6JFH/J4 core RNA转录体用脂质体介导转染Huh7.5.1细胞。转染细胞传代培养,第5天用荧光定量反转录-聚合酶链反应(fluorescent quantitative reverse transcriptase-polymerase chain reaction,FQ-RT-PCR)检测转染细胞内HCV RNA水平。FL-J6JFH/J4 core的RNA水平约为5.6×105GE/μg RNA,与野生型相当,无显著性差异(n=4,P>0.05)。结果如图3所示。

mean±SD.n=4,P>0.05.
图3转染第5天细胞内HCVRNA水平
Fig.3IntracellularHCVRNAlevelsatday5post-transfection
2.3 转染细胞内HCV蛋白阳性表达细胞的检测
FL-J6JFH1和FL-J6JFH/J4 core RNA转染细胞后第8天,以丙型肝炎患者血清为一抗,羊抗人-FITC为二抗,IFA检测转染细胞中HCV蛋白阳性细胞。Olympus倒置显微镜(CK40)观察并拍照。为更直观比较阳性细胞数目的差异,选择放大倍数为×100。结果表明,野生型FL-J6JFH1的阳性细胞数占细胞总数的50%,而FL-J6JFH/J4 core阳性细胞约占30%,低于核心置换前的野生型FL-J6JFH1株的水平(图4A)。
2.4 转染细胞上清液HCV感染性检测
FL-J6JFH1和FL-J6JFH/J4 core RNA转染细胞后第8天,收集细胞上清液,孵育 naïve Huh-7.5.1细胞,培养72 h后用IFA检测HCV的表达。观察同上。与对照FL-J6JFH1相比,FL-J6JFH/J4 core的阳性细胞数减少(图4B)。
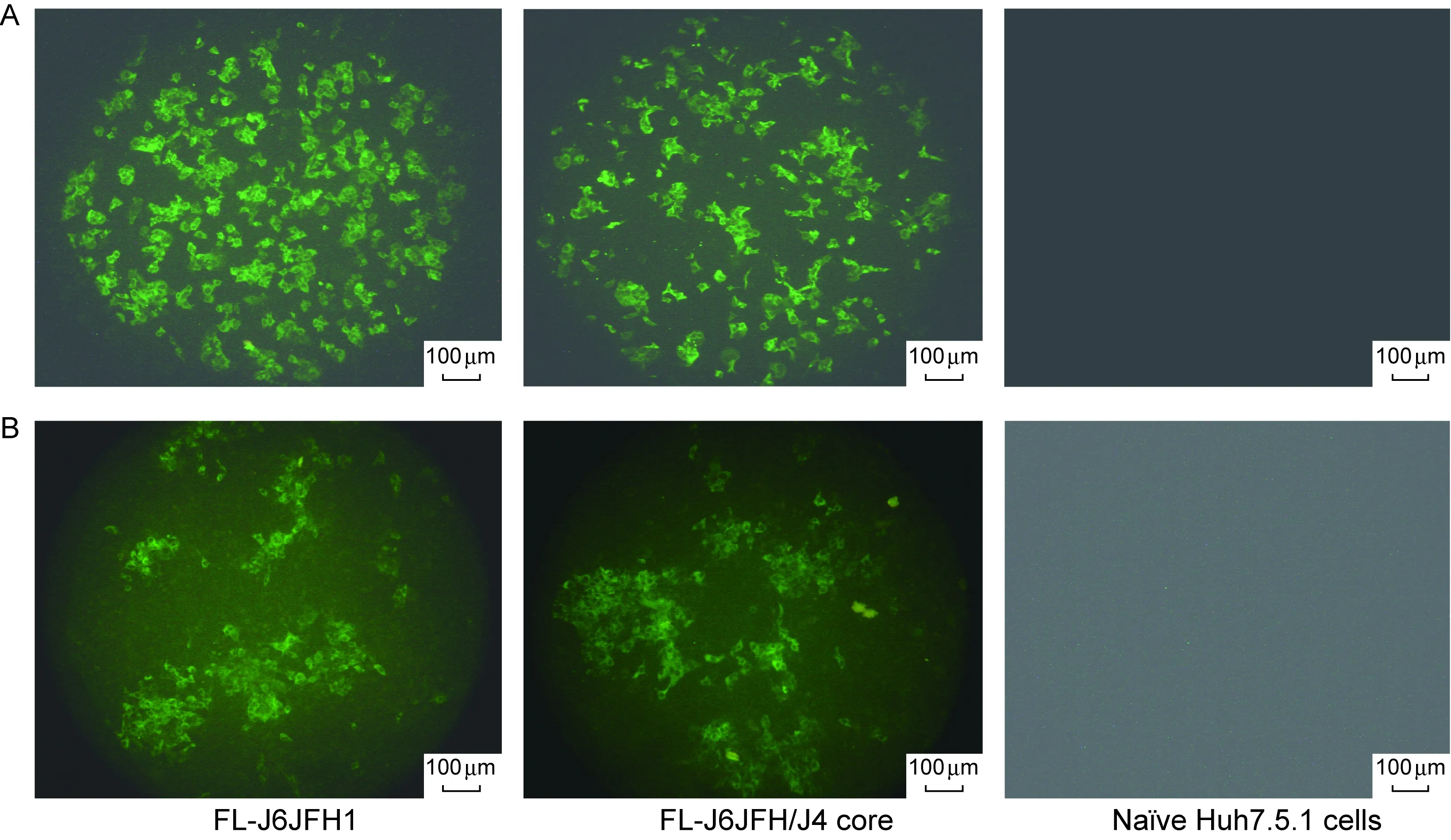
图4转染第8天和用收集的第8天上清液孵育naïveHuh7.5.1细胞后的HCV阳性细胞的免疫荧光检测
Fig.4ImmunofluorescenceassayofHCVproteinsinHuh7.5.1cellstransfectedwithRNA(A)orinfectedwithsupernatantsfromtransfectedcells(B)atday8
3 讨论
本研究用基因置换法构建了HCV FL-J6JFH/J4 core嵌合复制子,体外转录成RNA后转染Huh7.5.1肝癌细胞。Wakita等曾做过RNA 印迹(Northern blot)实验,结果在转染后12 h检测到降解的RNA转录体,24 h即可检测到新产生的HCV RNA[15]。本研究检测到转染后第5天FL-J6JFH/J4 core细胞内RNA水平与野生型FL-J6JFH1相当,这意味着在复制起始或延伸过程中,HCV J6核心和J4核心与其他复制相关蛋白结合的活力可能相当,高度保守的核心区在HCV各株间自由置换对HCV的复制能力不起或起较小作用。
HCV FL-J6JFH1和FL-J6JFH/J4 core RNA转染细胞后第8天,IFA检测转染细胞中HCV的表达。结果显示,FL-J6JFH/J4 core阳性细胞数占细胞总数的比例与野生型相比明显下降,提示J4核心基因替换后引起HCV蛋白翻译能力减弱,原因可能是在HCV结构或非结构蛋白翻译过程中,J4核心蛋白与翻译相关蛋白作用的活性弱于J6核心蛋白。说明核心蛋白可能对HCV蛋白的翻译有较大作用,各株之间相互置换,虽然氨基酸序列变化不大,但在一定程度上改变了HCV的蛋白翻译能力。
收集HCV FL-J6JFH1和FL-J6JFH/J4 core RNA转染细胞后第8天的细胞上清液,孵育naïve Huh7.5.1细胞。IFA检测发现,与对照FL-J6JFH1相比,FL-J6JFH/J4 core的阳性细胞数大幅减少。表明在HCV感染性病毒颗粒的包装或释放过程中,J4核心基因置换后会降低与其他相关蛋白的作用,使整体感染力降低。在IFA检测实验中,我们尝试使用NS3或NS5A的单克隆抗体,但效果不如用丙型肝炎患者血清敏感。Kato等[16]研究发现,HCV的开放阅读框架(open reading frame,ORF)编码的多聚蛋白前体,在宿主细胞信号肽酶和病毒蛋白酶作用下,裂解产生核心蛋白前体(p23),再经膜内蛋白酶SPP(信号肽酶)作用,形成成熟的p21[17,18]。成熟的p21有明显的二级结构,并能自折叠形成大至24个单体的多聚体,其中C端的125~179位氨基酸起重要作用。核心蛋白多聚体的具体功能还不清楚。Kunkel等[6]认为,核心蛋白11S可能是HCV病毒颗粒组装的中间体形式,组装时核心蛋白积聚,可加速病毒核衣壳的形成,降低形成感染性病毒颗粒所必需的核心蛋白与其他蛋白的相互作用。用J4核心区平行置换野生型FL-J6JFH的核心区后,有可能影响核心蛋白的加工、成熟、自组装和功能性11S蛋白作用的发挥。
上述初步研究表明,HCV 核心蛋白很可能在HCV感染性病毒颗粒的释放中起重要作用。目前,我们正在长时间观察FL-J6JFH/J4 core RNA转染细胞后RNA的消长,以及核心基因置换后对感染性病毒颗粒的影响,以进一步明确核心蛋白在HCV复制和感染性颗粒产生中的作用。
[1] Teo M, Hayes P. Management of hepatitis C [J]. Br Med Bull, 2004, 70: 51-69.
[2] Boulant S, Vanbelle C, Ebel C, Penin F, Lavergne JP. Hepatitis C virus core protein is a dimeric alpha-helical protein exhibiting membrane protein features [J]. J Virol, 2005, 79 (17): 11353-11365.
[3] Irshad M, Dhar I. Hepatitis C virus core protein: an update on its molecular biology, cellular functions and clinical implications [J]. Med Princ Pract, 2006, 15(6): 405-416.
[4] Klein KC, Dellos SR, Lingappa JR. Identification of residues in the hepatitis C virus core protein that is critical for capsid assembly in a cell-free system [J].J Virol, 2005, 79(11): 6814-6826.
[5] Murray CL, Jones CT, Tassello J, Rice CM. Alanine scanning of the hepatitis C virus core protein reveals numerous residues essential for production of infectious virus [J].J Virol, 2007, 81(19): 10220-10231.
[6] Kunkel M, Lorinczi M, Rijnbrand R, Lemon SM, Watowich SJ. Self-assembly of nucleocapsid-like particles from recombinant hepatitis C virus core protein [J]. J Virol, 2001, 75 (5): 2119-2129.
[7] Kunkel M, Watowich SJ. Conformational changes accompanying self-assembly of the hepatitis C virus core protein [J]. Virology, 2002, 294(2): 239-245.
[8] Duvignaud JB, Savard C, Fromentin R, Majeau N, Leclerc D, Gagné SM. Structure and dynamics of the N-terminal half of hepatitis C virus core protein: an intrinsically unstructured protein [J]. Biochem Biophys Res Commun, 2009, 378(1): 27-31.
[9] Majeau N, Gagné V, Boivin A, Bolduc M, Majeau JA, Ouellet D, Leclerc D. The N-terminal half of the core protein of hepatitis C virus is sufficient for nucleocapsid formation [J]. Gen Virol, 2004, 85(Pt 4): 971-981.
[10] Santolini E, Migliaccio G, La Monica N. Biosynthesis and biochemical properties of the hepatitis C virus core protein [J]. J Virol, 1994, 68(6): 3631-3641.
[11] Ray RB, Lagging LM, Meyer K, Steele R, Ray R. Transcriptional regulation of cellular and viral promoters by the hepatitis C virus core protein [J]. Virus Res, 2009, 37(3): 209-220.
[12] Goh PY, Tan YJ, Lim SP, Lim SG, Tan YH, Hong WJ. The hepatitis C virus core protein interacts with NS5A and activates its caspase-mediated proteolytic cleavage [J]. Virology, 2001, 290(2): 224-236.
[13] Fan Z, Yang QR, Twu JS, Sherker AH. Specific in vitro association between the hepatitis C viral genome and core protein [J]. Med Virol, 1999, 59(2): 131-134.
[14] Winer J, Jung CK, Shackel I, Williams PM. Development and validation of real-time quantitative reverse transcriptase-polymerase chain reaction for monitoring gene expression in cardiac myocytes in vitro [J]. Anal Biochem, 1999, 270(1): 41-49.
[15] Wakita T, Pietschmann T, Kato T, Date T, Miyamoto M, Zhao Z, Murthy K, Habermann A, Krausslich HG, Mizokami M, Bartenschlager R, Liang TJ. Production of infectious hepatitis C virus in tissue culture from a cloned viral genome [J]. Nat Med, 2005, 11(7):791-796.
[16] Kato T, Miyamoto M, Furusaka A, Date T, Yasui K, Kato J, Matsushima S, Komatsu T, Wakita T. Processing of hepatitis C virus core protein is regulated by its C-terminal sequence [J]. Med Virol, 2003, 69 (3):357-366.
[17] Okamoto K, Moriishi K, Miyamura T, Matsuura Y. Intramembrane proteolysis and endoplasmic reticulum retention of hepatitis C virus core protein [J]. J Virol, 2004, 78(12):6370-6380.
[18] McLauchlan J. Properties of the hepatitis C virus core protein: a structural protein that modulates cellular processes [J]. J Viral Hepat, 2000, 7(1): 2-14.
