应用DGGE技术分析扇贝养殖海区真核浮游生物种群多样性*
2010-01-05蔡玉勇王崇明
王 娜,蔡玉勇,,李 赟**,王崇明,崔 红
(1.中国海洋大学海水养殖教育部重点实验室,山东青岛266003;2.中国水产科学研究院黄海水产研究所,山东青岛266071;3.山东省潍坊市畜牧检测中心,山东潍坊261061)
应用DGGE技术分析扇贝养殖海区真核浮游生物种群多样性*
王 娜1,蔡玉勇1,2,李 赟1**,王崇明2,崔 红3
(1.中国海洋大学海水养殖教育部重点实验室,山东青岛266003;2.中国水产科学研究院黄海水产研究所,山东青岛266071;3.山东省潍坊市畜牧检测中心,山东潍坊261061)
为分析扇贝养殖海区真核浮游生物种类和丰度,本研究首先以实验室培养的9种饵料微藻总DNA为模板PCR扩增18S rDNA V 1~V 3区的高变区序列,筛选用于DGGE分析该扩增序列的变性剂浓度,结果表明,扩增序列长560 bp,DGGE分析可将不同藻种分离开。随后对青岛沙子口扇贝养殖海区9个月份真核浮游生物多样性进行了DGGE分析,结果从9个月份2 m深的海水中总共扩增出38条谱带,其中共有谱带3条,占总数的7.69%,不同月份的特征性谱带11个,占总谱带数的28.2%。不同月份水样中的谱带数显著不同,香农多样性指数为0.4632。相似性分析表明,2009年3和4月养殖海区真核微生物种类相似性最高,为89.7%。基于不同月份水样微生物种类多样性进行UPGMA聚类分析,结果表明,2008年5,6,8月和2009年1,2,3,4月分别聚为一支,显示取样时间相近的月份微生物种类相似度最高。
PCR-DGGE;18S rDNA序列;真核浮游生物多样性
海洋中的浮游生物是海洋生态系统的重要组成部分,是水系统中占优势的初级生产者,浮游微藻通过光合作用将无机物(如硝酸盐,磷酸盐)转化成新的有机物(脂肪,蛋白质等),为多种海洋动物幼体或成体提供丰富多样的饵料,是海洋生态食物链的重要组成部分。扇贝是滤食性贝类,通过滤食海水中大小合适的浮游生物,养殖海区浮游生物的种类及丰度直接影响到扇贝摄取食物的质量和数量,从而影响到养殖扇贝的生长,因此,分析养殖海区浮游生物的群落结构及季节性变化对于扇贝养殖具有重要意义。
自1979年Fischer和Lerman提出了DGGE(变性梯度凝胶电泳)技术以来,基于16S rDNA多变区序列,已有研究对土壤[1-2]、湖泊[3-4]、动物肠道[5]及蓝藻的群落变化[6]进行了分析,结果证明该技术可以对扩增片段碱基的微小差异进行准确的分离,对微生物的种类和丰度进行定性和定量的研究。在真核浮游生物的研究方面,Beatriz Díez等对真核浮游生物[7]、鲍磊等对厦门西海域的超微型浮游植物[8]、Yan Qingyun等对三峡库区的原核和真核浮游生物[9]、Rebecca J.Gast等对原生动物[10]、马晓军等对冰川的真核微生物的群落结构[11]分别进行了分析,探讨了不同生态环境真核微生物的遗传多样性。与传统的镜鉴技术相比,该技术对采集样品中的高丰度和低丰度生物、微型和超微型生物均可以进行分析,相对传统方法分析更为全面,而且分析效率也大大提高,显示该技术在研究真核浮游生物方面的潜在优势。有关扇贝摄食饵料的研究,仅有王如才等[12]利用镜鉴统计方法进行的研究,本研究首先通过9种培养微藻的分析筛选了PCR-DGGE分析的合适凝胶浓度和变性剂梯度,随后对青岛沙子口扇贝养殖海区9个月份的真核浮游生物种类多样性进行了分析。
1 材料和方法
1.1 材料
用于本研究DGGE分析的样品包括实验室培养微藻和养殖海区采集的海水样品。
实验室培养微藻有9种,分别为1:绿色巴夫藻Pav lova viridis;2:小新月菱形藻N itzschia closterium m inutissim a;3:绿色杜氏藻D unaliella viridis;4:中肋骨条藻Skeletonem a costatum;5:湛江叉边金藻D icrateria inornata;6:小亚德里亚共生藻Gym nodinium m icroadriaticum;7:新月菱形藻N itzschia closterium;8:扁藻Platymonas subcordiform is;9:三角褐指藻Phaeodacty lum tricornutum。所有微藻分别培养在f/2培养基中,光周期为12 h∶12 h。培养至指数生长期离心收集。
海水样品为2009年1月6日、2009年2月23日、2009年3月17日、2009年4月10日、2008年5月11日、2008年6月24日、2008年8月19日、2008年10月10日、2008年12月15日分别采自青岛沙子口扇贝养殖海区2 m深的海水。采集的5 L水样立即在实验室以6 000 r/min离心45min收集,沉淀物分装5个2 m L小离心管再经10 000 r/min离心30 min,弃上清后-80℃保存。
1.2 实验方法
1.2.1 基因组总DNA提取 取1 L海水样收集的浮游生物沉淀,加入65℃预热的提取缓冲液[13](3%CTAB,1%PVP,1.4 mol/L NaCl,0.1 mol/L Tris-HCl(p H=8.0),0.1 mol/L EDTA,0.2%β-巯基乙醇)500μL,65℃水浴1 h后加入蛋白酶K(终浓度为0.2 mg/mL),55℃温浴30 min。随后加入1/3体积的5 mol/L KAc(p H=8.0)[13]混匀,放置20 min,加入等体积酚∶氯仿∶异戊醇(25∶24∶1),轻缓摇匀10 min,10 000 r/min离心10 min。上清液移至新管,并加入2/3体积异丙醇,于4℃冰箱中过夜,经12 000 r/min离心15 min沉淀DNA,弃上清。70%的乙醇洗涤2~3次,无菌风下吹干后溶于50μL TE缓冲液(10 mmolTris-HCl,1 mmol EDTA),-20℃保存备用。
1.2.2 基因组DNA的PCR扩增 以提取的基因组DNA作为反应模板,基于Beatriz Díez等[14]报道,扩增18S rDNA基因V 1~V 3可变区的引物为Euk1A和Euk516r,序列分别是,Euk1A(5′-CTGGTTGA TCCTGCCA G-3′),Euk516r(5′-ACCA GACTTGCCCTCC-3′)。为DGGE分析,在引物Euk516r的5′附加40 bp的GC发卡结构,序列为5′-CGCCCGGGGCGCGCCCCGGGCGGGGCGGGGGCACGGGGGG-3′。预期扩增片段长度为560 bp。
50μL的PCR反应体系包括:5μL 10×buffer(含M g2+),4μL 2.5 mmol/L的dN TP混合物以及1.5μL 10μmol/L的正反向引物,0.5μL 5 U的Taq DNA聚合酶,3μL DNA模板(约30 ng),双蒸水补足50μL。PCR反应条件为,94℃130 s预变性,然后35个循环的94℃30 s变性,56℃45 s退火,72℃130 s延伸,最后72℃延伸7 min。扩增产物经1.0%(质量浓度)琼脂糖凝胶电泳进行初步分析。
1.2.3 DGGE电泳
(1)变性胶的制备 聚丙烯酰胺凝胶浓度为10%,变性剂梯度范围是25%~60%(100%变性剂为7 mol/L尿素和40%去离子甲酰胺混合物),变性剂浓度从上向下依次递增。
(2)加样 胶经1 h凝固后,移去梳子,将胶板移入已盛有60℃预热的1×TAE(40 mmol/LTris,20 mmol/L冰醋酸,1 mmol/L EDTA,p H=7.4)电泳缓冲液的电泳槽,电泳缓冲液清洗加样孔。加入实验室培养的微藻PCR反应混合物15μL(约3 000 ng),或加入海水样PCR反应混合物40μL(约12 000 ng)。(3)电泳及染色 采用Bio-Rad公司DcodeTM基因突变检测系统进行。在60℃恒定温度,100 V恒压下电泳15 h。电泳结束后剥胶,凝胶在0.4μg/mL的EB染色缓冲液中染色20~30 min[15],蒸馏水中脱色3 min。
1.2.4 观察及分析 用JS-380A自动凝胶图像分析仪观察电泳结果并拍照。用Quantity One软件分析条带数目及相对光密度,进行各月份条带数目的相似性分析。计算各月份的真核浮游生物多样性的香农多样性指数(Shannon index)。
2 结果与分析
2.1 提取基因组DNA纯化及完整性检测
收集的海水样品中含有成分复杂的杂质,这些杂质都会不同程度影响DNA的提取效率和纯度,从而影响后续的PCR-DGGE分析。本研究在DNA提取过程中加入聚维酮(PVP),蛋白酶K及醋酸钾,通过分析提取DNA在260,280和230 nm的吸光值,结果表明(见表1),提取DNA的260/280值在1.6~1.8之间,260/230值在1.8~2.5之间。利用引物从培养微藻及海水样品(见图1)提取的总DNA中均扩增到1条片段大小在560 bp左右的扩增产物,无非特异性扩增现象。说明提取DNA的纯度可以满足本研究要求。

表1 9个月份海水样品DNA纯度Table 1 The purity of DNA of nine monthsπmarine samp les
2.2 培养微藻18S rDNA V 1~V 3可变区序列的DGGE电泳
为了优化DGGE电泳条件,本研究先用实验室培养的9种微藻进行DGGE电泳条件筛选(见图2),结果发现,变性剂的浓度范围是影响DGGE电泳的关键因素,当变性剂浓度范围过大时,在变性剂低浓度区,扩增片段移动慢,电泳结束时,大多电泳谱带多滞留凝胶板上层。而变性剂浓度范围过窄,导致扩增片段泳动速率过快(均为低浓度变性剂)或过慢(均为高浓度变性剂),最终也不能将碱基差别小的扩增片段完全分开。通过比较,变性剂浓度范围在30%~48%比较理想。

图1 9个月海水样品18S rDNA的PCR扩增产物Fig.1 PCR amp lification of 18S rDNA of nine monthsπmarine samp les
从优化的DGGE电泳条件获得的电泳图可以看到,9个微藻的扩增片段在凝胶中处于不同位置,并且只有1条主带。其中,新月菱形藻扩增片段移动距离最小,位于凝胶上部,小亚德里亚共生藻扩增片段移动距离最大,位于凝胶底部,小新月菱形藻和新月菱形藻移动距离,处于比较接近位置,其它7种藻均位于凝胶的不同位置,根据DGGE电泳结果可将9种微藻清楚分开。

图2 9个微藻种的DGGE分离图谱Fig.2 DGGE fingerp rints of 9 microalgae species
2.3 沙子口养殖海区9个月的海水样品18S rDNA V 1~V 3可变区扩增片段的DGGE指纹图谱
9个月的海水样品DGGE电泳结果如图3所示。从图中可以看到,2008年5月11日采集的样品电泳谱带最为丰富,因此,本研究以2008年5月11日海水样品为标准,用Quantity One软件对9个海水样品的DGGE图谱进行分析(见图4)。图中显示9个月中共有38条谱带,各个月份的谱带数目明显不同,其中2008年5月11日样品的谱带数为21个,2008年12月15日的样品为13个,月份间真核浮游生物种类的香农多样性指数为0.4632。
在9个月的DGGE电泳谱带中,13,22和29号条带在分析的9个月均存在,为共有谱带,占总谱带的7.69%,9,20,21和30号在8个月中出现且密度较高,说明这些谱带显示的真核浮游生物具有广泛适应性。另外,也存在仅在1个或几个月份出现的特异性谱带,如14,31,36和37号分别是2009年1月6日,2008年8月30日,2008年10月10日,2008年2月15日采集水样的特有谱带,类似这些具有月份特征性的谱带数目为11条,占28.2%。
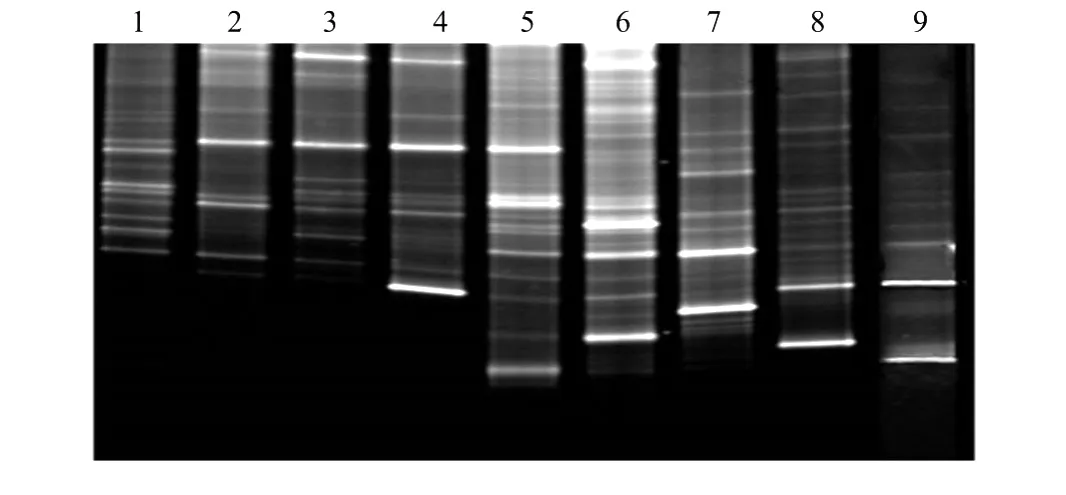
图3 9个月份海水样品DGGE分离图谱Fig.3 DGGE fingerprints of nine months’marine samples

图4 DGGE泳道/条带识别图Fig.4 Compare lane images
根据不同月份DGGE电泳谱带的对比结果,本研究用戴斯系数Cs(Dice coefficient)法计算各月份真核浮游生物种类的相似性(见表2),结果表明相似度最大为2009年3月和4月的样品,达89.7%,最小为2008年6月和12月的样品,为38.9%。基于相似性数据,构建UPGMA聚类图(见图5),结果显示2008年5,6,8月份聚为一大支,2008年12,10月和2009年1,2,4,3月份聚成一大支,显示月份相近的样品浮游生物的种类也较为接近。

表2 沙子口扇贝养殖海区各月份真核浮游生物相似性Table 2 Similarity of eukaryotic p lankton assemblages of ninemonths at the culture scallop area of Shazikou
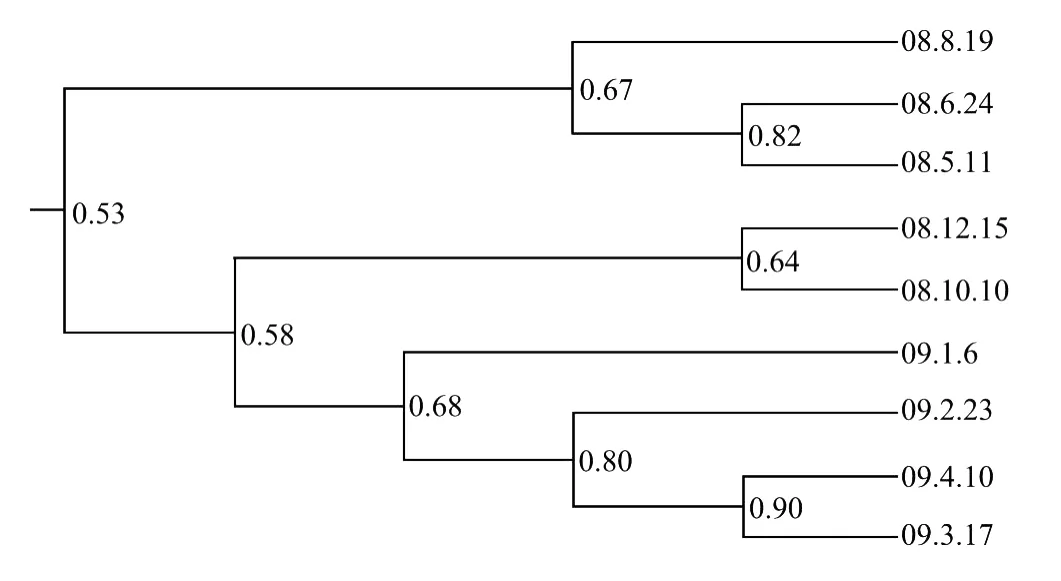
图5 沙子口养殖海区各月份真核浮游生物UPGMA聚类图Fig.5 UPGMA clustering analysis of eukaryotic plankton assemblages of nine months at the culture scallop area of Shazikou
3 讨论
本实验是对养殖海水中真核浮游生物多样性研究方法的探讨。利用PCR-DGGE技术,基于真核生物核糖体18S rDNA可变区序列,Beatriz Díez等[14],Marie Lefranc等[16]对海水和湖水中的真核浮游生物种类多样性进行了分析,认为该技术可用于水生环境中浮游生物种类多样性和丰度的定量分析。在国内,鲍磊等[8]对海水环境中的真核浮游生物,陈美军等[17]对太湖不同湖区的真核浮游生物,马晓军等[11]对不同类型冰川雪坑的真核微生物分别进行了种类多样性分析,本研究对实验室纯化培养的9种饵料微藻的DGGE分析显示,该方法可将不同微藻种清楚分开,这些研究一致表明PCR-DGGE技术用于研究真核微生物群落结构在时间和空间上的动态变化是可行的。该方法的优点是可对采自不同环境的大量样品进行快速的分析,DGGE电泳谱带的数量和亮度可相应反映环境中微生物种的数量和优势种群[18],它不仅可对易培养微生物进行分析,也可对不易培养微生物进行研究,与传统显微镜观察分析方法相比具有显著的优越性。
关于扇贝摄食饵料种类分析,已有的研究并不多,王如才等[12]的研究表明,栉孔扇贝的食料以硅藻为主,不同季节不同地区扇贝摄食饵料存在很大变化,与养殖海区浮游生物种群结构显著相关。因而研究养殖海区真核浮游生物的变化对于反映养殖期间贝类的食料变化具有一定意义。通过离心收集水样中大小在几微米到上百微米的浮游生物,本研究DGGE分析的结果表明,不同月份的真核浮游生物种的数量存在很大的不同,其中5月最多,而12月数量最少。比较不同月份微生物种类的相似性,结果发现,相近月份水样中的真核微生物种类的相似性也最高,其中2008年5,6和8月的种类最为相似,2009年1~4月的种类相似性最高。2008年5月和2009年4月水样中真核微生物的种类差异较大,可能与采样时间间隔较长,微生物群落变化较大有关。因此如要对整个养殖季节海区真核生物的种类多样性进行分析,有必要连续采样。本研究的结果表明,通过PCR扩增真核细胞18S rDNA可变区序列利用DGGE技术分析扇贝养殖海区浮游真核饵料生物的种类和丰度的季节性变化是可行的。本研究初步的结果表明,5,6,8月真核微生物种类数量最为丰富,说明这一时期是扇贝摄取饵料的种类最多,扇贝营养最为全面,因而是最有利扇贝生长的时期。
[1] 罗海峰,齐鸿雁,薛凯,等.在PCR-DGGE研究土壤微生物多样性中应用GC发卡结构的效应[J].生态学报,2003,23(8):1570-1575.
[2] 王光华,刘俊杰,齐晓宁,等.Biolog和PCR-DGGE技术解析施肥对德惠黑土细菌群落结构和功能的影响[J].生态学报,2008,28(1):220-226.
[3] 赵兴青,杨柳燕,陈灿,等.PCR-DGGE技术用于湖泊沉积物中微生物群落结构多样性研究[J].生态学报,2006,26(11):3610-3616.
[4] 吴利,余育和,张堂林,等.牛山湖浮游生物群落DNA指纹结构与理化因子的关系[J].湖泊科学,2008,20(2):235-241.
[5] 黄俊文,冯定远,林映才.PCR-DGGE技术及其在动物肠道微生态学研究中的应用[J].中国畜牧杂质,2006,42(17):47-50.
[6] Erik J van Hannen,Gabriel Zwart,Miranda P van Agterveld,et al.Changes in bacterial and eukaryotic community structure after mass lysis of filamentous cyanobacteria associated with viruses[J].Applied and Environmental Microbiology,1999,65(2):795-801.
[7] Diez B,Pedros-Alio C,Marsh T L,et al.Application of denaturing gradient gel electrophoresis(DGGE)to study the diversity of marine picoeukaryotic assemblages and comparison of DGGE with other molecular techniques[J].Applied and Environmental Microbiology,2001,67(7):2942-2951.
[8] 鲍磊,陈纪新,黄邦钦.应用变性梯度凝胶电泳研究厦门西海域超微型真核浮游生物多样性[J].海洋环境科学,2007,26(6):504-509.
[9] Yan Qingyun,Yu Yuhe,Feng Weisong,et al.Plankton community composition in the three gorges reservoir region revealed by PCR-DGGE and its relationships with environmental factors[J].Journal of Environmental Sciences,2007,20(2008):732-738.
[10] Gast R J,Dennett M R,Caron D A.Characterization of protistan assemblages in the ross Sea,Antarctica,by denaturing gradient gel electrophoresis[J].Applied and Environmental Microbiology,2004,70(4):2028-2037.
[11] 马晓军,刘炜,侯书贵,等.不同类型冰川雪坑中真核微生物多样性变化与环境因子关系研究[J].自然科学进展,2008,18(3):254-261.
[12] 王如才,兰锡禄,刘丽辉,等.栉孔扇贝的食料分析[J].青岛海洋大学学报,1989,S2:43-55.
[13] 杨君,王茜,刘美华,等.一种简便的海藻DNA提取方法[J].生物技术,9(4):39-42.
[14] Diez B,Pedros-Alio C,Marsh T L,et al.Application of denaturing gradient gel electrophoresis(DGGE)to study the diversity of marine picoeukaryotic assemblages and comparison of DGGE with other molecular techniques[J].Applied and Environmental Microbiology,2001,67(7):2942-2951.
[15] Yu Z,Morrison M.Comparisons of different hypervariable regions of rrs genes for use in fingerp ring of microbial communities by PCR-denaturing gradient gel electrophoresis[J].Applied and Environmental Microbiology,2004,70(8):4800-4806.
[16] Marie Lefranc,Aurelie Thenot,Cecile Lepere,et al.Genetic diversity of small eukaryotes in lakes differing by their trophic status[J].Applied and Environmental Microbiology,2005,71(10):5935-5942.
[17] 陈美军,孔繁翔,陈非洲,等.太湖不同湖区真核微型浮游生物基因多样性的研究[J].环境科学,2008,29(3):769-775.
[18] Fromin N,Hamelin J,Tarnawaski S,et al.Statistical analysis of denaturing gel electrophoresis(DGGE)fingerp rinting patterns[J].Environmental Microbiology,2002,4(11):634-643.
Diversity of Eukaryotic Plankton Assemblages at Culture Area fo r Scallop by Denaturing Gradient Gel Electrophoresis(DGGE)
WANG Na1,CA I Yu-Yong1,2,LI Yun1,WANG Chong-Ming2,CU IHong3
(1.The Key Lab of Mariculture,Ministry of Education,Ocean University of China,Qingdao 266003,China;2.Yellow Sea Fisheries Research Institute,Academy of Fisheries Sciences,Qingdao 266071,China;3.Center of Farming Detection of Weifang,Weifang 261061,China)
The 18S rRNA genes(V 1 to V 3 region)was amp lified by two universal primers(Euk516r-GC and Euk1A)from 9 cultured microalgae species.Then,the denaturing gradient gel electrophoresis(DGGE)with denaturing gradient of 25%to 65%urea were optimized.The result indicated that the primer sets gave a single band with the length of amplified fragment approximately 560 bp,and the DGGE with denaturing gradient of 35%to 48%urea could separated nine cultured microalgae species in different position.The diversity of eukaryotic planktons from scallop cultivation area was analyzed by DGGE,and total 38 electropho ric bands were showed from the 9 months water collected from 2 meters deep,of which 3 bands existed in all nine months,taking 7.69 percent of total bands number,and there were 11 characteristic bands in different month,taking 28.2 percent.The result indicated that the diversity of eukaryotic planktons was various significantly among different months,Shannon index is 0.4632.Similarity analysis showed that the highest similarity was 89.2%between M arch and April 2009.UPGMA clustering analysis based on different eukaryotic plankton assemblages showed that May,June and August of 2008 were grouped into a clad,January,February,March,April of 2009 into the other clad,which implied that similarity of the eukaryotic plankton diversity among close month was high.
PCR-DGGE;18S r DNA;eukarytic plankton diversity
S917.1
A
1672-5174(2010)12-051-06
国家高技术研究发展计划项目(2006AA 100307);现代农业产业技术体系建设专项(nycytx-4)资助
2009-07-13;
2010-05-20
王 娜(1983-),女,硕士,主要从事水产病害学研究.E-mail:www-wangna@163.com
**通讯作者:E-mail:sxsdlw l@ouc.edu.cn
责任编辑 于 卫
