基于DNA条形码的天津渤海湾鱼类资源鉴定及遗传分析
2025-02-28何晓旭白晓慧汪笑宇孟一耕



摘 要:为探索DNA条形码对渤海湾部分鱼类物种鉴定的有效性并进行相关的遗传分析,对天津渤海湾10种海水鱼类共177条线粒体COI基因序列进行扩增测序和比对分析。结果表明,10种海水鱼类COI基因T、C、A和G碱基平均含量分别为30.0%、27.9%、24.1%、18.0%,具有碱基偏倚性,177个基因序列共检测到70个单倍型。遗传多样性分析显示,总体的单倍型多样性为0.432 7~0.947 4,核苷酸多样性为0.000 8~0.006 3,其中焦氏舌鳎(Cynoglossus joyneri)的变异位点、单倍型数和单倍型多样性最高。遗传距离结果显示,种间遗传距离为0.237 5~0.357 5,其中种间平均遗传距离是种内平均遗传距离的77倍。系统进化树具有明显的分化谱系,与形态学的鉴定结果相吻合。研究结果表明,DNA条形码技术可以对天津近岸海域常见的10种鱼类进行有效鉴定,相关遗传分析可为天津渤海湾鱼类资源保护提供数据支持。
关键词:DNA条形码;COI基因;遗传多样性;鱼类
中图分类号:S917.4 """"文献标志码:A
文章编号:1004-6755(2025)01-0025-07
我国海洋生物资源丰富,海洋生物多样性是检测生态系统健康的一个重要指标。鱼类作为其中的重要组成部分,在海洋生态系统中占据了关键地位。在传统的鱼类物种鉴定中,多以形态学为基础进行分类鉴定,不仅难以准确辨别形态相似性很高的物种,同时还必须在样本完整的前提下完成。而DNA条形码技术可以利用生物体DNA中的一个或多个标准化短遗传标记来识别其属于特定物种[1-2]。DNA条形码在2003年被引入作为动物生物物种识别的方法,Hebert等[3]建议将线粒体细胞色素c氧化酶亚基1(COI)基因的650 bp左右片段作为动物物种全球生物学鉴定的通用标记或“DNA条形码”。目前该技术已成为一种高效的分子分类工具,越来越多的研究表明其可快速、准确地鉴定鱼类物种并进行遗传多样性分析[4-5]。
天津海域位于半封闭的渤海湾顶部,所辖海域面积约2 146 km2,海岸线北起天津河北行政区域北界线与海岸线交点(涧河口以西约2.4 km处),南至歧口,全长153.2 km。其自然条件优越,基础饵料丰富,曾是渤海湾多种经济洄游鱼、虾类产卵场,潮间带更是蕴藏着丰富的贝类生物资源,历史上有“天然渔场”之称。
据近几年的渔业资源调查结果显示,天津渤海湾渔业资源无论是种类还是数量上,都明显少于历史调查数据[6]。全面了解天津近岸海域经济海洋水产生物资源和生态环境状况是开展水生生物资源养护,合理开发和利用海洋渔业资源的前提。本研究在形态学鉴定基础上对天津渤海湾10种鱼类177条线粒体COI基因序列进行分析,探讨COI基因序列作为DNA条形码在天津渤海湾鱼类分类中的应用,建立条形码数据库,了解和掌握天津渤海湾鱼类分布规律,为制定合理的渔业资源保护措施提供数据支持。
1 材料与方法
1.1 样品采集
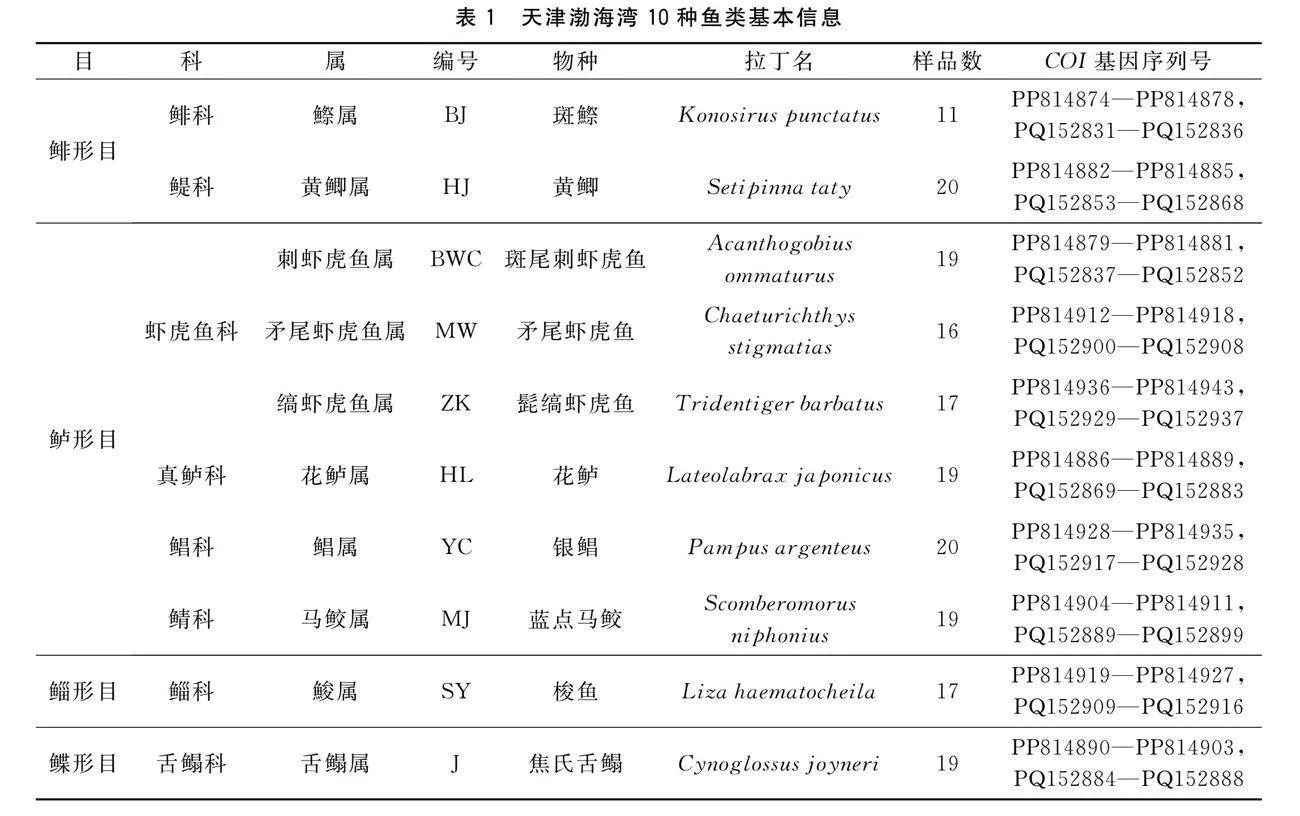
根据海岸线的走向及其包括的海域,共设8个站点,均匀分布于整个渤海湾(见图1)。租用247 kW渔业生产船只,使用单拖网(网口宽12 m,囊网网目20 mm),在设定的站位拖网0.5 h,拖速2~3 kn,对游泳动物进行采样。每个种类样本数量见表1,剪取鳍条组织放入装有无水乙醇的离心管中,对样本进行编号,放入低温冰箱-80 ℃保存备用。
1.2 样品分类
根据《天津水域鱼类资源种类名录及原色图谱》[7]进行形态学的初步鉴定,177个样品个体归属于4目,8科,10属,10种(表1)。
1.3 DNA提取、PCR扩增及序列测定
本实验采用醋酸铵沉淀法提取基因组DNA[8],NanoDrop ND-1000紫外分光光度计检测DNA浓度和质量,-20 ℃保存备用。扩增引物为鱼类DNA条形码扩增的通用引物[9],由上海生工生物工程有限公司合成。引物序列为Fish11F:TCAACCAACCACAAAGACATTGGCAC;Fish11R:TAGACTTCTGGGTGGCCAAAGAATCA。PCR扩增反应总体系为50 μL,含5 μL 10×Buffer,5 μL dNTP(10 mmol/L)、正反向引物各1 μL(10 μmol/L),DNA模板2.5 μL,TaqDNA聚合酶(5 U/μL)0.5 μL ,ddH2O 35 μL。PCR反应条件为:94 ℃ 5 min,94 ℃ 30 s,60 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃ 10 min,4 ℃保存。用1%琼脂糖凝胶电泳检测PCR扩增产物后,送至上海生工生物工程有限公司进行双向测序。
1.4 序列分析
将测序结果用BioEdit 7.0.5软件进行编辑校正。用MEGA v11软件[10]进行同源比对,确定序列长度,并统计DNA序列的碱基组成。统计得到序列碱基分布及变异情况,计算种内及种间遗传距离,构建分子系统树,经过1 000次重复抽样检测的Bootstrap值表示树上各分支的置信度。用DNAsp v6软件[11]计算单倍型数、单倍型多样性指数、核苷酸多样性指数。
2 结果与分析
2.1 COI基因序列分析与单倍型分布
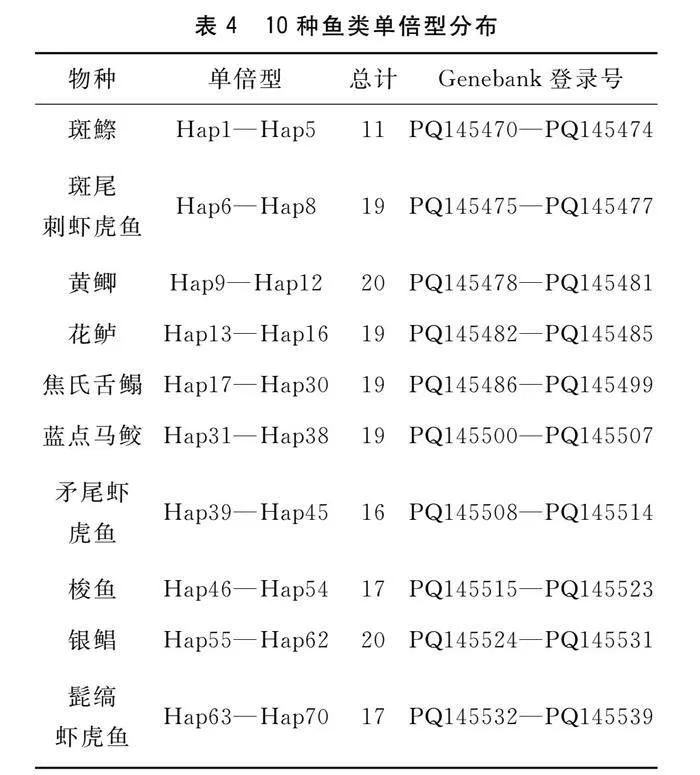
测序结果经过校正和比对分析,获得10种鱼类177个个体的COI基因序列,长度为631 bp。MEGA 7.0软件分析结果显示,序列的T、C、A和G碱基平均含量分别为30.0%、27.9%、24.1%、18.0%,A+T含量(54.1%)略高于G+C含量(45.9%)。其中G+C在密码子第1位点的含量最高(55.4%),除C以外,其余3个碱基在密码子第1、2、3位点分布都不平均(表2)。COI基因密码子的变异统计结果显示,共有不变位点509个,转换位点(69个)多于颠换位点(52个),转换与颠换比为5.36(表3),说明在序列中转换多于颠换。10种鱼类177个个体,共检测到70个单倍型,单倍型Genebank序列号见表4。
2.2 遗传多样性分析
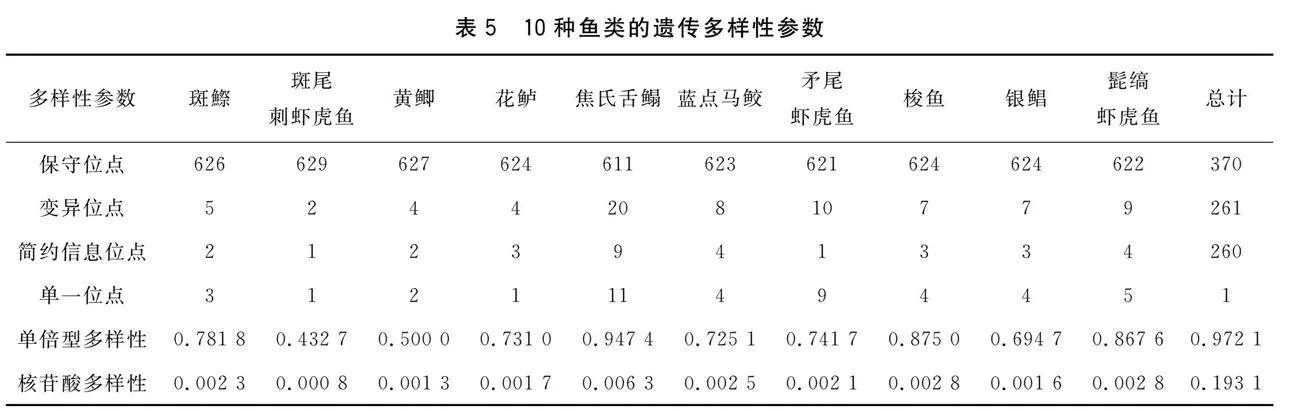
10种鱼类的遗传多样性分析结果显示,所比对序列中共检测出保守位点370个、多态性位点261个,其中简约信息位点260个、单突变位点1个。总体的单倍型多样性为0.972 1,核苷酸多样性为0.193 1。其中焦氏舌鳎的变异位点、单倍型数和单倍型多样性均最高,斑尾刺虾虎鱼的最低(表5)。
2.3 种内距离与种间距离
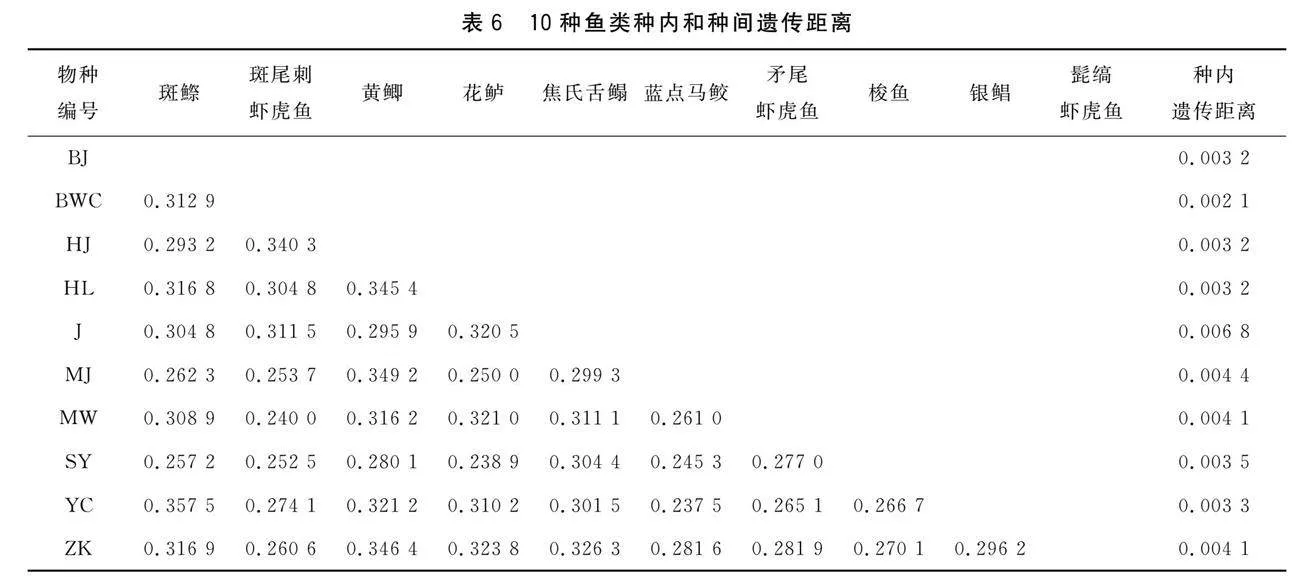
对于本次实验所收集的177条COI序列,利用MEGA v11软件,基于K2P双参数模型,计算出177条鱼的种间遗传距离和种内遗传距离(见表6)。结果显示,10种鱼类的种内平均遗传距离为0.003 8,并且10种鱼类的种内遗传距离均小于DNA条形码规定的最小种内遗传距离0.020 0。其中焦氏舌鳎的种内遗传距离最大,为0.006 8;斑尾刺虾虎鱼的种内遗传距离最小,为0.002 1。
10种鱼类的种间平均遗传距离0.293 6,是种内平均遗传距离的77倍。其中银鲳和斑鰶的种间遗传距离最大,为0.357 5;银鲳和蓝点马鲛的种间遗传距离最小,为0.237 5。
2.4 系统进化树
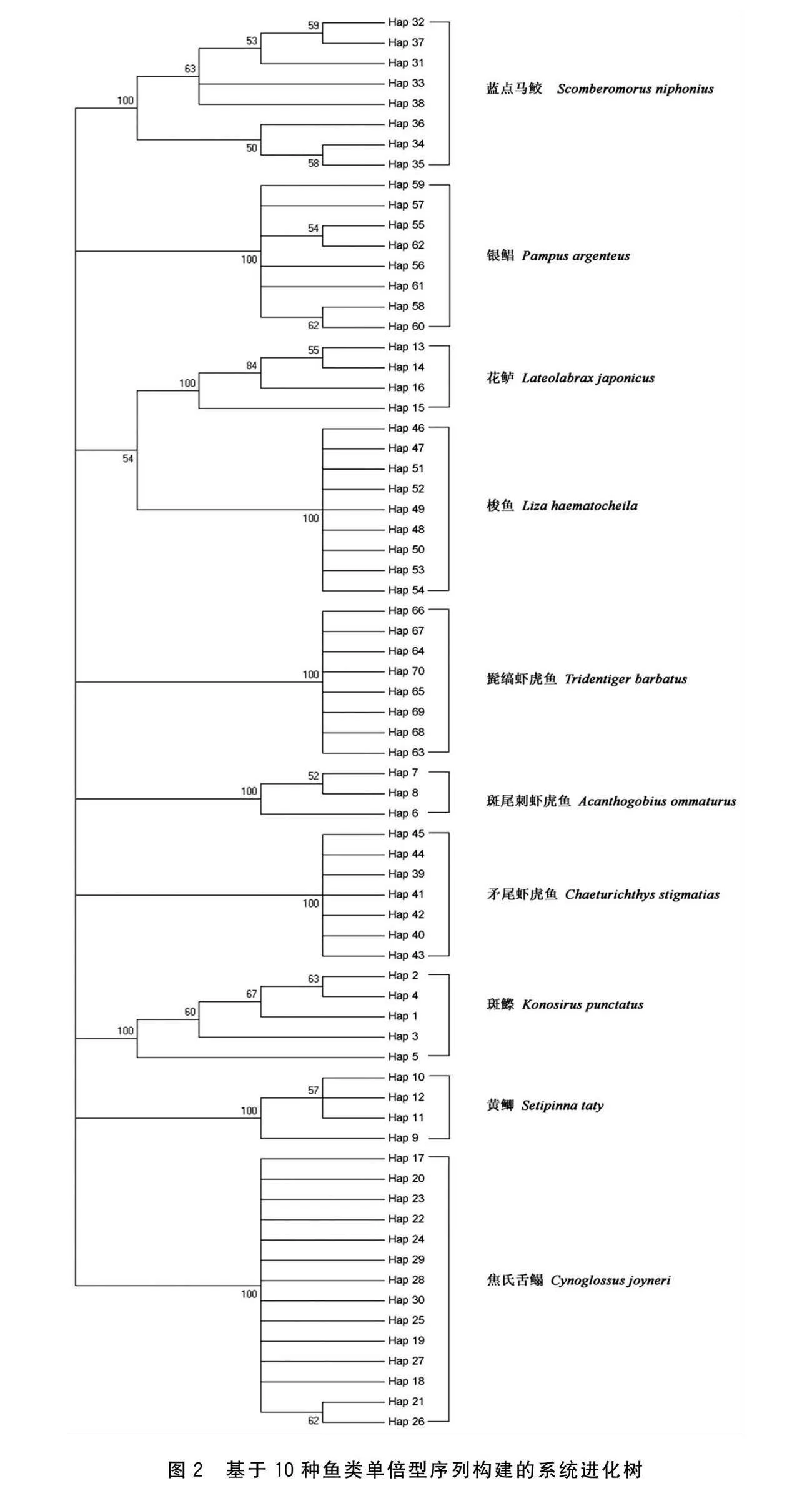
通过邻接法NJ(Neighbor-Joining)构建了10种鱼类70个单倍型的系统进化树(图2),结果显示同种鱼类的不同单倍型均聚为一支,具有明显的分化谱系,与形态学的鉴定结果相吻合。
3 讨论
天津渤海湾位于渤海西部,是众多鱼、虾、蟹、贝等海洋生物的产卵场、索饵场和育肥场,素有“天然鱼池”之称,在我国渔业水域中占有非常重要的地位。近年来,由于海洋污染和过度捕捞等因素的影响,渤海湾的渔业资源量曾经历显著下降。有研究表明2019年和2020年的游泳动物平均密度和生物量均低于2015年和2016年,种群特征显示生物多样性水平并不高[6]。近几年通过开展人工增殖放流、投放人工鱼礁、设立禁渔期等多项措施,监测评估渔业资源正在逐步恢复。在大多数硬骨鱼中,COI基因序列的AT含量均高于GC含量,具有一定的AT偏倚性[12]。本研究结果A+T含量(54.1%)略高于G+C含量(45.9%),与已有研究结果一致。其中密码子的第一位点碱基GC分布频率最高,为55.4%,这与刘凯莹[13]研究结果相似。在分子进化研究中,转换颠换比值可以体现DNA序列的进化方向和物种间的关系,同时也可反映进化频率[14]。本研究中碱基变异的转换与颠换比为5.36,转换多于颠换,说明序列可能在进化过程中经历了较少的突变。
遗传多样性是评估种群资源状况的一个重要依据,因此加强鱼类遗传多样性的保护和研究具有重要意义[15]。遗传多样性指的是物种群体内所有遗传信息的总和,鱼类遗传多样性是鱼类生存、适应和进化的基础,对于维护水生生态系统的平衡和稳定具有重要意义。一般遗传多样性较高的物种,具有更强的适应生存能力和进化潜力。遗传多样性主要包括线粒体DNA的单倍型多样性和核苷酸多样性指标[16]。Grant等[17]研究结果表明,单倍型多样性的临界值为0.5,核苷酸多样性的临界值为0.005。两者的值越高,遗传多样性程度就越高。本研究中的10种鱼类总体的单倍型多样性达到0.972 1,除斑尾刺虾虎鱼和黄鲫的单倍型多样性较低(0.432 7和0.5000 )外,其他9种鱼类的单倍型多样性都在0.5以上,其中斑鰶(0.781 8)、焦氏舌鳎(0.947 4)、髭缟虾虎鱼(0.867 6)和梭鱼(0.875 0)的单倍型多样性相对更高。从渤海湾渔业资源近年来的调查中可以看出,斑鰶、焦氏舌鳎和髭缟虾虎鱼为优势种[6],具有较强的适应环境能力和进化潜力,而梭鱼的高多样性可能与天津市开展的增殖放流工作有关。核苷酸多样性结果显示总体的核苷酸多样性为0.193 1,焦氏舌鳎最高(0.006 3),其余9种鱼类的核苷酸多样性均较低,在0.000 8~0.002 8之间。在10种海水鱼中,焦氏舌鳎的单倍型多样性大于0.5,核苷酸多样性大于0.005,属于高单倍型多样性和高核苷酸多样性类型,说明该种群中存在多种遗传变异,具有较高的遗传多样性。这些变异可能是由一个大而稳定的种群经过长时间历史演化所导致,也可能是被两个不同系群的种群二次接触影响产生的结果[18]。其余9种鱼类均属于高单倍型多样性和低核苷酸多样性类型,说明这些种群曾经历过瓶颈效应后,发生了种群的快速扩张与变异的不断积累[19]。
通过对天津渤海湾10种鱼类的COI基因进行分析,我们发现不同物种之间COI基因序列组成有很大的差异性,说明该基因在不同物种之间有一定的代表性。利用DNA条形码技术进行物种分类和鉴定的关键在于两点,一个是不同物种在系统发育树上可以检测到物种的单系性,本研究中的系统进化树分析结果表明同种鱼类的不同单倍型均聚为一大支;另一个是物种之间的遗传距离应明显大于物种内的遗传距离,而且种间的遗传距离约为种内的遗传距离的10倍以上。Hebert等[3]分析研究了一万多个物种,结果发现绝大多数物种的种内距离低于0.01。本研究的10种海水鱼类,种内的遗传距离在0.002 1~0.006 8,均小于0.01;而种间的平均遗传距离是种内平均遗传距离的77倍,结果证明COI基因可以对天津近岸海域常见的10种海鱼类进行有效鉴定。
本研究利用DNA条形码技术中COI基因对天津渤海湾的10种海水鱼类进行了物种分子鉴定可行性和有效性的测定,结果显示鉴定结果快速有效。同时我们也初步掌握了渤海湾部分鱼类的遗传数据信息,为后续的鱼类遗传多样性的保护和研究奠定基础,为天津渤海湾鱼类资源保护提供数据支持。
参考文献:
[1]
轩中亚,薛竣仁,陈修报,等.基于线粒体COI序列的中华绒螯蟹养殖群体与野生群体遗传多样性与遗传结构分析[J].水产学杂志,2021,34(2):15-22.
[2] ZENG X S,SUN C H,HUANG X Y,et al.DNA barcoding of Scomberomorus (Scombridae, Actinopterygii) reveals cryptic diversity and misidentifications[J].Zookeys,2022,1135(4):157-170.
[3] HEBERT P D,RATNASINGHAM S,DEWAARD J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proc Biol Sci. 2003,270(S1):S96-S99.
[4] 陈大伟,朱鹏灿,刘士力,等.用线粒体COI基因序列分析黄尾鲴养殖群体的遗传结构[J].水产学杂志,2024,37(3):1-6.
[5] SHAN B,LIU Y,YANG C,et al.DNA barcoding of fish in mischief reef:fish diversity of a reef fish community from Nansha Islands[J].Frontiers in Marine Science, 2021,7:618954.
[6] 孙万胜,白明,于洁,等.天津渤海湾渔业生物资源状况及变化分析[J].河北渔业,2022(4):29-33.
[7] 谷德贤,宁鹏飞,王婷,等.天津水域鱼类资源种类名录及原色图谱[M].北京:海洋出版社,2021:1-78.
[8] 白晓慧,杨华,孟一耕,等.大鳞副泥鳅群体线粒体DNA D-loop序列遗传变异分析[J].水产科学,2018,37(5):658-664.
[9] WARD R D,ZEMLAK T S,INNES B H,et al.DNA barcoding Australia's fish species[J].Philos Trans R Soc Lond B Biol Sci,2005,360(1462):1847-1857.
[10] TAMURA K,STECHER G,KUMAR S.MEGA11: Molecular evolutionary genetics analysis version 11[J].Mol Biol Evol,2021,38(7):3022-3027.
[11] ROZAS J,FERRER-MATA A,SNCHEZ-DELBARRIO J C,et al.DnaSP 6: DNA sequence polymorphism analysis of large data sets[J].Mol Biol Evol. 2017,34(12):3299-3302.
[12] 刘红艳,熊飞,翟东东,等.鲿科鱼类DNA条形码鉴定及系统进化研究[J].水产科学,2023,42(4):575-584.
[13] 刘凯莹.鲀形目鱼类DNA条形码及物种快速鉴定技术研究[D].上海:上海海洋大学,2023.
[14] 梁宏伟,孟彦,罗相忠,等.线粒体COI基因条形码在鲿科鱼类物种鉴定中的应用[J].中国水产科学,2018,25(4):772-782.
[15] 吕海敏,冯慧敏,汤永涛,等.基于cyt b基因分析河南省4大水系红鳍原鲌种群的遗传多样性[J].四川动物,2024,43(2):163-171.
[16] 朱威霖,肖珊,杨春玲,等.基于线粒体Cytb基因序列的华南沿海黄鳍棘鲷种群遗传结构分析[J].西南农业学报,2021,34(2):446-454.
[17] GRANT W S, BOWEN B W l.Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J].Journal of Heredity,1998,89(5):415-426.
[18] 李大命,刘燕山,唐晟凯,等.基于Cytb基因序列的江淮下游湖泊鲢群体遗传多样性分析[J].海洋渔业,2023,45(4):423-433.
[19] 张翠萍.华南地区花鰶的个体生物学特征及种群遗传多样性研究[D].广州:广州大学,2023.
Identification and genetic analysis of fish resources in Tianjin Bohai Bay based on DNA barcodes
HE Xiaoxu, BAI Xiaohui, WANG Xiaoyu, MENG Yigeng
(Tianjin Fisheries Research Institute, Tianjin 300221,China)
Abstract:In order to explore the effectiveness of DNA barcode on the identification of some fish species in the Bohai Bay and conduct relevant genetic analysis, an amplified sequencing and comparison analysis were carried out on a total of 177 mitochondrial COI gene sequences of 10 species of marine fish in Bohai Bay, Tianjin.The results showed that the average content of bases T, C, A, and G of COI gene of 10 kinds of marine fish was 30.0%, 27.9%, 24.1%, and 18.0% respectively, with base bias, and a total of 70 haplotypes were detected in 177 gene sequences. Genetic diversity analysis showed that the overall haploid diversity was 0.432 7~0.947 4, and the nucleotide diversity was 0.000 8~0.006 3, among which Cynoglossus joyneri had the highest mutation sites, haplotypes, and haploid diversity. The results of genetic distance showed that the genetic distance between species was 0.237 5~0.357 5, of which the average genetic distance between species was 77 times that of the average genetic distance within the species. The systematic evolutionary tree also had an obvious differentiation spectrum, which coincides with the morphological identification results. The results of this study showed that DNA barcode technology can effectively identify 10 species of fish commonly found in the coastal waters of Tianjin, and relevant genetic analysis can provide data support for the protection of fish resources in Tianjin Bohai Bay.
Key words:DNA barcode; COI gene; genetic diversity; fish
(收稿日期:2024-09-24)
