正向和反向蛋白质结构预测破解生命密码
2025-01-28郭晓强郭贝一
蛋白质是生物体的基本组成部分,也是最重要的生物功能大分子,拥有多样的生物学功能,几乎每一步生命过程都有它们的身影,从物质代谢到能量生成,从生长发育到细胞通信等。结构是功能的基础,蛋白质结构的奥秘是生命科学领域最迫切需要解决的核心问题之一,至今已有十余项蛋白质相关成果荣获诺贝尔化学奖[1],其重要性不言而喻。
蛋白质结构
蛋白质是一种由基本单位——氨基酸构成的生物大分子。为研究方便蛋白质结构被赋予层次化属性:有一级、二级、三级、四级结构之分。
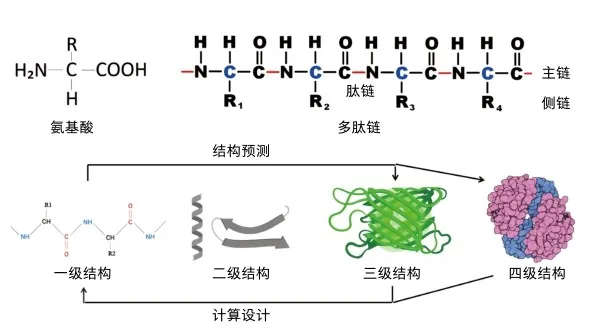
氨基酸
氨基酸是蛋白质的基本组成单位。顾名思义,氨基酸就是既有氨基又有羧基的有机物。构成蛋白质的天然氨基酸共20种(差别在于侧链的R基团不同),除甘氨酸外,其他都为L-α-氨基酸。一个氨基酸的氨基可与另一个氨基酸的羧基发生脱水缩合反应生成二肽,形成的化学键称肽键。多个氨基酸发生类似反应生成多肽,分子量较大的多肽通常称为蛋白质,二者间并无严格界限。
蛋白质一级结构
一级结构又称初级结构,指蛋白质链状分子中氨基酸的线性排列顺序,它是不同蛋白质差异的基础。1955年,英国生物化学家桑格(F.Sanger)完成了胰岛素两条多肽链一级结构的测序及二硫键位置的确定,荣获1958年诺贝尔化学奖。
蛋白质二级结构
二级结构指蛋白质部分主链形成的局部结构。1951年,美国理论化学家鲍林(L.Pauling,1954年诺贝尔化学奖获得者)和生物化学家科里(R.B.Corey)提出了二级结构的两种主要形式:α-螺旋和β-折叠。
蛋白质三级结构
三级结构指一条多肽链构成的完整三维结构,即包括主链形成的框架结构,并涵盖侧链之间的相对位置。1957年,英国生物化学家肯德鲁(J.C.Kendrew)首次确定肌红蛋白三级结构,分享了1962年诺贝尔化学奖。
蛋白质四级结构
四级结构指两条或两条以上独立多肽链构成的完整空间结构。1959年,英国生物化学家佩鲁茨(M.F.Perutz)首次确定血红蛋白四级结构,分享1962年诺贝尔化学奖。
蛋白质空间结构的实验研究
为解析蛋白质空间结构(三级和四级结构),而先后开发出的X-射线晶体衍射、磁共振波谱、冷冻电镜等众多技术,由于其高昂的费用、漫长的时间和高技术门槛等限制,使得蛋白质空间结构研究主要由少数实验室完成,至今实验解析的蛋白质空间结构只有几十万种。
相较而言,一级结构测定较为容易,特别是多个物种基因组计划的完成和运用中心法则从DNA碱基序列推导蛋白质中氨基酸的序列,至今已破译上亿种蛋白质的一级结构,二者之间的巨大差异使研究人员决定另辟蹊径。
一级结构决定高级结构
1961年,美国生物化学家安芬森(C.B.Anfinsen)借助核糖核酸酶变性-复性实验得出结论:蛋白质初级结构决定高级结构(安芬森定则),他因此分享了1972年诺贝尔化学奖。安芬森定则清晰地说明了蛋白质空间结构并非随机形成,而是根植于蛋白质一级结构,这一规律成为蛋白质结构预测的理论基础。
蛋白质结构预测
1970年代开始,研究人员开始尝试进行蛋白质结构预测研究,但一直进展缓慢,直到21世纪初,随着计算机的应用和人工智能技术的发展,该领域取得了一系列重大突破,尤以2024年3位诺贝尔奖获得者的贡献最为突出。蛋白质结构预测有两种策略:一是正向预测,即从一级结构推测空间结构;一是反向预测,即从三维结构反推氨基酸序列,又称蛋白质计算设计。
正向蛋白质结构预测
蛋白质理论预测发展已有较全面描述[1],这里简要介绍。
蛋白质数据库(proteindatabank,PDB)
1971年建立PDB,目的在于随时收集采用实验方法解析的各种蛋白质空间结构信息,以供全球研究人员方便使用,它是蛋白质结构预测的重要基础。最初只有7种蛋白质结构信息,2014年已突破10万种,且还在快速增加,已成为结构预测取得一系列重大突破的原动力。
结构预测基本策略
策略主要有同源建模和从头计算。同源建模基于不同蛋白质间一级结构保守性,从结构已被解析的蛋白质推测新蛋白质结构。从头计算是依据最终形成的蛋白质三维结构能量最低原理,利用能量函数计算不同结构的能量值获得最终结构。两种方法相辅相成,在结构预测发展过程中取得了一定成功,但整体效果并不理想。
蛋白质结构预测关键评估(CASP)
1994年,为推动蛋白质结构预测而发起的每两年举办一次的全球竞赛,为研究人员创建了聚集一堂分享该领域新进展的机会。主办方将未解结构的蛋白质氨基酸序列交给参赛者进行预测,同时与实验专家合作进行测定,最终将预测结构与实验测定的结构进行对比,判定预测准确度。评判标准为全局距离测试(globaldistancetest,GDT)评分,GDT采用百分制,分数越高则意味着理论预测精确度越高。该项赛事为研究人员提供了一个广泛交流的平台,促使大家群策群力,共同推进结构预测的进步。
自CASP举办以来,蛋白质结构预测方面确实取得一系列进展,但距离预期仍差距巨大,如2016年第12届CASP(CASP12)中,对无模板蛋白质预测的GDT得分只有40分。但2018年CASP迎来了革命性突破,GDT一跃达到60多分,其原因是基于人工智能技术AlphaFold(简称AF)的横空出世。
第一代AF的成功
哈萨比斯(D.Hassabis)是计算机和认知神经领域的专家,于2010年主导成立DeepMind公司,最初是想开发棋类游戏的人工智能模型,公司于2014年被谷歌收购,但基本架构不变。两年后,DeepMind引起全球轰动,源于开发的人工智能工具AlphaGo击败了围棋世界冠军。哈萨比斯决定挑战更大的难题,那就是用人工智能方法解决蛋白质的折叠问题,因此成立AF项目组。2018年,哈萨比斯领导的团队基于卷积神经网络深度学习策略构建第一代AF(AF1),使预测准确度大幅提升。AF1利用PDB已有的结构进行训练,生成基于多重序列比对所得氨基酸间空间距离图(确切地说,是距离概率分布图),它们提供的信息足够充足,从而可以在此基础上构建出蛋白质三维结构。相较于先前的方法,AF1确实是一个巨大进步,但所得结果与实验测定的蛋白质结构在精度方面仍有较大差距。哈萨比斯团队继续完善AF1,期待进一步突破,但残酷的现实是该方法遇到了瓶颈。无论如何改进都无法更近一步,江珀(J.M.Jumper)的加入破解了这一难题。
第二代AF重大突破
江珀是一位理论化学博士,专长于蛋白质动力学的深度学习策略研究,2017年10月加入DeepMind公司并于2018年7月成为AF项目主管,开启新征程。江珀对AF1进行了大刀阔斧的改进,摈弃卷积神经网络而启用转换器(transformer)架构,同时引入深度学习中广泛使用的注意力机制(attentionmechanism)。第二代AF(AF2)包括两个主要模块,分别是进化转换器(evoformer)和结构(structure)。进化转换器同时处理多序列比对表示(不同物种比对序列构成的二维矩阵)和氨基酸对表示(20种氨基酸对构成的二维矩阵)产生的信息,重要的是深度学习过程中两个数集之间相互交换信息并及时更新,从而实现共同进化。然后,结构模块直接应用氨基酸对表示和目标氨基酸序列构建三维骨架结构,每种氨基酸的核心原子(N-Cα-C)定义为一种三角形。随后通过空间旋转和位置移动产生主链结构,再进一步确定侧链构象,从而初步得到预测的三维结构,此结构多次回传到进化转换器进行改进,最终直接输出三维结构[2]。
在2020年的CASP14中,AF2展现出更大成功,不仅大幅领先其他预测方法,复杂蛋白GDT得分达到90(这一数值预示与实验所测结构相当),且在分辨率方面(约1?)也媲美实验所得结构。AF2的成功表明,哈萨比斯和江珀已将人工智能神经网络工具在蛋白质结构预测方面应用得出神入化,可视为对蛋白质结构重大科学问题(困扰蛋白质结构学家达60年之久)的首次突破。AF2源代码的公开进一步增加了其影响力,促使它在全球被广泛测试和验证,从而在基础科学问题解决和药物研发等领域发挥了推动作用。2024年,DeepMind公司进一步推出第三代AF,在原有精准度基础上增加了使用范围,如预测蛋白质复合物、蛋白质-核酸/小分子配体等三维结构。
反向蛋白质结构预测
蛋白质在自然界数量众多,其神奇的功能引起科学界的极大兴趣,其巨大的应用潜力更是成为关注的焦点。但天然蛋白存在诸多缺陷,如催化反应有限、稳定性差和半衰期短等,因此新型蛋白质就成为科学家研究的重要方向。目前新型蛋白质制备主要有两种策略:一是定向进化(天然蛋白质的修改),这一方法的实现使美国科学家阿诺德(F.H.Arnold)分享了2018年诺贝尔化学奖;另一种是理性设计。

蛋白质理性设计又有两种方式,一种是组合策略,另一种是从头设计。组合策略就是把两种或多种蛋白质的局部结构进行合理的组合,从而产生嵌合体,这种策略在实际中已广泛应用,像基因编辑工具ZFN和TELAN技术,免疫学的人源化抗体和CART技术等,这种方法可看作对自然界已有蛋白质的“简单”拼接。从头设计则是设计并制备出自然界中不存在的具有全新氨基酸序列的蛋白质。
分子设计一直是化学家研究的重要内容,如2016年诺贝尔化学奖授予绍瓦热(J.Sauvage)、斯托达特(J.F.Stoddart)和费林加(B.L.Feringa)三位科学家就是表彰他们在“分子机器的设计与合成”方面的贡献。这些都是小分子设计,与此相比,作为大分子的蛋白质其设计难度就相当巨大,因此长期不被看好。
蛋白质从头设计的初期探索
蛋白质从头设计研究开始于1970年代,长期以来进展缓慢,一是蛋白质设计本身就是一种巨大挑战,二是当时蛋白质结构解析刚起步,被阐明的蛋白质结构极为有限,从而大大限制了结构信息的借鉴。所以研究之初,从头设计被认为是不可能完成的任务,直到1980年代才有所改观,先后经历了手动设计、计算设计和模型设计三个阶段。
手动设计
利用简单生物物理原理并结合经验进行设计。20种氨基酸可归类为疏水性(h)和极性(p)两类,其不同类别的组合与蛋白质二级结构存在一定对应关系(基于经验)。如“hpphppp”匹配α-螺旋,“hphp”匹配β-折叠等。1979年,格特(B.Gutte)基于此设计出一个35个氨基酸的RNA结合蛋白。1988年,里根(L.Regan)和德格拉多(W.F.DeGrado)构建了一个4螺旋蛋白[3],其在水溶液中具有协同折叠的球状结构,且非常稳定,成为蛋白质设计史上的第一个里程碑。
计算设计
通过数学方程定义主链构象,并结合侧链重排算法设计完整序列。1997年,达希亚特(B.I.Dahiyat)和梅奥(S.L.Mayo)利用这一策略设计出人工锌指结构,与天然锌指结构的28个氨基酸相比,仅有6个氨基酸相同(表明为全新结构),磁共振波谱证明最终结构与设定非常符合,这标志着蛋白质从头设计史上又一个重要里程碑。但是,该法主要局限于短序列。
模型设计
又称基于碎片和生物信息学的计算设计,主要源于PDB中越来越多的蛋白质结构信息。PDB中特定蛋白质的完整结构信息被分解成不同形状的结构碎片,每个碎片都赋予序列偏好和相互作用模式,这些碎片可根据人类需求重新组装出新型结构的蛋白质。这一阶段始于21世纪初,多位科学家做出了卓越贡献,其中以贝克(D.Baker)的工作最为显著。
模型设计的首度突破
1962年,贝克出生于美国西雅图的一个学术家庭,父亲马歇尔(MarshallBaker)是物理学家,母亲马西娅(MarciaBaker)是地球物理学家,可谓名副其实“学二代”。在哈佛大学期间,贝克最初的专业是哲学和社会科学,然而在修完发育生物学课程并阅读《双螺旋》一书后,他的兴趣在最后一学年转向了生物学。1984年,他进入加州大学伯克利分校,师从细胞生物学家谢克曼(R.Schekman,2013年诺贝尔生理学或医学奖获得者),以酵母为材料研究囊泡的运输机制。
1989年博士毕业后,贝克进入加州大学旧金山分校结构生物学家阿加德(D.Agard)的实验室进行博士后研究。他在细胞组织和分子结构方面的训练为将来从事蛋白质设计工作奠定了基础。1993年,贝克进入西雅图华盛顿大学医学院的生物化学系建立团队,采用常规实验方法如质谱和磁共振波谱研究蛋白质结构。与此同时,他还将研究范围扩大到计算机建模,这为蛋白质折叠产生三维结构,以及这些结构与蛋白质功能的相关性研究提供了关键见解。此时,他决定挑战生物化学领域的重大难题之一——蛋白质结构预测。
贝克在蛋白质结构预测研究过程中开发出计算平台罗塞塔(Rosetta)[4]。罗塞塔将PDB中具有相似局部序列的无关结构碎片进行组装,同时优化序列和结构以适应目标主链构象;计算中使用了蒙特卡洛优化、能量函数,并考虑范德华力、氢键和溶剂效应等因素。罗塞塔参加了1998年的CASP3,比赛中表现良好,成功预测了给定序列的空间结构,算得上一大进步,但整体效果不佳。提升罗塞塔的效果就需要消耗更多计算资源,此时小科研团队就显得捉襟见肘,难以应付,贝克为此启动了一个名为“罗塞塔@家园”的项目,让全球志愿者在自己电脑上协助计算,群策群力逐步改进程序。但遗憾的是,罗塞塔在结构预测方面迟迟没有大的突破。贝克团队却意外发现反向使用罗塞塔却较为成功,就是根据给定蛋白质的结构信息来获取氨基酸序列。贝克的研究也从最初的结构预测转向理性设计,并率先获得成功,算得上“失之东隅,收之桑榆”。

2003年,贝克及同事完成了93个氨基酸的蛋白Top7从头设计和合成,并得到实验确认[5]。这是蛋白质从头设计领域第一个真正意义上的突破,理由是:一是分子量大,93个氨基酸远超以前的设计;二是精确性高,预定结构与最终结构高度一致;三是创新性强,Top7蛋白与天然蛋白质同源性极低,是一种自然界不存在的全新人工蛋白;四是设计难度大,PDB中可供参考的信息非常有限,真正的从头设计。这次成功使科学界普遍接受了蛋白质从头设计的可行性,但这一策略是否具有通用性,特别是能否用于具生理功能蛋白质的设计呢?
砥砺前行
贝克团队随后进一步改进罗塞塔程序,提高性能,并尝试从头设计功能蛋白,首先选择具有催化功能的酶。2008年,贝克等人首次实现了酶的计算设计,最终获得的人工酶催化反应速率远快于非酶促反应,但与天然酶相比,整体效率尚存在不足[6],借助阿诺德定向进化策略最终获得更高活性酶。这一成果说明从头设计功能蛋白的可行性,但在策略上尚需进一步完善。
贝克团队结合从头设计领域的最新进展和自己团队的经验,对设计策略进行了全面提升,并通过合成一系列新性能的人工蛋白质证明其方法的可行性。他们首先尝试设计配体结合蛋白,获得的类固醇结合蛋白具有高亲和力和高选择性,进一步结合其他方法将亲和力从纳摩尔提升至皮摩尔的水平。2016年,他们创造出具有自我组装能力的二十面体病毒样颗粒,蛋白质分子量达兆道尔顿,直径24~40纳米[7]。他们还在多个应用领域设计出新功能蛋白,如蛋白质开关和传感器等[8],特别需要提及的是,新型冠状病毒感染流行期间,他们还设计出用于诊断和治疗的新蛋白质,为疫苗研发提供了重要的帮助。
天然蛋白质都是基于适应特定自然环境而长期选择的产物,不可避免会存在诸多缺陷,而从头设计则可有效规避部分问题,所设计和合成的人工蛋白质具有更好的稳定性和新性能,增加了实用性[9]。
需补充的是,为更好地进行计算设计,天然结构的借鉴是必不可少的一环。因此,贝克团队也在时刻关注蛋白质结构预测方面的进展。在哈萨比斯和江珀引入人工智能开发出有效的AF2后,贝克也将人工智能策略引入到罗塞塔程序,从而获得自己的蛋白质结构预测工具[10],进一步反哺蛋白质设计方面的工作。
造福人类
2024年诺贝尔化学奖的两项成果堪称完美互补,一项是认识蛋白质,一项是改造蛋白质,且都是在更高纬度实现突破。在化学家眼中,蛋白质是一种略显“不普通”的重要生物大分子,而蛋白质在生命科学乃至医学领域则具有更为举足轻重的地位,所以解决蛋白质结构问题无疑对化学和生理学与医学均具有重要价值,因此他们获得诺贝尔奖实至名归。化学更看重普适性(蛋白质整体研究),而生理学或医学更偏向特殊性(某种或某类蛋白质研究),看来授予化学奖显然更合适。
任何科学突破都非一朝一夕或单纯极少数天才人物的贡献,而是一代代科学家前赴后继、长期探索和积累的结果。从上面的介绍可清晰看出结构生物学、蛋白质合成、计算机科学、人工智能等领域的科学家均发挥了重要作用,2024年的三位获奖者正是在借鉴和吸收前人成果基础上并结合自身敏锐的洞察力和卓越的创造力实现了集成式创新,而这些成果又为将来的新突破奠定了重要基础。
蛋白质结构预测的成功为揭开众多生命奥秘和开发新型药物提供了强大工具,而蛋白质计算设计则为这种原本重要的生物大分子赋予了更强大的功能,拓展了其应用范围。后续的研究和应用都将在推动人类健康事业发展和提升生活质量方面发挥重大作用,从而体现诺贝尔奖颁发的真谛——造福人类。
总之,贝克、哈萨比斯和江珀在蛋白质计算设计与蛋白质结构预测领域的卓越贡献,其影响极为深远(有些很难预测),他们开启了生物化学和生物学研究的新纪元,使我们能够以前所未有的方式理解和利用蛋白质,这将对人类的生活和生产带来重大改变。
(本文部分内容参考了诺贝尔奖官方网站的信息,在此表示感谢!)
[1]郭贝一,郭晓强.AlphaFold和蛋白质结构预测.科学,2024,76(5):39-44.
[2]JumperJ,EvansR,PritzelA,etal.HighlyaccurateproteinstructurepredictionwithAlphaFold.Nature,2021,596(7873):583-589.
[3]ReganL,DeGradoWF.Characterizationofahelicalproteindesignedfromfirstprinciples.Science,1988,241(4868):976-978.
[4]SimonsKT,BonneauR,RuczinskiI,etal.AbinitioproteinstructurepredictionofCASPIIItargetsusingROSETTA.Proteins,1999,Suppl3:171-176.
[5]KuhlmanB,DantasG,IretonGC,etal.Designofanovelglobularproteinfoldwithatomic-levelaccuracy.Science,2003,302(5649):1364-1368.
[6]JiangL,AlthoffEA,ClementeFR,etal.Denovocomputationaldesignofretro-aldolenzymes.Science,2008,319(5868):1387-1391.
[7]BaleJB,GonenS,LiuY,etal.Accuratedesignofmegadaltonscaletwo-componenticosahedralproteincomplexes.Science,2016,353(6297):389-394.
[8]LanganRA,BoykenSE,NgAH,etal.Denovodesignofbioactiveproteinswitches.Nature,2019,572(7768):205-210.
[9]HuangPS,BoykenSE,BakerD.Thecomingofageofdenovoproteindesign.Nature,2016,537(7620):320-327.
[10]BaekM,DiMaioF,AnishchenkoI,etal.Accuratepredictionofproteinstructuresandinteractionsusingathree-trackneuralnetwork.Science,2021,373(6557):871-876.
关键词:诺贝尔化学奖蛋白质结构预测从头设计■
