基于宏基因组分析鸡爪熟肉制品中微生物污染与基因信息
2024-11-05宋晟郭焜鹏郝琴王芳严礼李政张继红







摘 要:采集10 种鸡爪熟肉制品,用传统微生物培养法和宏基因组学方法分析鸡爪熟肉制品中微生物污染水平、群落组成及相关遗传信息。结果表明:7号样品菌落总数为1.3×105 CFU/g,不符合GB 2726—2016《食品安全国家标准 熟肉制品》限量要求,其他样品菌落总数结果均<10 CFU/g,微生物DNA浓度检测结果与菌落总数结果基本一致,可有效评估样品中微生物污染水平;1~6号和8~10号样品主要污染微生物为流产衣原体、大肠杆菌、肺炎克雷伯氏菌和鹦鹉衣原体,相对丰度分别为60.87%~75.49%、13.20%~18.41%、8.29%~16.41%和1.67%~3.67%,7号样品主要污染微生物为短乳杆菌、流产衣原体,相对丰度分别约为63%和8%,所有样品共检测到7 种病毒,主要污染病毒为禽白血病病毒;共鉴定出12 种抗生素耐药基因、10 种BactMet基因,对四环素和吖啶橙等多种抗生素与化合物存在抗性;共鉴定出30 种毒力因子基因,编码外膜受体、磷脂酶C等毒力因子;鲍氏不动杆菌携带7 种抗生素耐药基因、5 种BactMet基因及28 种毒力因子基因,是超级耐药与致病菌。
关键词:宏基因组;鸡爪熟肉制品;毒力因子;抗生素耐药基因
Metagenomic Analysis of Microbial Contamination and Genetic Information in Cooked Chicken Feet Products
SONG Sheng1, GUO Kunpeng1, HAO Qin2, WANG Fang1, YAN Li1, LI Zheng1, ZHANG Jihong1,*
(1. Hunan Provincial Key Laboratory of Food Safety Monitoring and Early Warning,
Hunan Provincial Institute of Product and Goods Quality Inspection, Changsha 410000, China;
2. Miluo City Food and Drug Inspection Institute, Yueyang 414400, China)
Abstract: A traditional culture method and metagenomic analysis were used to determine the level of microbial contamination, community composition and related genetic information in 10 samples of cooked chicken feet products. The results showed that the total bacterial count of sample 7 was 1.3 × 105 CFU/g, which did not meet the requirements of the
GB 2726-2016 National Food Safety Standard for Cooked Meat Products. The total bacterial counts of the other samples were all less than 10 CFU/g. The results of microbial DNA concentration and total bacterial count were basically consistent, thus being valid to assess the level of microbial contamination in the samples. The main contaminant bacteria in samples 1–6 and 8–10 were Chlamydia abortus, Escherichia coli, Klebsiella pneumoniae, and Chlamydia psittaci, with relative abundances of 60.87%–75.49%, 13.20%–18.41%, 8.29%–16.41%, and 1.67%–3.67%, respectively. The main contaminant bacteria in sample 7 were Lactobacillus brevis and C. abortus, with relative abundances of about 63% and 8%, respectively. A total of 7 viruses were detected in all samples, the main one being Avian leukemia virus. A total of 12 antibiotic resistance genes and 10 BactMet genes were identified, indicating resistance to various antibiotics and compounds such as tetracycline and acridine orange. A total of 30 virulence factor genes were identified, encoding outer membrane receptor, phospholipase C, and other virulence factors. Acinetobacter baumannii was found to carry 7 antibiotic resistance genes, 5 BactMet genes, and 28 virulence factor genes, being a super antibiotic-resistant and pathogenic bacterium.
Keywords: metagenome; cooked chicken feet products; virulence factors; antibiotic resistance genes
DOI:10.7506/rlyj1001-8123-20240713-183
中图分类号:TS251.6 文献标志码:A 文章编号:1001-8123(2024)11-0001-11
引文格式:
宋晟, 郭焜鹏, 郝琴, 等. 基于宏基因组分析鸡爪熟肉制品中微生物污染与基因信息[J]. 肉类研究, 2024, 38(11): 1-11. DOI:10.7506/rlyj1001-8123-20240713-183. http://www.rlyj.net.cn
SONG Sheng, GUO Kunpeng, HAO Qin, et al. Metagenomic analysis of microbial contamination and genetic information in cooked chicken feet products[J]. Meat Research, 2024, 38(11): 1-11. (in Chinese with English abstract) DOI:10.7506/rlyj1001-8123-20240713-183. http://www.rlyj.net.cn
近年来,随着食品工业技术的发展,鸡爪等禽副产品生产逐渐规模化、标准化[1-2],但仍存在鸡爪糜烂变质、微生物污染严重等报道[3]。熟肉制品中微生物污染检测主要采用传统微生物培养法和分子生物学方法。Korkeala等[4]采用微生物培养法证明乳杆菌是熟肉制品中的主要腐败菌;Syne等[5]对肉类加工厂中即食鸡肉和培根进行取样分析,发现金黄色葡萄球菌是样品中最常见的食源性致病菌;Yost等[6]采用多重聚合酶链式反应(polymerase chain reaction,PCR)鉴定出肉品中主要腐败菌为乳酸菌。宏基因组学是近年来兴起的研究微生物多样性的新方法,即从样品中提取所有微生物的DNA,构建宏基因组文库,经数据库比对与生物信息学研究,分析样品中所有微生物的遗传组成和功能[7-8],较传统微生物研究方法能更准确反映样品中微生物组成[9-10]。
Cauchie等[11]对288 个猪肉样品进行嗜冷菌、乳酸杆菌计数及高通量测序,证实传统微生物培养法与宏基因组学方法能相互验证与互补;Liu Jie等[12]采用宏基因组测序技术研究肥瘦鸡盲肠微生物群差异,揭示不同脂肪沉积能力的鸡品种之间肝脏和血浆代谢物的差异;Fang Shaoming等[13]利用16S rRNA和宏基因测序方法揭示兔断奶至育肥期间肠道微生物群落与平均日增体质量间的关系。目前,结合传统微生物培养与宏基因组学方法研究鸡爪熟肉制品微生物污染的研究报道较少[14]。
为深入研究鸡爪熟肉制品中微生物污染情况及来源,本研究收集10 种鸡爪熟肉制品,包括泡椒凤爪、泡椒柠檬凤爪和卤凤爪等,通过平板计数法检测样品中菌落总数,光密度(optical density,OD)法检测样品中微生物DNA,综合评价微生物污染程度;基于宏基因组学方法分析微生物组成,进行主要污染微生物鉴定,分析其可能来源,通过解读遗传物质信息,对抗性和毒力基因进行统计分析,以期为鸡鸭养殖、屠宰和生产过程中实现微生物可追溯、可控提供理论参考。
1 材料与方法
1.1 材料与试剂
鸡爪熟肉制品(10 个)购自京东,均为销量榜排名靠前的具有代表性的产品,其中辐照杀菌技术生产的泡椒凤爪8 个、高温高压杀菌技术生产的五香卤凤爪2 个(表1)。
每种样品约1.2 kg,一部分用于检测菌落总数,约1 kg,-20 ℃保存,另一部分用于DNA提取和测序,约0.2 kg,-80 ℃保存[15]。
平板计数琼脂 北京陆桥技术股份有限公司;E.Z.N.ATM Mag-Bind磁珠DNA提取试剂盒 美国Omega Bio-Tek公司;Qubit3.0 DNA检测试剂盒 美国Invitrogen公司。
1.2 仪器与设备
Masticator Silver拍击式均质器 西班牙IUL公司;MIR-254L-PC低温培养箱 日本三洋公司;Qubit® 3.0荧光计 美国Invitrogen公司;ETC 811 PCR仪 北京
东胜创新生物科技有限公司;FR-980生物电泳图像分析系统 上海复日科技有限公司。
1.3 方法
1.3.1 菌落总数测定
将样品切成小于1 cm3小块,无菌混合均匀,称取25.0 g样品,放入均质袋中。倒入225 mL无菌磷酸盐缓冲液并均质1 min。梯度稀释后,将1 mL溶液移入培养皿中,倒入约15 mL平板计数琼脂。平板旋转混合均匀,待琼脂完全固化后,翻转,(36±1)℃培养48 h,计数[16]。
1.3.2 微生物基因组DNA提取与浓度测定
采用E.Z.N.A™ Mag-Bind磁珠DNA提取试剂盒提取样品中微生物基因组DNA,检测DNA完整性与浓度。
1.3.3 宏基因组测序
将第2代测序原始图像数据文件转换为原始测序序
列[17],经质量评估和过滤获得洁净数据[18-19]。开放阅读框(open reading frame,ORF)预测后生成候选基因集,并对样品中所有预测基因序列进行聚类,以每类中最长的基因作为代表序列,构建非冗余基因目录。对基因数和基因长度进行分析,得到基因丰度信息[20]。
1.3.4 物种分类和基因功能注释
使用德国波恩大学Crystal Impact GbR公司开发的DIAMOND V0.8.20软件对基因集的蛋白质序列与美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)非冗余蛋白质序列进行比较,筛选条件:可靠性评价值(E值)<1.0×10-5,得分>60。获得基因的物种分类注释信息后,计算界、门、纲、目、科、属和种等不同分类级别的物种相对丰度[21-23]。将基因集的蛋白质序列与抗菌杀菌剂和金属抗性基因数据库(antibacterial biocide and metal resistance genes database,BacMet)、毒力因子数据库(virulence factor database,VFDB)和抗生素耐药基因数据库(antibiotic resistance genes database,ARDB)进行比较,获得功能基因的相对丰度[24-27]。
1.4 数据处理与分析
采用Excel对数据进行处理与分析。
2 结果与分析
2.1 菌落总数和DNA质量浓度
由表2可知,7号样品菌落总数为1.3×105 CFU/g,超过GB 2726—2016《食品安全国家标准 熟肉制品》限量要求,属不合格产品,存在严重的微生物污染。其他9 个样品菌落总数均小于10 CFU/g。由此可知,熟制鸡爪食品安全风险相对较低,与Lewis[28]、Benedito[29]等研究结果一致。DNA浓度检测结果与菌落总数检测结果一致,其中除6号ad7e4fd3cca8d755990dade80a5fd9f9样品外,其余样品的生产原料均存在一定程度的微生物污染,但经过杀菌,微生物已被有效灭活。
2.2 样品污染微生物组成
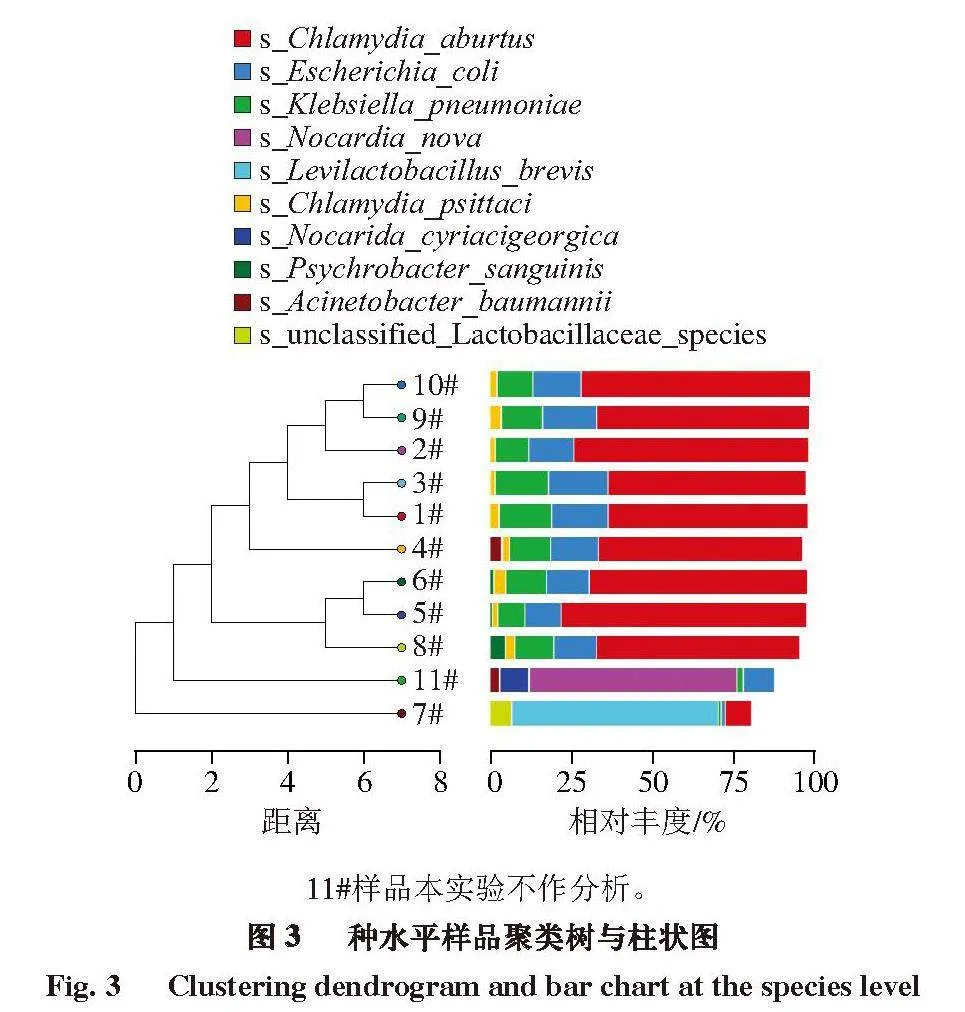
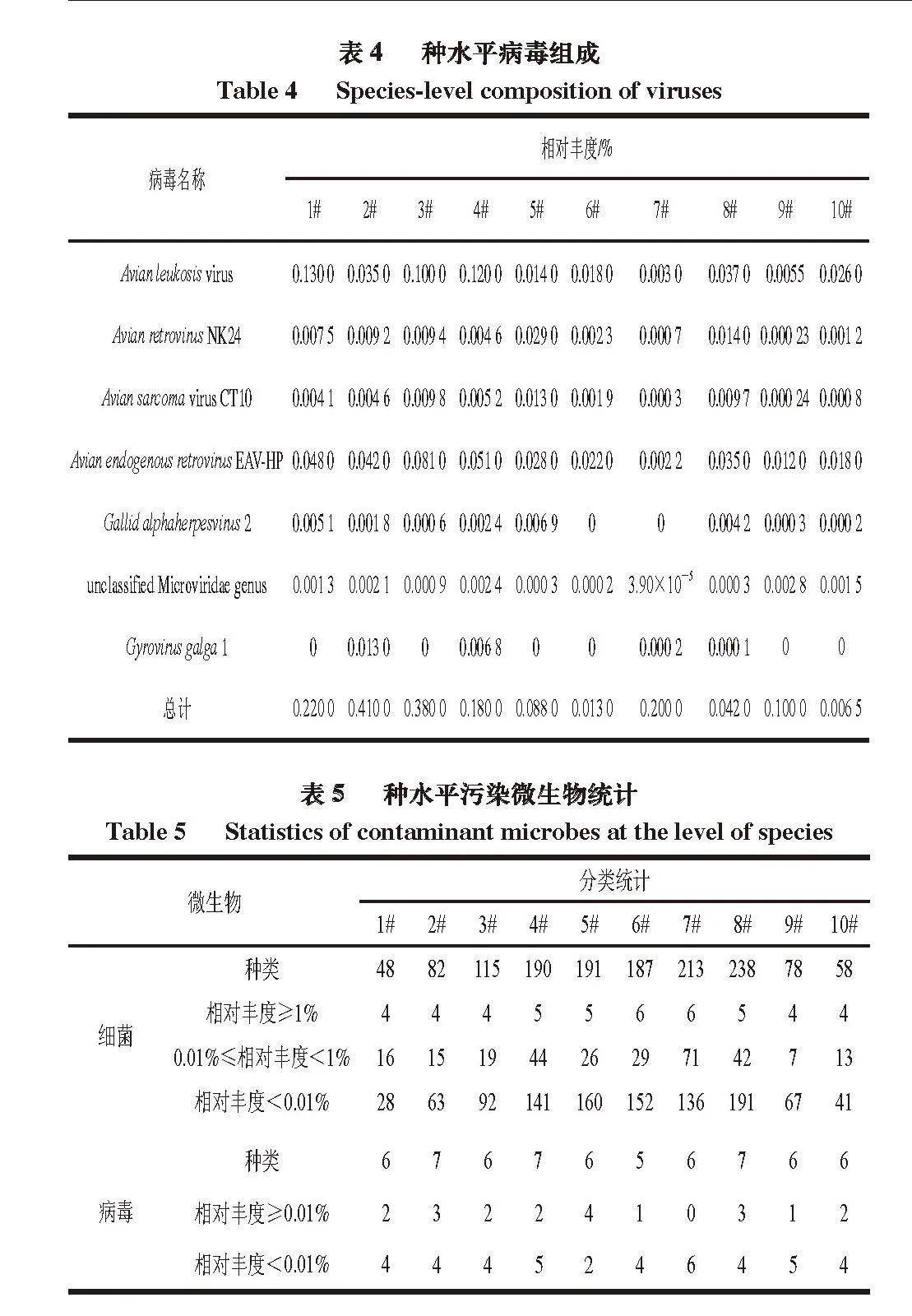
在种水平上,所有样品中共检出368 种微生物,7、8号样品检出210多种微生物;4~6号样品检出约190 种微生物;3号样品检出115 种微生物;2、9号样品检出约80 种微生物,1、10号样品检出约50 种微生物。在所有样品中,相对丰度≥1%的微生物数量在4~6 种之间。如图1及表3~5所示,1~6、8~10号样品主要污染微生物为流产衣原体(Chlamydia abortus)、大肠杆菌(Escherichia coli)、肺炎克雷伯氏菌(Klebsiella pneumoniae)和鹦鹉热衣原体(Chlamydia psittaci),相对丰度分别为60.87%~75.49%、13.20%~18.41%、8.29%~16.41%和1.67%~3.67%;7号样品主要污染微生物为短乳杆菌(Lactobacillus brevis)、流产衣原体、unclassified Lactobacillaceae species、植物乳植杆菌(Lactiplantibacillus plantarum)、unclassified Levilactobacillus species和大肠杆菌,其相对丰度分别约为63%、8%、6%、4%、1%和1%,与已有研究[30-32]结果相似。在种水平上,所有样品共检出7 种病毒,2、4、8号样品检出7 种病毒;1、3、5、7、9、10号样品检出6 种病毒,6号样品检出5 种病毒。主要污染病毒为禽白血病病毒(Avian leukosis virus),1、3、4号样品禽白血病病毒相对丰度分别为0.130 0%、0.100 0%、0.120 0%,样品2、5、6、8、10中禽白血病病毒相对丰度分别为0.035 0%、0.014 0%、0.018 0%、0.037 0%、0.026 0%,与Fadly[33]、Sung[34]等研究结果相似。
仅统计相对丰度前10 位微生物。
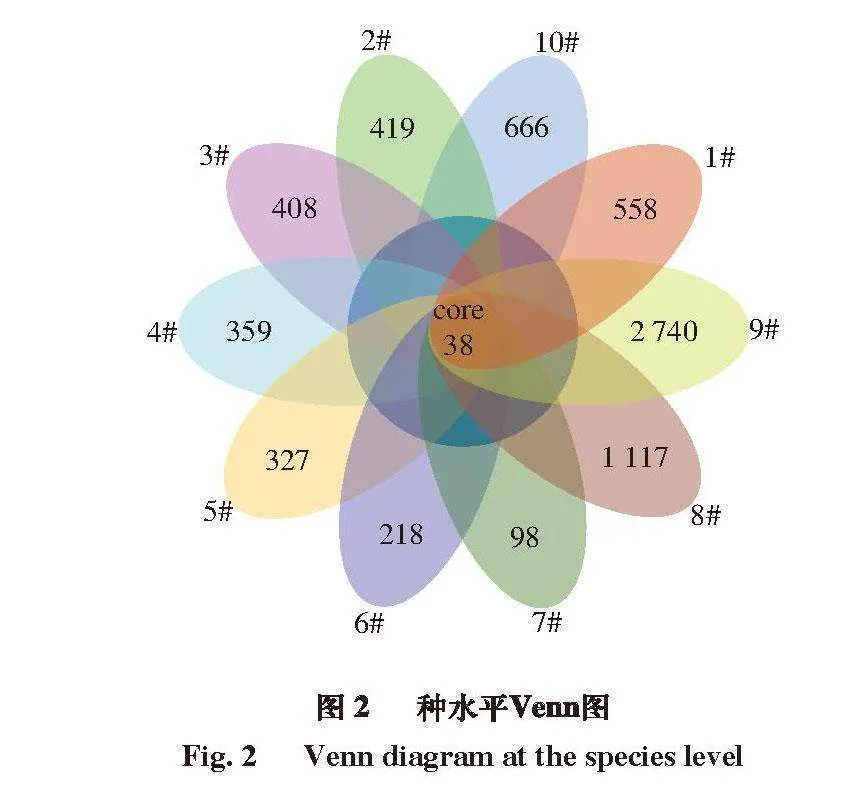
采用Venn图分析不同样品中共有或特有微生物种类,将微生物种类组成相似性和重叠性信息可视化。由图2可知,10 个样品均存在共有和特有的微生物种类,按门、纲、目、科、属和种水平分析,分别有7、14、24、31、31、38 种共有微生物。包括常见于畜禽肉及其制品中的肠道沙门氏菌、大肠杆菌、空肠弯曲杆菌等多种致病菌[35-37],详见表6。
在种水平基于相似性结果绘制聚类树,由图3可知,
7号样品微生物组成与其他样品完全不同。其主要由短乳杆菌组成,而1~6、8~10号样品主要由流产衣原体组成。
11#样品本实验不作分析。
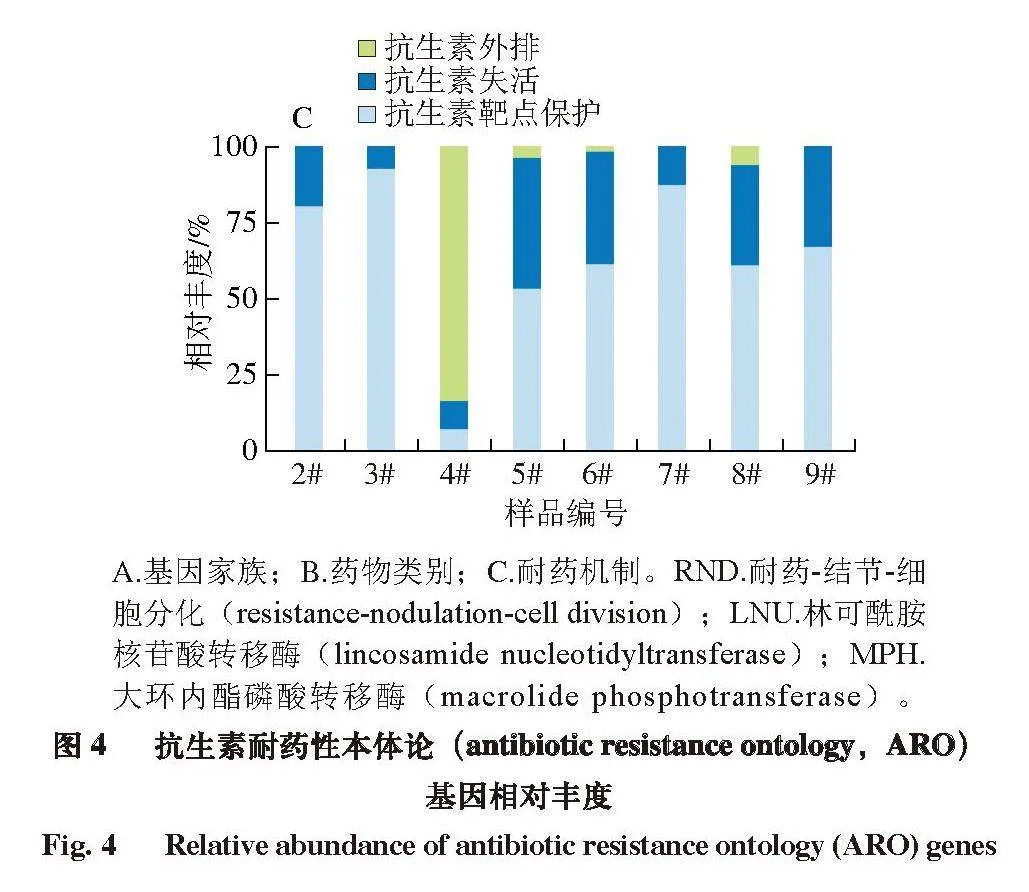
2.3 抗生素耐药基因分析
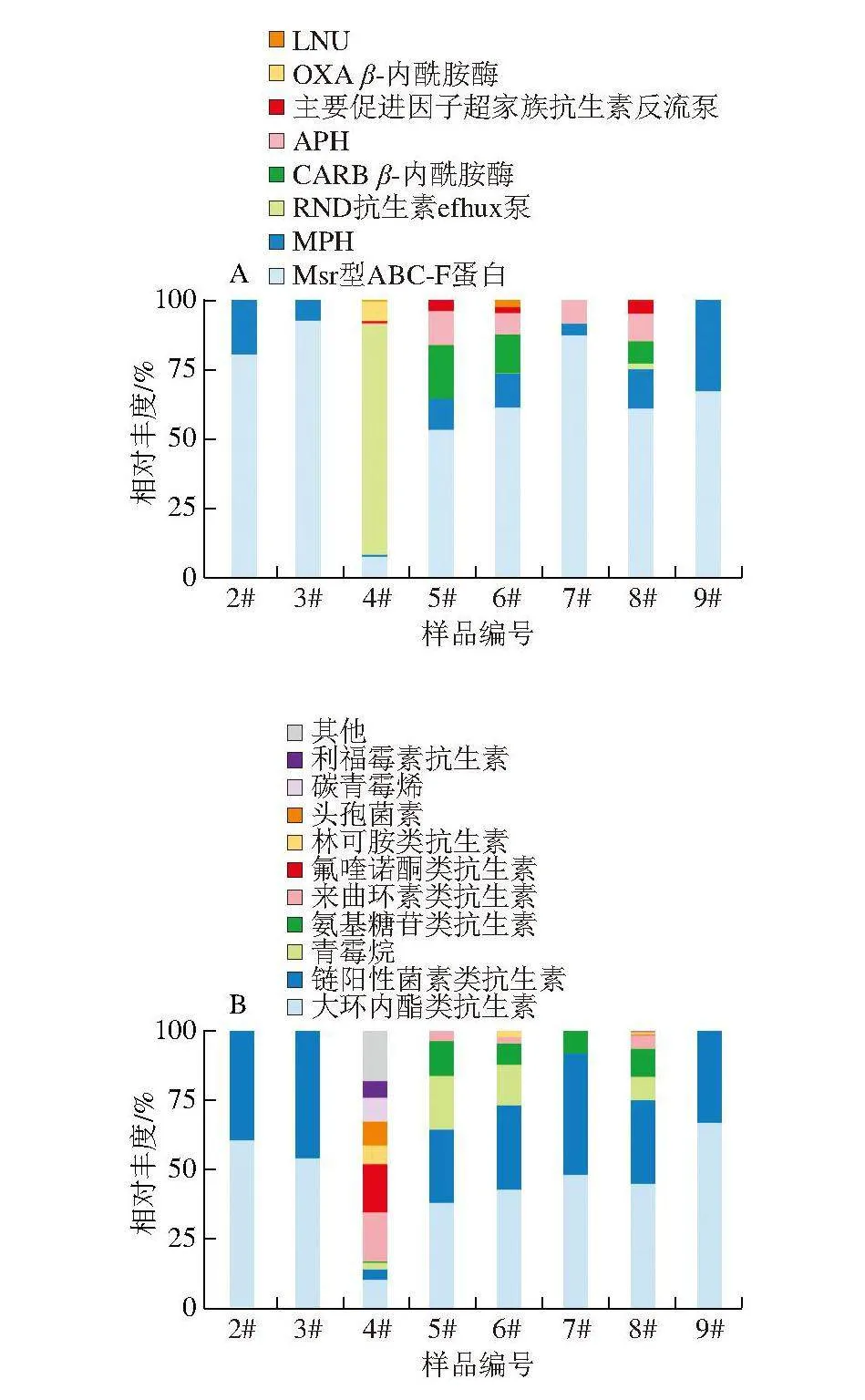
如图4、表7所示,10 个样品中共发现12 种抗生素耐药基因,涉及13 种抗生素耐药性。根据基因功能注释,tet(H)、tet(39)、adeF、adeH和adeJ的耐药机制为抗菌外排;OXA-69、CARB-16、APH(3’’)-Ib、
APH(6)-Id、lnuA和mphE的耐药机制为抗生素失活;msrE的耐药机制为抗生素靶点保护。鲍氏不动杆菌(Acinetobacter baumannii)是携带抗生素耐药基因最多的细菌,包括adeF、adeH、adeJ、OXA-69和APH(3’’)-Ib,
对多种药物具有耐药性,如氨基糖苷类抗生素、碳青霉烯类抗生素和四环素类抗生素等[38-39]。肺炎克雷伯氏菌携带adeH、adeJ、OXA-69和msrE 4 种抗生素耐药基因,对大环内酯类抗生素和链阳性菌素类抗生素等多种药物具有耐药性[40]。unclassified Pseudomonadota species携带adeJ、APH(3”)-Ib和mphE 3 种抗生素耐药基因,对多种药物具有耐药性,如青霉烷和大环内酯类抗生
素等[41]。肠沙门氏菌(Salmonella enterica)携带adeJ和msrE,对林可胺类抗生素等具有耐药性。睡眠嗜组织菌(Histophilus somni)携带tet(H)和adeJ,对四环素类抗生素等具有耐药性[27,42]。除1、10号样品外,其余样品均检出抗生素耐药基因。其中,4、8号样品检出9 种抗生素耐药基因,6号样品检出7 种抗生素耐药基因,
5号样品检出6 种抗生素耐药基因,7号样品检出3 种抗生素耐药基因,2、3、9号样品检出2 种抗生素耐药基因。
A.基因家族;B.药物类别;C.耐药机制。RND.耐药-结节-细胞分化(resistance-nodulation-cell division);LNU.林可酰胺核苷酸转移酶(lincosamide nucleotidyltransferase);MPH.大环内酯磷酸转移酶(macrolide phosphotransferase)。
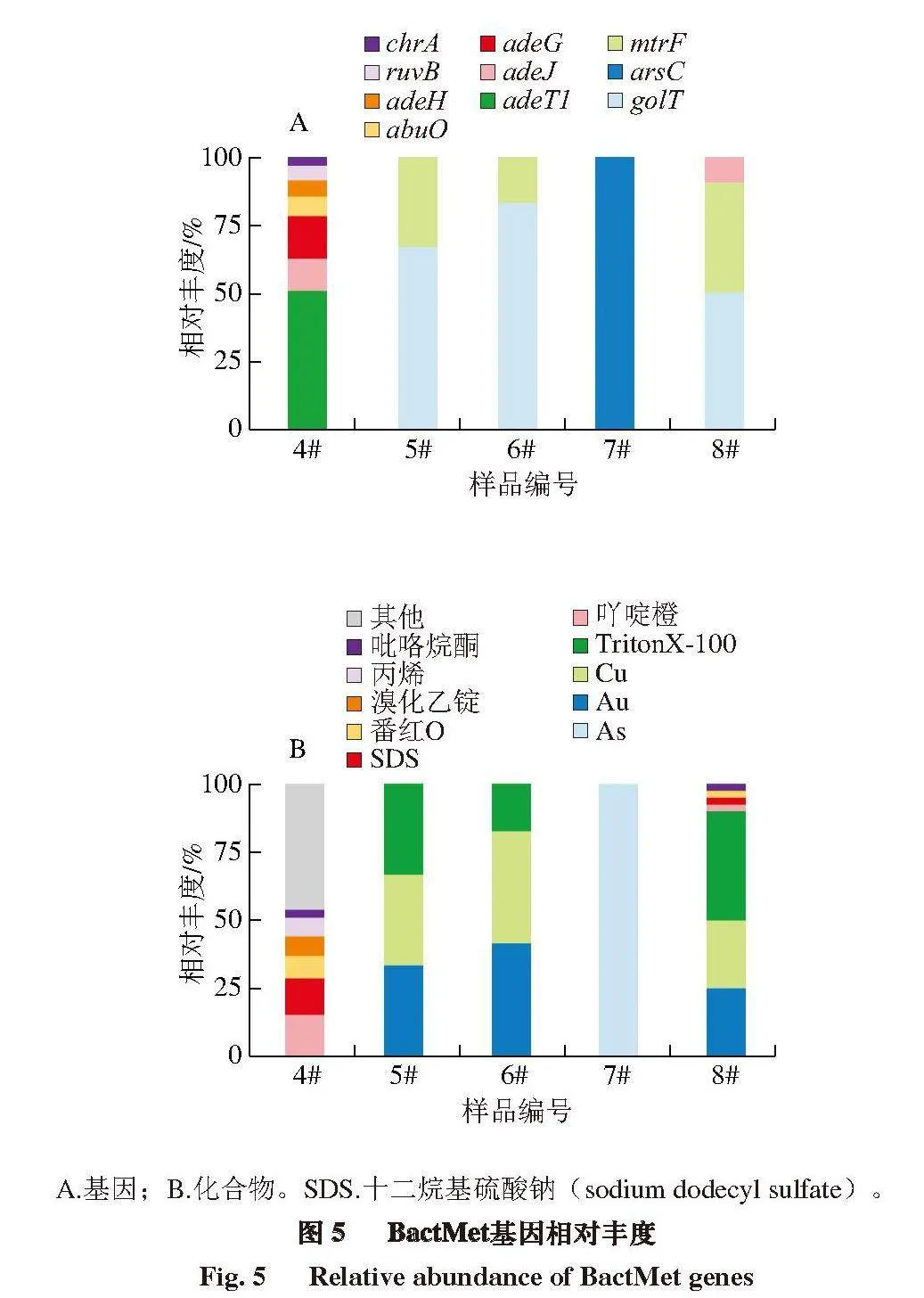
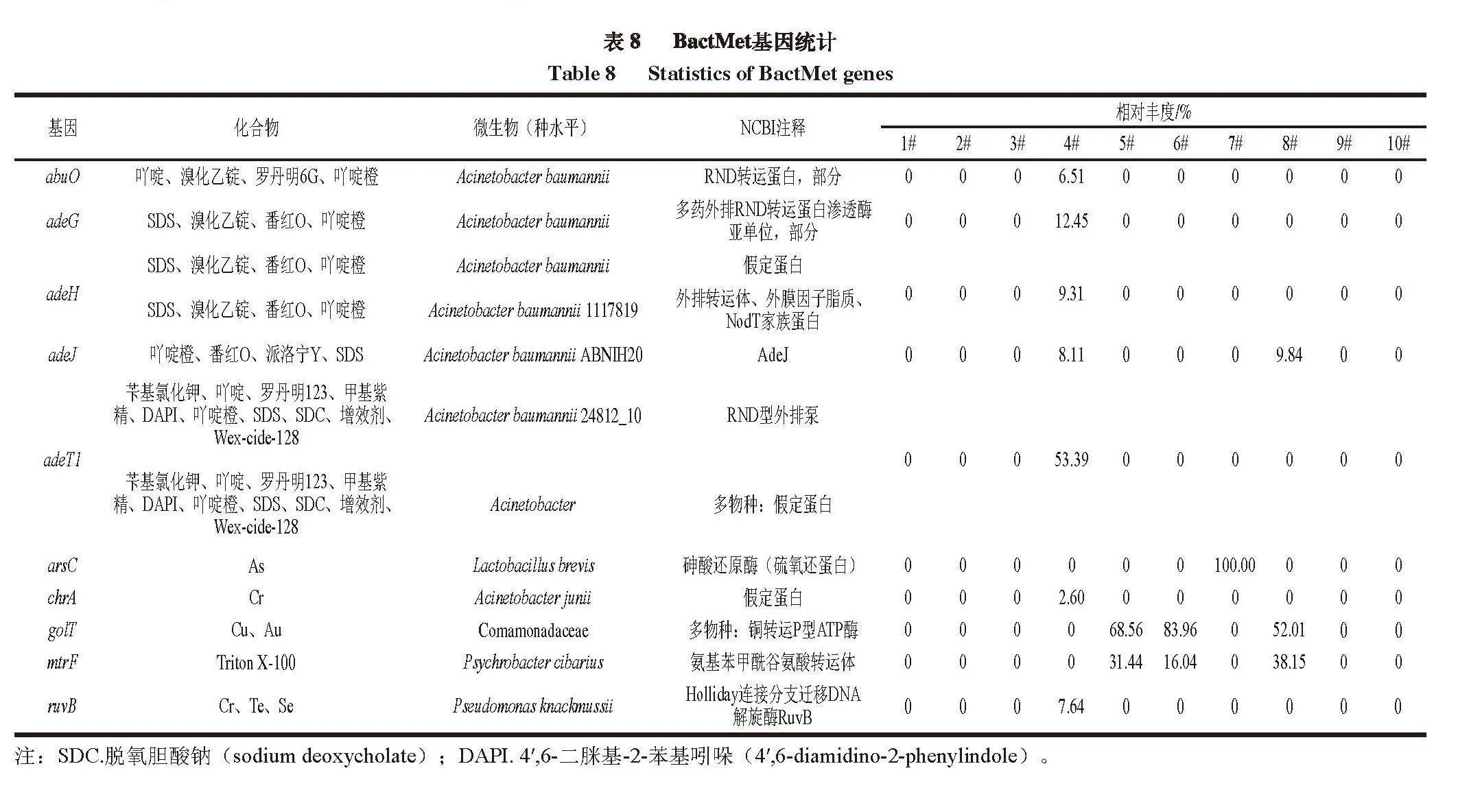
2.4 BactMet基因分析结果
如图5、表8所示,10 个样品中共发现10 种BactMet基因,涉及3 种类型的抗生素耐药机制,包括抗生素外排、抗微生物失活和抗微生物靶标保护。10 个样品中共发现21 种类型的化合物抗性。鲍氏不动杆菌是携带最多BactMet基因的细菌,包括abuO、adeG、adeH、adeJ和adeT1,对多种化合物具有抗性,如吖啶等;
琼氏不动杆菌(Acinetobacter junii)携带chrA,对Cr具有抗性;短乳杆菌携带arsC,对As具有抗性;丛毛单孢菌(Comamonadaceae)携带golT,对Cu和Au具有抗性;克氏假单胞菌(Pseudomonas knackmussii)携带ruvB,
对Cr、Te和Se具有抗性[25,43]。养料嗜冷杆菌(Psychrobacter cibarius)携带mtrF,对Triton X-100具有抗性。4~8号样品中
检测到BactMet基因。其中,4号样品检出7 种BactMet基因,8号样品检出3 种BactMet基因,5、6号样品检出2 种BactMat基因,7号样品检出1 种BactMeta基因。
A.基因;B.化合物。SDS.十二烷基硫酸钠(sodium dodecyl sulfate)。
2.5 毒力因子分析结果
VFDB包括2 个子数据库:setA数据库和setB数据库,其中setA数据库是核心库,包含已验证的毒力基因,setB是全数据库,在setA的基础上增加了预测的毒力基因。
根据VFDB(setA数据库)的同源性比较结果,
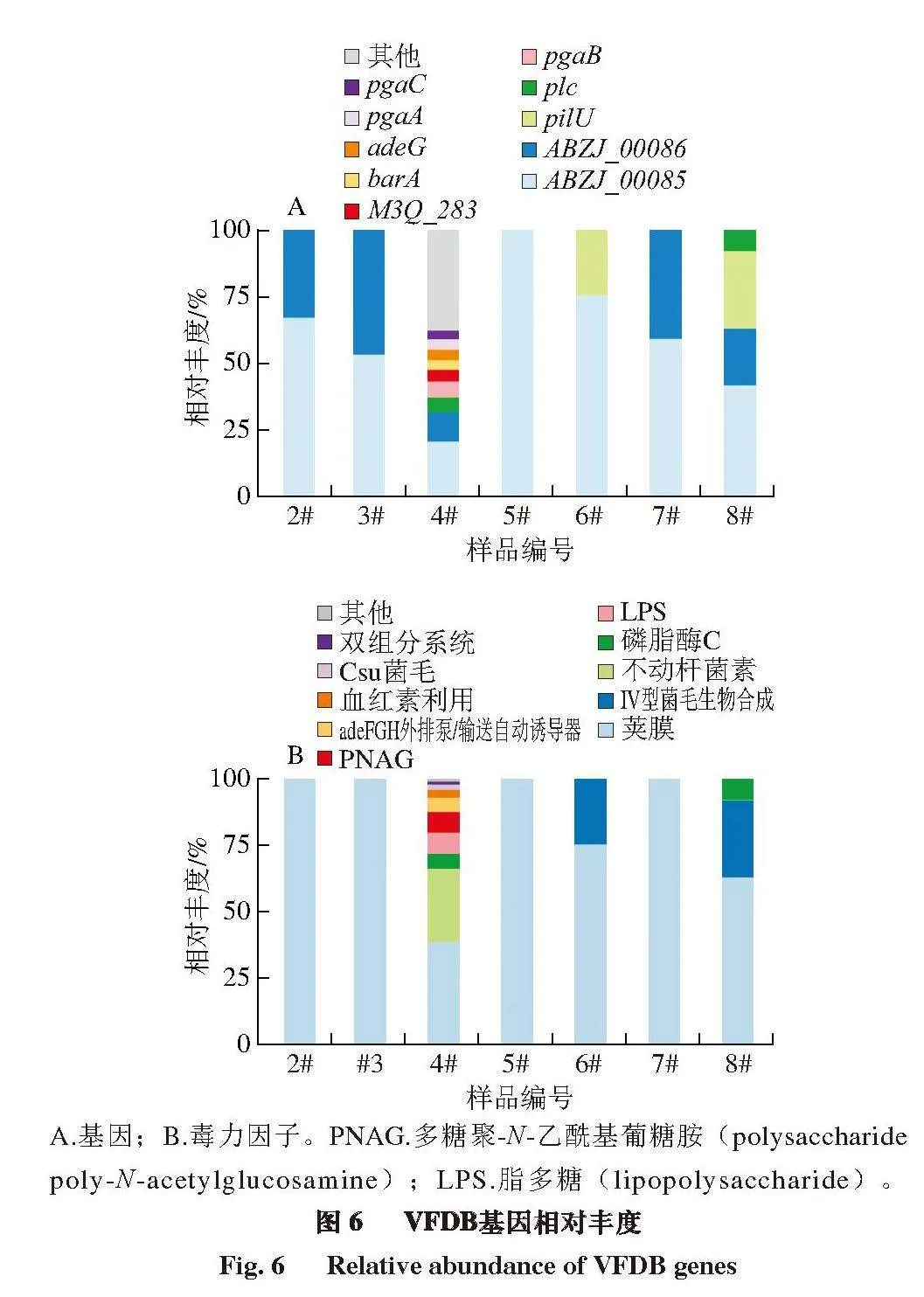
在4号样品中鉴定出7 种毒力因子基因,而在其余样品中未发现毒力因子基因。鲍氏不动杆菌ACICU是携带毒力因子基因最多的细菌,其携带的毒力因子基因包括bauA、barA、csuE、basB、bfmS和plc,与Harding等[39]结果一致。bauA、barA和basB编码的蛋白质有外膜受体、多药ABC转运蛋白ATP酶、渗透酶及非核糖体肽合成酶模块,与不动杆菌素有关,其功能是高亲和力儿茶酚-羟肟酸铁载体与宿主细胞竞争铁;bfmS编码信号转导组氨酸激酶,其功能是控制生物膜的形成和细胞形态,参与细胞黏附、抗血清杀伤和抗生素敏感性;csuE编码的蛋白质为假定蛋白,与Csu菌毛有关,其功能是在生物膜形成的最初步骤中发挥作用,使细菌细胞黏附在非生物表面,并在生物膜结构完全发育之前开始形成微菌落;plc编码的蛋白质是磷脂酶C,其功能是通过裂解宿主细胞膜中的磷脂及降解黏膜屏障中的磷脂促进细菌入侵,有助于宿主细胞的裂解,从而促进发病机制。嗜肺军团菌
亚种携带katB,其编码的蛋白质为过氧化物酶katB,其表达在指数期后达到最高,对细胞内存活和传播很重要,如图6、表9所示。
A.基因;B.毒力因子。PNAG.多糖聚-N-乙酰基葡糖胺(polysaccharide poly-N-acetylglucosamine);LPS.脂多糖(lipopolysaccharide)。
根据VFDB(setB数据库)的同源性比较结果,共鉴定出30 种毒力因子基因,除1、9、10号样品外,其余样品均鉴定出毒力因子基因。在4号样品中鉴定出28 种毒力因子基因,在8号样品中鉴定出4 种毒力因子基因,在2、3、6、7号样品中鉴定出2 种毒力因子基因,而在5号样品中鉴定出1 种毒力因子基因。在鲍氏不动杆菌ATCC 17978中鉴定出8 种毒力因子基因,包括plc、bauB、pgaA、pgaB、pgaC、adeG、adeH和bfmR,它们相应的功能蛋白分别为磷脂酶C、铁不动杆菌素结合蛋白、假定蛋白、血红素储存信号肽蛋白、血红素储存系统HmsR蛋白、阳离子/多药外排泵、RND外排转运体和NarL系列双组分反应调节器。在鲍氏不动杆菌AB0057中鉴定出5 种毒力因子基因,包括barA、basD、bauC、AB57_0984和csuE,相应功能蛋白分别为ABC转运蛋白、非核糖体肽合成酶BasD、铁不动杆菌素转运系统渗透酶、转录调节因子、LysR蛋白家族和CsuE蛋白。在鲍氏不动杆菌TYTH-1中鉴定出3 种毒力因子基因adeH、M3Q_282和M3Q_283,相应的功能蛋白分别为NodT家族外排转运体外膜脂蛋白、蛋白酪氨酸磷酸酶和假定蛋白。鲍氏不动杆菌D1279779中鉴定出2 种毒力因子基因basB和basA,相应的功能蛋白分别为非核糖体肽合酶和不动杆菌素生物合成蛋白。在鲍氏不动杆菌MDR-ZJ06中鉴定出2 种毒力因子基因ABZJ_00085和ABZJ_00086,其相应的功能蛋白分别为IS4家族转座酶ORF1和IS4家族转座酶ORF2。鲍氏不动杆菌AYE中鉴定出1 种毒力因子基因bauA,其相应的功能蛋白为铁不动杆菌素受体BauA,鲍曼不动菌BJAB0715中鉴定出1 种毒力因子基因barB,其相应功能蛋白为ABC型多药转运系统、ATP酶和渗透酶组分,鲍氏不动杆菌MDR-TJ中鉴定出1 种毒力因子基因ABTJ_03743,其相应作用蛋白为磷酸甘露聚糖突变酶,斯氏假单胞菌(Pseudomonas stutzeri)A1501中鉴定出1 种毒力因子基因pilU,其相应功能蛋白为抽动运动蛋白PilU[26],如图6、表10所示。
3 讨 论
10 种鸡爪熟肉制品经菌落总数检测与宏基因组分析可知,1~6号和8~9号样品主要污染微生物是流产衣原体、大肠杆菌、肺炎克雷伯氏菌和鹦鹉衣原体,这些微生物已在鸡鸭养殖场、鸡鸭肉、鸡鸭肠或粪便中检测到,说明产品中的污染微生物主要来自冻鸡爪,因清洗过程未完全去除,导致终产品中检测到相关基因,建议相关企业加强原料的验收工作,确保购入的冻鸡爪符合食品安全标准,并在加工过程中加强清洗工作,以有效去除微生物污染。7号样品菌落总数不符合食品安全国家标准要求,主要污染微生物为短乳杆菌、流产衣原体、unclassified Lactobacillus species、植物乳杆菌和大肠杆菌,表明污染源可能是泡椒等发酵蔬菜,主要增殖阶段为浸泡过程,建议相关企业加强泡椒等原料的微生物检测与控制,确保原料符合食品安全标准。同时,应高度重视泡椒泡制过程,将其视为危害分析与关键控制点中的重要环节,严格监控卫生条件,防止微生物的污染和增殖。所有样品均检出禽白血病病毒,表明该病毒在家禽养殖中广泛存在,存在潜在危害。禽白血病病毒对家禽健康和生产性能构成威胁,可能导致生产损失和经济影响。因此,家禽养殖工作者需要高度重视该病毒的防治工作。
通过对污染微生物遗传信息分析可知,样品中共鉴定出12 种抗生素耐药基因,对13 种抗生素具有耐药性,涉及3 种类型的抗生素耐药机制。这表明多种抗生素在家禽养殖环节广泛使用,导致不同来源的鸡爪原料存在多种耐药基因,其中鲍氏不动杆菌携带7 种抗生素耐药基因,对7 种抗生素具有耐药性,属于超级耐药菌;共鉴定出10 种BactMet基因,对21 种化合物具有抗性,鲍氏不动杆菌携带最多的BactMet基因,对吖啶、溴化乙锭等多种化合物存在抗性;共鉴定出30 种毒力因子基因,鲍氏不动杆菌中鉴定出28 种毒力因子基因,毒力因子包括磷脂酶C等。鲍氏不动杆菌是本研究中鉴定出的超级致病菌与超级耐药菌,且多个样品中均有检出,说明禽肉中普便存在鲍曼超级致病菌与超级耐药菌,具有极大的食品安全风险。对于普通消费者而言,购买和食用禽肉及其制品时,应高度关注交叉污染风险;对于禽肉加工企业而言,应建立完善的微生物控制体系,包括原料验收、加工过程卫生控制、产品检测等,以确保最终产品的微生物指标符合食品安全标准。同时,食品安全监管机构也应加强对鸡爪熟肉制品等禽肉产品的抽检力度,及时发现并排除食品安全风险。通过加强监管和抽检,促使企业更加重视产品质量和食品安全,从而保障消费者的健康权益。
通过本研究可知,鸡爪中的胶原蛋白、油、盐等不会显著干扰微生物DNA提取,与Cauchie等[11]对猪肉样品分析情况相似,证明传统微生物分析方法与宏基因组分析方法可结合用于肉制品中的微生物污染研究。下一步,将在确保样品的代表性、时间的连贯性、养殖到消费的完整性前提下,收集禽用饲料、养殖用水、养殖场环境样品、鸡鸭粪便、屠宰环境样品、屠宰样品、禽肉生产环境样品、禽肉生产过程样品及终产品,采用现有技术解析微生物群落结构,捕捉微生物群落的动态变化,追踪污染微生物来源,为污染防控提供建设性建议。
4 结 论
7号样品菌落总数为1.3×105 CFU/g,不符合GB 2726—2016限量要求,其他样品菌落总数结果
均<10 CFU/g;1~6号和8~10号样品主要污染微生物为流产衣原体、大肠杆菌、肺炎克雷伯氏菌和鹦鹉衣原体,7号样品主要污染微生物为短乳杆菌、流产衣原体,所有样品共检测到7 种病毒,主要污染病毒为禽白血病病毒;共鉴定出12 种抗生素耐药基因、10 种BactMet基因、30 种毒力因子基因。其中鲍氏不动杆菌携带7 种抗生素耐药基因、5 种BactMet基因及28 种毒力因子基因,是超级耐药与致病菌。基于逐渐补充、完善的生物信息学数据库的第2代测序宏基因组研究方法在食品微生物研究领域具有广泛的应用前景,可用于各类食品中污染微生物研究,以及包括发酵豆制品、酱类等发酵食品中功能微生物组成研究,各类基因的鉴定、统计与分析对基因功能及可能存在的代谢物研究具有重要的指导意义。
参考文献:
[1] KALOGEROPOULOS N, CHIOU A, IOANNOU M, et al. Nutritional evaluation and bioactive microconstituents (phytosterols, tocopherols, polyphenols, triterpenic acids) in cooked dry legumes usually consumed in the Mediterranean countries[J]. Food Research International, 2010, 43(8): 2006-2013. DOI:10.1016/j.foodchem.2010.01.005.
[2] SANTOS F F, AQUINO M H C, NASCIMENTO E R, et al. Chicken feet bacteriological quality at 4 steps of technological processing[J]. Poultry Science, 2011, 90: 2864-2868. DOI:10.3382/ps.2011-01588.
[3] CHEN M H, WANG S W, WANG W Z, et al. Contamination of Salmonella Schwarzengrund cells in chicken meat from traditional marketplaces in Taiwan and comparison of their antibiograms with those of the human isolates[J]. Poultry Science, 2010, 89: 359-365. DOI:10.3382/ps.2009-00001.
[4] KORKEALA H J, BJRKROTH K J. Microbiological spoilage and contamination of vacuum-packaged cooked sausages[J]. Journal of Food Protection, 1997, 60: 724-731. DOI:10.4315/0362-028X-60.6.724.
[5] SYNE S M, RAMSUBHAG A, ADESIYUN A A. Microbiological hazard analysis of ready-to-eat meats processed at a food plant in Trinidad, West Indies[J]. Infection Ecology & Epidemiology, 2013, 3(1): 20450-20459. DOI:10.3402/iee.v3i0.20450.
[6] YOST C K, NATTRESS FM. The use of multiplex PCR reactions to characterize populations of lactic acid bacteria associated with meat spoilage[J]. Letters in Applied Microbiology, 2000, 31: 129-133. DOI:10.1046/j.1365-2672.2000.00776.x.
[7] HANDELSMAN J, RONDON MR, BRADY SF, et al. Goodman molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10): R245-R249. DOI:10.1016/S1074-5521(98)90108-9.
[8] THOMAS T, GILBERT J, MEYER F. Metagenomics: a guide from sampling to data analysis[J]. Microbial Informatics and Experimentation, 2012, 2(1): 3. DOI:10.1186/2042-5783-2-3.
[9] TRINGE S G, RUBIN E M. Metagenomics: DNA sequencing of environmental samples[J]. Nature Reviews Genetics, 2005, 6(11): 805-814. DOI:10.1038/nrg1709.
[10] CHEN K, PACHTER L. Bioinformatics for whole-genome shotgun sequencing of microbial communities[J]. PLoS Computational Biology, 2005, 1(2): 106-112. DOI:10.1371/journal.pcbi.0010024.
[11] CAUCHIE E, DELHALLE L, TAMINIAU B, et al. Assessment of spoilage bacterial communities in food wrap and modified atmospheres-packed minced pork meat samples by 16S rDNA metagenetic analysis[J]. Frontiers in Microbiology, 2020, 10: 3074. DOI:10.3389/fmicb.2019.03074.
[12] LIU J, WANG J, ZHOU Y, et al. Integrated omics analysis reveals differences in gut microbiota and gut-host metabolite profiles between obese and lean chickens[J]. Poultry Science, 2022, 101(11): 102165. DOI:10.1016/j.psj.2022.102165.
[13] FANG S M, CHEN X, PAN J H, et al. Dynamic distribution of gut microbiota in meat rabbits at different growth stages and relationship with average daily gain (ADG)[J]. BMC Microbiology, 2020, 20(1): 116-119. DOI:10.1186/s12866-020-01797-5.
[14] 杨昌林, 刘灿灿. 四川省近年来食品安全状况分析[J]. 食品工程, 2022(2): 23-27. DOI:10.3969/j.issn.1673-6044.2022.02.007.
[15] 国家卫生健康委员会, 国家市场监督管理总局. 食品安全国家标准 食品微生物学检验 肉与肉制品采样和检样处理: GB 4789.17—2024[S]. 北京: 中国标准出版社, 2024.
[16] 国家卫生健康委员会, 国家市场监督管理总局. 食品安全国家标准 食品微生物学检验 菌落总数测定: GB 4789.2—2022[S]. 北京: 中国标准出版社, 2022.
[17] CHEN S F, ZHOU Y Q, CHEN Y, et al. Fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890. DOI:10.1093/bioinformatics/bty560.
[18] PENG Y, LEUNG H C M, YIU S M, et al. IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth[J]. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174.
[19] LANGMEAD B, SALZBERG S L. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923.
[20] LIU Y C, GUO J T, HU G Q, et al. Gene prediction in metagenomic fragments based on the SVM algorithm[J]. BMC Bioinformatics, 2013, 14(Suppl 5): S12. DOI:10.1186/1471-2105-14-S5-S12.
[21] LI W Z, GODZIK A. Cd-hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences[J]. Bioinformatics, 2006, 2(13): 1658-1659. DOI:10.1093/bioinformatics/btl158.
[22] ALTSCHUL S F, MADDEN T L, ZHANG J, et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389.
[23] LI H, HANDSAKER B, WYSOKER A, et al. The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp352.
[24] EDDY S R. A new generation of homology search tools based on probabilistic inference[J]. Genome Informatics, 2009, 23(1): 205-211. DOI:10.1142/9781848165632_0019.
[25] PAL C, BENGTSSON-PALME J, RRENSING C, et al. BacMet: antibacterial biocide and metal resistance genes database[J]. Nucleic Acids Research, 2014, 42: D737-D743. DOI:10.1093/nar/gkt1252.
[26] CHEN L H, YANG J, YU J, et al. VFDB: a reference database for bacterial virulence factors[J]. Nucleic Acids Research, 2005, 33: 325-328. DOI:10.1093/nar/gki008.
[27] LIU B, POP M. ARDB: antibiotic resistance genes database[J]. Nucleic Acids Research, 2009, 37: D443-D447. DOI:10.1093/nar/gkn656.
[28] LEWIS S J, VELASQUEZ A, CUPPETT S L. Effect of electron beam irradiation on poultry meat safety and quality[J]. Poultry Science, 2002, 81(6): 896-903. DOI:10.1093/ps/81.6.896.
[29] BENEDITO J, CAMBERO M I, ORTUNO C. Modeling and optimization of sensory changes and shelf-life in vacuum-packaged cooked ham treated by E-beam irradiation[J]. Radiation Physics and Chemistry, 2011, 80(3): 505-513. DOI:10.1016/j.radphyschem.2010.11.001.
[30] STURDEVANT G L, GARDNER D L, KARI L, et al. Frameshift mutations in a single novel virulence factor alter the in vivo pathogenicity of Chlamydia trachomatis for the female murine genital tract[J]. Infection and Immunity, 2010, 78: 3660-3668. DOI:10.1128/IAI.00386-10.
[31] ROSSI F, RIZZOTTI L, FELIS G E, et al. Horizontal gene transfer among microorganisms in food: current knowledge and future perspective[J]. Food Microbiology, 2014, 42(1): 232-243. DOI:10.1016/j.fm.2014.04.004.
[32] KIM H E, LEE J J, LEE M J, et al. Analysis of microbiome in raw chicken meat from butcher shops and packaged products in South Korea to detect the potential risk of foodborne illness[J]. Food Research International, 2019, 122: 517-527. DOI:10.1016/j.foodres.2019.05.032.
[33] FADLY A M, SMITH E J. Isolation and some characteristics of a subgroup J-like Avian leukosis virus associated with myeloid leukosis in meat-type chickens in the United States[J]. Avian Diseases, 1999, 43(3): 391-400. DOI:10.2307/1592636.
[34] SUNG H W, KIM J H, REDDY S, et al. Isolation of subgroup J Avian leukosis virus in Korea[J]. Journal of Veterinary Science, 2002, 3: 71-74. DOI:10.1093/chromsci/45.1.38.
[35] FOLEY S L, JOHNSON T J, RICHE S C, et al. Salmonella pathogenicity and host adaptation in chicken-associated serovars[J]. Microbiology and Molecular Biology Reviews, 2013, 77(4): 582-607. DOI:10.1128/MMBR.00015-13.
[36] HERMANS D, VAN DEUN K, MESSENS W, et al. Campylobacter control in poultry by current intervention measures ineffective: urgent need for intensified fundamental research[J]. Veterinary Microbiology, 2011, 152: 3-4. DOI:10.1016/j.vetmic.2011.03.010.
[37] KIM J J, SEO K W, MO I P, et al. Genetic characterization of fluoroquinolone resistance in Salmonella enterica Serovar Gallinarum isolates from Chicken in Korea[J]. Avian Diseases, 2019, 63(4): 584-590. DOI:10.1637/aviandiseases-D-19-00095.
[38] RAMIREZ M S, BONOMO R A, TOLMASKY M E. Carbapenemases: transforming Acinetobacter baumannii into a yet more dangerous menace[J]. Biomolecules, 2020, 10(5): 720. DOI:10.3390/biom10050720.
[39] HARDING C M, HENNON S W, FELDMAN M F. Uncovering the mechanisms of Acinetobacter baumannii virulence[J]. Nature Reviews Microbiology, 2018, 16(2): 91-102. DOI:10.1038/nrmicro.2017.148.
[40] WANG G Y, ZHAO G, CHAO X Y, et al. The characteristic of virulence, biofilm and antibiotic resistance of Klebsiella pneumoniae[J]. International Journal of Environmental Research and Public Health, 2020, 17(17): 6278. DOI:10.3390/ijerph17176278.
[41] KHAMIS F, AL-ZAKWANI I, MOLAI M, et al. Demographic, clinical, and outcome characteristics of carbapenem-resistant Enterobacteriaceae over a 10-year period (2010—2020) in Oman[J]. International Journal of Infectious Diseases, 2022, 4: 165-170. DOI:10.1016/j.ijregi.2022.08.001.
[42] POSSEBON F S, TIBA CASAS M R, NERO L A, et al. Prevalence, antibiotic resistance, PFGE and MLST characterization of Salmonella in swine mesenteric lymph nodes[J]. Preventive Veterinary Medicine, 2020, 179: 105024. DOI:10.1016/j.prevetmed.2020.105024.
[43] MORADALI M F, GHODS S, REHM B H. Pseudomonas aeruginosa lifestyle: a paradigm for adaptation, survival, and persistence[J]. Frontiers in Cellular & Infection Microbiology, 2017, 7: 39. DOI:10.3389/fcimb.2017.00039.
收稿日期:2024-07-13
基金项目:湖南省自然科学基金项目(2022JJ90028)
第一作者简介:宋晟(1979—)(ORCID: 0009-0002-1349-572X),男,高级工程师,硕士,研究方向为食品安全与食品
微生物学。E-mail: 55074740@qq.com
*通信作者简介:张继红(1967—)(ORCID: 0000-0003-3162-2235),女,教授级高级工程师,学士,研究方向为食品安全与食品安全风险预警。E-mail: 383302005@qq.com
