益生菌防控单核细胞增生李斯特氏菌生物膜形成的机制及应用研究进展
2024-11-05武桐煊杨慧轩胡安祺刘昀阁张一敏
摘 要:单核细胞增生李斯特氏菌是一种具有高度环境适应性的食源性致病菌,生物膜的形成是导致其在食品工业中持久性存留、食品污染和疾病传播的重要原因。益生菌作为一种有益宿主健康的微生物,同时也对单核细胞增生李斯特氏菌有较强的抑制和清除作用,逐渐被开发为一种生物保护策略应用于单核细胞增生李斯特氏菌生物膜的防控。然而,由于单核细胞增生李斯特氏菌生物膜形成受多种因素调控,这种基于益生菌的生物保护策略应用及抑制生物膜机理尚未得到完全阐明,缺乏全面、系统的总结。因此,本文基于单核细胞增生李斯特氏菌的群体感应、泳动能力、疏水性、自聚集性、胞外聚合物生成等生物膜形成途径,详细总结益生菌保护策略在不同加工表面防控单核细胞增生李斯特氏菌生物膜的研究现状。同时,对单核细胞增生李斯特氏菌在肉品工业中的持久性存留和生物膜向肉品转移问题,以及相对应的生物抑菌策略进行讨论。最后,针对益生菌防控单核细胞增生李斯特氏菌生物膜过程中可能存在的细菌信号交流机制做出展望,以期为在食品工业中设计更安全、有效的生物防护策略提供理论依据和参考。
关键词:益生菌;单核细胞增生李斯特氏菌;生物膜;群体感应;肉品工业
Mechanisms of Action and Application of Probiotics for the Prevention and Control of Listeria monocytogenes Biofilm Formation: An Update
WU Tongxuan, YANG Huixuan, HU Anqi, LIU Yunge*, ZHANG Yimin*
(College of Food Science and Engineering, Shandong Agricultural University, Tai’an 271018, China)
Abstract: Listeria monocytogenes is a foodborne pathogen with high environmental adaptability, and the formation of its biofilm is an important cause of its persistent residues in the food industry leading to food contamination and disease transmission. Probiotics, as microorganisms that are beneficial to host health and have a strong ability to inhibit and eliminate L. monocytogenes, are gradually being developed as a bioprotective strategy against L. monocytogenes biofilm. However, since L. monocytogenes biofilm formation is regulated by multiple factors, the inhibitory mechanism of probiotic-based bioprotective strategies against L. monocytogenes biofilm has not been fully clarified and a comprehensive and systematical review of related studies is lacking. Therefore, the current status of research on probiotic-based bioprotective strategies against the biofilm formation of L. monocytogenes on different processing surfaces is reviewed with respect to quorum sensing, swimming motility, hydrophobicity and auto-aggregation, and extracellular polymer production. Meanwhile, the problems of persistent retention of L. monocytogenes and transfer of its biofilm to meat in the meat industry are discussed as well as the biocontrol strategies for solving these problems. Finally, an outlook on the possible mechanism of bacterial signal communication involved in L. monocytogenes biofilm control by probiotics is given with a view to providing a theoretical basis for designing safer and more effective bioprotective strategies in the food industry.
Keywords: probiotics; Listeria monocytogenes; biofilm; quorum sensing; meat industry
DOI:10.7506/rlyj1001-8123-20240619-157
中图分类号:TS251.5 文献标志码:A 文章编号:1001-8123(2024)11-0064-10
引文格式:
武桐煊, 杨慧轩, 胡安祺, 等. 益生菌防控单核细胞增生李斯特氏菌生物膜形成的机制及应用研究进展[J]. 肉类研究, 2024, 38(11): 64-73. DOI:10.7506/rlyj1001-8123-20240619-157. http://www.rlyj.net.cn
WU Tongxuan, YANG Huixuan, HU Anqi, et al. Mechanisms of action and application of probiotics for the prevention and control of Listeria monocytogenes biofilm formation: an update[J]. Meat Research, 2024, 38(11): 64-73. DOI:10.7506/rlyj1001-8123-20240619-157. http://www.rlyj.net.cn
单核细胞增生李斯特氏菌(Listeria monocytogenes)(下文简称单增李斯特菌)是世界卫生组织公认的四大食源性致病菌之一,具有抗逆性强、传播速度快等特点,人类感染后可导致败血症、脑膜炎和肠胃炎等症状,特别是新生儿、孕妇、老人及免疫功能低下的个体,有20%~30%的致死风险[1]。其中,食品是单增李斯特菌的主要传播媒介之一[2],根据2023年欧盟食品安全局报道,即食食品中,水产品、肉及肉制品、乳及乳制品以及水果蔬菜和果汁的总体检出率分别为7.1%、2.1%、0.37%和2.6%[3]。这主要是由于单增李斯特菌具有很强的生物膜形成能力,其可以附着在食品加工表面,抵抗胁迫环境,并造成持续的污染。Li Xin等[4]的一项针对中国吉林省肉类零售市场和屠宰场的调研发现,所有分离株都含有生物膜相关基因,有助于单增李斯特菌在恶劣的环境条件下长期生存,增强其致病性和持久性。
单增李斯特菌通过分泌胞外聚合物(extracellular polymeric substances,EPS)形成一定的空间结构,进而形成生物膜[5-6],将其自身包绕其中并形成大量细菌聚集体,细菌群落可以通过种间相互作用抵抗外界胁迫环境,从而产生耐药性,有利于单增李斯特菌长期存活,促进其繁殖和传播,加剧食品污染[7]。然而,单纯物理或机械方法并不能完全减少或消除生物膜的黏附,而与抗生素协同的抑菌策略实施则需要考虑抗生素的耐药性;化学方法的功效则受限于所用杀菌剂的类型和浓度、接触时间、pH值以及生物活性的类型和水平等因素,且部分存在较严重的副作用,如氯化消毒剂能够腐蚀金属表面,增加癌症风险,其他方法如酶去除生物膜等也由于生产成本较高而受到限制。近年来,基于微生物保护策略,利用益生菌抑制单增李斯特菌生物膜形成受到关注,这种生物抑菌技术具备天然性、益生性、生产周期短且抑菌途径广泛等优势,具有较大的生物膜
抑制潜力[8]。
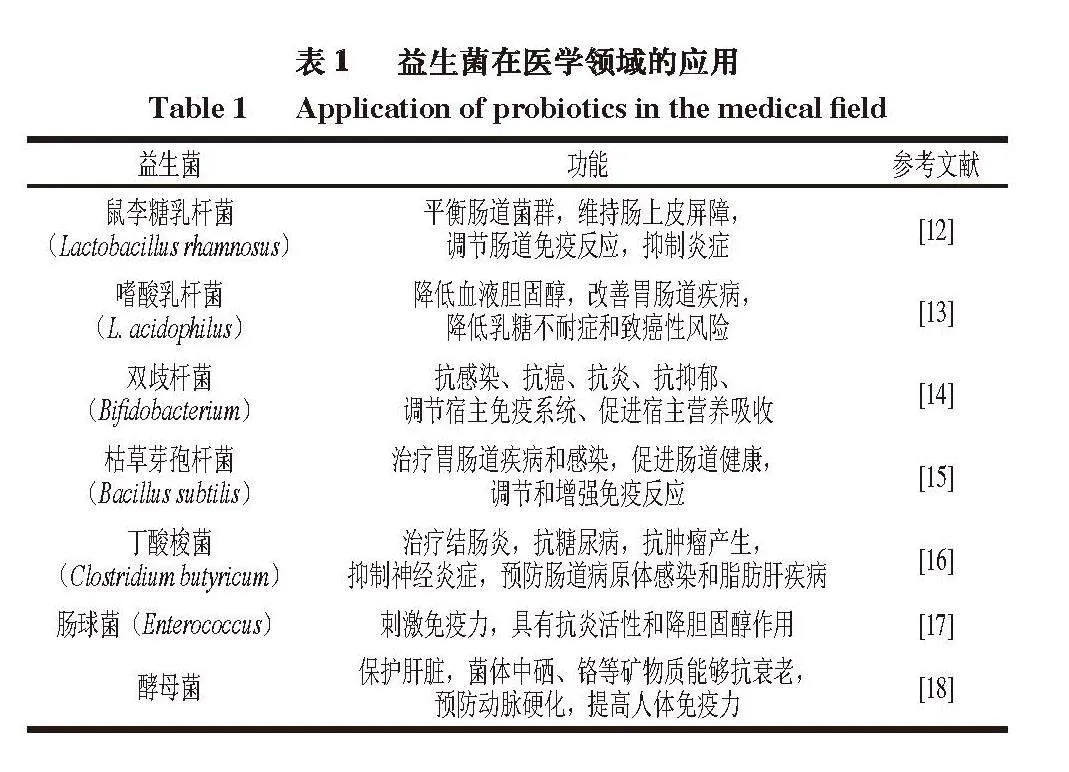
联合国粮农组织和世界卫生组织将益生菌定义为:给予一定数量、能对宿主健康产生有益作用的活微生
物[9]。这些微生物具备调节机体肠道微生态平衡、心血管健康和提高免疫力等生理功能,在医学领域已有广泛应用(表1)。近年来,随着人们对于生物膜细胞持久性存留问题的关注,大量研究学者基于益生菌生物保护策略,探究对单增李斯特菌生物膜形成的抑制作用,并在不同非生物表面如聚苯乙烯、不锈钢和玻璃上均取得显著的生物清除效果。然而,生物膜具有复杂的形成机制,其过程受到多种因素调节,这使益生菌对单增李斯特菌的生物防控机制尚未得到系统阐明。首先,鞭毛介导的细菌运动有助于单增李斯特菌附着在表面和随后的生物膜形成[10]。单增李斯特菌一旦接触非生物表面,即通过信使分子环二聚鸟苷单磷酸开始转向基质生产[11]。同时,群体感应系统和介导一般应激反应的σ因子SigB也被证明在单增李斯特菌生物膜形成过程中被激活,并赋予生物膜细胞对抗菌剂更强的耐药性。综上所述,本文重点基于单增李斯特菌生物膜形成的主要机制,对益生菌防控单增李斯特菌生物膜形成的应用研究进行系统综述,以期为基于益生菌的生物保护策略的发展提供理论依据。
1 益生菌的主要抑菌途径及应用方式
益生菌能够在生长过程中或裂解后分泌可溶性物质,具有多种功能活性。其中细菌素[19]、有机酸[20]、过氧化氢[21]和脂肪酸[22]等具有广泛的抗菌活性。乳酸菌和枯草芽孢杆菌产生的细菌素是抑制病原菌的主要途径之一。细菌素能干扰细菌细胞壁的合成,并在细胞膜表面形成孔隙,导致膜电位耗散和内容物流出,最终使细胞死亡[23]。乳酸菌还能产生乳酸、乙酸、甲酸、琥珀酸、柠檬酸和丁酸等有机酸,其在细胞外通过解聚细菌外膜组分导致细胞外膜通透性增加,抑制病原菌生长。有机酸在胞内则解离出质子和酸根离子,导致病原体向胞外泵出质子,造成其额外能量消耗;留在胞内的酸根离子则能够抑制病原菌遗传信息传递和破坏胞内渗透压平衡[24]。
此外,益生菌和致病菌具有相似的碳水化合物利用底物时,二者对同一营养素生态位存在竞争效应,其通过与病原菌竞争营养物质和黏附位点限制定植[25]。
尽管这些抑菌机制被证明具有有效的抑菌活性,然而在食品中实际应用时,无论是活的益生菌还是其分离纯化的抗微生物组分被掺入食品基质中都有一定的局限性[26]。例如,细菌的生长和持久存活受外部参数控制,如食物类型、包装方式、温度和pH值等,并且纯化的细菌素产量低,生产成本相对较高,通常仅对相近亲缘菌株有效[27]。随着人们对益生菌研究的不断深入,学者们发现益生菌的功能活性不完全来自活体益生菌,一些细菌的细胞膜、细胞壁及代谢物同样具有相同或类似的功能活性,这些“对宿主健康有益的无生命微生物和/或其成分的制剂”在益生菌研究领域也被称为后
生元[28]。后生元具备安全、易用、贮藏性好、在广泛
pH值和温度范围内具有稳定性及广谱抗菌活性等特点。目前,对其制备和分析的方法较为普遍的是通过离心制取其无细胞上清液(cell-free supernatant,CFS)[29],因此广泛的研究在亚抑菌浓度,即低于最小抑菌浓度(minimal inhibit concentration,MIC)下以CFS的形式进行抑菌应用,这几乎能够涵盖所有的益生菌化合物,可以满足消费者的需求并确保产品的安全性。此外,它们还有助于延长保质期,控制腐败和病原微生物的生长,不受耐药性的限制,并为食品提供更好的感官特性,具有作为食品添加剂的潜力[30]。这些代谢产物还可以生物表面活性剂的方式应用,其助溶性能强,能快速穿透生物膜结构,具有毒性低、生物兼容性好和可降解等优点[31];
或与纳米材料结合,制成抗菌纳米膜以适应多种食品加工用途并增强抗菌潜力[32]。
2 益生菌抑制单增李斯特菌生物膜形成的机制及应用
2.1 群体感应系统
群体感应是细菌通过感受胞外信号调控基因表达的一种群体密度依赖性胞间信号转导系统[33-34]。单增李斯特菌自身主要存在2 种群体感应调控系统,分别是自诱导剂介导的LuxS/AI-2和Agr/AIP系统,可调控生物膜形成、毒力和侵袭性等生理特性[35]。首先,益生菌CFS具备干扰信号分子AI-2产生的潜力。Wei Jinyuan等[36]基于植物乳植杆菌Z102的CFS,使用乙酸乙酯等比混合萃取得到乳酸菌乙酸乙酯萃取物,发现经过其不同质量浓度萃取物处理后,单增李斯特菌产AI-2信号分子数量受到抑制,萃取物质量浓度与生物膜清除率表现出量效关系,质量浓度为3.2、1.6 mg/mL的萃取物在24 h时的生物膜清除率分别为64.97%、54.78%;并且随着抑菌时间延长,在48 h时的生物被膜清除率分别可达到65.97%、56.38%。这些萃取物能有效清除单增李斯特菌预成型的生物被膜,且清除率与萃取物的质量浓度呈正相关性。类似的研究中,黄湘湄等[37]用同样的方法制得海洋源乳酸菌乙酸乙酯萃取物,这些萃取物可使信号分子AI-2的产生几乎完全受到抑制,活性仅保留1.5%。并且萃取物对单增李斯特菌生物膜的抑制作用存在浓度依赖性,经125、250、500 μg/mL的乙酸乙酯萃取物作用48 h后单增李斯特菌生物膜的抑制率分别为达到40.85%、66.16%、89.94%。因此,益生菌既能抑制AI-2分子的产生从而减少产量,也可以抑制已产生的信号分子功能使其失活。
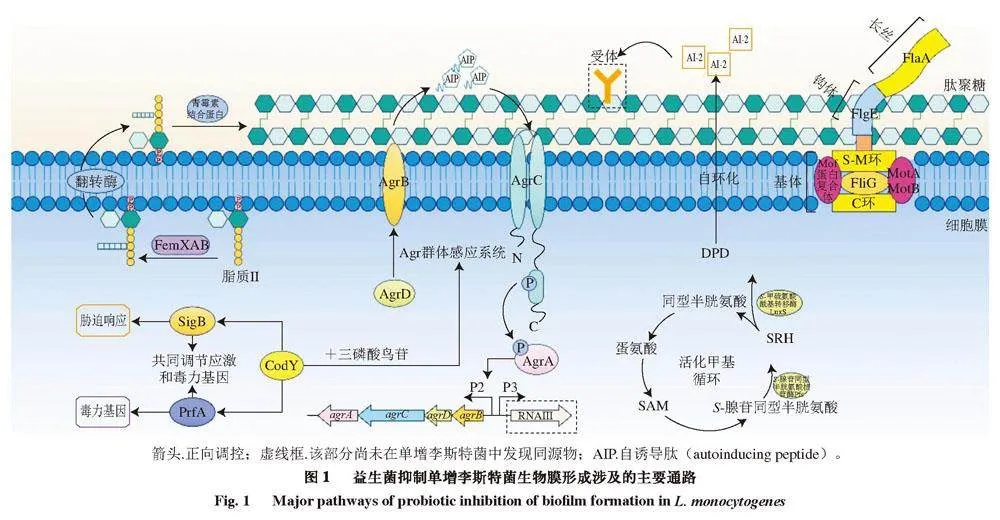
值b51a31f232933c9cc3fa2b008d158d8489e34bbd58d17e99eaf79ff3756ed5f5得注意的是,Rezzonico等[38]研究表明,事实上在单增李斯特菌中尚未发现已知的AI-2同源8aed03c913a47ac8ff9f428d52b5eaab73450ecfcae02b695c621ad0617ee7ba受体,因此对于益生菌影响AI-2信号分子从而抑制单增李斯特菌生物被膜形成机制,以及单增李斯特菌产生的AI-2信号分子在单增李斯特菌中的功能尚不明确。AI-2的前体物质是4,5-二羟基-2,3-戊二酮(4,5-dihydroxy-2,3-pentanedion,DPD),DPD是细胞甲基化供体S-腺苷甲硫氨酸(S-adenosylhomocysteine,SAM)的副产物(图1)[39]。
Belval等[40]在luxS基因突变株中加入S-核糖同型半胱氨酸(S-ribosylhomocysteine,SRH)和AI-2后,观察发现外源添加的AI-2对该突变株的生物膜形成没有影响,而加入SRH使生物膜更加致密并且增加了附着细胞的数量。因此,后续的研究应对益生菌CFS在LuxS/AI-2系统中的关键作用靶点及单增李斯特菌AI-2信号分子的调控作用做进一步探究。
相较于LuxS/AI-2系统,Agr群体感应系统已被广泛证实其在单增李斯特菌中具有调节生物被膜初始黏附的作用,因此基于该系统抑制生物膜形成的研究较为广泛。Agr群体感应系统由4 个基因操纵子agrB、agrD、agrC、agrA组成,在agr操纵子编码的4 种蛋白质中,AgrB是一种膜结合肽酶,可切割和加工AgrD衍生的前肽,影响AIP的输出和释放[41-42]。信号分子AIP在细胞外空间中积累后,由受体组氨酸激酶AgrC接受信号,磷酸化反应调节因子AgrA进行下游基因调控[43]。益生菌可以通过降低Agr群体感应系统中信号分子AIP产生和信号接收相关的基因表达量从而抑制生物膜的形成。例如,枯草芽孢杆菌KU43细菌素能使agrB和agrD的转录水平分别下调为原来的36.6%和31.6%,减弱信号分子的加工及跨膜蛋白转运能力[44]。Qiao Zhu等[45]研究中使用甲壳乳杆菌MN 047的CFS处理后则影响了反应调节因子AgrA和组氨酸激酶AgrC组成的双组分信号调控系统,从而干扰群体感应系统的信号传递。此外,Marques等[46]对益生菌的分泌蛋白研究发现,益生菌的丝氨酸型D-Ala-D-Ala羧肽酶和L,D-转肽酶能分别与单增李斯特菌膜上蛋白AgrB和AgrC的细胞外部分结合,这可以阻止环状AIP的加工和易位出细胞及信号的接收,导致非磷酸化的AgrA不能触发靶基因的表达,从而阻止信号转导的发生。
2.2 泳动能力
细菌的泳动能力主要依靠鞭毛相关基因介导,主要分为单一泳动能力和群集泳动能力[47],细菌组成聚集细胞团在基质表面运动,有利于生物被膜的形成[48]。因此,益生菌也可以通过控制鞭毛基因的表达,从而影响鞭毛介导的泳动能力,以此干扰细菌向非生物表面运动和初步附着。Park等[44]发现,枯草芽孢杆菌KU43的CFS能够降低影响构成鞭毛马达/开关组件蛋白FliG[49]和鞭毛钩蛋白FlgE的合成,限制其泳动扩散,显著抑制单增李斯特菌生物膜形成。Hossain等[20]研究表明,1/2 MIC的乳杆菌后生元能够明显下调编码鞭毛丝蛋白基因flaA,使其表达量降低至约为原来的40%,使泳动能力减弱,干扰生物膜形成前期鞭毛泳动。除上述类型外,益生菌还可以改变鞭毛转录调节因子,从而干扰鞭毛表达的过程。在单增李斯特菌中,鞭毛表达由运动基因阻遏蛋白MogR和抗阻遏蛋白GmaR以温度依赖性方式进行转录协调。GmaR蛋白在低于30 ℃的温度下与MogR中和,单增李斯特菌表达鞭毛蛋白并通过基于鞭毛的运动在宿主外移动[50]。DegU是低温下鞭毛表达所必需的调节因子,其通过识别FliN-GmaR启动子中的操作位点转录促进GmaR表达,还可以增强鞭毛基因的转录,是鞭毛运动的正调节因子[51]。Qiao Zhu等[45]研究发现,甲壳乳杆菌MN 047细菌素在亚抑菌浓度下可使单增李斯特菌中degU的表达量下调,影响鞭毛表达过程中的调控因子,减弱鞭毛介导的泳动能力。
2.3 疏水性和自聚集性
细菌表面疏水性是指细菌在极性水中所呈现的不稳定状态引起的一系列菌体的重新分布及排列的变化,与菌体对环境的附着力有关,因此可能会影响菌株的初始黏附和生物被膜形成。而自动聚集能力是指菌体在培养过程中大量并快速繁殖,从而导致其自行聚集成团的现象,是细菌在非生物表面沉积和微菌落形成的重要机制,有助于菌体初始黏附和EPS分泌[52-53]。研究[44,54-55]证实,益生菌如酿酒酵母和枯草芽孢杆菌的CFS可以降低疏水性和自聚集能力,减弱细菌黏附表面的能力,抑制单增李斯特菌生物膜形成的初始阶段。胡安祺等[56]研究表明,枯草芽孢杆菌CFS对单增李斯特菌的细胞疏水性和自聚集性具有显著抑制作用,抑制率最高分别为75%和65%左右。枯草芽孢杆菌CFS即使在1.562 5 mg/mL(1/64 MIC)下仍对疏水性具有较强的抑制效果。同样地,自聚集性和疏水性也与益生菌的定殖和黏附能力有关[57]。Lee等[21]研究中的乳杆菌KU200656同样具有较好的细胞表面疏水性和自聚集性,因此具有优越的黏附能力,从而加以利用竞争营养和宿主肠道细胞结合位点来抑制病原体的黏附。益生菌能够利用细菌之间的自聚集能力,可在浮游状态下与单增李斯特菌结合成共聚集体,随后抑制单增李斯特菌的定植和生物膜形成,但是应用益生菌与单增李斯特菌的共聚集体阻碍单增李斯特菌生物膜形成时,要考虑两者之间的种间相互作用及对其毒力的影响,并且共聚集体是否易被常规手段清除需要进一步探索研究,以选择合适的共聚体组合。
2.4 EPS
EPS是微生物分泌出的一种复杂的大分子聚合物,主要成分为多糖和蛋白,还包含少量的脂肪酸、核酸、腐植酸、氨基酸和其他分子[58]。生物膜形成的早期阶段涉及EPS的合成,其促进细菌黏附于表面和细胞间黏附,聚集能够导致不可逆结合的稳定结构形成,在定植和生物膜成熟期间也有助于保持生物膜的结构稳定性。Qiao Zhu等[45]发现,亚抑菌浓度的甲壳乳杆菌MN 047细菌素可以显著抑制胞外多糖的产生,减少不可逆黏附,控制单增李斯特菌早期生物膜形成。胞外多糖作为一种强大而具有黏性的框架是EPS聚集的关键物质。Zhuang Zheng等[59]研究表明,胞外多糖过表达突变株生物膜比对照菌株含有更多的EPS,即形成更厚、更致密的生物膜,并且胞外多糖的增加也增强了生物膜中的整体细胞活力。同时在该研究中,多糖生物合成基因操纵子相关基因过表达突变株的氧化还原活性电位范围更宽,有助于增强电流的产生,利于信号交流。
EPS的生产与肽聚糖的合成密切相关,很多关于肽聚糖合成编码基因突变的研究(基因pbp1a、lafB和dltD等)[60-61]和小分子肽聚糖组装的干扰(基因pbp4等)[62-63]证实了肽聚糖合成在生物膜发育中的重要作用。因此,益生菌对于肽聚糖合成途径相关基因的阻碍对减少生物膜的形成非常重要。Park[44]、Kim[55]等分别发现酿酒酵母菌株及枯草芽孢杆菌KU43和KU201的CFS可下调与EPS产生有关的基因dltB[64-65],降低EPS产量,减弱成熟生物膜和细胞间相互作用,抑制单增李斯特菌生物膜扩展。Jara等[66]还利用乳杆菌生物膜作为表面“调理剂”来调节单增李斯特菌生物膜的黏附。该研究发现,发酵乳杆菌MP26和唾液乳杆菌MP14形成的双物种生物膜虽然不能降低单增李斯特菌的黏附力,但物种相互作用似乎干扰了EPS的合成和生物膜内的物种分布。为使用乳酸菌属生物膜作为“天然”固定方式来控制单增李斯特菌的传播开辟了新的可能性。此外,最近的研究还发现群体感应系统与EPS产生之间可能存在调节关系。Lee等[67]发现,参与活化甲基循环的SAM和基于Agr群体感应的信号可能在单增李斯特菌中同步调节营养物质可用性、EPS的合成和生物膜形成,而且这些调控联系在单增李斯特菌的特定生长模式中被激活。在营养丰富、鸟苷三磷酸和SAM浓度较高的条件下,SAM及代谢调节因子CodY的激活均使Agr群体感应系统的操纵子基因表达上调,并且SAM的增加还能诱导murE和pbpA1的转录以合成肽聚糖。单增李斯特菌代谢和群体感应系统上的相互联系被不断揭示,也为生物膜抑制研究提供了新的关键作用位点。
2.5 包膜结构
由于益生菌CFS中主要抑菌物质为细菌素,因此即便采用亚抑菌浓度,也能够改变单增李斯特菌细胞膜结构,降低单增李斯特菌活力,减弱其代谢水平甚至杀死生物膜中的部分细菌个体。Behbahani等[54]使用枯草芽孢杆菌CFS处理单增李斯特菌生物膜细胞后发现,扫描电子显微镜明显观测到生物膜细胞出现细胞壁褶皱、凹陷甚至破裂等形态变化。同样,枯草杆菌素JS-4通过促进膜孔的形成、空泡化和细胞质流出,从而增加渗透性并最终导致膜完整性的丧失。细胞膜的渗透压降低同时会导致K+、核酸和蛋白质流出,这可以破坏渗透平衡以及代谢、复制、转录和翻译等关键过程[68]。针对单增李斯特菌生物膜结构,搭载细菌素的生物表面活性剂被广泛开发,其具有良好的乳化、分散、增溶等特性,更有利于快速渗透生物膜。de Araujo等[69]研究表明,基于枯草芽孢杆菌ATCC 21332细菌素的生物表面活性剂具有很好的两亲性,可以与磷脂相互作用,有干扰细胞质膜的作用,并改变单增李斯特菌的渗透性,从而导致细胞损伤。Mendoza等[70]研究表明,类似表面活性剂乳化程度可达到80%以上,能在生物膜外稳定和均匀存在,促进聚苯乙烯表面生物膜的透化或溶解,并且在香肠生产系统中具有良好的应用效果。
2.6 生物膜细胞毒力
益生菌在有效防控生物膜形成的同时还可进一步降低生物膜内残存细胞的毒力基因表达,降低单增李斯特菌生物膜细胞的潜在风险。细菌表面蛋白ActA是单增李斯特菌最关键、最具特征的毒力因子之一[71],ActA锚定在细菌膜或线粒体膜上,诱导肌动蛋白细胞骨架的形成,介导肌动蛋白聚合驱动的细菌运动[72],如侵袭宿主细胞和免疫逃逸等。枯草芽孢杆菌KU43和KU201的CFS[44]能够下调基因actA,干扰肌动蛋白聚合,使单增李斯特菌毒力下降。基因prfA编码其他毒力基因转录激活因子,是单增李斯特菌跨越胃肠道屏障、发挥其致病性的关键基因,参与生物膜的形成[73]。研究表明,植物乳杆菌KU200656[21]和嗜酸乳杆菌[74]的CFS通过下调基因prfA,从而减弱单增李斯特菌毒力效应。SigB是单增李斯特菌重要的中枢调控因子,主要负责调控单增李斯特菌在不利环境中的各种抗逆反应,其也参与在胁迫环境下的适应过程并促进生物膜的形成[75-76],而经枯草芽孢杆菌GS3(DS-1)CFS处理后,单增李斯特菌中调节应激反应因子SigB的表达显著降低34.80%[54]。另外,研究[77]表明单增李斯特菌为在不同环境中生存,需要不断重新调节基因表达以适应环境并存活繁殖。其中,PrfA和SigB蛋白起主导作用,代谢调节因子CodY则是SigB和PrfA网络之间的纽带,在养分充足的条件下,CodY抑制sigB的表达,因此在营养胁迫条件下可激活该应激反应调节因子,并且CodY通过直接上调prfA导致毒力基因表达增加,同时也导致sigB表达增加。SigB和PrfA也在多种调控系统的交叉网络中实现微调,为不同微环境提供了适当的基因表达模式,从而形成一个信号转导系统,控制多种基因表达[78]。因此,益生菌对于广泛的毒力基因的抑制,例如磷脂酰胆碱磷脂酶编码基因plcB[79]、溶血素O毒素编码基因hlyA[80]、表面蛋白内化蛋白编码基因inlA、inlB[45]等,可能首先通过抑制基因prfA和sigB从而下调其他毒力基因。
除上述类型外,Menezes等[81]通过观察巴西饮料源酿酒酵母菌株及单增李斯特菌在Caco-2细胞表面上的结合发现,该菌株与单增李斯特菌竞争黏附位点,使单增李斯特菌黏附和感染的几率下降。表明益生菌还可以与单增李斯特菌竞争黏附位点,影响单增李斯特菌早期的黏附与定植,从而削弱单增李斯特菌黏附能力,降低单增李斯特菌侵袭效率[82]。Lee等[21]以ht-29细胞为依附介质的研究也同样佐证乳杆菌KU200656可附着在肠道上与单增李斯特菌竞争营养和结合位点,并且延长停留在肠道中与宿主相互作用的时间,防止单增李斯特菌感染,从而减轻毒力作用。
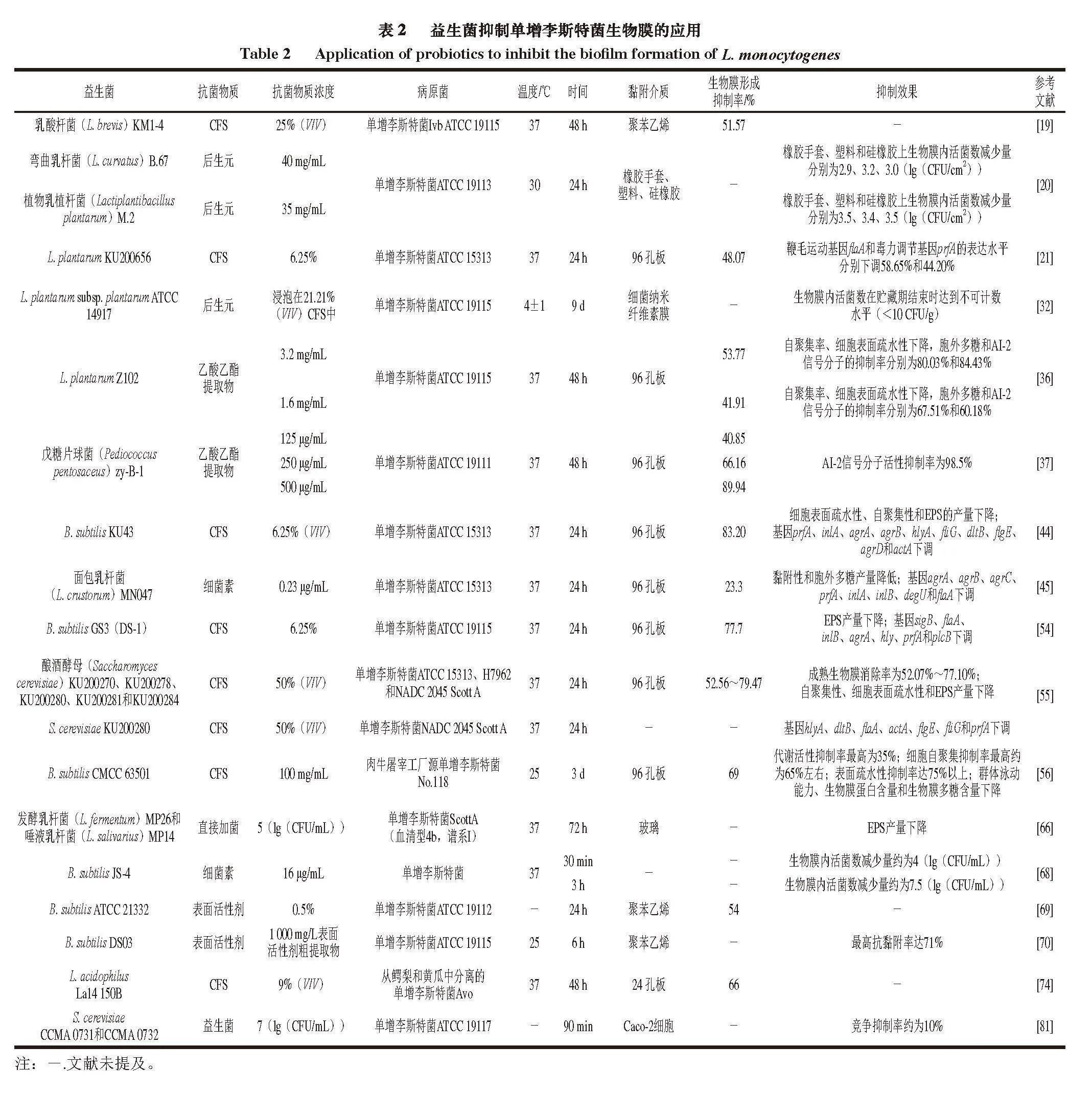
益生菌抑制单增李斯特菌生物膜的应用如表2所示。
3 单增李斯特菌在肉品工业中持久性存留及生物防控对策
肉制品中丰富的营养物质为单增李斯特菌的持久性存留提供了十分有利的条件。Fagerlund等[83]调研挪威多家肉制品加工厂后发现,生产线开始生产后的1 周内,在不同类型的切片熟食肉产品、产品残留物及与切片机生产线相关的加工设备中均能检测到单增李斯特菌。并且,同一肉类加工厂中在数年(4~9 年)的时间里检测到相同的分离株。细菌在浮游状态很容易被工厂的清洁环节清除,而一旦形成生物膜,其在黏附状态下则会变得难以被完全清除,造成持久性存留问题。汪忆梦等[84]
随机采集中国山东省潍坊市市售肉及肉制品,进行单增李斯特菌的分离鉴定后发现单增李斯特菌总检出率高达18.82%。在肉及肉制品加工过程中,由于加工流程的复杂性和连续性,容易引发单增李斯特菌的交叉污染。Schäfer等[85]发现,冷却之后禽体样本初始仅有5%的样本受到单增李斯特菌的污染,经过工厂自动化切割环节,鸡胸肉和鸡大腿在5 h后污染率分别高达25%和70%。在肉制品深加工过程中,食物和设备之间产生的交叉污染现象最严重。Alía等[86]对干腌火腿去骨、压缩和切片过程中单增李斯特菌的多样性进行调查,结果显示,在720 个样本中,干腌火腿去骨和切片区域中有66 个样本(9.16%)被鉴定为含有单增李斯特菌,复杂的加工生产线更容易引起交叉污染。有充足的证据表明,单增李斯特菌能在众多肉制品加工环境材料如不锈钢、橡胶[20]、玻璃[66]、塑料[69]等表面形成生物膜[87]。从非生物表面转移到食物的微生物数量取决于生物膜的特性,包括微生物群的表面密度、生物膜的结构、其产生胞外多糖的能力及微生物细胞的附着强度等[88]。如Jeon等[89]对单增李斯特菌在不同食物接触表面上生物膜的形成以及向烘烤鸭肉和水煮鸭肉的转移能力进行研究,结果表明,单增李斯特菌生物膜从聚丙烯接触面上到2 种鸭肉的转移率最高,不锈钢最低,原因与不同接触面上的黏附能力相关,其次在所有接触面上,生物膜到烘烤鸭肉的转移率较高,也说明生物膜的转移受食物水分含量的影响。生物膜的产生会使食品的污染水平提升,生物膜的转移也受到接触面类型及食物本身性质的影响,极强的生存适应能力可能是生物膜在各场所中导致交叉污染的重
要原因。
在肉及肉制品中,乳酸菌作为主要的益生菌对单增李斯特菌的防治起到了重要作用。首先,在不同的肉制品加工介质表面,乳酸菌已被证实可以有效抑制单增李斯特菌,可以阻止其表面生物膜的初步黏附。例如,Hossain等[20]分别在橡胶手套、塑料、硅橡胶上实验证实,乳酸菌在3 种介质中均有抑制单增李斯特菌的效果。Dinçer[19]通过研究乳酸菌对食品接触表面聚苯乙烯上的单增李斯特菌抑制效果发现,单增李斯特菌的生物膜抑制率高达51.57%。益生菌在防控单增李斯特菌生物膜方面具有很大的潜力。对于肉制品而言,益生菌也可继续作为产品的栅栏因子提供对单增李斯特菌的防控作用。Ravyts等[90]发现添加具有产细菌素特性的清酒乳杆菌能够有效控制发酵香肠中单增李斯特菌的生长,比利时香肠和意大利腊肠被人工污染约3.5(lg(CFU/g))的单增李斯特菌,而在生产过程完成后,单增李斯特菌分别减少1.4、0.6(lg(CFU/g))。此外,纯化的益生菌代谢产物在包装材料中也具有广泛的应用前景,特别是对于肉及肉制品。在智能包装系统中乳酸菌产生的细菌素主要针对革兰氏阳性菌,其中乳酸链球菌素是唯一被美国、澳大利亚等国家批准作为肉制品添加剂的细菌素,现今已经基于乳酸链球菌素和其他药物的组合开发了多种肉类包装,以延缓或抑制单增李斯特菌的生长[91],如利用乳酸链球菌素和山梨酸钾的协同作用制造的木薯淀粉-羟丙基甲基纤维素薄膜包装[92],以及使用含有乳酸链球菌素和乳酸钾的交织剂对煮熟、切片的火腿进行真空包装等[93]。此外,还有学者关注乳酸链球菌素与合成聚合物(聚乙烯、聚丙烯、聚氯乙烯等)组合的包装材料,为了使特定的抗菌化合物实施过程可控,一般将合成聚合物作为涂层涂覆,从而避免高温或剪切力的影响。合成聚合物涂层可以作为抗菌化合物的载体,将抗菌化合物快速、均匀地扩散到食品表面,其比浸渍和喷洒抗菌化合物更具优势,能够高效抑制单增李斯特菌[94]。
4 结 语
随着我国对食源性疾病监测体系的不断完善,食源性致病菌在食品工业中持久性存留被广泛关注。单增李斯特菌在食品加工领域通过基因分型等方法被广泛检出,其中生物膜形成能力是其自我防御的重要机制,使其难以被去除。基于益生菌的生物保护策略在应对生物膜形成方面显示了巨大潜力,其在多种食品加工表面如塑料、不锈钢和橡胶等都展现了良好的生物膜抑制效果,并具备多种应用形式适应食品加工过程。益生菌及其代谢产物对单增李斯特菌群体感应系统、泳动和自聚集能力的干扰及黏附位点的竞争,影响了其在非生物表面的初始黏附过程。同时这种生物保护策略还进一步抑制了已发生黏附细胞的EPS产生和毒力因子的表达,降低了残存菌株的食品危害风险。在肉类工业中,单增李斯特菌存在持久的留存问题,并可能在加工过程中导致进一步的交叉污染。许多益生菌防控生物膜的研究表明,这种生物抑制策略在单增李斯特菌于非生物表面黏附期间或在肉制品中均展现了很好的应用潜力,并在肉制品包装中也具备多种应用形式。然而值得注意的是,益生菌在应用期间会不可避免地与单增李斯特菌细胞接触,其中LuxS/AI-2种间交流系统中部分组件尚未在单增李斯特菌中发现同源物,该系统的精确调控机制仍不明确。因此,在直接应用益生菌过程中,其与单增李斯特菌形成的共聚集体之间是否存在信号交流,是否存在双物种生物膜形成的风险,均有待进一步阐明。单种和多种细菌信号交流机制的探究将是未来重要的待解决问题,这对更好地设计生物保护策略、降低生物膜带来的食品安全风险至关重要。
参考文献:
[1] LIU Y T, SUN W X, SUN T M, et al. The prevalence of Listeria monocytogenes in meat products in China: a systematic literature review and novel meta-analysis approach[J]. International Journal of Food Microbiology, 2020, 312: 108358. DOI:10.1016/j.ijfoodmicro.2019.108358.
[2] FAN Z L, XIE Jing, LI Y, et al. Listeriosis in mainland China: a systematic review[J]. International Journal of Infectious Diseases, 2019, 81: 17-24. DOI:10.1016/j.ijid.2019.01.007.
[3] European Food Safety Authority. The European union one health 2022 zoonoses report[J]. European Food Safety Authority Journal, 2023, 21(12): e8442. DOI:10.2903/j.efsa.2023.8442.
[4] LI X, SHI X N, SONG Y, et al. Genetic diversity, antibiotic resistance, and virulence profiles of Listeria monocytogenes from retail meat and meat processing[J]. Food Research International, 2022, 162: 112040. DOI:10.1016/j.foodres.2022.112040.
[5] ABDALLAH M, BENOLIEL C, DRIDER D, et al. Biofilm formation and persistence on abiotic surfaces in the context of food and medical environments[J]. Archives of Microbiology, 2014, 196(7): 453-472. DOI:10.1007/s00203-014-0983-1.
[6] FAN Y, QIAO J J, LU Z X, et al. Influenccdaadbfdc9c5f4c08fc7ca435c0d52c7e of different factors on biofilm formation of Listeria monocytogenes and the regulation of cheY gene[J]. Food Research International, 2020, 137: 109405. DOI:10.1016/j.foodres.2020.109405.
[7] 李虎良, 张蕾. 抗生素耐药性的分子机制及抑菌策略[J]. 中国生物化学与分子生物学报, 2024, 40(6): 759-769. DOI:10.13865/j.cnki.cjbmb.2024.01.1365.
[8] OLOKETUYI S F, KHAN F. Inhibition strategies of Listeria monocytogenes biofilms-current knowledge and future outlooks[J]. Journal of Basic Microbiology, 2017, 57(9): 728-743. DOI:10.1002/jobm.201700071.
[9] 董娜, 张晓, 田彦辉, 等. 走近益生菌[J]. 生物学教学, 2013, 38(9): 79. DOI:10.3969/j.issn.1004-7549.2013.09.041.
[10] LEMON K P, HIGGINS D E, KOLTER R. Flagellar motility is critical for Listeria monocytogenes biofilm formation[J]. Journal of Bacteriology, 2007, 189(12): 4418-4424. DOI:10.1128/JB.01967-06.
[11] KOLTER R, GREENBERG E P. The superficial life of microbes[J]. Nature, 2006, 441: 300-302. DOI:10.1038/441300a.
[12] 林晨越, 林洁楠, 文刚, 等. 鼠李糖乳杆菌对新生儿坏死性小肠结肠炎保护作用的研究进展[J]. 中国现代医生, 2024, 62(7): 104-107. DOI:10.3969/j.issn.1673-9701.2024.07.025.
[13] GAO H J, LI X, CHEN X T, et al. The functional roles of Lactobacillus acidophilus in different physiological and pathological processes[J]. Journal of Microbiology and Biotechnology, 2022, 32(10): 1226-1233. DOI:10.4014/jmb.2205.05041.
[14] CHEN J, CHEN X Y, HO C L. Recent development of probiotic bifidobacteria for treating human diseases[J]. Frontiers in Bioengineering and Biotechnology, 2021, 9: 770248. DOI:10.3389/fbioe.2021.770248.
[15] AKINSEMOLU A A, ONYEAKA H, ODION S, et al. Exploring Bacillus subtilis: ecology, biotechnological applications, and future prospects[J]. Journal of Basic Microbiology, 2024, 64(6): 614. DOI:10.1002/jobm.202300614.
[16] STOEVA M K, GARCIA-SO J, JUSTICE N, et al. Butyrate-producing human gut symbiont, Clostridium butyricum, and its role in health and disease[J]. Gut Microbes, 2021, 13(1): 1-28. DOI:10.1080/19490976.2021.1907272.
[17] BONDI M, LAUKOVA A, DE NIEDERHAUSERN S, et al. Controversial aspects displayed by enterococci: probiotics or pathogens?[J]. Biomed Research International, 2020, 2020: 9816185. DOI:10.1155/2020/9816185.
[18] 魏长浩, 邓泽元. 益生菌及其应用研究进展[J]. 乳业科学与技术, 2018, 41(1): 26-32. DOI:10.15922/j.cnki.jdst.2018.01.006.
[19] DINÇER E. Impact of lactic acid bacteria strains against Listeria monocytogenes biofilms on various food-contact surfaces[J]. Archives of Microbiology, 2024, 206(2): 80. DOI:10.1007/s00203-023-03811-6.
[20] HOSSAIN M I, MIZAN M F R, ROY P K, et al. Listeria monocytogenes biofilm inhibition on food contact surfaces by application of postbiotics from Lactobacillus curvatus B.67 and Lactobacillus plantarum M.2[J]. Food Research International, 2021, 148: 11054Xtdgkqf9vRz1D5ldwt26Q==95. DOI:10.1016/j.foodres.2021.110595.
[21] LEE J E, LEE N K, PAIK H D. Antimicrobial and anti-biofilm effects of probiotic Lactobacillus plantarum KU200656 isolated from kimchi[J]. Food Science and Biotechnology, 2020, 30(1): 97-106. DOI:10.1007/s10068-020-00837-0.
[22] 王利红, 周罗雄, 杨明明. 浅谈益生菌的抗菌机制[C]//中国畜牧兽医学会家禽生态学分会学术研讨会论文集. 北京: 中国畜牧兽医学会家禽生态学分会, 2014.
[23] HUANG F Q, TENG K L, LIU Y Y, et al. Bacteriocins: potential for human health[J]. Oxidative Medicine and Cellular Longevity, 2021, 2021: 5518825. DOI:10.1155/2021/5518825.
[24] 张军, 田子罡, 王建华, 等. 有机酸抑菌分子机理研究进展[J]. 畜牧兽医学报, 2011, 42(3): 323-328.
[25] 吕秀莉, 岳莹雪, 平丽筠, 等. 益生菌黏附机制及其拮抗肠道致病菌研究进展[J]. 食品科学, 2023, 44(9): 313-320. DOI:10.7506/spkx1002-6630-20220522-282.
[26] HARTMANN H A, WILKE T, ERDMANN R. Efficacy of bacteriocin-containing cell-free culture supernatants from lactic acid bacteria to control Listeria monocytogenes in food[J]. International Journal of Food Microbiology, 2011, 146(2): 192-199. DOI:10.1016/j.ijfoodmicro.2011.02.031.
[27] MARIA J L. A review of food-grade vectors in lactic acid bacteria: from the laboratory to their application[J]. Critical Reviews in Biotechnology, 2017, 37(3): 296-308. DOI:10.3109/07388551.2016.1144044.
[28] 张明, 孙宇航. 食品中后生元的研发和应用研究进展[J]. 食品科学技术学报, 2024, 42(2): 19-31. DOI:10.12301/spxb202300813.
[29] 李誉, 张竞文, 郑亚平, 等. 后生元的功能及其应用[J]. 中国食品学报, 2024, 24(2): 373-381. DOI:10.16429/j.1009-7848.2024.02.034.
[30] BARCENILLA C, DUCIC M, LÓPEZ M, et al. Application of lactic acid bacteria for the biopreservation of meat products: a systematic review[J]. Meat Science, 2022, 183: 108661. DOI:10.1016/j.meatsci.2021.108661.
[31] 姚芙蓉, 李军, 张莹, 等. 生物表面活性剂生产及应用研究进展[J]. 微生物学通报, 2022, 49(5): 1889-1901. DOI:10.13344/j.microbiol.china.210561.
[32] SHAFIPOUR YORDSHAHI A, MORADI M, TAJIK H, et al. Design and preparation of antimicrobial meat wrapping nanopaper with bacterial cellulose and postbiotics of lactic acid bacteria[J]. International Journal of Food Microbiology, 2020, 321: 108561. DOI:10.1016/j.ijfoodmicro.2020.108561.
[33] FUQUA W C, WINANS S C, GREENBERG E P. Quorum sensing in bacteria: the LuxR-LuxI family of cell density-responsive transcriptional regulators[J]. Journal of Bacteriology, 1994, 176(2): 269-275. DOI:10.1128/jb.176.2.269-275.1994.
[34] WATERS C M, BASSLER B L. Quorum sensing: cell-to-cell communication in bacteria[J]. Annual Review of Cell and Developmental Biology, 2005, 21: 319-346. DOI:10.1146/annurev.cellbio.21.012704.131001.
[35] 胡丽丽, 董庆利, 夏阳, 等. 单增李斯特菌生物膜形成及其调控机制研究进展[J]. 食品与发酵工业, 2021, 47(8): 276-282. DOI:10.13995/j.cnki.11-1802/ts.025678.
[36] WEI J Y, ZHANG X G, ISMAEL M, et al. Anti-biofilm effects of Z102-E of Lactiplantibacillus plantarum against Listeria monocytogenes and the mechanism revealed by transcriptomic analysis[J]. FCkE5j+K6DZ+J32UILU8WDonNPFJdxiwEQXyIPfZQkyU=oods, 2024, 13(16): 2495. DOI:10.3390/foods13162495.
[37] 黄湘湄, 吴雅茜, 刘颖, 等. 海洋源乳酸菌AI-2类群体感应抑制剂对单增李斯特菌抑制效果研究[J]. 生物技术通报, 2019, 35(4): 36-42. DOI:10.13560/j.cnki.biotech.bull.1985.2018-0934.
[38] REZZONICO F, DUFFY B. Lack of genomic evidence of AI-2 receptors suggests a non-quorum sensing role for LuxS in most bacteria[J]. BMC Microbiology, 2008, 8: 1-19. DOI:10.1186/1471-2180-8-154.
[39] 刘蕾, 桂萌, 武瑞赟, 等. LuxS/AI-2型群体感应系统调控细菌生物被膜形成研究进展[J]. 食品科学, 2016, 37(19): 254-262. DOI:10.7506/spkx1002-6630-201619043.
[40] BELVAL S C, GAL L, MARGIEWES S, et al. Assessment of the roles of LuxS, S-ribosyl homocysteine, and autoinducer 2 in cell attachment during biofilm formation by Listeria monocytogenes EGD-E[J]. Applied and Environmental Microbiology, 2006, 72(4): 2644-2650. DOI:10.1128/AEM.72.4.2644-2650.2006.
[41] GRAY B, HALL P, GRESHAM H. Targeting agr-and agr-like quorum sensing systems for development of common therapeutics to treat multiple gram-positive bacterial infections[J]. Sensors, 2013, 13(4): 5130-5166. DOI:10.3390/s130405130.
[42] ZETZMANN M, SÁNCHEZ-KOPPER A, WAIDMANN M S, et al. Identification of the agr peptide of Listeria monocytogenes[J]. Frontiers in Microbiology, 2016, 7: 206813. DOI:10.3389/fmicb.2016.00989.
[43] 涂春田, 汪洋, 易力, 等. 信号分子调控细菌生物被膜形成的分子机制[J]. 生物工程学报, 2019, 35(4): 558-566. DOI:10.13345/j.cjb.180326.
[44] PARK Y J, KIM Y J, YU H H, et al. Cell-free supernatants of Bacillus subtilis and Bacillus polyfermenticus inhibit Listeria monocytogenes biofilm formation[J]. Food Control, 2023, 144: 109387. DOI:10.1016/j.foodcont.2022.109387.
[45] QIAO Z, GUO X, WANG T, et al. Effects of sub-minimum inhibitory concentrations of bacteriocin BM173 on Listeria monocytogenes biofilm formation[J]. Probiotics and Antimicrobial Proteins, 2023: 1-11. DOI:10.1007/s12602-023-10192-1.
[46] MARQUES P H, JAISWAL A K, DE ALMEIDA F A, et al. Lactic acid bacteria secreted proteins as potential Listeria monocytogenes quorum sensing inhibitors[J]. Molecular Diversity, 2023: 1-16. DOI:10.1007/s11030-023-10722-7.
[47] 张晨曦, 刘鼎阔. 细菌群集运动特性的研究进展[J]. 生物工程学报, 2023, 39(8): 3188-3203. DOI:10.13345/j.cjb.220892.
[48] RUHAL R, KATARIA R. Biofilm patterns in gram-positive and gram-negative bacteria[J]. Microbiological Research,3ce6399481f1d8a94b7157c54cf1ef1e 2021, 251: 126829. DOI:10.1016/j.micres.2021.126829.
[49] DESVAUX M, HÉBRAUD M. The protein secretion systems in Listeria: inside out bacterial virulence[J]. FEMS Microbiology Reviews, 2006, 30(5): 774-805. DOI:10.1111/j.1574-6976.2006.00035.x.
[50] CHO S Y, NA H W, OH H B, et al. Structural basis of flagellar motility regulation by the MogR repressor and the GmaR antirepressor in Listeria monocytogenes[J]. Nucleic Acids Research, 2022, 50(19): 11315-11330. DOI:10.1093/nar/gkac815.
[51] OH H B, LEE S J, YOON S I. Structural and biochemical analyses of the flagellar expression regulator DegU from Listeria monocytogenes[J]. Scientific Reports, 2022, 12(1): 10856. DOI:10.1038/s41598-022-14459-5.
[52] 赵维俊. 益生菌表面疏水性与自动聚集能力的研究[D]. 西安: 陕西科技大学, 2012.
[53] YANG S J, LEE J E, LIM S M, et al. Antioxidant and immune-enhancing effects of probiotic Lactobacillus plantarum 200655 isolated from kimchi[J]. Food Science and Biotechnology, 2019, 28(2): 491-499. DOI:10.1007/s10068-018-0473-3.
[54] BEHBAHANI B A, NOSHAD M, VASIEE A, et al. Probiotic Bacillus strains inhibit growth, biofilm formation, and virulence gene expression of Listeria monocytogenes[J]. LWT-Food Science and Technology, 2024, 191: 115596. DOI:10.1016/j.lwt.2023.115596.
[55] KIM Y J, YU H H, SONG Y J, et al. Anti-biofilm effect of the cell-free supernatant of probiotic Saccharomyces cerevisiae against Listeria monocytogenes[J]. Food Control, 2021, 121: 107667. DOI:10.1016/j.foodcont.2020.107667.
[56] 胡安祺, 杨慧轩, 姚现琦, 等. 枯草芽孢杆菌无细胞上清液抑制单增李斯特菌生物被膜形成的研究[J]. 食品与发酵工业, 2024, 50(13): 1-8. DOI:10.13995/j.cnki.11-1802/ts.035957.
[57] RODRÍGUEZ P F P, ARÉVALO-VILLENA M, ROSA I Z, et al. Selection of potential non-sacharomyces probiotic yeasts from food origin by a step-by-step approach[J]. Food Research International, 2018, 112: 143-151. DOI:10.1016/j.foodres.2018.06.008.
[58] 谢淑仪, 陈姗姗, 栾天罡. 电活性微生物胞外聚合物的特征与应用[J]. 微生物学报, 2023, 63(2): 540-552. DOI:10.13343/j.cnki.wsxb.20220428.
[59] ZHUANG Z, YANG G Q, MAI Q J, et al. Physiological potential of extracellular polysaccharide in promoting Geobacter biofilm formation and extracellular electron transfer[J]. Science of the Total Environment, 2020, 741: 140365. DOI:10.1016/j.scitotenv.2020.140365.
[60] WEN Z T, BITOUN J P, LIAO S M. PBP1a-deficiency causes major defects in cell division, growth and biofilm formation by Streptococcus mutans[J]. PLoS ONE, 2015, 10(4): e0124319. DOI:10.1371/journal.pone.0124319.
[61] OUYANG Y, LI J, DONG Y Q, et al. Genome-wide screening of genes required for Listeria monocytogenes biofilm formation[J]. Journal of Biotech Research, 2012, 4: 13-25.
[62] KOLODKIN-GAL I, ROMERO D, CAO S G, et al. D-amino acids trigger biofilm disassembly[J]. Science, 2010, 328(5978): 627-629.M+xRAPnfeAnKC/SimzHGzw== DOI:10.1126/science.1188628.
[63] PARSONS C, COSTOLO B, BROWN P, et al. Penicillin-binding protein encoded by pbp4 is involved in mediating copper stress in Listeria monocytogenes[J]. FEMS Microbiology Letters, 2017, 364(20). DOI:10.1093/femsle/fnx207.
[64] ABACHIN E, POYART C, PELLEGRINI E, et al. Formation of D-alanyl-lipoteichoic acid is required for adhesion and virulence of Listeria monocytogenes[J]. Molecular Microbiology, 2002, 43(1): 1-14. DOI:10.1046/j.1365-2958.2002.02723.x.
[65] PIERCEY M J, HINGSTON P A, TRUELSTRUP HANSEN L. Genes involved in Listeria monocytogenes biofilm formation at a simulated food processing plant temperature of 15 ℃[J]. International Journal of Food Microbiology, 2016, 223: 63-74. DOI:10.1016/j.ijfoodmicro.2016.02.009.
[66] JARA J, PÉREZ-RAMOS A, DEL SOLAR G, et al. Role of Lactobacillus biofilms in Listeria monocytogenes adhesion to glass surfaces[J]. International Journal of Food Microbiology, 2020, 334: 108804. DOI:10.1016/j.ijfoodmicro.2020.108804.
[67] LEE Y J, WANG C. Links between S-adenosylmethionine and Agr-based quorum sensing for biofilm development in Listeria monocytogenes EGD-E[J]. MicrobiologyOpen, 2020, 9(5): e1015. DOI:10.1002/mbo3.1015.
[68] WEI Z H, SHAN C J, ZHANG L X, et al. A novel subtilin-like lantibiotics subtilin JS-4 produced by Bacillus subtilis JS-4, and its antibacterial mechanism against Listeria monocytogenes[J].
LWT-Food Science and Technology, 2021, 142: 110993. DOI:10.1016/j.lwt.2021.110993.
[69] DE ARAUJO L V, GUIMARÃES C R, DA SILVA MARQUITA R L,
et al. Rhamnolipid and surfactin: anti-adhesion/antibiofilm and antimicrobial effects[J]. Food Control, 2016, 63: 171-178. DOI:10.1016/j.foodcont.2015.11.036.
[70] MENDOZA I C, VILLAVICENCIO-VASQUEZ M, AGUAYO P,
et al. Biosurfactant from Bacillus subtilis DS03: properties and application in cleaning out place system in a pilot sausages processing[J]. Microorganisms, 2022, 10(8): 1518. DOI:10.3390/microorganisms10081518.
[71] TRAVIER L, LECUIT M. Listeria monocytogenes ActA: a new function for a ‘classic’ virulence factor[J]. Current Opinion in Microbiology, 2014, 17: 53-60. DOI:10.1016/j.mib.2013.11.007.
[72] VÁZQUEZ-BOLAND J A, KUHN M, BERCHE P, et al. Listeria pathogenesis and molecular virulence determinants[J]. Clinical Microbiology Reviews, 2001, 14(3): 584-640. DOI:10.1128/cmr.14.3.584-640.2001.
[73] 李孟华, 闫帅帅, 李德志, 等. 单增李斯特菌生物膜形成调控机制的研究进展[J]. 生物工程学报, 2021, 37(9): 3151-3161. DOI:10.13345/j.cjb.200734.
[74] MASEBE R D, THANTSHA M S. Anti-biofilm activity of cell free supernatants of selected lactic acid bacteria against Listeria monocytogenes isolated from avocado and cucumber fruits, and from an avocado processing plant[J]. Foods, 2022, 11(18): 2872. DOI:10.3390/foods11182872.
[75] HU Y W, OLIVER H F, RAENGPRADUB S, et al. Transcriptomic and phenotypic analyses suggest a network between the transcriptional regulators HrcA and σB in Listeria monocytogenes[J]. Applied and Environmental Microbiology, 2007, 73(24): 7981-7991. DOI:10.1128/AEM.01281-07.
[76] ZHOU Q C, WANG L, YIN X J, et al. SigB-dependent tolerance to protein synthesis-inhibiting antibiotics in Listeria monocytogenes EGDe[J]. Current Microbiology, 2012, 64(3): 234-241. DOI:10.1007/s00284-011-0058-3.
[77] SIBANDA T, BUYS E M. Listeria monocytogenes pathogenesis: the role of stress adaptation[J]. Microorganisms, 2022, 10(8): 1522. DOI:10.3390/microorganisms10081522.
[78] GABALLA A, GUARIGLIA-OROPEZA V, WIEDMANN M, et al.
Cross talk between SigB and PrfA in Listeria monocytogenes facilitates transitions between extra-and intracellular environments[J]. Microbiology and Molecular Biology Reviews, 2019, 83(4). DOI:10.1128/mmbr.00034-19.
[79] SARGHALEH S J, BEHBAHANI B A, HOJJATI M, et al. Evaluation of the constituent compounds, antioxidant, anticancer, and antimicrobial potential of Prangos ferulacea plant extract and its effect on Listeria monocytogenes virulence gene expression[J]. Frontiers in Microbiology, 2023, 14: 1202228. DOI:10.3389/fmicb.2023.1202228.
[80] PRICE R, JAYEOLA V, NIEDERMEYER J, et al. The Listeria monocytogenes key virulence determinants hly and prfA are involved in biofilm formation and aggregation but not colonization of fresh produce[J]. Pathogens, 2018, 7(1): 18. DOI:10.3390/pathogens7010018.
[81] MENEZES A G T, DE SOUSA MELO D, RAMOS C L, et al. Yeasts isolated from Brazilian fermented foods in the protection against infection by pathogenic food bacteria[J]. Microbial Pathogenesis, 2020, 140: 103969. DOI:10.1016/j.micpath.2020.103969.
[82] SALMINEN S, BOULEY C, BOUTRON-RUAULT M C, et al.
Functional food science and gastrointestinal physiology and function[J]. The British Journal of Nutrition, 1998, 80(Suppl 1): S147-S171. DOI:10.1079/bjn19980108.
[83] FAGERLUND A, LANGSRUD S, MØRETRØ T. In-depth longitudinal study of Listeria monocytogenes ST9 isolates from the meat processing industry: resolving diversity and transmission patterns using whole-genome sequencing[J]. Applied and Environmental Microbiology, 2020, 86(14): e00579. DOI:10.1128/AEM.00579-20.
[84] 汪忆梦, 张凤娟, 于明明, 等. 2022年潍坊市肉与肉制品中单增李斯特菌污染状况分析[J]. 食品安全导刊, 2023(32): 13-15; 19. DOI:10.16043/j.cnki.cfs.2023.32.008.
[85] SCHÄFER D F, STEFFENS J, BARBOSA J, et al. Monitoring of contamination sources of Listeria monocytogenes in a poultry slaughterhouse[J]. LWT-Food Science and Technology, 2017, 86: 393-398. DOI:10.1016/j.lwt.2017.08.024.
[86] ALÍA A, ANDRADE M J, RODRÍGUEZ A, et al. Prevalence and characterization of Listeria monocytogenes in deboning and slicing areas of Spanish dry-cured ham processing[J]. LWT-Food Science and Technology, 2020, 128: 109498. DOI:10.1016/j.lwt.2020.109498.
[87] BERESFORD M R, ANDREW P W, SHAMA G. Listeria monocytogenes adheres to many materials found in food-processing environments[J]. Journal of Applied Microbiology, 2001, 90(6): 1000-1005. DOI:10.1046/j.1365-2672.2001.01330.x.
[88] MIDELET G, CARPENTIER B. Transfer of microorganisms, including Listeria monocytogenes, from various materials to beef[J]. Applied and Environmental Microbiology, 2002, 68(8): 4015-4024. DOI:10.1128/AEM.68.8.4015-4024.2002.
[89] JEON H R, KWON M J, YOON K S. Control of Listeria innocua biofilms on food contact surfaces with slightly acidic electrolyzed water and the risk of biofilm cells transfer to duck meat[J]. Journal of Food Protection, 2018, 81(4): 582-592. DOI:10.4315/0362-028x.jfp-17-373.
[90] RAVYTS F, BARBUTI S, FRUSTOLI M A, et al. Competitiveness and antibacterial potential of bacteriocin-producing starter cultures in different types of fermented sausages[J]. Journal of Food Protection, 2008, 71(9): 1817-1827. DOI:10.4315/0362-028x-71.9.1817.
[91] REALINI C E, MARCOS B. Active and intelligent packaging systems for a modern society[J]. Meat Science, 2014, 98(3): 404-419. DOI:10.1016/j.meatsci.2014.06.031.
[92] BASCH C Y, JAGUS R J, FLORES S K. Physical and antimicrobial properties of tapioca starch-HPMC edible films incorporated with nisin and/or potassium sorbate[J]. Food and Bioprocess Technology, 2013, 6: 2419-2428. DOI:10.1007/s11947-012-0860-3.
[93] JOFRE A, GARRIGA M, AYMERICH T. Inhibition of 99c5dafdca1c3aebea4a11985b86aab7c8c2694a2c2309fa2efe1b8f71df1c60Listeria monocytogenes in cooked ham through active packaging with natural antimicrobials and high-pressure processing[J]. Journal of Food Protection, 2007, 70(11): 2498-2502. DOI:10.4315/0362-028x-70.11.2498.
[94] COMA V. Bioactive packaging technologies for extended shelf life of meat-based products[J]. Meat Science, 2008, 78(1/2): 90-103. DOI:10.1016/j.meatsci.2007.07.035.
收稿日期:2024-06-19
基金项目:国家现代农业(肉牛牦牛)产业技术体系建设专项(CARS-37)
第一作者简介:武桐煊(2003—)(ORCID: 0009-0006-9583-1000),女,本科生,研究方向为食品科学。
E-mail: 1460845786@qq.com
*通信作者简介:刘昀阁(1992—)(ORCID: 0000-0001-5955-3660),女,讲师,博士,研究方向为肉品科学。
E-mail: liuyunge@meatsci.com
张一敏(1985—)(ORCID: 0000-0001-5240-7126),女,教授,博士,研究方向为肉品科学。
E-mail: ymzhang@sdau.edu.cn
