动物源性食品真实性鉴别技术研究进展
2024-11-05郭亚宁刘亚轩周剑李凯李亮

摘 要:动物源性食品富含多种优质蛋白、必需脂肪酸及矿物质,对人体健康具有重要作用。近年来,动物源性食品掺假现象屡有发生,传统的鉴别技术因存在操作复杂、检出限高、准确度低的局限性,已无法满足人们对食品质量安全的需求,因此,为保障消费者的合法权益,研发快速、准确的鉴别技术对改善动物源性食品掺假掺杂乱象具有重要意义。本文从无损检测和分子生物学两方面概述当前鉴别动物源性食品真伪的主要方法,归纳这些方法在食品掺假、品种鉴定和原产地溯源等应用中的优缺点,分析该领域在研究中所面临的问题,为开发新型动物源性食品鉴定方法提供参考依据,为食品安全监督监管提供有力的技术支持。
关键词:动物源性食品;掺假;鉴别技术;食品质量安全
Research Progress on Authentication Techniques for Foods of Animal Origin
GUO Yaning1,2, LIU Yaxuan2, ZHOU Jian1,*, LI Kai1, LI Liang1
(1. Institute of Quality Standard and Testing Technology for Agro-products, Chinese Academy of Agricultural Sciences, Beijing 100081, China;
2. College of Food Science and Bioengineering, Tianjin Agricultural University, Tianjin 300384, China)
Abstract: Animal-derived foods are an excellent source of high-quality proteins, essential fatty acids and minerals, which play an important role in human health. In recent years, there have been numerous instances of adulteration of foods of animal origin. Traditional identification techniques have proven inadequate in meeting consumer demands for food quality and safety due to the limitations of complex operation, high detection limit and low accuracy. In order to protect the legitimate rights and interests of consumers, the development of rapid and accurate identification technology is importance for reducing adulteration of foods of animal origin. This paper outlines the current major methods for identifying the authenticity of animal-derived foods, including non-destructive testing methods and those based on molecular biology, with particular reference to their advantages and disadvantages when applied in food adulteration, species identification and geographical origin traceability. Finally, it analyses problems facing this field of research in order to provide a reference for the development of new methods for animal-derived food identification and to provide strong technical support for food safety supervision and regulation.
Keywords: foods of animal origin; adulteration; identification techniques; food quality and safety
DOI:10.7506/rlyj1001-8123-20240713-184
中图分类号:TS207.3 文献标志码:A 文章编号:1001-8123(2024)11-0055-09
引文格式:
郭亚宁, 刘亚轩, 周剑, 等. 动物源性食品真实性鉴别技术研究进展[J]. 肉类研究, 2024, 38(11): 55-63. DOI:10.7506/rlyj1001-8123-20240713-184. http://www.rlyj.net.cn
GUO Yaning, LIU Yaxuan, ZHOU Jian, et al. Research progress on authentication techniques for foods of animal origin[J]. Meat Research, 2024, 38(11): 55-63. DOI:10.7506/rlyj1001-8123-20240713-184. http://www.rlyj.net.cn
经加工和制作后供人类食用的各类动物组织、蛋、乳制品、肉及肉制品(包括动物内脏)和水生动物产品等统称为动物源性食品,以其丰富多样的种类、多元独特的口感赢得了人们的广泛喜爱[1]。近年来,随着经济的迅猛增长,人们的生活水平有了明显的提升,世界各国对动物源性食品的需求量也逐年攀升,产品结构发生巨大变化。据统计,2023年我国的猪肉、牛肉、羊肉、禽肉总产量高达9 641万 t,同比增长4.5%,猪肉、牛肉、羊肉、禽肉分别增长4.6%、4.8%、1.3%、4.9%[2]。但不法商家为了从中牟取非法利益,不断提高掺假手段,使消费者对动物源性食品的质量失去信心,如羊肉掺假[3]、牛肉掺假[4-5]事件,这主要是利用廉价的猪肉、鹅肉、鸭肉等替代品冒充高价值的牛肉和羊肉进行售卖,以此获得不正当利益。伪造、替换、稀释、篡改、对食品原产国或配料虚假申报、过度处理和侵犯知识产权等一系列非法行为均被列入食品掺假。动物源性食品在经深度加工或普遍使用添加剂浸泡进入市场后,仅凭气味、色泽及质地等传统感官评价方法难以精准区分其真实性。然而,随着科学技术的不断进步,基于无损检测技术与分子生物学原理的先进鉴别方法在国内外动物源性食品真实性鉴别领域脱颖而出,这些方法不仅弥补了传统方法的局限性,还通过深入剖析遗传物质的特征,实现了对动物源性食品的高精度鉴别,对保障食品安全、保护消费者合法权益、促进行业健康发展等具有重要意义。
本文聚焦近年来市面上掺假掺杂的热点问题,归纳总结动物源性食品真实性鉴别领域常用方法的优缺点,分析该领域研究面临的主要问题,以期对科研人员有所帮助,为实现动物源性食品安全监管监督提供检测依据。
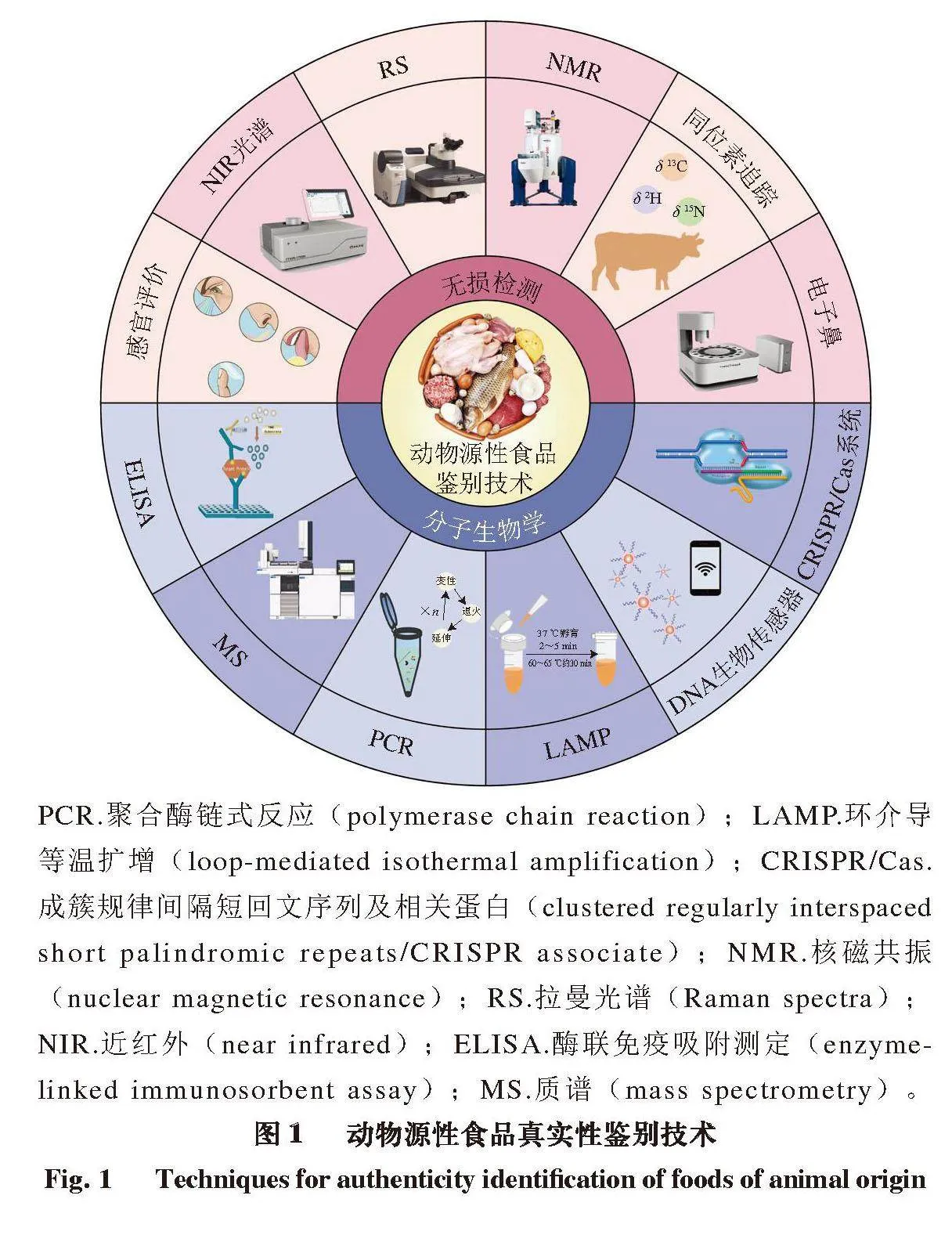
目前,国内外常用的动物源性食品真实性鉴别技术见图1。
PCR.聚合酶链式反应(polymerase chain reaction);LAMP.环介导等温扩增(loop-mediated isothermal amplification);CRISPR/Cas.成簇规律间隔短回文序列及相关蛋白(clustered regularly interspaced short palindromic repeats/CRISPR associate);NMR.核磁共振(nuclear magnetic resonance);RS.拉曼光谱(Raman spectra);NIR.近红外(near infrared);ELISA.酶联免疫吸附测定(enzyme-linked immunosorbent assay);MS.质谱(mass spectrometry)。
1 基于无损检测的动物源性食品鉴别技术
无损检测技术有别于传统破坏性检测技术,可以很大程度上保留被检样品的原始状态,且样品的化学特性不会受到影响,无损检测技术相较于传统破坏性检测技术省去了复杂、费时的前处理步骤[6],只需对待测样品少量取样后,经数据及信号采集、数据处理及信号控制3 个步骤即可实现对待检样品质量的评估[7],在食物细菌检
测[8-9]、重金属检测[10]、真假鉴别[11]中应用广泛。
1.1 感官评价
感官评价是一门依托于人的触觉、味觉、视觉和听觉等感官测量、分析和解释食物的科学技术,对企业产品的研发、优化及持续改进具有重要意义[12]。食品的风味[13]和色泽[14]不仅是其品质的重要体现,还能直接刺激消费者的感官,进而影响消费者的购买决策。
胡煌等[15]指出,不同种类的生鲜肉在色泽上略有差别,羊肉比牛肉略浅,猪肉呈粉红色,然而在肉的贮藏、销售及消费过程中,这些色泽会因氧化作用而逐渐趋于棕红色。Kumari等[16]利用感官评价方法比较动物肉和植物肉在颜色、质地、风味上的差异。但在经济利益的驱动下,不法商贩使用大量的香精香料改变肉的风味、使用色素改变肉的色泽,以至消费者无法凭借感官评价来鉴别动物源性食品的真实性,因此,检测速度快、准确度高、现场易操作的鉴别技术亟待开发。
1.2 NIR光谱
NIR光谱分析技术基于光谱学的工作原理被广泛应用。当处在780~2 500 nm范围内的光照射待测物质时,这些物质中的分子会对特定波长的光有所吸收,这个过程会导致电子的跃迁或分子振动能级的变化,从而为分析物质的化学和物理特性提供重要信息。目前,NIR光谱技术与自主学习能力强、建模速度快、非线性拟合能力强的机器学习模型相结合的方法已被研究学者广泛关注,促进了从传统的经验判断到仪器分析检测手段的转变,提高了检测方法的准确性、可靠性。例如,
Wang Li等[17]发现560 nm处的肌红蛋白可能是区别肉质真伪的关键信息,于是将深度学习模型与可见光-近红外二维光谱相结合用于牛羊肉的掺假鉴定,此方法相比于传统的机器学习方法展示出了更高的准确度。Windarsih等[18]利用傅里叶变换红外光谱和机器学习辅助技术相结合的方法,开发出能鉴别和预测金枪鱼油中猪油掺假的最佳模型。此外,将NIR光谱与机器学习算法相结合的方法,在品种分类鉴别[19]、样品来源区分[20]等多个领域均被广泛应用。Leng Tuo等[21]对色谱解读发现,6 250~5 580 cm-1区域的变化与脂肪酸有关,后利用快速、无损的NIR光谱技术和化学计量技术检测碎牛肉与猪肉的二元掺假及碎牛肉与猪肉、鸭肉的三元掺假情况,结果发现,采用选定波长的原始光谱建立的判别分析模型对二元掺假系统的判别准确率高达100%,三元掺假系统的误差低于8%,NIR无损检测技术为肉糜产品掺假检测提供了全新思路。将NIR光谱、荧光光谱和激光诱导击穿光谱的特征变量数据融合起来,建立随机森林模型,对不同产地食用明胶的掺假情况进行鉴定,其准确率、回收率和F分数均达到100%[22]。此外,Pereira等[23]将NIR光谱技术运用于山羊奶中痕量牛奶成分的鉴别,准确率高达100%。NIR光谱作为一种高效、无损、低成本、多组分的检测技术,在化学、生物学、食品科学等领域均有广泛的应用前景,尽管NIR光谱技术具有明显的优势,但其对复杂模型的高度依赖及对操作人员专业技能的要求却成为了制约其发展的关键因素。
1.3 RS
RS是由印度科学家C.V.拉曼首次发现的,其本质是光在分子水平上的非弹性散射[24],即通过分析不同频率下的散射光谱,就能够提取关于分子振动和转动的关键信息,这些信息对于深入研究和分析分子结构至关重要。韩娅红等[25]基于不同种属之间蛋白质的差异,通过RS对87 种肉骨粉(猪、鸡、牛、羊肉骨粉)进行鉴定,证明所开发的偏最小二乘判别分析(partial least squares discriminant analysis,PLS-DA)模型对鸡和哺乳动物、猪和反刍动物、牛肉骨粉和羊肉骨粉鉴别效果明显,并且PLS-DA模型在肉骨粉种属来源鉴别中展现出了卓越的性能,其灵敏度和特异性均超过93.8%。Boyaci等[26]根据不同动物脂肪成分的差异,利用RS与主成分分析相结合的方法鉴别不同肉类(猪肉、牛肉、鱼肉、绵羊肉、家禽肉、水牛肉和山羊肉)及其腊肠脂肪的特征振动频率,该方法成功规避了复杂肉类中基质的干扰,并以其高效的分析速度在动物源性食品真伪鉴别领域展现出巨大的应用前景。Forooghi等[27]证明,RS与数据驱动的类比软独立建模和PLS-DA相结合的全新策略能够明显区分牛黄油和人造奶油。该技术不仅可以检测样品中的痕量成分,还可以对不同状态下的食品进行检测,如固体、液体和气体,尽管如此,RS在实际应用中仍需补
充相应的标准库、排除样品中水分含量及荧光背景的潜在干扰。
1.4 NMR
NMR属于新型的无损检测技术,以分析原子核的磁性为基础[28],即将具有磁偶极子(相当于具有南极和北极的条形磁铁)的原子核置于强磁场后,应用特定射频的脉冲激发原子核,通过检测回到基态的原子核所释放的信号进行数据分析,以获得样品内部的化学信息。利用NMR技术对食品成分进行分析的研究已被许多科研工作者广泛讨论,如蛋白质[29-30]、脂肪[31]、糖[32]等。Leng Tuo等[33]利用主成分分析模型得出蛋氨酸和谷氨酰胺
可能是判别牛肉与猪肉、鸭肉的关键氨基酸,其次,根据所建立的正交OPLS-DA模型对牛肉中的二元和三元掺假情况进行鉴别,其准确率达100%。Hajjar等[34]利用高分辨率1H NMR技术、结合光谱去卷积技术和多元统计分析成功描绘了鸡蛋黄中的三酰甘油氢谱,为鸡蛋的来源、鸡种和养殖系统的识别提供了生物标记物。NMR检测技术因不损伤细胞且具有高分辨率和高灵敏度等特点,在食品成分分析、来源追踪、添加物检测及真伪鉴别等方面具有相当大的潜力,但在食品领域广泛应用的道路上,有两大挑战不容忽视:一是操作人员需要具备深厚的专业知识和技能;二是后期磁场维护的成本相对较高。
1.5 同位素追踪
同位素追踪法旨在通过精密仪器检测待检食品样本中稳定同位素比率的细微差异。该方法涉及到将样本中的同位素特征精准量化,并随后与经过严格校准、来源已知的同位素比率数据库进行比对,进而确保食品质量的真实性和安全性。吴浩等[35]运用稳定同位素指纹分析技术,对16 份进口三文鱼样品与2 份国产三文鱼样品进行系统考察,结果表明,相较于骨骼和肌肉组织,三文鱼鳞片及表皮中同位素比率的差异对于其产地的溯源展现出更显著的指示效果。白婷等[36]深入分析15 种矿物质在区分白羽肉鸡和黑水凤尾鸡品种特征上的作用,并指出锰、钼及铯具有完全的鉴别能力。此外,稳定同位素分析技术也被广泛应用在乳制品掺假方面,
Joachim等[37]将δ 13C和α-亚麻酸作为有机乳制品的鉴别指标,Ehtesham等[38]通过分析牛奶样本中脂肪酸组成、固形物含量及δ 2H同位素比值,并与相应饮用水源和饲料中的δ 2H同位素比值进行关联性研究,成功实现对乳制品地理起源的精确追溯。同位素追踪技术以其高度的精确性和卓越的抗干扰能力,在特定元素同位素比率的测定中展现出显著的优势。然而,这一技术的实施依赖于高精尖的仪器设备和专业的操作人员,从而导致分析成本的显著提升,此外,鉴于同位素追踪技术主要聚焦于少数几种特定元素的检测,在成分复杂的食品鉴别中受到一定程度的限制。
1.6 电子鼻
电子鼻是一个集成多个化学传感器的设备,这些高敏感的传感器可以对食物中的气味或挥发性化合物进行检测,当这些物质释放出来时,传感器会将其信号转换成电信号,之后通过对不同传感器输出信号的模式进行分析,进而对食品进行识别[39]。Kalinichenko等[40]创新性地将石英晶体微天平传感器电子鼻技术与2 种先进的化学计量学方法相融合,旨在精确区分不同种类香肠中大豆蛋白的含量,对保障食品标签信息的真实性及防止食品欺诈行为具有重要意义。同时,通过化学计量学原理的优化,构建了一套基于比色传感器的低成本电子鼻系统,在高效、经济地识别与检测牛肉中掺假猪肉方面展现了非凡的潜力与应用前景[41]。此外,电子鼻技术凭借化学计量分析模块与气体传感器阵列的重要程序,已成为乳制品品质鉴别[42-45]及蜂蜜质量评估[46-47]等多领域不可或缺的技术手段。但鉴于单一检测技术在食品掺假鉴定中可能存在局限性,其结果的准确性往往受到质疑。为了提升羊肉中鸭肉掺假检测的准确度,Wang Qian等[48]采用气相色谱-质谱联用技术,通过精准鉴定肉类样本中的挥发性有机化合物,有效验证电子鼻技术的初步筛选结果,增强了检测的可靠性;与此同时,Jia Wenshen等[49]
将NIR光谱与电子鼻相结合用以检测羊肉中鸭肉的掺假行为,此外,为了对所得的数据进行全面分析,还引入了基于F1分数的模型可靠性估计(F1-score-based model reliability estimation,F1-score-MRE)数据融合。结果表明,F1-score-MRE数据融合方法在羊肉掺假方面的准确率高达98.58%(F1分数:0.985 5)。陈鹏羽等[50]基于电子鼻与顶空固相微萃取结合气相色谱-嗅闻-质谱联用技术,为3 种不同品质羊肉的评估、地理来源追溯及真伪鉴别提供了参考数据,极大丰富了肉制品检测领域的研究方法。电子鼻因具有无需前处理、对操作人员的知识要求低等优势,在食品品质控制、鉴别、品种识别、快速检测和警报系统等方面均有应用,未来,随着传感器技术的持续进步与人工智能算法的日臻完善,电子鼻对不同物质、不同模式识别算法敏感度不同、模型不通用、不便携带等诸多问题将会被解决[51]。
2 基于分子生物学的动物源性食品鉴别技术
2.1 基于蛋白质的动物源性食品鉴别技术
蛋白质在动物营养中占据核心地位,它不仅构成动物体的重要组织基础,还在动物的生长发育及酶、激素的产生过程中发挥着不可或缺的作用。此外,蛋白质的独特性源于其复杂的空间结构、基因编辑及碱基排列的多样性,这种特异性在医学、生物技术和食品检测等多个领域均得到广泛应用。作为蛋白质组学探索领域内的前沿工具,蛋白质分子标记物正逐步成为科研人员深入探究生物体内蛋白质表达、功能特性和互作关系的关键,并在高通量筛选技术[52-53]的精细化应用、跨物种间蛋白质组差异的比较[54-56]、生物信息学分析策略的发展[57-58]等多个领域取得了令人瞩目的研究成果。
2.1.1 ELISA
ELISA是一种普遍采用的蛋白质免疫检测技术,其核心原理在于每种抗体与其相应的抗原之间存在高度的特异性结合能力。根据实际应用中检测需求与条件的差异,ELISA技术被细化为多种类型,包括间接法、竞争法和夹心法等,以满足不同领域的检测需要[59]。
骆训国等[60]将种属特异性强、易于获取的免疫球蛋白G作为检测标记物,采用ELISA夹心法检测混合生肉中猪肉成分,该方法最低能检测到1%(m/m)的猪肉掺杂,且在牛肉、羊肉、鸡肉和兔肉中具有特异性。在单克隆抗体夹心ELISA实验的框架下,Jiang Xingyi等[61]确定2 种哺乳动物骨骼肌蛋白单克隆抗体的特异性,并通过对缓冲液进行优化后发现,该步骤能显著提高从猪肉中提取目标分析物的效率和抗原性,且对加热哺乳动物掺杂检出限可低至1%(m/m)。ELISA技术是识别动物源性食品中欺诈行为的一种有效且低成本的方法,多年来的实践证明了这种方法的有效性。尽管如此,在食品加工中蛋白质可能会出现不可逆的变化,这些变化会影响基于蛋白质的ELISA检测方法的有效性。因此,这种方法不但不适用于高度加工的食品,还在多物种真伪鉴定中存在局限性。
2.1.2 MS
MS技术在化学分析领域发挥重要作用,其基本原理是将待测样品引入MS仪后将其转化为气态离子,并利用电场或磁场对离子质荷比筛选能力的不同进行分离,最后将分离后的离子引入检测器中记录下离子的信号强度并转化成MS图,进行数据分析。蛋白质经过翻译修饰后会形成特异性较好的N-链寡糖,施姿鹤[62]基于此采用高效液相色谱-电喷雾MS分析,构建牛肉中掺假猪肉和牛肉中掺假鸭肉的多对同晶置换定性定量鉴别方法,该方法准确性高,线性方程R2>0.99,检出限依次为0.88%、0.26%。Zhang Yingying等[63]将高分辨率MS采集得到的数据进行分析后,通过计算线性相关系数和回收率,确定了能准确定量狐狸的肽段,建立了液相色谱-串联质谱方法用于鉴定和准确定量实际样品中的狐狸肉掺假。Zhang Jiukai等[64]创新性地将串联质谱的枪式蛋白质组学技术与预定多反应监测方法相结合,根据牛奶样本筛选出的4 组肽定量离子对,实现了对牛奶样品中微量掺假成分的精准定量分析。该技术具有灵敏度高、检测速度快的特点,被研究学者广泛关注,但前处理时间长、不能现场检测、检测成本高、蛋白质易变性且只能用于检测可电离的物质,在一定程度上限制了其应用。
2.2 基于核酸分子的动物源性食品鉴别技术
动物源性食品真实性鉴别技术主要是基于无损检测和蛋白质分子,然而,随着消费者对食品风味和营养价值的要求不断提高,现有的检测方法越来越难以验证高度加工食品的真实性和质量。以核酸分子为基础的动物源性食品真实性鉴别技术打破了以往鉴别技术的局限性,其凭借核酸序列在深加工过程中的稳定性已在生态、医疗和食品等多个领域得到了广泛应用。
2.2.1 PCR
PCR是一种体外扩增技术,能够在特定的条件下从少量DNA/RNA模板中扩增出大量特定的DNA/RNA片段。引物、DNA聚合酶、缓冲液和4 种脱氧核苷酸是PCR实验所需的反应物,近年来,技术的发展促使科研工作者能更快速、更准确地在实验室中进行DNA/RNA片段的扩增,该技术主要包括变性、退火、延伸3 个关键步骤[65]。
依据检测原理可将其分为常规PCR、荧光实时定量PCR(real-time quantitative PCR,real-time PCR)、数字PCR(digital PCR,dPCR)等。常规PCR无需使用特殊的设备和试剂,成本较为低廉,但其缺陷在于无法实现对扩增产物的实时监测,若想进行半定量分析还需要借助凝胶电泳等方法。楼叶青等[66]对大西洋鳕鱼、阿拉斯加狭鳕鱼和白姑鱼的线粒体细胞色素c氧化酶亚基I进行分析、扩增后发现,通过扩增产物片段的大小可对大西洋鳕鱼、阿拉斯加狭鳕鱼与白姑鱼进行辨别,而大西洋鳕鱼与阿拉斯加狭鳕鱼需要借助Bst EII限制性内切酶进行鉴别。Doroudian等[67]提出了将PCR技术与组织学分析相结合的新方法,来弥补单一检测方法的局限性。线粒体DNA在组织中无处不在且序列高度保守,因此是用于鉴定肉及肉制品掺假的主要目标片段[68]。Li Jiapeng等[69]
利用线粒体上的DNA设计双重real-time PCR并根据扩增产物在熔融曲线上的出峰数量、位置,对牛与非牛物种进行初步筛选并对牛源性成分进行半定量。杨瑶等[70]用筛选得到的猪细胞核单拷贝基因作为扩增靶标进行real-time PCR扩增,成功检出肉制品中的猪源性成分。dPCR将连续稀释的样品包裹成数百或数万个小液滴,然后根据泊松分布对起始浓度进行绝对定量。刘立兵等[71]利用dPCR技术,建立肉粉质量与所含基因拷贝数浓度的线性关系,可准确定量检测香肠中鸡、猪、牛源性成分。该方法与传统细菌培养方法相比,反应速度快、自动化程度高,虽然PCR技术具有以上优势,但为确保PCR技术的准确性和可靠性,亟需有效应对和解决当前存在的引物设计不合理、样品处理效率低、引物探针特异性不足及污染干扰等一系列挑战。
2.2.2 LAMP
2000年,日本荣研株式会社首次提出LAMP技术,LAMP在模板提取、预变性、反应时间等方面均做出了优化[72],相比于传统的PCR,一方面LAMP规避了因反复加热而导致的DNA模板变性问题,另一方面实现了在常温条件下就能对目标分子进行连续快速扩增,在食品真实性鉴别[73]中应用广泛。Kumari等[74]设计了6 种LAMP引物,在64 ℃下以牛的线粒体细胞b为目标基因实现了特异性扩增,且灵敏度为0.1 pg。Peng Jiangsong等[75]将水蛭作为研究对象,通过与real-time PCR比较发现LAMP的灵敏度明显较高、耗时短,可用于现场检测,但LAMP扩增产物需借助凝胶电泳、荧光测量等技术进一步分析。Qin Panzhu等[76]将LAMP与荧光偏振技术将结合,用于鉴定食源性动物中猪肉掺假,大大提高了LAMP的实用性,检测限可达0.01%(m/m)。
2.2.3 DNA生物传感器
DNA生物传感器凭借其卓越的特异性配对和分子识别能力,巧妙地融合了先进的传感器技术,实现了对目标分子的高灵敏度、高精确度检测。根据传感器类型的不同,可分为质量敏感型、热敏型、场效应型等,其中电化学生物传感器和荧光生物传感器应用最为广泛[77]。在食品检测领域,DNA生物传感器具有快速、高灵敏、高通量、可重复性等优势。Zhang Chao等[78]利用物种间的特异性开发了多重LAMP耦合三叉戟式横向流动生物传感器方法,大大缩短了检测时间,且整个过程不需要精密仪器,适用于现场检测。Mansouri等[79]开发了一种表面等离子体共振增强型生物传感器,该方法可对低至1.0 nmol/L的目标驴肉DNA进行定量检测,相对标准偏差为0.85%,提高了检测的灵敏度和可重复性。然而,DNA生物传感器在应用于实际动物源性食品鉴别技术时仍然面临一些挑战,如开发困难、前处理复杂、成本高等问题。
2.2.4 CRISPR/Cas系统
CRISPR/Cas作为基因编辑剪刀可以对单链DNA实施精准、高效的剪切,后来人们不断扩展其在物种
改良[80]、肉类掺假鉴定[81]、食源性微生物监管[82]等领域的应用范围。不同的CRISPR相关蛋白在PAM识别位点、作用机制和应用领域等方面具有不同的特点。例如,Cas9在医疗领域发挥着重要作用,而Cas12在快速检测领域有着广泛的应用[83]。Chen Jiahui等[84]把具有特异性和反式裂解活性的CRISPR/Cas与无需标记的DNA-银纳米簇荧光探针相结合,扩宽线性范围的同时也显著增强了荧光探针的稳定性,将鸭的掺假量直接转化为荧光信号的强弱。Zhao Gang等[85]尝试结合重组聚合酶扩增技术与CRISPR/Cas12a技术检测肉制品中是否掺假猪肉,该研究可凭借肉眼在30 min内得到检测结果,且检出限为0.001 ng。精准、快速、低成本[8V8jtmHzduFdRbq+pCl+RuQsWZNVHWK0DX/SVHsKrvy8=6]的优势极大促进了CRISPR/Cas系统在复杂食品中痕量目标物检测方面的应用,并为此提供了一条全新的途径。
综上所述,不同鉴别技术应用于动物源性食品中的优缺点比较见表1。
3 结 语
近年来,动物源性食品掺假事件持续走高引发了人们的广泛关注,针对这一现状,我国陆续颁布了许多有关肉类真实性鉴别技术的国家标准。本文总结近5 年常用于鉴别动物源性食品真伪的技术,讨论各种技术的优缺点,其中,基于蛋白质ELISA开发的试剂盒和便于携带的光谱设备具有检测速度快、成本低的优势,深受市场监管部门和企业生产厂家的青睐。然而,蛋白质作为生物标记物用于深加工食品检测时容易发生变性和降解,这严重降低了检测的灵敏度;与之相比,核酸分子特有的种间多态性和热稳定性保证了其检测结果的可靠,在食品质量安全检测与执法机关应用较多,但该技术对操作人员和实验室要求较高,且现如今没有专门的机构对核酸标准物质进行研制,导致各企业生产的核酸标准物质缺乏可追溯性。未来应在快速、准确、低成本的前提下,不断完善动物源性食品的特异性蛋白数据库、丰富光谱和电子鼻等指纹信息,建立最佳的数据模型,优化PCR反应中核酸提取的限速步骤,在实际应用中应选择适当的鉴别技术或将多种鉴别技术联用,以更好地满足对复杂样品中目标成分的分析,这对预防食品掺假、保障食品安全、促进食品行业持续健康发展具有重要作用。
参考文献:
[1] 戴义国, 刘光辉, 颜丙利, 等. 动物源性食品安全检测技术探析[J]. 现代畜牧科技, 2024(2): 115-117. DOI:10.19369/j.cnki.2095-9737.2024.02.031.
[2] 国家统计局. 中华人民共和国2023年国民经济和社会发展统计公报[N]. 人民日报, 2024-03-01(010).
[3] 白鑫燚, 李晓毅. 济南门店大多已不见羊肉卷[N]. 山东商报, 2023-08-12(7).
[4] 唐穗平, 张燕, 黄景辉. 广东省牛羊肉及其制品中掺杂掺假情况的调查分析[J]. 食品安全质量检测学报, 2016, 7(5): 1882-1886. DOI:10.19812/j.cnki.jfsq11-5956/ts.2016.05.025.
[5] 凌莉, 李志勇, 高东微, 等. 从肉类掺假案例反思我国进出口食品安全监管[J]. 安徽农业科学, 2013(20): 8711-8713; 8715. DOI:10.3969/j.issn.0517-6611.2013.20.091.
[6] 曾雪晴, 李洪军, 王兆明, 等. 无损检测技术在掺假肉及肉制品中的应用进展[J]. 食品与发酵工业, 2019, 45(1): 252-258. DOI:10.13995/j.cnki.11-1802/ts.017198.
[7] 马昕. 无损检测在食品品质检测中的运用[J]. 现代食品, 2020(14): 123-125. DOI:10.16736/j.cnki.cn41-1434/ts.2020.14.039.
[8] 贺巧玲, 曹敏, 孙在兴, 等. 光谱技术及电子感官系统在食源性致病菌无损检测中的应用[J]. 食品研究与开发, 2023, 44(20): 219-224. DOI:10.12161/j.issn.1005-6521.2023.20.030.
[9] 苗小雨, 柴春祥, 鲁晓翔. 无损检测技术在食品微生物检测中的应用与展望[J]. 食品与发酵工业, 2022, 48(8): 311-319. DOI:10.13995/j.cnki.11-1802/ts.028633.
[10] 石洪玮. 单波长激发-能量色散X射线荧光光谱法检测食品中重金属的研究[D]. 邯郸: 河北工程大学, 2023: 1-2.
[11] SILVA D L M M, ADRIANO L F, MÔNICA G C, et al. Portable near-infrared (NIR) spectrometer and chemometrics for rapididentification of butter cheese adulteration[J]. Food Chemistry, 2023, 425: 136461. DOI:10.1016/j.fod3ce0901144b062dbf4153cd147f477afacb0aa031cb21454b88787894b01607odchem.2023.136461.
[12] 吴木兰, 宋萧萧, 崔武卫, 等. 基于近红外光谱技术对南阳豌豆产地鉴别研究[J]. 光谱学与光谱分析, 2023, 43(4): 1095-1102. DOI:10.3964/j.issn.1000-0593(2023)04-1095-08.
[13] 李卿, 马盛凯, 张悦, 等. 感官评定在新食品开发中的应用[J]. 食品工程, 2021(2): 42-45. DOI:10.3969/j.issn.1673-6044.2021.02.013.
[14] 孟新涛, 张婷, 许铭强, 等. 基于气相离子迁移谱的羊肉掺伪快速鉴别方法[J]. 新疆农业科学, 2019, 56(10): 1939-1947. DOI:10.6048/j.issn.1001-4330.2019.10.020.
[15] 胡煌, 吕飞, 丁玉庭. 肉制品的呈色机理和色泽评定研究进展[J]. 肉类研究, 2016, 30(12): 48-53. DOI:10.15922/j.cnki.rlyj.2016.12.009.
[16] KUMARI S, ALAM N A, HOSSAIN J M, et al. Sensory evaluation of plant-based meat: bridging the gap with animal meat, challenges and future prospects[J]. Foods, 2024, 13(1): 108. DOI:10.3390/foods13010108.
[17] WANG L, LIANG J, LI F, et al. Deep learning based on the vis-NIR two-dimensional spectroscopy for adulteration identification of beef and mutton[J]. Journal of Food Composition and Analysis, 2024, 126: 105890. DOI:10.1016/j.jfca.2023.105890.
[18] WINDARSIH A, JATMIKO H T, ANGGRAENI S A, et al. Machine learning-assisted FT-IR spectroscopy for identification of pork oil adulteration in tuna fish oil[J]. Vibrational Spectroscopy, 2024, 134: 103715. DOI:10.1016/j.vibspec.2024.103715.
[19] LEI F, BAOHUA W, SUSU Z, et al. Application of visible/infrared spectroscopy and hyperspectral imaging with machine learning techniques for identifying food varieties and geographical origins[J]. Frontiers in Nutrition, 2021, 8: 680357. DOI:10.3389/fnut.2021.680357.
[20] 张浩, 刘振, 王玲, 等. 基于近红外光谱结合机器学习算法检测食用明胶品种溯源的研究[J]. 河南农业大学学报, 2021, 55(3): 460-467. DOI:10.16445/j.cnki.1000-2340.20210409.003.
[21] LENG T, LI F, XIONG L, et al. Quantitative detection of binary and ternary adulteration of minced beef meat with pork and duck meat by NIR combined with chemometrics[J]. Food Control, 2020, 113: 107203. DOI:10.1016/j.foodcont.2020.107203.
[22] ZHANG H, LIU Z, ZHANG J, et al. Identification of edible gelatin origins by data fusion of NIRS, fluorescence spectroscopy, and LIBS[J]. Food Analytical Methods, 2021, 14(3): 525-536. DOI:10.1007/s12161-020-01893-2.
[23] PEREIRA S D V E, FERNANDES S D D D, ARAÚJO D U C M, et al.
Simultaneous determination of goat milk adulteration with cow milk and their fat and protein contents using NIR spectroscopy and PLS algorithms[J]. LWT-Food Science and Technology, 2020, 127: 109427. DOI:10.1016/j.lwt.2020.109427.
[24] ZHANG W Y, JI M, WEN D S. Raman spectroscopic techniques for detecting structure and quality of frozen foods: principles and applications[J]. Critical Reviews in Food Science and Nutrition, 2021, 61(16): 2623-2639. DOI:10.1080/10408398.2020.1828814.
[25] 韩娅红, 蹇林, 杜瑞铭, 等. 基于骨蛋白拉曼光谱特异性的肉骨粉种属鉴别方法[J]. 农业工程学报, 2020, 36(16): 267-273. DOI:10.11975/j.issn.1002-6819.2020.16.032.
[26] BOYACI I H, UYSAL R S, TEMIZ T, et al. A rapid method for determination of the origin of meat and meat products based on the extracted fat spectra by using of Raman spectroscopy and chemometric method[J]. European Food Research and Technology, 2014, 238(5): 845-852. DOI:10.1007/s00217-014-2168-1.
[27] FOROOGHI E, ZADE S V, JANNAT B, et al. Detection of sheep butter adulteration with cow butter and margarine by employing Raman spectroscopy and multivariate data analysis[J]. International Dairy Journal, 2024, 157: 106010. DOI:10.1016/j.idairyj.2024.106010.
[28] VEEMAN W S. Nuclear magnetic resonance, a simple introduction to the principles and applications[J]. Geoderma, 1997, 80(3): 225-242. DOI:10.1016/S0016-7061(97)00054-2.
[29] 冯翠萍, 刘一诺, 樊双喜, 等. 核磁共振磷谱法测定乳制品中的酪蛋白含量[J]. 食品科学, 2021, 42(4): 221-226. DOI:10.7506/spkx1002-6630-20200527-324.
[30] XING M J, LIU F J, LIN J Z, et al. Origin tracing and adulteration identification of bird’s nest by high- and low-field NMR combined with pattern recognition[J]. Food Research International, 2024, 175: 113780. DOI:10.1016/j.foodres.2023.113780.
[31] 李少华, 李申, 李翠翠. 食用油脂鉴别及掺假检测技术研究进展[J].
食品工业科技, 2022, 43(20): 430-436. DOI:10.13386/j.issn1002-0306.2021090293.
[32] 高西贝, 杜欣玥, 王琪琦, 等. 蜂蜜的真实性鉴别方法研究进展[J]. 食品安全质量检测学报, 2022, 13(18): 5842-5848. DOI:10.19812/j.cnki.jfsq11-5956/ts.2022.18.038.
[33] LENG T, HU X Y, CHEN Y, et al. Rapid identification and quantitation of pork and duck meat of binary and ternary adulteration in minced beef by 1H NMR combined with multivariate data fusion[J]. Food Control, 2023, 154: 110018. DOI:10.1016/j.foodcont.2023.110018.
[34] HAJJAR G, HADDAD L, RIZK T, et al. High-resolution 1H NMR profiling of triacylglycerols as a tool for authentication of food from animal origin: application to hen egg matrix[J]. Food Chemistry, 2021, 360: 130056. DOI:10.1016/j.foodchem.2021.130056.
[35] 吴浩, 周秀雯, 陈海泉, 等. 不同产地三文鱼的稳定同位素指纹特征及原产地溯源[J]. 食品科学, 2021, 42(16): 304-311. DOI:10.7506/spkx1002-6630-20200723-321.
[36] 白婷, 蔡浩洋, 邓银华, 等. 基于微量元素指纹图谱对黑水凤尾鸡进行产地溯源的研究[J]. 中国测试, 2018, 44(9): 57-62; 74. DOI:10.11857/j.issn.1674-5124.2018.09.011.
[37] JOACHIM M, ANETTE G. Follow-up of stable isotope analysis of organic versus conventional milk[J]. Analytical and Bioanalytical Chemistry, 2010, 398(3): 1493-1500. DOI:10.1007/s00216-010-3995-y.
[38] EHTESHAM E, HAYMAN A, HALE V R, et al. Influence of feed and water on the stable isotopic composition of dairy milk[J]. International Dairy Journal, 2015, 47: 37-45. DOI:10.1016/j.idairyj.2015.02.008.
[39] PERIS M, ESCUDER-GILABERT L. Electronic noses and tongues to assess food authenticity and adulteration[J]. Trends in Food Science & Technology, 2016, 58: 40-54. DOI:10.1016/j.tifs.2016.10.014.
[40] KALINICHENKO A, ARSENIYEVA L. Electronic nose combined with chemometric approaches to assess authenticity and adulteration of sausages by soy protein[J]. Sensors and Actuators B: Chemical, 2020, 303: 127250. DOI:10.1016/j.snb.2019.127250.
[41] HAN F, HUANG X, AHETO H J, et al. Detection of beef adulterated with pork using a low-cost electronic nose based on colorimetric sensors[J]. Foods, 2020, 9(2): 193. DOI:10.3390/foods9020193.
[42] TOHIDI M, GHASEMI-VARNAMKHASTI M, GHAFARINIA V, et al.
Development of a metal oxide semiconductor-based artificial nose as a fast, reliable and non-expensive analytical technique for aroma profiling of milk adulteration[J]. International Dairy Journal, 2018, 77: 38-46. DOI:10.1016/j.idairyj.2017.09.003.
[43] AYARI F, GHALEH M E, RABBANI H, et al. Using an E-nose machine for detection the adulteration of margarine in cow ghee[J]. Journal of Food Process Engineering, 2018, 41(6): 12806. DOI:10.1111/jfpe.12806.
[44] 廖美燕, 龙鸣, 洪晶, 等. 电子鼻在乳及其制品检测中的应用研究进展[J]. 中国乳业, 2022(10): 57-64. DOI:10.12377/1671-4393.22.10.11.
[45] WANG B, XU S, SUN D W. Application of the electronic nose to the identification of different milk flavorings[J]. Food Research International, 2010, 43(1): 255-262. DOI:10.1016/j.foodres.2009.09.018.
[46] 陈芳, 黄玉坤, 苑阳阳, 等. 基于电子鼻无损鉴别掺假蜂蜜[J]. 西华大学学报(自然科学版), 2018, 37(5): 56-60. DOI:10.3969/j.issn.1673-159x.2018.05.012.
[47] 陈晓明, 初叶心, 乔江涛, 等. 蜂蜜品种识别和掺假鉴别的研究进展[J].
食品工业, 2020, 41(2): 225-230.
[48] WANG Q, LI L, DING W, et al. Adulterant identification in mutton by electronic nose and gas chromatography-mass spectrometer[J]. Food Control, 2019, 98: 431-438. DOI:10.1016/j.foodcont.2018.11.038.
[49] JIA W S, QIN Y D, ZHAO C T. Rapid detection of adulterated lamb meat using near infrared and electronic nose: a F1-score-MRE data fusion approach[J]. Food Chemistry, 2024, 439: 138123. DOI:10.1016/j.foodchem.2023.138123.
[50] 陈鹏羽, 张德权, 李少博, 等. 基于电子鼻和顶空固相微萃取结合气相色谱-嗅闻-质谱联用技术分析不同品种羊肉煮制风味特征[J]. 食品与发酵工业, 2024, 50(10): 298-305. DOI:10.13995/j.cnki.11-1802/ts.037157.
[51] 曲爱玲, 马长路. 电子鼻在食品安全中的应用及发展趋势[J]. 食品工业, 2022, 43(8): 320-325.
[52] YU Y, GAO Q L, SONG Y, et al. High-throughput screening of potential allergens from complex proteins of large yellow croaker (Larimichthys crocea) by mass spectrometry[J]. Chinese Journal of Analytical Chemistry, 2017, 45(5): e1713-e1718. DOI:10.1016/S1872-2040(17)61016-8.
[53] XIANG X, LU J, TAO M, et al. High-throughput identification 417707b48843bfe095b8634b8b6019a24e58e3fa5a8defc9e289c1fe4a3afbe7of meat ingredients in adulterated foods based on centrifugal integrated purification-CRISPR array[J]. Food Chemistry, 2024, 443: 138507. DOI:10.1016/j.foodchem.2024.138507.
[54] WANG X, ZHAO X, HUANG D, et al. Proteomic analysis and cross species comparison of casein fractions from the milk of dairy animals[J]. Scientific Reports, 2017, 7(1): 43020. DOI:10.1038/srep43020.
[55] YANG Y, ZHENG N, WANG W, et al. N-glycosylation proteomic characterization and cross-species comparison of milk fat globule membrane proteins from mammals[J]. Proteomics, 2016, 16(21): 2792-2800. DOI:10.1002/pmic.201500361.
[56] YANG Y, ZHENG N, ZHAO X, et al. N-glycosylation proteomic characterization and cross-species comparison of milk whey proteins from dairy animals[J]. Proteomics, 2017, 17(9): 1600361. DOI:10.1002/pmic.201600361.
[57] TU M, CHENG S, LU W, et al. Advancement and prospects of bioinformatics analysis for studying bioactive peptides from food-derived protein: sequence, structure, and functions[J]. Trends in Analytical Chemistry, 2018, 105: 7-17. DOI:10.1016/j.trac.2018.04.005.
[58] 左惠心, 韩玲, 余群力, 等. 牦牛与黄牛肌肉差异蛋白质组及生物信息学分析[J]. 农业机械学报, 2017, 48(4): 313-320. DOI:10.6041/j.issn.1000-1298.2017.04.041.
[59] 范莉. 酶联免疫吸附法在食品检验中的实践应用研究[J]. 食品安全导刊, 2021(27): 135-136. DOI:10.16043/j.cnki.cfs.2021.27.074.
[60] 骆训国, 栗绍文, 周蕾蕾, 等. 夹心ELISA方法检测生肉混合物中的猪肉成分的研究[J]. 动物医学进展, 2010, 31: 20-22. DOI:10.3969/j.issn.1007-5038.2010.z1.005.
[61] JIANG X Y, RAO Q C, MITTL K, et al. Monoclonal antibody-based sandwich ELISA for the detection of mammalian meats[J]. Food control, 2020, 110: 107045. DOI:10.1016/j.foodcont.2019.107045.
[62] 施姿鹤. 肉中蛋白质N-糖基化分析及其在常见肉类掺假鉴别中的应用[D]. 南京: 南京农业大学, 2020: 1-11.
[63] ZHANG Y Y, LIU M Y, WANG S, et al. Identification and quantification of fox meat in meat products by liquid chromatography-tandem mass spectrometry[J]. Food Chemistry, 2022, 372: 131336. DOI:10.1016/j.foodchem.2021.131336.
[64] ZHANG J K, WEI L Y, MIAO J L, et al. Authenticity identification of animal species in characteristic milk by integration of shotgun proteomics and scheduled multiple reaction monitoring (MRM) based on tandem mass spectrometry[J]. Food Chemistry, 2024, 436: 137736. DOI:10.1016/j.foodchem.2023.137736.
[65] 柴晓茹, 刘超, 孟军涛, 等. PCR技术分类及其在复杂样本检测中的应用研究进展[J]. 沧州师范学院学报, 2024, 40(1): 47-52. DOI:10.3969/j.issn.2095-2910.2024.01.009.
[66] 楼叶青, 胡艺凡, 郑方媛, 等. 应用聚合酶链式反应和限制性内切酶酶切技术鉴别鳕鱼[J]. 肉类研究, 2019, 33(10): 57-62. DOI:10.7506/rlyj1001-8123-20190802-172.
[67] DOROUDIAN M, ANBARA H, SOEZI M, et al. Improving fraud detection in processed meats: a histology-PCR approach[J]. Journal of Food Composition and Analysis, 2023, 123: 105593. DOI:10.1016/j.jfca.2023.105593.
[68] LI Y C, LIU S Y, MENG F B, et al. Comparative review and the recent progress in detection technologies of meat product adulteration[J]. Comprehensive Reviews in Food Science and Food Safety, 2020, 19(4): 2256-2296. DOI:10.1111/1541-4337.12579.
[69] LI J P, WEI Y X, LI J C, et al. A novel duplex SYBR green real-time PCR with melting curve analysis method for beef adulteration detection[J]. Food Chemistry, 2021, 338: 127932. DOI:10.1016/j.foodchem.2020.127932.
[70] 杨瑶, 斯能武, 严钰澳, 等. 实时荧光PCR定量检测肉制品中猪源性成分[J]. 食品工业科技, 2022, 43(3): 268-274. DOI:10.13386/j.issn1002-0306.2021050232.
[71] 刘立兵, 陈敏娜, 孙晓霞, 等. 微滴式数字聚合酶链式反应对香肠制品中鸡、猪、牛源性成分的定量分析[J]. 肉类研究, 2020, 34(8): 51-56. DOI:10.7506/rlyj1001-8123-20200508-115.
[72] 杨粤, 付博宇, 张蕴哲, 等. 环介导等温扩增检测技术的应用与方法改进[J]. 食品安全质量检测学报, 2016, 7(4): 1526-1530. DOI:10.19812/j.cnki.jfsq11-5956/ts.2016.04.034.
[73] WANG S, SONG H S, WANG T, et al. Recent advancements with loop-mediated isothermal amplification (LAMP) in assessment of the species authenticity with meat and seafood products[J]. Critical Reviews in Food Science and Nutrition, 2024: 1-22. DOI:10.1080/10408398.2024.2329979.
[74] KUMARI S, KUMAR R R, MENDIRATTA S K, et al. On-site detection of tissues of buffalo origin by loop-mediated isothermal amplification (LAMP) assay targeting mitochondrial gene sequences[J]. Food Analytical Methods, 2020, 13(5): 1060-1068. DOI:10.1007/s12161-020-01718-2.
[75] PENG J S, LI Y, DENG X L, et al. Loop-mediated isothermal amplification
for on-site visual identification of leech species[J]. Food Analytical Methods, 2024, 17(4): 618-627. DOI:10.1007/s12161-024-02597-7.
[76] QIN P Z, LI Y L, YAO B B, et al. Rational incorporating of loop-mediated isothermal amplification with fluorescence anisotropy for rapid, sensitive and on-site identification of pork adulteration[J]. Food Control, 2022, 137: 108863. DOI:10.1016/j.foodcont.2022.108863.
[77] 刘萍, 任有良, 狄燕清. DNA生物传感器研究综述[J]. 商洛学院学报, 2011, 25(2): 35-41. DOI:10.3969/j.issn.1674-0033.2011.02.008.
[78] ZHANG C, ZHANG X, LIAO G, et al. Species-specific TM-LAMP and Trident-like lateral flow biosensor for on-site authenticity detection o0b4248de281d407379ec2deb4cb99da4f horse and donkey meat[J]. Sensors and Actuators B: Chemical, 2019, 301: 127039. DOI:10.1016/j.snb.2019.127039.
[79] MANSOURI M, FATHI F, JALILI R, et al. SPR enhanced DNA biosensor for sensitive detection of donkey meat adulteration[J]. Food Chemistry, 2020, 331: 127163. DOI:10.1016/j.foodchem.2020.127163.
[80] RAI M, MAGUDEESWARI P, TYAGI W. CRISPR/Cas genome editing: strategies and potential for crop improvement[M]. Cham: Springer International Publishing, 2020: 53-67.
[81] LIU H, WANG J, ZENG H, et al. RPA-Cas12a-FS: a frontline nucleic acid rapid detection system for food safety based on CRISPR-Cas12a combined with recombinase polymerase amplification[J]. Food Chemistry, 2021, 334: 127608. DOI:10.1016/j.foodchem.2020.127608.
[82] STOUT E, KLAENHAMMER T, BARRANGOU R. CRISPR-Cas technologies and applications in food bacteria[J]. Annual Review of Food Science and Technology, 2017, 8: 8413-8437. DOI:10.1146/annurev-food-072816-024723.
[83] 王炀坤, 纪世新, 苏莹莹, 等. 基因编辑工具CRISPR-Cas9与CRISPR-Cas12a的比较与应用[J]. 长春师范大学学报, 2023, 42(12): 107-112. DOI:10.3969/j.issn.1008-178X.2023.12.018.
[84] CHEN J H, LIU J H, WU D, et al. CRISPR/Cas precisely regulated dna-templated silver nanocluster fluorescence sensor for meat adulteration detection[J]. Journal of Agricultural and Food Chemistry, 2022, 70(44): 14296-14303. DOI:10.1021/acs.jafc.2c04500.
[85] ZHAO G, WANG J, YAO C, et al. Alkaline lysis-recombinase polymerase amplification combined with CRISPR/Cas12a assay for the ultrafast visual identification of pork in meat products[J]. Food Chemistry, 2022, 383: 132318. DOI:10.1016/j.foodchem.2022.132318.
[86] TAO D, XIAO X, LAN X, et al. An inexpensive CRISPR-based point-of-care test for the identification of meat species and meat products[J]. Genes, 2022, 13(5): 912. DOI:10.3390/genes13050912.
[87] 白京, 李家鹏, 邹昊, 等. 近红外特征光谱定量检测羊肉卷中猪肉掺假比例[J]. 食品科学, 2019, 40(2): 287-292. DOI:10.7506/spkx1002-6630-20180531-452.
[88] 白京, 李家鹏, 邹昊, 等. 近红外光谱定性定量检测牛肉汉堡饼中猪肉掺假[J]. 食品科学, 2019, 40(8): 287-292. DOI:10.7506/spkx1002-6630-20180802-016.
[89] LOGAN B G, HOPKINS D L, SCHMIDTKE L M, et al. Authenticating common Australian beef production systems using Raman spectroscopy[J]. Food Control, 2021, 121: 107652. DOI:10.1016/j.foodcont.2020.107652.
[90] BOYACI I H, TEMIZ H T, UYSAL R S, et al. A novel method for discrimination of beef and horsemeat using Raman spectroscopy[J]. Food Chemistry, 2014, 148: 37-41. DOI:10.1016/j.foodchem.2013.10.006.
[91] BAI Y, LIU H, ZHANG B, et al. Research progress on traceability and authenticity of beef[J]. Food Reviews International, 2021, 39: 1645-1665. DOI:10.1080/87559129.2021.1936000.
[92] 易冰清, 郭秀秀, 颜治, 等. 乳制品掺假现状与稳定同位素鉴别技术研究进展[J]. 同位素, 2020, 33(5): 293-303. DOI:10.7538/tws.2020.33.05.0293.
[93] 王静娴, 张昂. 基于稳定同位素分析技术的蜂蜜产地鉴别研究[J]. 现代食品, 2024, 30(4): 207-210. DOI:10.16736/j.cnki.cn41-1434/ts.2024.4.064.
[94] 裴高璞, 史波林, 赵镭, 等. 典型掺假蜂蜜的电子鼻信息变化特征及判别能力[J]. 农业工程学报, 2015, 31(S1): 325-331. DOI:10.3969/j.issn.1002-6819.2015.z1.039.
[95] MANDLI J, EL FATIMI I, SEDDAOUI N, et al. Enzyme immunoassay (ELISA/immunosensor) for a sensitive detection of pork adulteration in meat[J]. Food Chemistry, 2018, 255: 380-389. DOI:10.1016/j.foodchem.2018.01.184.
[96] ASENSIO L, GONZÁLEZ I, GARCÍA T, et al. Determination of food authenticity by enzyme-linked immunosorbent assay (ELISA)[J]. Food Control, 2008, 19(1): 1-8. DOI:10.1016/j.foodcont.2007.02.010.
[97] STACHNIUK A, SUMARA A, MONTOWSKA M, et al. Liquid chromatography-mass spectrometry bottom-up proteomic methods in animal species analysis of processed meat for food authentication and the detection of adulterations[J]. Mass Spectrometry Reviews, 2021, 40(1): 3-30. DOI:10.1002/mas.21605.
[98] KITPIPIT T, SITTICHAN K, THANAKIATKRAI P. DZ2juoo0r8q0keEwCTvqU0MP53OTh1qbowiy1f0tXoFo=irect-multiplex PCR assay for meat species identification in food products[J]. Food Chemistry, 2014, 163: 77-82. DOI:10.1016/j.foodchem.2014.04.062.
[99] NESVADBOVA M, KRALIK P, DZIEDZINSKA R, et al. An integrated system of four multiplex qPCR assays for the precise and sensitive identification of animal species in food and feed[J]. Food Control, 2022, 135: 108781. DOI:10.1016/j.foodcont.2021.108781.
[100] VISHNURAJ M R, ARAVIND KUMAR N, VAITHIYANATHAN S,
et al. Authentication issues in foods of animal origin and advanced molecular techniques for identification and vulnerability assessment[J]. Trends in Food Science & Technology, 2023, 138: 164-77. DOI:10.1016/j.tifs.2023.05.019.
[101] BENJAKUL S, SAETANG J. Development of loop-mediated isothermal amplification (LAMP) assays for the rapid authentication of three swimming crab species[J]. Foods, 2022, 11(15): 2247. DOI:10.3390/foods11152247.
[102] THANAKIATKRAI P, BURANACHAI C, KITPIPIT T.
A novel biosensor method to detect cow DNA in milk and dairy products[J]. Food Control, 2024, 155: 110096. DOI:10.1016/j.foodcont.2023.110096.
[103] CHEN Y, YANG T, QIAN S, et al. Multiple crRNAs-assisted CRISPR/Cas12a assay targeting cytochrome b gene for amplification-free detection of meat adulteration[J]. Analytica Chimica Acta, 2022, 1231: 340417. DOI:10.1016/j.aca.2022.340417.
收稿日期:2024-07-13
基金项目:“十四五”国家重点研发计划重点专项(2023YFF0614602)
第一作者简介:郭亚宁(2003—)(ORCID: 0009-0006-6313-1819),女,硕士研究生,研究方向为农业领域核酸标准物质。
E-mail: gyn93810802@163.com
*通信作者简介:周剑(1983—)(ORCID: 0000-0002-8110-1908),男,研究员,博士,研究方向为农业领域标准物质。
E-mail: zhoujian_8382@163.com
