非洲猪瘟病毒O174L 蛋白的真核表达载体构建及分子特征分析
2024-06-28沙嘎那日·吉日木图林晓沈兆基郭肖蓉李奎贾红周荣
沙嘎那日·吉日木图 林晓 沈兆基 郭肖蓉 李奎 贾红 周荣
摘要:为分析非洲猪瘟病毒O174L 基因,通过同源重组方式将O174L 基因连接至pRK5M-C-2×Strep载体,构建重组质粒,经PCR扩增和测序鉴定正确后,将重组质粒转染至猪小肠上皮细胞系(porcine intestinal columnarepithelial cells,IPEC-J2)中,通过免疫荧光和Western blot检测O174L蛋白的表达情况。PCR及测序结果显示,pRK5M-C-2×Strep-O174L 重组质粒构建成功。免疫荧光和Western blot检测结果显示,O174L蛋白能够在IPEC-J2细胞中稳定表达。生物信息学分析结果显示,基于O174L 基因和B646L(p72)基因序列构建各分离毒株的2个系统发育树间排列高度相似。来自中国的16株分离株中,O174L 基因序列的相似性高达96.76%~100.00%。其中,与中国爆发的其他Ⅱ型分离株相比,China/2018/AnhuiXCGQ在O174L蛋白的第67、75及110位氨基酸存在差异,GZ201801在第110位氨基酸存在差异。Ⅰ型分离株 SD/DY-I/2021和 HeN/ZZ-P1/2021的O174L蛋白的氨基酸序列分别在第13、73、93、95、113和114位上与其他中国Ⅱ型分离株存在差异。O174L蛋白为稳定的亲水蛋白,没有信号肽和跨膜区;其二级结构由α螺旋、β折叠和无规则卷曲组成,三级结构预测结果与二级结构预测相符。以上结果为深入研究ASFV O174L蛋白与宿主间的相互作用和遗传进化提供了基础。
关键词:非洲猪瘟病毒;O174L 基因;真核表达;分子特征
doi:10.13304/j.nykjdb.2023.0162
中图分类号:S858.28;Q78 文献标志码:A 文章编号:10080864(2024)04011414
非洲猪瘟(African swine fever,ASF)是由非洲猪瘟病毒(African swine fever virus,ASFV)引起的一种烈性、高度接触性传染病,可感染家猪、野猪,其天然宿主为软蜱(Ornithodoros moubata)。ASFV通过空气、食物、环境和虫媒等多个途径传播。其强毒力株对生猪致病率高,致死率可高达100%,临床表现主要为发热、皮肤发绀及淋巴结、肾、胃肠粘膜明显出血等[1]。ASF在全球的爆发和流行对养猪产业和食品安全产生了巨大威胁。自2018年传入我国后,给我国养猪业造成了毁灭性打击[1]。
ASFV 是非洲猪瘟病毒属的唯一成员,为一种大型的线性双链DNA 病毒[2]。基因组大小约170~190 kb,编码150多种蛋白质,参与ASFV 生命周期的不同阶段,包括进入宿主细胞、抑制宿主免疫反应、病毒复制及病毒自身DNA 损伤修复等[3]。ASFV感染的主要靶细胞类型是单核-吞噬系统的细胞,包括特定组织巨噬细胞(macrophages,Mφ)和网状细胞的特定谱系[4]。其中,巨噬细胞作为重要的固有免疫细胞之一,是机体抵御病原微生物入侵的第一道防线[5]。巨噬细胞的主要特点是能够产生活性氧(reactive oxygenspecies,ROS)和提高一氧化氮合成酶(nitric oxidesynthetase,iNOS)活性,使其清除胞内感染的能力增强[6-9]。
ASFV基因组DNA是ROS攻击的重要靶分子之一。DNA受ROS氧化损伤的方式主要有2种:一种是DNA双链中的碱基直接被氧化,如链断裂和自发脱嘌呤/脱嘧啶;另一种是脱氧核苷三磷酸(dNTPs)池中的游离碱基被氧化。然而,包括病毒在内的所有生物体基本都无法避免来自外界和生物体自身内部的DNA损伤,若不及时修复这些损伤,将会对生物体产生灾难性的后果,比如DNA复制和转录过程将不能正常进行,严重影响基因组的完整性和稳定性,进而引起病毒自身复制和组装停止、突变以及死亡等一系列危害[10]。为了有效克服这些DNA损伤,ASFV 进化出了自己的修复系统。ASFV 修复系统包括由自身编码的Ⅱ类AP内切酶(E269R)、修复性DNA 聚合酶(O174L)、DNA连接酶(NP419 L)3个修复酶参与的碱基切除修复通路[1112]。O174L 蛋白是一种类似细胞DNA聚合酶β(DNA polymerase beta,Pol β)的修复性DNA 聚合酶,能够有效修复单核苷酸缺口DNA[1314]。O174L 蛋白的三维结构表明,与其他DNA聚合酶不同,O174L仅由1个具有催化位点的手掌结构域和参与脱氧核苷三磷酸(dNTP)选择的C末端结构域组成,缺乏Pol β具有的带有dRP裂解酶活性位点的N末端8-kD结构域[1516]。然而关于O174L 的保真度却存在争议,研究报道,O174L 错误插入核苷酸的频率为10-4~10-5,与Pol β的值相当[1718]。相比之下,O174L在其他研究中的保真度值低40~700倍[19]。尽管对O174L蛋白有了一定的了解,但是ASFV整个DNA修复系统以及O174L蛋白在病毒自身DNA损伤修复中的作用机制仍未知。因此,本研究通过真核系统表达ASFV的DNA聚合酶O174L,进一步利用生物信息学方法分析O174L蛋白的分子特征,以期对该基因及其蛋白的功能研究提供参考。
1 材料与方法
1.1 试验材料
pRK5M-C-2×Strep 载体、DH5α 感受态和IPEC-J2细胞由本实验室保存。
1.2 试剂
高保真DNA聚合酶 2×Phanta Max Master Mix(P515)、快速克隆试剂盒 Clon Express Ⅱ OneStepCloning Kit(C112)、DNA 聚合酶2×Rapid TaqMaster Mix(P222)、小量提取质粒试剂盒 FastPure Plasmid Mini Kit(DC201)购自诺唯赞生物科技股份(南京)有限公司。限制性内切酶NotⅠ(R0189V)和BamHⅠ(R0136V)购自New EnglandBiolabs 公司( 美国)。DNA 回收试剂盒(DR010250)购自浙江易思得生物科技有限公司(杭州)。聚乙烯亚胺(polyethylenimine, PEI)转染试剂(23966)购自Polyscience 公司(美国)。抗Streptavidin标签单克隆抗体(BE2076-100)购自柏奥易杰(北京)科技有限公司。辣根过氧化物酶标记羊抗兔&鼠IgG(M210035)购自艾比玛特医药科技(上海)有限公司。蛋白显色液ImmobilonWestern HRP(WBKLS0100)购自 Merck 公司(美国)。BCA蛋白质检测试剂盒PierceTM BCA ProteinAssay Ki(t WL338065)购自Thermo公司(美国)。
1.3 试验方法
1.3.1 生物信息学分析
查询GeneBank数据库中已有注释的ASFV基因组数据,下载 O174L 基因序列以及相应分离株的B646L(p72)基因序列,利用 DNAstar7.1和DNAMAN7等分析软件对其进行多序列比对。使用CLUSTALW程序进行核苷酸序列的多重比对。使用MEGAX 软件采用邻接(neighbor-joining,NJ)法基于O174L 和B646L 基因序列构建系统发育树,通过1 000 个自展值(Bootstraps)确定统计学显著性。从NCBI 下载ASFV分离株CADC_HN09中O174L蛋白的氨基酸序列,通过ExPASy在线分析蛋白质的理化性质,采用SignalP和TMHMM法分别预测蛋白质的信号肽和跨膜区,采用PSIPRED法在线分析蛋白质的二级结构,通过同源建模SWISS-MODEL法在线预测蛋白质的三级结构。
1.3.2 引物设计及合成
根据NCBI GenBank 中ASFV CADC_HN09 基因组(GenBank ID:MZ614662.1)的核苷酸序列合成O174L 基因,由生工生物工程(上海)股份有限公司合成。并设计含有载体和O174L 基因同源序列的特异性引物O174L-F(5-TGAATTAAGCTTGGTGGATCCATGTTAACGCTTATTCAAGGAAAA-3,下划线部分为Not Ⅰ酶切位点)和 O174L-R( 5-TGCGGGTGGCTCCATGCGGCCGCTTATAAACGTTTCTTAGGTATGCG-3,下划线部分为BamH Ⅰ酶切位点),并加入保护性碱基,扩增片段大小为525 bp。引物由生工生物工程(上海)股份有限公司合成。
1.3.3 O174L 基因的扩增
以合成的O174L 基因为模板进行PCR 扩增目的基因。PCR 体系:2×Phanta Max Master Mix 10 μL,ddH2O 5 μL,上、下游引物各1 μL,模板3 μL,共20 μL。PCR程序:95 ℃3 min;95 ℃ 15 s,60 ℃ 15 s,72 ℃ 40 s,35个循环;72 ℃ 7 min。PCR产物经1%琼脂糖凝胶电泳鉴定后,用DNA回收试剂盒纯化PCR产物。
1.3.4 重组质粒的构建
用限制性内切酶Not Ⅰ和BamH Ⅰ双酶切pRK5M-C-2×Strep 载体,用快速克隆试剂盒将纯化回收的O174L 基因PCR产物及双酶切载体pRK5M-C-2×Strep 进行连接,连接产物转化至DH5α感受态细胞内。挑取单菌落置于LB液体培养基中,在37 ℃恒温摇床内振荡培养12 h,再进行菌液PCR鉴定及质粒小量提取,送生工生物工程(上海)股份有限公司测序鉴定。
1.3.5 细胞培养及转染
用含有10% 的胎牛血和1%的青霉素、链霉素双抗的DMEM培养基培养IPEC-J2 细胞,待细胞长至约80%密度时使用PEI转染试剂将重组质粒转染至IPEC-J2细胞中,加入无血清、无抗性DMEM培养基培养4~6 h后,更换新的完全培养基继续培养24 h。
1.3.6 免疫荧光试验
使用12 孔细胞培养板接种细胞,转染重组质粒并培养24 h后收取细胞。采用4% 多聚甲醛室温固定细胞20 min,加入0.5% Triton X-100对细胞通透15 min,随后使用3% BSA室温封闭1 h。4 ℃条件下加入Strep标签抗体(1∶8 000)孵育过夜,随后加入荧光二抗,室温避光孵育1 h,最后加入4,6-二脒基-2-苯基吲哚[2-(4-amidinophenyl)-6-indole‐carbamidine dihydrochloride,DAPI]进行避光染色,在荧光显微镜下观察。
1.3.7 Western blot 检测
用添加蛋白酶抑制剂的RIPA(radio immunoprecipitation assay)裂解缓冲液提取细胞蛋白,并使用BCA蛋白质检测试剂盒测定蛋白质含量。将蛋白质样品与1×SDSloading buffer 混合并煮沸10 min。取上清进行SDS-PAGE 凝胶电泳,然后转印至PVDF 膜(Merck,美国)。将膜在室温下用5%脱脂牛奶溶液封闭2 h,加入一抗(1∶10 000)4 ℃孵育过夜;随后加入二抗(1∶5 000)室温孵育45 min;最后通过化学发光试剂和成像系统检测目的蛋白信号。使用Image J(v.1.53k)对结果进行灰度值分析。
1.4 数据分析
采用GraphPad Prism 处理数据并作图,采用独立样本t 检验进行差异显著性分析。
2 结果与分析
2.1 O174L 系统发育分析
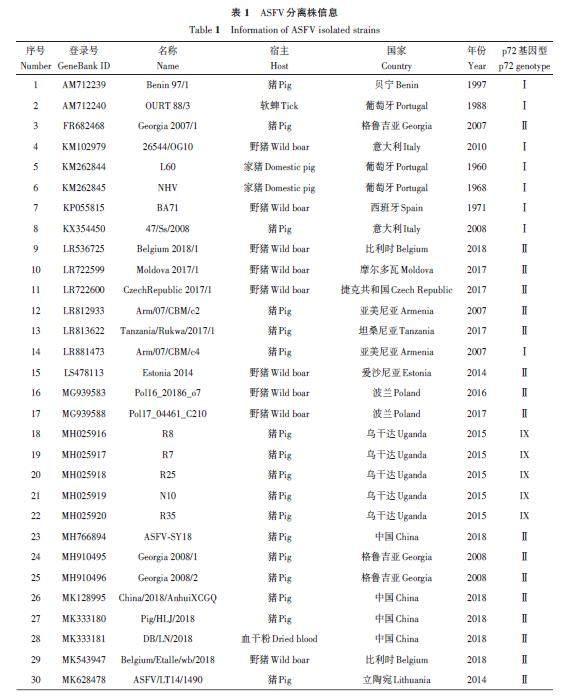
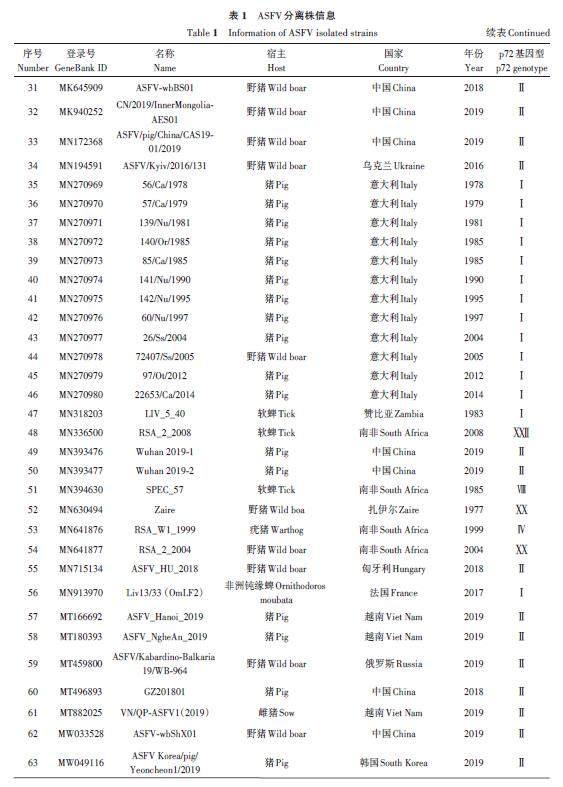
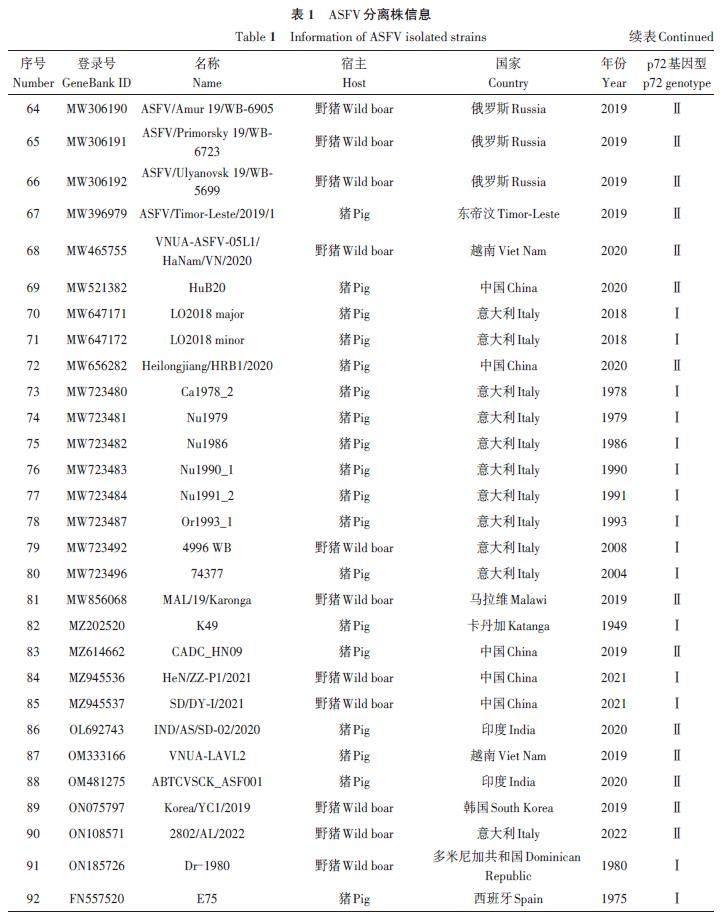
在GeneBank 数据库检索ASFV 分离株基因组,共获得92 个不同分离株的O174L 基因序列和相应分离株的B646L(p72)基因序列,包括完整的分离时间、地点、宿主和p72 基因型等相关信息,如表1所示。参考NCBI,获得的分离株有7 个不同的p72 基因型,分别是Ⅰ、Ⅱ、Ⅳ、Ⅷ、Ⅸ、ⅩⅩ和ⅩⅫ型。
系统发育树结果(图1)显示,基于O174L 和B646L 基因序列的2 个系统发育树中各分离株的排列和基因型分型高度吻合。基因型Ⅰ(除LIV_5_40外)、Ⅱ和Ⅸ的ASFV分离株在2个系统发育树中相同的基因型均聚类在同一个进化枝上。基因型为Ⅳ、Ⅷ、XX和XXII 的分离株聚类在同一个大进化枝上,说明这几个基因型分离株相互之间的遗传关系与Ⅰ、Ⅱ和Ⅸ型更近。赞比亚分离株(LIV_5_40,MN318203)虽然是Ⅰ型,但基于O174L 和B646L 基因序列的2个系统发育树中均与其他Ⅰ 型分离株的距离较远。
基于O174L 基因和B646L(p72)基因的2个系统发育树存在2个细微差异。首先是基于O174L基因的系统发育树中(图1A),相同的基因型大进化枝下面出现了子进化枝,其中子进化支上的Ⅰ型分离株为Liv13/33 (OmLF2)(MN913970)、K49(MZ202520)、NHV(KM262845)、OURT 88/3(AM712240)、Arm/07/CBM/c4(LR881473)、Pig/HeN/ZZ-P1/2021(MZ945536)和Pig/SD/DY-I/2021(MZ945537);Ⅱ 型分离株为ASFV/Ulyanovsk 19/WB-5699(MW306192)、GZ201801(MT496893) 和China/2018/AnhuiXCGQ(MK128995)。虽然基于O174L 基因序列的系统发育树Ⅰ型和Ⅱ型分支也出现了子进化枝,但这些子进化枝上的分离株O174L 基因序列与其他大进化枝上的分离株O174L 基因序列相比有着99%以上的序列一致性,而基于B646L(p72)基因序列构建的系统发育树上没有出现类似的子进化枝(图1B)。
由图2可知,来自中国的16个ASFV分离株包括2个基因Ⅰ型和14个基因Ⅱ型,通过多序列比对,各分离株间的O174L 基因序列相似性高达96.76%~100.00%。在氨基酸序列比对中,与中国爆发的其他Ⅱ型分离株相比,China/2018/AnhuiXCGQ在O174L蛋白的第67、75以及110位氨基酸上存在差异,GZ201801在第110位氨基酸上存在差异;基因Ⅰ型分离株SD/DY-I/2021 和HeN/ZZ-P1/2021 的O174L蛋白氨基酸序列分别在第13、73、93、95、113和114位上与其他中国Ⅱ型分离株存在差异。
2.2 O174L蛋白质的理化性质、信号肽与跨膜区预测
ExPASy在线预测分析显示,O174L 基因编码蛋白长度为174 aa,理论分子量为20.33 kD,预估半衰期为30 h,不稳定指数为25.44,表明O174L蛋白为稳定蛋白。亲水性平均值为-0.101,为亲水蛋白。经SignalP 和TMHMM 法预测(图3)显示,O174L蛋白质不存在信号肽和跨膜区。
2.3 O174L 蛋白质二级和三级结构预测
分析蛋白的二级结构(图4)显示,O174L蛋白由8 个α 螺旋(α helix)、7 个β 折叠(β strand)和16 个无规则卷曲(random coil)组成(图4A)。蛋白三级结构预测(图4B)显示,依然是α螺旋、β折叠和无规则卷曲为该蛋白质的主要组成结构,与二级结构预测相符。
2.4 O174L 基因扩增产物的鉴定
以合成的O174L 基因为模板,通过PCR扩增目的基因,PCR产物经1%琼脂糖凝胶电泳分析,在525 bp处有明亮的特异性条带,条带大小与预期一致(图5A)。
2.5 重组表达质粒的鉴定
将O174L 基因连接至线性化载体pRK5M-C2×Strep 中(图5B)。重组表达质粒pRK5M-C-2×Strep-O174L 经菌液PCR鉴定,可见525 bp的目的基因片段(图5C),测序结果正确,表明重组质粒构建成功。
2.6 O174L 蛋白在IPEC-2 细胞中的表达
用PEI转染试剂将重组表达质粒pRK5M-C-2×Strep-O174L 转染至IPEC-J2 细胞。通过免疫荧光和Western blot 检测O174L 蛋白的表达,结果显示,转染试验组在荧光显微镜下显示大量绿色荧光,而未转染重组质粒的对照组细胞未见绿色荧光(图6A)。Western blot 分析IPEC-J2细胞出现大小为20.33 kD的特异性条带,与重组蛋白O174L 的预期大小相符。灰度值结果显示,对照组无特异性条带(图6B和C),进一步证明目的蛋白O174L 能在IPEC-J2 细胞系中稳定表达。
3 讨论
1921年肯尼亚首次报道ASF疫情,随后蔓延到欧洲,又传播到南美洲和加勒比地区[20];2007年,进入格鲁吉亚,逐步影响东欧等国家[21];2018年,我国沈阳地区首次爆发[22],并逐渐向其他国家蔓延,如巴布亚新几内亚、印度[23]、德国[24]、多米尼加[25]和海地[26]。因ASFV基因组复杂、编码蛋白较多,很多蛋白功能未知以及交叉保护免疫力差等因素严重阻碍了ASF疫苗的研发,到目前为止,还没有有效的治疗措施。
本研究根据ASFV 分离株CADC_HN09 基因组序列合成O174L基因,成功构建了O174L基因的真核表达载体,通过免疫荧光与Western blot验证了O174L基因能够在猪小肠上皮细胞(IPEC-J2)中稳定表达。O174L 是聚合酶PolX 家族成员[13]。PolX家族显示出一种典型的聚合酶结构,因为它与人手相似,被称为拇指、手掌和手指。枯草芽孢杆菌PolX(BsPolX)、嗜热细菌PolX(TtPolX)、大鼠PolX(RatPolβ)和人类Polβ(HsPolβ)中8-kD结构域和拇指结构域在同源蛋白中高度保守,但O174L与这些同源蛋白的序列相似性非常低,约为30%,一致性更低,仅约10%[2728],且O174L缺少8-kD结构域和拇指结构域,但多了1个专门与受损伤DNA碱基缺口位点下游5端寡核苷酸上的磷酸基团(5-P)结合的口袋结构域。当碱基缺口位点的下游具有5-P时,O174L的催化效率会比没有5-P时高出约14倍,由此表明,与5-P结合的口袋结构域在O174L的DNA修复过程中发挥着重要作用[29]。但目前O174L 的DNA 修复机制还未知,因此研究O174L的DNA修复机制,对研制ASFV疫苗以及抗病育种具有很大的理论指导作用。
本研究对O174L 基因进行了生物信息学分析,发现基于O174L 基因序列构建的系统发育树的分支和分离株的排列与基于B646L 基因序列构建的系统发育树结果高度相似,来自中国的16株分离株中,各分离株间的O174L 基因序列相似性高达96.76%~100.00%。其中,与中国爆发的其他Ⅱ 型分离株相比,China/2018/AnhuiXCGQ 在O174L 的第67、75以及110位氨基酸上存在差异,GZ201801 在第110 位氨基酸上存在差异。Ⅰ型分离株SD/DY-I/2021和HeN/ZZ-P1/2021的O174L氨基酸序列分别在第13、73、93、95、113和114位上与其他中国Ⅱ型分离株存在差异。O174L蛋白为稳定的亲水蛋白,无信号肽和跨膜区,其二级结构由α螺旋、β折叠和无规卷曲组成,三级结构预测结果与二级结构预测相符。对ASFV进行基因分型对于揭示病毒的起源和快速鉴定同源性毒株非常重要。随着基因克隆、PCR和测序技术的迅速发展,ASFV 的基因组研究也得到了进一步的发展。1984年,含有98% ASFV基因组信息的文库被成功建立[30],之后,关于ASFV基因组文库建设的文章数量逐渐增加。目前几个特定的基因已被用来评估ASFV 基因型,包括B646L(p72)、E183L(p54)、CP204L(p30)、EP402(CD2v)、O174L、KP86R 等[31]。迄今为止,基于C-末端p72编码基因(B646L)已经描述了24种基因型[32],然而,公开可用的完整基因组序列仅代表基因型Ⅰ至Ⅴ、Ⅶ至Ⅹ和XX,绝大多数序列对应于基因型Ⅰ、Ⅱ和Ⅸ[33-37],从而限制了对地理起源和爆发模式的理解。随着对ASFV基因组和分型技术,特别是全基因组测序和交叉保护试验数据的不断深入研究,对进化和选择过程将会有更好的理解,对不同DNA病毒和单独病毒基因的分化时间和起源也会有更准确的估计[38]。
赞比亚分离株LIV_5_40在B646L(p72)基因型分类中虽然属于Ⅰ型,但无论是在基于O174L和B646L 基因的系统发育树中还是在ASFV全基因组发育树中,与其他Ⅰ型分离株均不在同一个进化枝上,而是与Ⅲ和XXII 型聚在一起,且与Ⅳ和XX 型也较为接近[38]。通过DNAMAN7碱基序列比对,LIV_5_40分离株的O174L 和B646L 基因序列与本研究中的其他Ⅰ型分离株序列相比存在43~45 和102~109 个单核苷酸多态性(singlenucleotide polymorphisms,SNP)。这种数量较多的碱基突变可能是由于不同分离株间的基因重组。当不同亚种的毒株感染同一宿主,均需要利用宿主细胞的复制系统进行复制,不同毒株间虽然各有一些特殊基因片段,但很多基因片段非常相似,导致宿主细胞复制系统有时会将两者弄混,在复制不同毒株的相同基因片段时,连带把一种毒株的特殊基因片段重组到了另一种毒株上,形成“杂交”毒株。ASFV在赞比亚地区丛林中反复感染软蜱、疣猪、丛林猪等多种宿主,因此其面临的选择压力和环境压力导致赞比亚地区的ASFV毒株易发生基因重组,因此猜测赞比亚ASF 流行病学中可能存在丛林作用[39]。
研究表明,ASFV 聚合酶Ⅹ基因的缺失导致猪巨噬细胞内ASFV的DNA损伤积累和突变频率增加[40],表明该基因对维持ASFV基因的稳定必不可少。然而,O174L 基因序列的突变和缺失对其蛋白功能的影响仍是未知。本研究构建O174L 在猪消化道细胞(IPEC-J2)内表达的重组质粒,为今后O174L蛋白与宿主细胞内蛋白质的相互作用研究提供了工具。
参考文献
[1] CHAPMAN D A G, TCHEREPANOV V, UPTON C, et al ..Comparison of the genome sequences of non-pathogenic andpathogenic African swine fever virus isolates [J]. J. Gen. Virol,2008, 89(Pt2):397-408.
[2] NDLOVU S, WILLIAMSON A L, HEATH L, et al .. Genomesequences of three African swine fever viruses of genotypes IV andXX from zaire and south africa, isolated from a domestic pig (susscrofa domesticus), a warthog (phacochoerus africanus), and aEuropean wild boar (sus scrofa) [J/OL]. Microbiol. Resour.Announc., 2020, 932: e00341-20 [2023-02-15]. https://doi. org/10.1128/MRA.00341-20.
[3] P?REZ-N??EZ D, GARC?A-URDIALES E, MART?NEZ-BONETM, et al.. CD2v interacts with adaptor protein AP-1 during Africanswine fever infection [J/OL]. PLoS One, 2015, 104: e0123714[2023-02-15]. https://doi.org/10.1371/journal.pone.0123714.
[4] SáNCHEZ E G, PéREZ-N??EZ D, REVILLA Y. Mechanisms ofentry and endosomal pathway of African swine fever virus [J/OL].Vaccines (Basel), 2017, 54:42 [2023-02-15]. https://doi.org/10.3390/vaccines5040042.
[5] MILLS C D. M1 and M2 macrophages: oracles of health anddisease [J]. Crit. Rev. Immunol., 2012, 32(6):463-488.
[6] BILITY M T, CHENG L, ZHANG Z, et al.. Hepatitis B virusinfection and immunopathogenesis in a humanized mouse model:induction of human-specific liver fibrosis and M2-like macrophages[J/OL]. PLoS Pathog., 2014, 103:e1004032 [2023-02-15]. https://doi.org/10.1371/journal.ppat.1004032.
[7] MARTINEZ F O, GORDON S. The M1 and M2 paradigm ofmacrophage activation: time for reassessment [J/OL]. F1000PrimeRep., 2014, 6: 13 [2023-02-15]. https://doi. org/10.12703/P6-13.eCollection 2014.
[8] MARTINEZ F O, GORDON S, LOCATI M, et al.. Transcriptionalprofiling of the human monocyte-to-macrophage differentiation andpolarization: new molecules and patterns of gene expression [J]. J.Immunol., 2006, 177(10):7303-7311.
[9] VOGEL D Y, VEREYKEN E J, GLIM J E, et al .. Macrophagesin inflammatory multiple sclerosis lesions have an intermediateactivation status [J/OL]. J. Neuroinflamm., 2013, 10:35 [2023-02-15]. https://doi.org/10.1186/1742-2094-10-35.
[10] FORMAN H J, TORRES M. Redox signaling in macrophages [J].Mol. Aspects Med., 2001, 224(5):189-216.
[11] PEI D S, STRAUSS P R. Zebrafish as a model system to study DNAdamage and repair [J]. Mutatation Res., 2013, 743-744:151-159.
[12] PRZYBYLOWSKA K, KABZINSKI J, SYGUT A, et al .. Anassociation selected polymorphisms of XRCC1, OGG1 andMUTYH gene and the level of efficiency oxidative DNA damagerepair with a risk of colorectal cancer [J]. Mutatant Res., 2013,745-746:6-15.
[13] OLIVEROS M, Y??EZ R J, SALAS M L, et al .. Characterizationof an African swine fever virus 20-kDa DNA polymerase involvedin DNA repair [J]. J. Biol. Chem., 1997, 272(49):30899-30910.
[14] SHOWALTER A K, TSAI M D. A DNA polymerase withspecificity for five base pairs [J]. J. Am. Chem. Soc., 2001, 123(8):1776-1777.
[15] SHOWALTER A K, BYEON I J, SU M I, et al .. Solutionstructure of a viral DNA polymerase X and evidence for amutagenic function [J]. Nat. Struct. Biol., 2001, 81(1):942-946.
[16] MACIEJEWSKI M W, SHIN R, PAN B, et al .. Solutionstructure of a viral DNA repair polymerase [J]. Nat. Struct.Biol., 2001, 81(1):936-941.
[17] AHN J, KRAYNOV V S, ZHONG X, et al .. DNA polymerasebeta: effects of gapped DNA substrates on dNTP specificity,fidelity, processivity and conformational changes [J]. Biochem.J., 1998, 331(Pt1):79-87.
[18] CHAGOVETZ A M, SWEASY J B, PRESTON B D. Increasedactivity and fidelity of DNA polymerase beta on singlenucleotidegapped DNA [J]. J. Biol. Chem., 1997, 272(44):27501-27504.
[19] LAMARCHE B J, KUMAR S, TSAI M D. ASFV DNA polymerseX is extremely error-prone under diverse assay conditions andwithin multiple DNA sequence contexts [J]. Biochemistry, 2006,454(9):14826-14833.
[20] COSTARD S, WIELAND B, DE GLANVILLE W, et al .. Africanswine fever: how can global spread be prevented ? [J]. PhilosTrans. R Soc. Lond B Biol. Sci., 2009, 3641(530):2683-2696.
[21] ROWLANDS R J, MICHAUD V, HEATH L, et al .. Africanswine fever virus isolate, Georgia, 2007 [J]. Emerg. Infect. Dis.,2008, 141(2):1870-1874.
[22] ZHOU X, LI N, LUO Y, et al .. Emergence of African swinefever in china, 2018 [J]. Transbound Emerg. Dis., 2018, 65(6):1482-1484.
[23] RAJUKUMAR K, SENTHILKUMAR D, VENKATESH G, et al..Genetic characterization of African swine fever virus from domesticpigs in India [J]. Transbound Emerg. Dis., 2021, 68(5):2687-2692.
[24] SAUTER-LOUIS C, FORTH J H, PROBST C, et al .. Joiningthe club: first detection of African swine fever in wild boar inGermany [J]. Transbound Emerg. Dis., 2021, 68(4):1744-1752.
[25] GONZALES W, MORENO C, DURAN U, et al .. African swinefever in the Dominican Republic [J]. Transbound Emerg. Dis.,2021, 68(6):3018-3019.
[26] BLOME S, FRANZKE K, BEER M. African swine fever - areview of current knowledge [J/OL]. Virus Res., 2020, 287:198099[2023-02-15]. https://doi.org/10.1016/j.virusres.2020.198099.
[27] LINDAHL T, WOOD R D. Quality control by DNA repair [J].Science, 1999, 2865(446):1897-1905.
[28] BEARD W A, WILSON S H. Structural design of a eukaryoticDNA repair polymerase: DNA polymerase beta [J]. MutatantRes., 2000, 4603(4):231-244.
[29] CHEN Y, ZHANG J, LIU H, et al .. Unique 5-P recognitionand basis for dG: dGTP misincorporation of ASFV DNApolymerase X [J/OL]. PLoS Biol., 2017, 152: e1002599[2023-02-15]. https://doi.org/10.1371/journal.pbio.1002599.
[30] LEY V, ALMENDRAL J M, CARBONERO P, et al .. Molecularcloning of African swine fever virus DNA [J]. Virology, 1984,133(2):249-257.
[31] MAZUR-PANASIUK N, WO?NIAKOWSKI G. The uniquegenetic variation within the O174L gene of polish strains ofAfrican swine fever virus facilitates tracking virus origin [J].Arch. Virol., 2019, 164(6):1667-1672.
[32] QU H, GE S, ZHANG Y, et al .. A systematic review of genotypesand serogroups of African swine fever virus [J]. Virus Genes,2022, 58(2):77-87.
[33] BISHOP R P, FLEISCHAUER C, DE VILLIERS E P, et al ..Comparative analysis of the complete genome sequences ofkenyan African swine fever virus isolates within p72 genotypesⅨ and Ⅹ [J]. Virus Genes, 2015, 50(2):303-309.
[34] CHAPMAN D A, DARBY A C, SILVA M D A, et al .. Genomicanalysis of highly virulent Georgia 2007/1 isolate of Africanswine fever virus [J]. Emerg. Infect. Dis., 2011, 17(4):599-605.
[35] DE VILLIERS E P, GALLARDO C, ARIAS M, et al ..Phylogenomic analysis of 11 complete African swine fever virusgenome sequences [J]. Virology, 2010, 400(1):128-136.
[36] MASEMBE C, SREENU V B, SILVA FILIPE A D A, et al ..Genome sequences of five African swine fever virus genotypeIX isolates from domestic pigs in Uganda [J/OL]. Microbiol.Resour. Announc., 2018, 713:e01018-18 [2023-02-15]. https://doi.org/10.1128/MRA.01018-18.
[37] RODR?GUEZ J M, MORENO L T, ALEJO A, et al .. Genomesequence of African swine fever virus BA71, the virulentparental strain of the nonpathogenic and tissue-culture adaptedBA71V [J/OL]. PLoS One, 2015, 1011:e0142889 [2023-02-15].https://doi.org/10.1371/journal.pone.0142889.
[38] ASLANYAN L, AVAGYAN H, KARALYAN Z. Whole-genomebasedphylogeny of African swine fever virus [J]. Vet. World,2020, 13(10):2118-2125.
[39] SIMULUNDU E, LUBABA C H, VAN HEERDEN J, et al .. Theepidemiology of African swine fever in "nonendemic" regionsof Zambia (1989—2015): implications for disease preventionand control [J/OL]. Viruses, 2017, 99: 236 [2023-02-15].https://doi.org/10.3390/v9090236.
[40] REDREJO-RODRíGUEZ M, RODR?GUEZ J M, SU?REZ C,et al .. Involvement of the reparative DNA polymerase pol Ⅹ ofAfrican swine fever virus in the maintenance of viral genomestability in vivo [J]. J. Virol., 2013, 871(7):9780-9787.
(责任编辑:张冬玲)
基金项目:中央级公益性科研院所基本科研业务费专项(Y2021XK20);国家自然科学基金项目(31972541);六安市产学合作重大专项。
