小麦单株穗数的QTL定位及CAPS标记开发
2024-06-21万家乐吴美琴陈心怡赵晓雪常梦帅陈璨李亮卢杰
摘 要:穗数是小麦产量构成三要素之一,开展小麦成穗数研究,对高产育种及种质资源创制具有重要意义。本研究以安农859/武农988的135份DH群体为研究材料,分别测定两年五个环境下单株穗数表型值,并基于DH群体的55K芯片数据进行单株穗数QTL分析,开发CAPS分子标记。结果表明,在1B、1D、2A、3D、4A、4B、4D、6A、7D等染色体上共检测到21个与单株穗数相关的QTL,其中,位于4B染色体上的Qsn-ahau-4B.2在5个环境下均被检测到,侧翼标记为AX-95004669-AX-109580651,物理区间为2.65Mb,可解释16.92%~49.98%表现变异,加性效应为1.32~3.69个穗数,增效等位基因来自安农859,是一个主效、稳定的QTL。基于Qsn-ahau-4B.2区间内候选基因TraesCS4B01G042700开发一个多态性CAPS标记(T-0507)。本研究结果将为小麦单株成穗数研究提供参考。
关键词:小麦;单株穗数;QTL定位;标记开发
中图分类号:S512.1 ""文献标识码:A ""文章编号:1673-1794(2024)02-0016-05
作者简介:万家乐,安徽农业大学农学院硕士生,研究方向:小麦分子育种;吴美琴, 陈心怡,赵晓雪,常梦帅,安徽农业大学农学院硕士生;陈璨,安徽农业大学农学院讲师,博士;李亮,濉溪县五铺农场中级农艺师;通信作者:卢杰,安徽农业大学农学院副教授,博士,研究方向:小麦遗传育种(合肥 230036)。
基金项目:国家现代农业产业技术体系——国家小麦产业技术体系专项(CARS-03);安徽省重大科技专项“麦优异种质资源创新、重要新基因挖掘及分子设计育种应用”(2021d06050003)
收稿日期:2023-11-27
小麦是我国主要的粮食作物之一,种植面积占粮食作物的22%,随着人口增长及环境变化,高产已成为我国小麦育种及粮食生产的重要目标之一[1]。穗数是产量三要素之一[2-3],属数量性状,表现为连续变异,由多基因控制,受环境因素影响[4]。品种间单株成穗数存在差异,受基因与环境共同作用[5-6]。因此,开展小麦单株穗数的主效、稳定QTL鉴定,挖掘候选基因及开发实用性分子标记,对高产小麦新品种培育具有重要指导意义[4]。
前人对小麦单株成穗数开展了大量研究。王升星等[7]利用安农0711/烟农19 BC1F2回交群体在1A、5D上检测到2个控制单株穗数的QTL,分别解释2.86%和1.92%的表型变异。严峻等[8]利用硬粒小麦与野生二粒小麦构建的重组自交系在5B染色体上检测到两个控制单株穗数的QTL,合计表型解释率为44.7%。胡洋山[9]在CN18/T1208构建的RIL群体检测到26个单株穗数相关的QTL,将cQTN.sicau-2D.2定位于70.45~81.75Mb区间内,表型解释率为3.91%~13.25%。Naruoka等[10]构建的RIL群体定位到控制单株穗数QTn.mst-6B位点,解释9%~17%的表型变异。闫林等[11]将单株穗数定位4B染色体Xgwm495~Xgwm113区间内,其表型解释率为34.6%。Shah等[12]使用RIL群体在3A染色体定位到与单株穗数有关的一因多效的QTL。刘丽华等[13]利用248份育成品种检测到8个单株穗数QTL位点,其中,位于1B染色体的QTL可解释6.33%~8.73%的表型变异。王鑫等[14]利用矮杆小麦种质构建的分离群体在3A、6A、4B检测到三个控制单株穗数的QTL。Kazuhiro等[15]利用DH群体在1As染色体检测到控制单株穗数QTL,可解释62.9%表型变异。李俊周[16]利用郑州8761与川育35050构建的133个DH群体分别在3A、5A、7B染色体上检测到5个QTL位点,控制单株穗数QTL,可解释表型解释率为8.9%~12.1%。Wang等[17]利用小麦3228与京4839杂交的237份家系检测得到位于1A、1B、3B、3D、4D、5B、6A染色体上单株穗数QTL,其中位于3D染色体上的QTL可解释21.51%表型变异。上述研究表明,小麦单株穗数作为产量相关性状,受到广泛关注,研究亦表明该性状是受多基因控制的数量性状,目前尚无候选基因报道。尚需通过选择单株成穗数差异显著的研究材料,基于多年多点表型测定,进一步开展该性状遗传研究。
功能标记开发是基因内部的改变引起表型性状的变异多态性序列,而测序技术的进步对分子标记开发有着促进作用[18-19]。CAPS标记是利用限制性内切酶对扩增的特异引物PCR进行酶切的标记技术,其操作简单快速[20-21]。
本研究拟以单株穗数差异显著的小麦品种安农859和武农988构建的DH群体为研究材料,测定两年四个环境下双亲及DH群体单株穗数表型值,基于55K芯片进行单株穗数QTL分析,挖掘控制单株穗数的稳定主效QTL位点,开发CAPS分子标记,以期为小麦分子标记辅助选择育种提供有价值的参考。
1 材料和方法
1.1 试验材料
供试材料为单株穗数差异显著的小麦品种安农859和武农988及构建的135份DH群体,分别于2021—2022年度、2022—2023年度种植在合肥市庐阳区高新技术农业示范园(31.90N,117.20E)、淮北市濉溪县五铺农场(33.70N,116.70E)、亳州市涡阳县(33.75 N,116.40E)三地,每个家系2行区,2.0 m行长,0.22 m行距,常规大田管理。
1.2 表型数据测定
在小麦灌浆期从双亲及每家系中选择10株长势一致单株,统计单株穗数。
1.3 数据处理
利用Excel软件对表型数据进行描述性统计,相关性分析、方差分析利用IBM SPSS Statistics25软件完成,正态分布图由Origin2022绘制。DNA序列比对由SnapGene完成,广义遗传力和方差分析由R语言完成,使用U检验对开发的标记进行显著性检验。
1.4 遗传图谱构建和QTL定位
用小麦55K SNP芯片对135份家系进行基因分型,去除亲本中缺失、杂合、未分型、未连锁的标记,最终保留16445个纯合多态性标记,利用IciMapping4.1软件完备复合区间作图法(ICIM)进行QTL分析,将LOD阈值设置为2.5,将3个及3个以上环境、表型解释率大于15%的QTL定义为主效稳定位点,以“Q+sn+ahau+染色体编号+次序”为命名方式[22]。
1.5 CAPS标记开发
根据两端侧翼标记的信息,在中国春小麦参考基因组查询目标区间内注释基因,筛选出高信度的注释基因,并设计特异性扩增引物,克隆测序,使用SnapGene软件比对测序结果。将亲本中存在多态性位点进行标记开发,根据有差异位点在NEB网站寻找合适的限制性内切酶。对亲本和DH群体进行酶切检测,结合琼脂糖凝胶电泳结果,统计安农859与武农988带型,与安农859带型一致记作为2,与武农988带型一致记作为0,结合135份DH群体家系带型与单株穗数表型,进行U检验。
2 结果分析
2.1 单株穗数表型数据分析
由表1可知,两年四个环境下亲本安农859单株穗数表型均显著高于武农988(Plt;0.01),DH群体的单株穗数在各个环境下呈连续性分布。DH群体单株穗数表型均值介于双亲之间,5个环境偏度和峰度绝对值小于2,符合正态分布(如图1),符合QTL分析要求。表型变异系数介于32%~47%之间,广义遗传率为0.65。表明DH群体单株穗数表型变异广泛,存在超亲分离现象,属于多基因控制的数量性状。
相关分析表明(表2),2023GY与2023HB、2022HF、2022GY、BLUP达到极显著相关,相关系数分别为0.33、0.35、0.28、0.70。2022HF与2022GY达到极显著相关,相关系数为0.43。
2.2 小麦单株穗数QTL鉴定分析
基于小麦55K芯片筛选到16445个多态性SNP和4个环境下单株穗数表型值,利用ICIM算法,在DH群体中共检测到21个单株穗数QTL,分布于1B、1D、2A、3D、4A、4B、4D、6A、7D等9条染色体上,其中,只在1个环境中检测到的QTL共11个,占52.38%;Qsn-ahau-4D.2在2个环境中检测,表型解释率为17.66%~19.19%;Qsn-ahau-4D.1位点在3个环境被检测,表型解释率为11.20%~32.71%,物理区间为62.88~132.96Mb;Qsn-ahau-4B.2于5个环境下均被检测到,LOD值为5.43~20.16,表型解释率为16.92%~49.98%,加性效应为1.32~3.69个穗数,增效等位基因来自安农859,侧翼标记分别是AX-95004669和AX-109580651,物理区间为29.19~31.84Mb,表明该位点是一个主效、稳定的QTL。可见,单株有效穗数是受数量基因控制的性状,存在稳定主效的QTL。
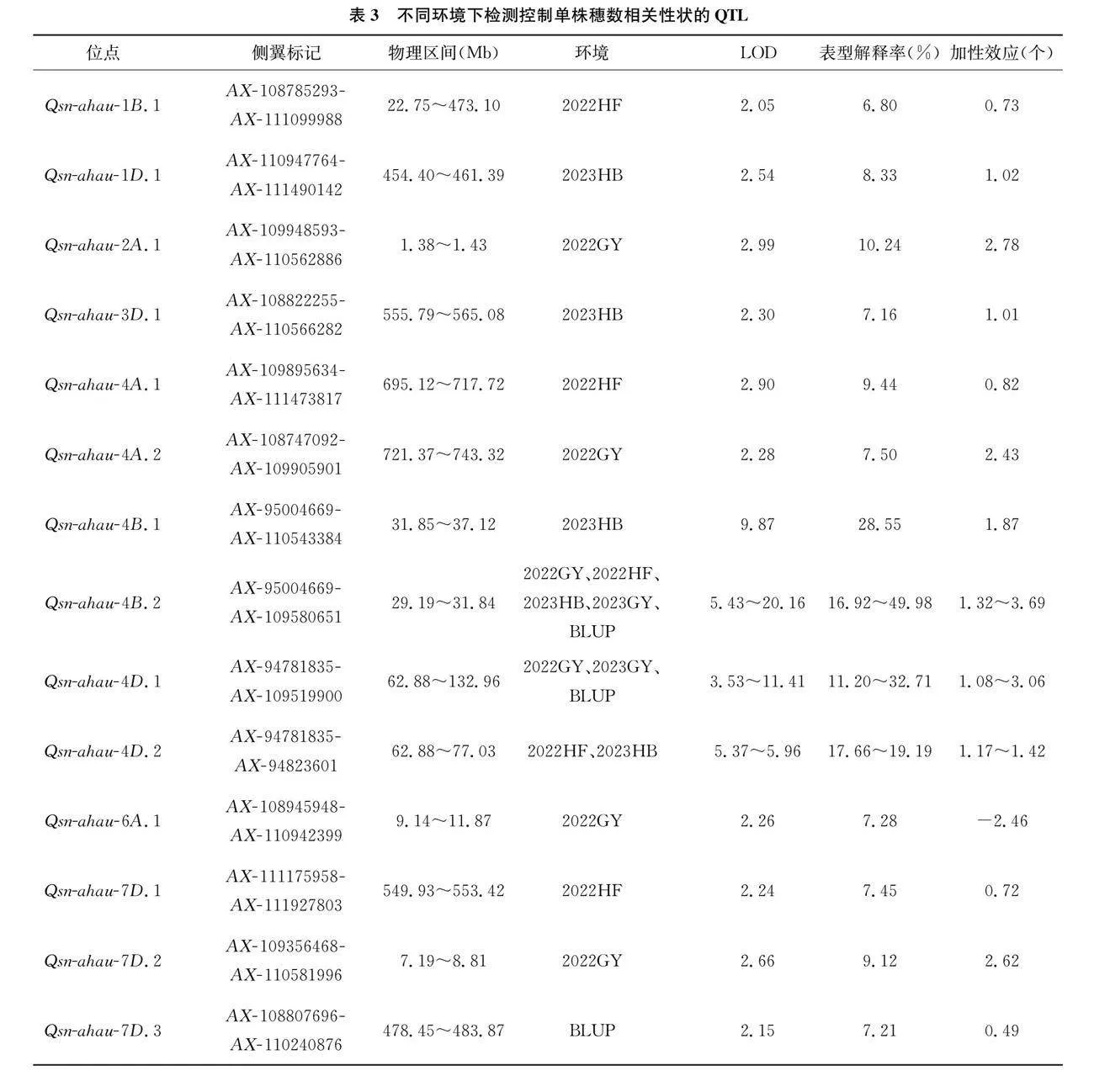
2.3 候选基因挖掘及CAPS标记开发
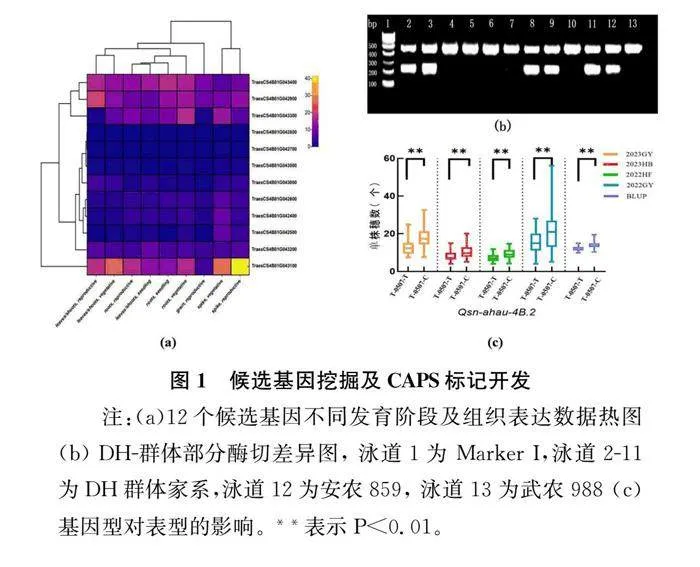
为进一步研究4B染色体上主效稳定QTL(Qsn-ahau-4B.2)候选基因,通过公共表达数据库expVIP (wheat-expression.com)获取该位点2.65Mb区段内不同发育阶段不同组织的表达数据,并利用R4.1.2进行热图绘制(图1a),并通过基因注释,共有12个高可信度的注释基因,基因序列克隆分析表明,DH群体双亲在TraesCS4B01G042700基因起始密码子ATG上游1769bp存在差异位点,安农859的变异类型T,武农988变异类型为C,利用NEB网站(https://www.neb.cn/),开发1个CAPS标记,命名为T-0507。对安农859、武农988及其DH群体家系基因型鉴定,其正向引物为:GCCTAGCACAAGCACGAGT,反向引物为:ATGAGGCCGCGTATACTGATG,PCR扩增的退火温度为59℃,限制性内切酶HhaI可以将安农859切割成257bp和232bp2个片段,在135份DH群体家系中分离比为1.25:1,其带型清晰,扩增稳定。在2023GY、2023HB、2022HF、2022GY环境中,含安农988等位基因的家系单株穗数的平均值分别为12.89、8.13、7.15、15.04,含武农859等位基因的家系单株穗数的平均值为18.01、10.43、9.19、21.59,U检验分析差异极显著,表明TraesCS4B01G042700是单株穗数的候选基因,有待于进一步深入研究。
4 讨论
单株穗数是产量相关性状,易受环境的影响,属于数量性状。本研究在4B染色体上检测到一个单株穗数主效QTL位点Qsn-ahau-4B.2,物理区间为29.19~31.84Mb。前人在4B染色体上也有检测到单株穗数QTL的报道,其中,Deng 等[23]使用亲本Am3在4B染色体物理区间442.59~482.82Mb检测到一个主效QTL位点QSn.sdau-4B,侧翼标记为Xwmc657-Xgwm11,可解释67.6%表型变异。苏志芳等[24]利用农大015 和复壮30 为亲本构建RIL群体检测到在4B染色体物理区间442.59~652.88Mb控制单株有效穗数的主效稳定QTL位点,侧翼标记为Xgwm107a~Xgwm513,表型解释率为13.91%。杨林等[2]检测到在4B染色体上标记区间Wms368~Wms113存在一个单株穗数主效QTL位点Qsp-4B-6,物理区间为59.60~256.31Mb。王婷婷等[25]利用RIL群体184份家系进行转录组测序,构建unigene遗传图谱,检测控制单株穗数QTL为 QSn-4B.1为主效QTL位点,贡献率大于23%。上述QTL位点与本研究定位QTL位点不同,表明本研究单株穗数主效QTL位点Qsn-ahau-4B.2是一个新主效QTL位点。
本研究表明TraesCS4B01G042700是单株穗数主效QTL(Qsn-ahau-4B.2)的一个候选基因。关于TraesCS4B01G042700基因功能,已有相关研究报道,Doebley等[26-27]发现在玉米中该基因编码转录因子Teosinte branched 1(Tb1),通过抑制主茎上腋芽的生长和雌花的发育,抑制蛋白的过度表达导致玉米分枝减少,Takeda等[28]过表达水稻Tb1同源物(OsTB1)导致顶端优势增加,腋芽生长减少,Aguilar-Martinez等[29]在拟南芥Tb1同源物 (BRC1)下调导致分支生长增加。在小麦中,Lewis等[30]发现Tb1过表达导致分蘖和穗数减少,Liu等[31]研究认为TraesCS4B01G042700参与了顶端优势和腋枝数量和长度的调控,进而调控小麦单株分蘖数。上述研究表明,TraesCS4B01G042700是调控单株茎蘖的基因,深入开展该基因调控单株穗数的机制研究对小麦产量形成及高产育种具有重要意义。
5 结论
本研究在4B染色体上定位到一个主效稳定的单株穗数QTL位点Qsn-ahau-4B.2,其侧翼标记为是AX-95004669和AX-109580651,单株穗数表型解释率为16.92%~49.98%,其物理区间为29.19~31.84Mb,根据QTL定位结果及CAPS标记在DH群体验证结果表明,增效等位基因来自多穗型亲本安农859。基于T-0507标记在群体材料分型结果,发现该标记在单株穗数U检验中显著相关,因此推测TraesCS4B01G042700可能为单株穗数的候选基因,并开发1个CAPS(T-0507)标记,可用于分子标记辅助育种。
[参 考 文 献]
[1]
孙果忠.我国小麦种业发展现状及未来建议[J].农业科技通讯,2021(7):4-8.
[2] 杨林,邵慧,吴青霞,等.小麦分蘖数和单株穗数 QTL 定位及上位性分析[J].麦类作物学报, 2013, 33(5): 875-882.
[3] 汤颖子.协调型小麦分蘖成穗规律及控制基因的QTL定位[D].成都:四川农业大学,2016:4-5.
[4] 刘静娴. “西农822×烟农999”重组自交系群体QTL定位与种质创新[D].泰安:山东农业大学,2023:2-10.
[5] 孙宇慧,刘天相,石善党,等.小麦穗粒数及千粒重主效QTL共定位区QC-7AL的精细定位及遗传效应分析[J].麦类作物学报,2018,38(11):1288-1292.
[6] 张广旭,王康君,谭一罗,等.小麦穗部产量性状研究进展与展望[J].农业与技术,2021,41(8):809-813.
[7] 王升星,牛影,陈聪灵,等.小麦单株产量及其相关性状的全基因组QTL分析[J].安徽农业大学学报,2017,44(4):720-725.
[8] 严俊,张玲玲,万兵,等.硬粒小麦与野生二粒小麦重组自交系群体穗部性状的QTL定位[J].四川农业大学学报,2011,29(2):147-153.
[9] 胡洋山, 汤颖子,李治, 等.小麦分蘖成穗数相关分子标记在重组自交系 (RIL) 群体中的有效性验证及实用性评价[J].麦类作物学报,2018,38(1): 8-15.
[10] Y N,E L T,P S L,et al.Identification of quantitative trait loci for productive tiller number and its relationship to agronomic traits in spring wheat[J].TAG. Theoretical and applied genetics. Theoretische und angewandte Genetik,2011,123(6):1047-1049.
[11] 闫林,王辉,孙道杰,等.小麦有效穗数的遗传分析及其SSR分子标记[J].中国农业大学学报, 2009 (5): 21-28.
[12] SHAH M M,GILL K S,BAENZIGER P S,et al.Molecular map of loci for agronomic traits on chromosome 3A of bread wheat[J].Crop Science,1999,39(6):1728-1732.
[13] 刘丽华,刘阳娜,周悦,等.基于高效SNP芯片的小麦产量相关性状全基因组关联分析[J].麦类作物学报,2023,43(11):1404-1416.
[14] 王鑫,马莹雪,杨阳,等.小麦矮秆种质SN224的鉴定及农艺性状QTL分析[J].作物学报,2016,42(8):1134-1142.
[15] SUENAGA K,KHAIRALLAH M,WILLIAM H M,et al.A new intervarietal linkage map and its application for quantitative trait locus analysis of\" gigas\" features in bread wheat[J].Genome,2005,48(1):65-75.
[16] 李俊周.小麦穗部相关性状的遗传与QTL分析[D].郑州:河南农业大学,2005:30-31.
[17] WANG J, LIU W, WANG H, et al. QTL map of yield-related traits in the wheat germplasm 3228[J]. Euphytica, 2011, 177: 277-292.
[18] 张福彦,范家霖,陈晓杰,等.小麦粒重相关基因的遗传定位和分子标记辅助育种进展[J].植物遗传资源学报,2020,21(3):507-516.
[19] LIU Y,HE Z,APPELS R,et al. Functional markers in wheat: current status and future prospects[J].Theoretical and applied genetics,2012,125:1-10.
[20] 杨保安,张建伟,张福彦,等.81份国外小麦品种(系)Puroindoline基因分子鉴定与分析[J].核农学报,2014,28(5):817-824.
[21] 赵雪,谢华,马荣才.植物功能基因组研究中出现的新型分子标记[J].中国生物工程杂志,2007(8):104-110.
[22] CHO Y G,EUN M Y,KIM Y K,et al. The semidwarf gene, sd-1, of rice (Oryza sativa L.). I. Linkage with the esterase locus, Estl-2[J].Theoretical and applied genetics,1994,89:49-53.
[23] DENG S,WU X,WU Y,et al.Characterization and precise map of a QTL increasing spike number with pleiotropic effects in wheat[J].Theoretical and Applied Genetics,2011,122:281-289.
[24] 苏志芳.小麦重要农艺性状QTL的定位[D].呼和浩特:内蒙古农业大学,2008:31-34.
[25] 王婷婷.小麦TL-RIL群体株型和穗部性状的QTL分析及应用[D].泰安:山东农业大学,2023:26-42.
[26] DOEBLEY J,STEC A,HUBBARD L.The evolution of apical dominance in maize[J]. Nature,1997,386(6624):485-488.
[27] DOEBLEY J.The genetics of maize evolution[J].Annu. Rev. Genet.,2004,38:37-59.
[28] TAKEDA T,SUWA Y,SUZUKI M,et al.The OsTB1 gene negatively regulates lateral branching in rice[J].The Plant Journal,2003,33(3):513-520.
[29] AGUILAR-MARTINEZ J A,SINHA N.Analysis of the role of Arabidopsis class I TCP genes At TCP7,At TCP8, At TCP22,and At TCP23 in leaf development[J].Frontiers in plant science,2013,4:406.
[30] LEWIS J M,MACKINTOSH C A,SHIN S,et al.Overexpression of the maize Teosinte Branched1 gene in wheat suppresses tiller development[J].Plant Cell Reports,2008,27:1217-1225.
[31] LIU J,LUO W,QIN N,et al.A 55 K SNP array-based genetic map and its utilization in QTL map for productive tiller number in common wheat[J].Theoretical and Applied Genetics,2018,131:2439-2450.
QTL Mapping and Development of CAPS Markers for Ear Number per Plant in Wheat
Wan Jiale,Wu Meiqin,Chen Xinyi,Zhao Xiaoxue,Chang Mengshuai,Chen Can,Li Liang,Lu Jie
Abstract: The number of ears is one of the three components of wheat yield, and conducting research on the number of ears in wheat is of great significance for high-yield breeding and germplasm resource creation. This research used 135 DH populations from Annong 859/Wunong 988 as research materials to determine the phenotypic values of spike number per plant in five environments over two years. QTL analysis of spike number per plant was conducted based on the 55K chip data of the DH population, and CAPS molecular markers were developed. The results showed that a total of 21 QTLs related to the number of spikes per plant were detected on chromosomes 1B, 1D, 2A, 3D, 4A, 4B, 4D, 6A, 7D, etc. Among them, Qsn-ahau-4B.2 located on chromosome 4B was detected in all five environments, labeled with AX-95004669-AX-109580651 on the wing, with a physical interval of 2.65Mb, which can explain 16.92% to 49.98% of the variation. The additive effect was 1.32-3.69 spikes, and the synergistic allele was from Annong 859, which is a major and stable QTL. A polymorphic CAPS marker (Z-2700) was developed based on the candidate gene TracesCS4B01G042700 within the Qsn-ahau-4B.2 interval. The results of this study will provide reference for the study of the number of spikelets per plant in wheat.
Key words:wheat;number of spikes per plant;QTL localization;marker development
责任编辑:陈星宇
