伪狂犬病病毒感染猪脾淋巴细胞的转录组学分析
2024-06-03陈家骥周家芳胡庭俊
陈家骥,赵 怡,周家芳,胡庭俊,3*
(1.广西大学动物科学技术学院,广西南宁 530004;2.广西壮族自治区兽用生物制品工程研究中心,广西南宁 530004;3.广西畜禽繁育与疾病防控重点实验室,广西南宁 530004)
伪狂犬病病毒(Pseudorabies virus,PRV)是猪伪狂犬病(Pseudorabies,PR)的病原,具有高度传染性,对妊娠母猪和仔猪危害巨大。妊娠母猪感染会导致流产、木乃伊胎、死胎或产弱仔[1-2]。新生仔猪感染后会出现神经系统症状,包括尖叫、共济失调、腹痛等,病死率极高[2]。2011年以前通过经典的BarthaK61疫苗较好地控制了PRV的流行,但由于病毒变异出现毒力更强的变异毒株,致使疫苗保护力下降,PR也重新在中国流行起来[3-5]。
PRV属于疱疹病毒科水痘病毒属,具有天然的免疫逃避机制,会抑制猪的免疫系统[6]。脾脏是机体内最大的外周免疫器官,聚集了大量T/B淋巴细胞,在启动免疫应答过程中扮演着至关重要的角色[7]。组蛋白乙酰化酶(histone acetyltransferase,HATs)和组蛋白去乙酰化酶(histone deacetylase,HDACs)可通过乙酰化或去乙酰化作用影响基因的转录从而影响各生物学进程。病毒依赖HATs和HDACs调节组蛋白结构进而调控其基因表达[8]。
目前,高通量测序技术已被广泛用于病毒感染致病机制的研究,对了解病毒感染后机体生物学进程、病毒致病机制具有重要意义,为病毒防控提供重要依据。本研究利用PRV体外感染猪脾淋巴细胞,应用ELISA以及RT-qPCR检测组蛋白乙酰化的相关基因的转录和表达水平,采用转录组测序技术对差异表达基因进行GO和KEGG富集分析,寻找PRV感染与组蛋白乙酰化之间的联系,为后续深入研究其作用分子机制提供参考。
1 材料与方法
1.1 材料
1.1.1 病毒 PRV-GXLB-2013株由广西大学动物科学技术学院预防兽医学教研室分离鉴定并保存。
1.1.2 主要试剂 RPMI-1640培养基,美国Gibco公司产品;胎牛血清(FBS),上海逍鹏生物科技有限公司产品;青霉素与链霉素混合液、红细胞裂解液、PBS,北京索莱宝科技有限公司产品;40 μm细胞筛网,浙江硕华生命科学研究股份有限公司产品;HAT、HDAC ELISA检测试剂盒,泉州九邦生物科技有限公司产品;RNAiso Plus,宝日医生物技术(北京)有限公司产品;All-in-One 5×RT Master Mix、BlasTaqTM2× qPCR Master Mix,上海爱必梦生物科技有限公司产品;细胞DNA提取试剂盒,南京诺唯赞生物科技股份有限公司;其他试剂均为分析纯级别试剂。
1.1.3 主要仪器设备 -80 ℃低温冰箱,普和希健康医疗器械(上海)有限公司产品;CO2培养箱,德国宾德公司产品;高速台式冷冻离心机,德国艾本德公司产品;梯度PCR仪、实时荧光定量PCR仪、电泳凝胶成像仪,美国Bio-Rad公司产品;琼脂糖水平电泳仪,北京六一生物科技有限公司产品。
1.2 方法
1.2.1 制备猪脾淋巴细胞悬液 试验所用猪购自广西南宁某猪场,经检验为PRV、PCV2、PRRSV等病原阴性,PRV抗体阴性。无菌剖检,取猪脾脏,置入超净台中用预冷的PBS清洗3次。加入少量PBS,用灭菌注射器内芯进行研磨,后经40 μm细胞筛网过滤后将所得细胞悬液转入15 mL离心管。
加入红细胞裂解液,裂解15 min,800 r/min离心5 min后弃上清;加入PBS轻轻吹打混匀沉淀细胞,800 r/min离心5 min,弃上清;加入含10% FBS的RPMI-1640培养液,轻轻吹打混匀,经筛网过滤。收集细胞滤液,取25 μL细胞悬液于细胞计数板中计算细胞数量,用含100 mL/L FBS的RPMI-1640培养液将细胞调整至5×106/mL进行铺板[9]。
1.2.2 试验分组及处理 试验设置细胞对照组(C组)和PRV感染组(P组),每组3个重复,为C-1、C-2、C-3和P-1、P-2、P-3。将细胞悬液接种于6孔板内,每孔2 mL,置于37 ℃、体积分数为5%的CO2条件下培养6 h后,吸弃1 mL培养液,C组加入1 mL培养液、P组加入1 mL PRV病毒液(TCID50为10-8.25/0.1 mL,MOI=0.1)孵育2 h后,吸弃1 mL培养液并加入1 mL培养液孵育8、12、24、48 h。
1.2.3 PRV感染猪脾淋巴细胞核酸扩增及鉴定 使用Primer 6.0软件设计引物,由南宁捷尼斯生物有限公司合成,引物序列见表1。按照诺唯赞细胞DNA提取试剂盒说明书,提取PRV感染8 h时猪脾淋巴细胞中的DNA进行PCR扩增。对扩增产物进行琼脂糖凝胶电泳,观察扩增片段位置。

表1 引物序列
1.2.4 ELISA检测HAT和HDAC酶活力水平 弃去1 mL培养液后吸取剩余培养液轻轻吹打沉降在6孔板底部的脾淋巴细胞使其悬浮在培养基中,转移至EP管中,12 000 r/min 4 ℃离心5 min,收集上清液即得样品。严格参照试剂盒说明书进行测定。
1.2.5 RT-qPCR检测HAT1和HDAC1 mRNA转录水平 弃去1 mL培养液后吸取剩余培养液,轻轻吹打沉降在6孔板底部的脾淋巴细胞使其悬浮在培养基中,转移至RNase Free EP管中,1 500 r/min 4 ℃离心5 min,弃上清;每管加入1 mL PBS吹散后再次1 500 r/min 4 ℃离心5 min,弃上清,加入1 mL的RNAiso Plus,反复吹吸,静置5 min,按照TRIzol法提取,经评估合格后即得总RNA样品。按照反转录试剂盒说明书进行反转录得到cDNA,放入-80 ℃冰箱保存。引物设计、合成同1.2.3所述,引物序列见表1。按照BlasTaqTM2×qPCR Master Mix试剂盒说明书进行RT-qPCR,结果以GAPDH作为内参,使用2-ΔΔCt法计算各基因表达水平。
1.2.6 测序样品收集 按照1.2.5方法获取总RNA样品后,放入-80 ℃冰箱保存。
1.2.7 测序文库构建与测序 取-80 ℃保存的样品,去除rRNA后,构建链特异性文库,文库质检合格后采用Illumina NovaseqTM6000进行转录组测序。
1.2.8 RNA-Seq数据处理 由于在测序过程中伴随着核酸片段的打断以及接头等过程会导致序列数据参差不齐,所以需要对原始数据进行数据质控处理,使用Cutadapt[10]过滤掉不合格的序列后得到有效数据,使用Hisat2[11-12]对预处理后的有效数据(Valid Data)与猪参考基因组进行比对,并获取在参考基因组或基因上的位置信息,以及测序样本特有的序列特征信息。根据基因长度计算每个基因的FPKM,并计算映射到该基因的读数。计算差异倍数以及进行差异显著性分析,最终以|log2(FC)|>1,P<0.05为条件筛选出差异表达基因(DEGs)进行后续GO、KEGG等生物信息学分析。
1.2.9 差异表达基因RT-qPCR验证 为了验证测序结果是否准确,从DEGs中随机挑选10个mRNA进行验证。引物设计、合成同1.2.3所述,引物序列见表1。
1.2.10 数据统计与分析 试验结果采用SPSS 22.0软件独立样本t检验进行显著性分析。结果以“平均值±标准差”表示,*(P<0.05)表示差异显著,**(P<0.01)表示差异极显著。
2 结果
2.1 PRV感染猪脾淋巴细胞核酸扩增及鉴定
图1所示,C组均未检测出PRV扩增片段(388 bp),P组均检测出PRV扩增片段。由此可知,PRV已成功感染猪脾淋巴细胞。
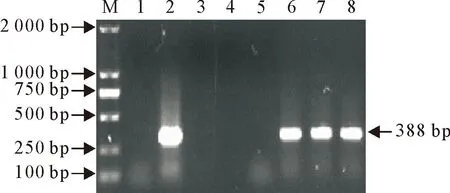
M.DNA标准DL 2 000;1.阴性对照;2.阳性对照;3~5.细胞对照组;6~8.PRV感染
2.1 ELISA检测HAT和HDAC酶活力水平
由图2A可知,PRV感染猪脾淋巴细胞8~24 h均能极显著上调HAT酶活力水平(P<0.01),而感染48 h时对HAT酶活力水平无显著影响(P>0.05)。

A.HAT酶;B HDAC酶A.HAT enzyme; B.HDAC enzyme
由图2B可知,PRV感染猪脾淋巴细胞8~24 h均能显著或极显著下调HDAC酶活力水平(P<0.05,P<0.01),而感染48 h时对HDAC酶活力水平无显著影响(P>0.05)。
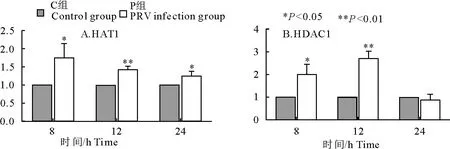
A.HAT1;B.RRV
2.2 RT-qPCR检测HAT1、HDAC1 mRNA转录水平
图3A所示,PRV感染猪脾淋巴细胞8~24 h均能显著上调HAT1 mRNA转录水平(P<0.05),其中12 h时能极显著上调(P<0.01)。
图3B所示,PRV感染猪脾淋巴细胞8 h时显著上调HDAC1 mRNA转录水平(P<0.05),12 h时极显著上调(P<0.01),24 h时则对HDAC1 mRNA表达水平无显著影响(P>0.05)。
2.3 PRV感染猪脾淋巴细胞转录组测序
2.3.1 原始数据质量分析结果 本研究对所测样品进行RNA质检合格后,进行测序,所得原始数据在去除接头和低质量的Reads后,样本的有效数据量分布在11.18 G~12.10 G,Q30%在96.78%~97.17%,GC碱基含量在50.5%~52.5%,具体信息见表2。以上结果说明测序数据质量良好,可用于后续的数据分析。

表2 测序数据质量统计
2.3.2 基因差异表达分析结果及差异基因表达聚类分析 通过对PRV感染组和细胞对照组比较,以|log2(FC)|>1且P<0.05为差异基因筛选条件。图4所示,共筛选出2 293个差异显著基因,其中726个基因表达上调,1 567个基因表达下调。并对所有基因进行聚类分析,结果如图5所示,本试验组内差异小,生物学重复性良好,组间差异大,表明PRV感染会引起猪脾淋巴细胞转录水平异常。
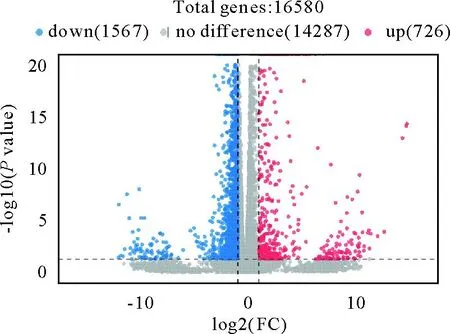
图4 PRV感染猪脾淋巴细胞的差异表达基因火山图
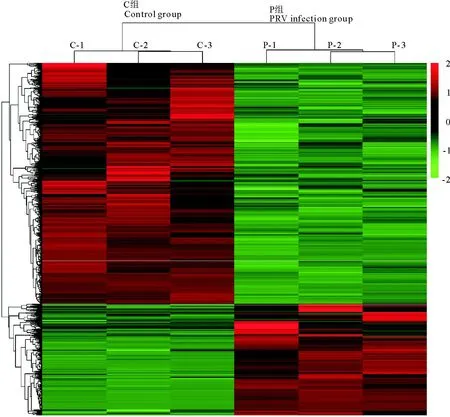
图5 差异转录基因的聚类分析(红色为表达上调,绿色为表达下调)
2.3.3 差异表达基因GO功能分析 根据GO注释对差异基因进行GO功能分析,并挑选显著性排名前20的功能制作GO富集因子气泡图,结果如图6所示。这些功能中有许多病毒感染和免疫相关的功能,例如病毒防御应答(defense response to virus)、免疫应答(immune response)、病毒基因组复制的负调控(negative regulation of viral genome replication)、炎症反应(inflammatory response)等功能。
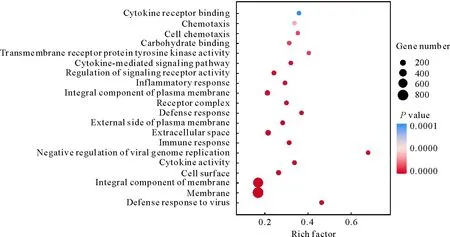
横坐标Rich factor为富集因子值(下同)The x-axis represents Rich factor values (the same applies to the subsequent text)
2.3.4 差异表达基因KEGG功能分析 根据KEGG注释对差异基因进行KEGG功能分析,挑选显著性排名前20的通路制作成KEGG通路富集因子气泡图。图7显示,这些差异表达基因主要被富集到单纯疱疹病毒感染(Herpes simplex virus 1 infection)等病毒感染通路;细胞凋亡(apoptosis)、坏死性凋亡(necroptosis)等程序性死亡通路;以及NOD样受体信号通路(NOD-like receptor signaling pathway)、Toll样受体信号通路(Toll-like receptor signaling pathway)、趋化因子信号通路(Chemokine signaling pathway)等免疫相关通路上。这些结果表明,PRV感染会影响宿主免疫防御反应相关信号通路,影响基因表达及蛋白质合成引起强烈的免疫反应。
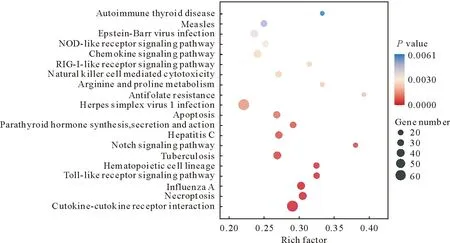
图7 KEGG富集因子气泡图
2.3.5 差异表达基因RT-qPCR验证 为验证高通量测序结果的可信度和准确性,随机挑选10条差异表达基因,其中3条基因测序结果为上调,7条基因测序结果为下调。通过RT-qPCR进行表达验证。结果如图8所示,10条基因趋势均与测序结果一致,表明本研究RNA-seq测序数据具有较高的准确性和可信度。
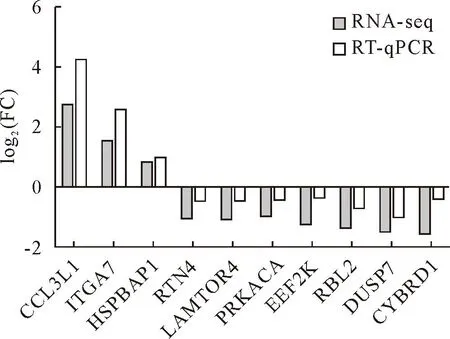
纵坐标为FC以2为底数的对数The y-axis represents the logarithm base 2 of FC
3 讨论
PRV感染猪后可在机体内建立终身感染机制,当机体抗体水平下降,潜伏的病毒就会导致机体发病。然而目前并没有有效的方法来清除潜伏的PRV,随着2011年PRV 2型毒株在我国卷土重来给我国猪养殖业造成了巨大的经济影响,亟需了解PRV的感染机制以及制定出相应的防控措施,因此对于PRV在宿主细胞中感染和复制的研究逐渐成为了研究热点。
组蛋白乙酰化是一种广泛存在于真核生物中的染色质修饰机制,主要通过HATs和HDACs对组蛋白上的赖氨酸进行修饰。HATs可增加组蛋白和非组蛋白调节蛋白中赖氨酸残基的乙酰基,而HDACs则具有相反的作用。前者的修饰通过开放的染色质结构激活RNA转录,而后者的修饰则抑制基因转录[13]。病毒感染可以影响宿主细胞的乙酰化修饰模式,调节蛋白质活性和相互作用,从而影响细胞生长、分化、代谢和免疫等生理和病理过程。有研究表明,抑制HDAC1可诱导DNA损伤反应导致双链DNA释放到细胞质中,激活cGMP-cAMP合成酶和下游的STING/TBK1/IRF3先天免疫信号通路从而抑制PRV感染[14]。也有学者研究发现,PRV感染可以诱导H2AX的磷酸化并导致DNA损伤反应敲除HDAC6可以抑制PRV感染,过表达HDAC6可以促进PRV感染[15];常雯茹发现过表达HDAC1促进PRV感染,沉默HDAC1抑制PRV感染[16]。上述研究表明,组蛋白乙酰化修饰在PRV感染过程中发挥着重要的调节作用。本研究中PRV感染早期12 h时HAT和HDAC修饰酶存在显著或极显著差异,这表明这一时间段是PRV感染和复制的关键节点。表明PRV感染会通过调节染色质上的组蛋白修饰来影响宿主细胞基因表达以及与宿主细胞互作,从而对细胞的生理和免疫进程产生一定的影响。这种变化可能与病毒侵入宿主细胞的免疫应答、病毒基因转录以及病毒复制等过程存在相关的调控通路和机制,结合本实验室前人研究,故选取12 h细胞样品进行转录组测序。
近年来高通量测序技术被广泛应用于病毒感染引起的差异转录组研究中,可为研究提供丰富的基础数据,进一步揭示病毒感染的机制。病毒感染宿主后,机体免疫相关通路会被激活,发挥抗病毒作用以清除病原并减轻自身损伤[17];李海敏等[18]通过高通量测序技术分析出PRV感染PK-15细胞差异基因聚焦于免疫等信号通路;有研究表明沉默lncA3可显著上调IRF1、IFNβ的转录水平,抑制PRV-Ⅱ的复制[19];PRV与细胞自噬存在关联,PRV感染早期促进细胞自噬,后期通过激活PI3K/AKT/mTORC1途径抑制自噬[20]。
本研究对PRV感染的猪脾淋巴细胞进行了转录组测序,对DEGs进行了生物信息学分析。结果显示,PRV感染后具有显著差异的基因有2 293个,其中转录显著上调的基因有726个,转录显著下调的基因1 567个。根据GO功能分析可以发现这些差异基因广泛分布于细胞膜、核、质中,调节细胞膜表面受体活性,参与信号转导,调节病毒基因组复制,影响细胞因子活性,介导炎症反应等生物学进程,这说明PRV感染会破坏宿主的物质代谢和生物合成过程从多个方面影响宿主细胞。KEGG功能分析则聚焦于免疫相关通路,包括Toll样受体信号通路、NOD样受体信号通路等通路。PRV感染后,通过激活TLR-NF-κB信号通路和AIM2炎症小体以增强小鼠的炎症反应[21]。NOD样受体能够激活NF-κB信号通路,引发促炎性因子的表达和释放,最终导致炎症的发生[22-23]。已有文献报道PRV感染会影响宿主免疫防御反应相关信号通路,影响基因表达及蛋白质合成,引起强烈的免疫反应,而这些通路也被证实与组蛋白乙酰化存在联系[24-26],但仍需要进一步研究其与PRV之间的联系。此外,KEGG结果显示DEGs还富集到了细胞凋亡、坏死性凋亡等程序性死亡通路,这可能与PRV感染导致的细胞损伤、炎症反应和免疫反应等多种机制相关。未来可对PRV感染导致的细胞死亡机制进行更深入的研究,更好地理解PRV的致病机制,为该病毒的防控提供理论基础。
本研究通过ELISA、RT-qPCR检测了PRV感染猪脾淋巴细胞HAT、HDAC的表达水平和转录水平,对PRV感染猪脾淋巴细胞12 h的样本进行了高通量测序并进行了生物信息学分析。这些差异表达基因大多被富集到了病毒性疾病、免疫、代谢和凋亡等生物学过程以及Toll样受体信号通路、NOD样受体信号通路等通路,可为进一步了解PRV感染、致病的分子机制提供参考。
