基于GEPIA 数据库分析胃癌和正常组织的差异表达基因及对预后影响
2024-05-11张紫涵陈明明桑圣刚张荣光
张紫涵,陈明明,刘 君,桑圣刚,张荣光,
(1.海南医学院公共卫生与全健康国际学院,海南 海口 571199;2.海南医学院基础医学与生命科学学院,海南 海口 571199;3.海南医学院第一附属医院检验科,海南 海口 570102)
胃癌细胞起源于消化道上皮细胞,患病率高且患病年龄年轻化,由于初期表现不明显确诊难度较大[1]。最新研究表明,2020 年全球范围内新增的胃癌病例已超百万,且发病率已经超越肺癌、乳腺癌、结直肠癌和前列腺癌[2]。每年约76.9 万人死于胃癌,死亡率位于癌症的前列[3,4]。目前综合治疗方案主要为手术、化学疗法和免疫治疗,这些方法虽有助于提高患者的生存率和生活质量,但中晚期胃癌的转移或复发常导致治疗失败[5-7]。因此深入了解胃癌的分子机制和生物学特性,寻找早期生物标记,并开发精准的治疗手段,可提高早期胃癌患者的治疗效果,减少中晚期胃癌的复发和转移风险,从而为患者争取更好的生存和生活质量。GEPAI数据库具有基因表达分析和自定义数据分析的功能,能提供预后分析、肿瘤及正常组织差异表达分析、基因相关分析等数据,可以对各种癌症和正常组织的基因表达差异及整体生存率等进行分析。本研究通过GEPIA 生物信息数据库筛选出胃癌与正常组织表达具有差异的前10 个基因,并对筛选的差异基因进行生存预后分析,讨论其在胃癌发生、发展以及疾病进展中的作用,为胃癌的诊断治疗提供参考。
1 资料与方法
1.1 差异基因的获得
访问GEPAI 数据库(http://gepia.cancer.pku.cn/),点击“Differential Genes”(差异基因)功能按钮,定义筛选条件为“Dataste”(数据集),疾病类型设为“STAD”[胃癌(Stomach adenocarcinoma,STAD)],调整|log2FC|和q-value 的cut off 值,分别为1.0 和0.01,最后,在“Differential Methods”(差异方式)部分选择“ANOVA”作为方法,并在“Chromosomal Distribution”(染色体分布)下拉菜单中选择“over-expressed”(过表达),对数据进行相关分析。
1.2 对筛选基因进行生存分析
进入GEPAI 数据库首页选择“Surval”(生存)与“Surval Plots”(生存图)选项,在Gene 框中依次输入肿瘤组织和正常组织中表达差异较大的基因。然后从“Methods” (方法)中选取“Overall Survival and Disease Free Survival” (总体生存期和无病生存期),设定Group cut off(截断值)为 “Median” (中位数),同时设置cut off-High(%)(最高截断)和cut off-Low(%)(最低截断)分别为“50”,对于“Hazards Ratio” (风险比率)和 “95% Confidence Interval”(95%置信区间)分别勾选“Yes” ,确定“Axis Units”(横轴的单位)为“Months”(月),“Datasets Selection” (数据集选择)选择“STAD”,最后点击“Plot”绘制在肿瘤和正常组织中差异表达基因的生存曲线和无病生存期(RFS)曲线。
2 结果
2.1 在胃癌组织和正常组织中基因的表达存在差异
在GEPAI 数据集中筛选了符合TCGA(癌症基因组图谱)、正常组织和GTEx(基因型和基因表达量数据库)一致性的样本619 例,其中包括408 例胃癌组织样本和211 例正常组织样本。筛选出了4 644 个胃癌组织与正常组织存在差异的基因。前10 名基因依次为:PRR11、CLDN7、TPX2、GNL3L、SKA3、ADHFE1、MTDH、CEP55、KIF11、KIF20B。对408 例胃癌组织和211 例正常胃组织样本进行基因表达差异分析。与正常胃组织相比,胃癌组织中PRR11、CLDN7、TPX2、GNL3L、SKA3、MTDH、CEP55、KIF11、KIF20BmRNA 表达水平升高,见图1~10,ADHFE1mRNA 表达水平降低,见图6。图1~10 中红色模块代表胃癌组织,灰色模块代表正常组织。

图1 PRR11 基因在胃癌和正常组织的表达Fig 1 Expression of PRR11 gene in gastric cancer and normal tissue

图2 CLDN7 基因在胃癌和正常组织的表达Fig 2 Expression of CLDN7 gene in gastric cancer and normal tissue

图3 TPX2 基因在胃癌和正常组织的表达Fig 3 Expression of TPX2 gene in gastric cancer and normal tissue
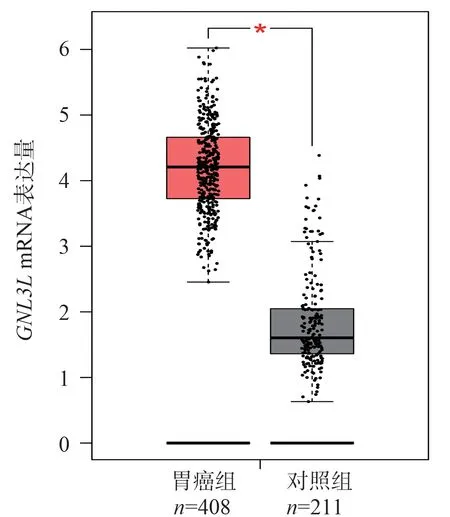
图4 GNL3L 基因在胃癌和正常组织的表达Fig 4 Expression of GNL3L gene in gastric cancer and normal tissue

图5 SKA3 基因在胃癌和正常组织的表达Fig 5 Expression of SKA3 gene in gastric cancer and normal tissue
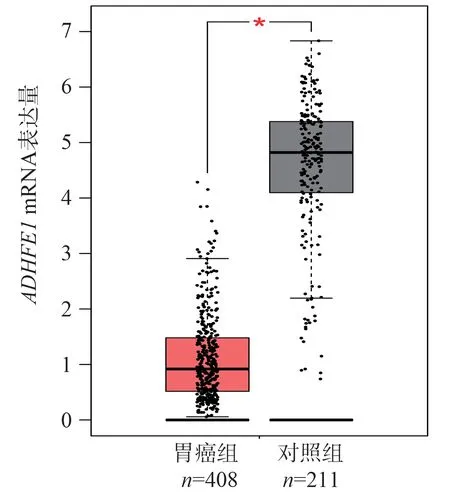
图6 ADHFE1 基因在胃癌和正常组织的表达Fig 6 Expression of ADHFE1 gene in gastric cancer and normal tissue
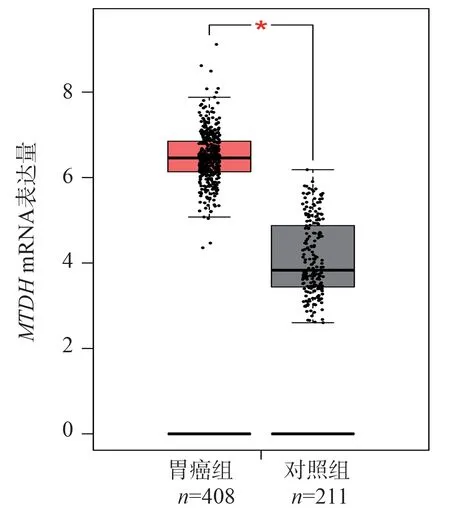
图7 MTDH 基因在胃癌和正常组织的表达Fig 7 Expression of MTDH gene in gastric cancer and normal tissue
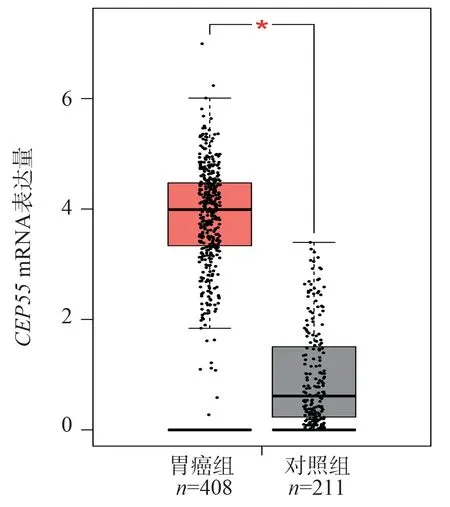
图8 CEP55 基因在胃癌和正常组织的表达Fig 8 Expression of CEP55 gene in gastric cancer and normal tissue
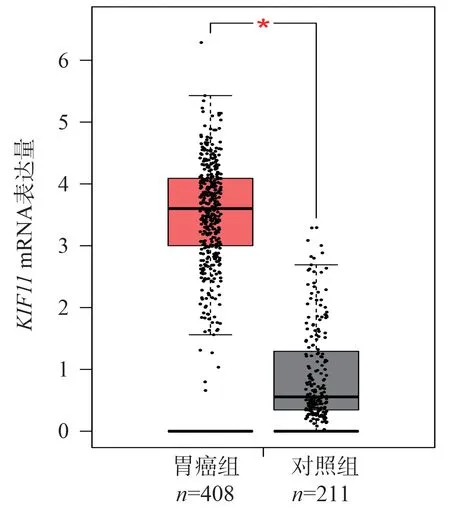
图9 KIF11 基因在胃癌和正常组织的表达Fig 9 Expression of KIF11 gene in gastric cancer and normal tissue

图10 KIF20B 基因在胃癌和正常组织的表达Fig 10 Expression of KIF20B gene in gastric cancer and normal tissue
2.2 基因差异表达对肿瘤患者生存期的影响
通过GEPIA 分析差异基因表达水平与胃癌患者预后的关系,对胃癌患者进行生存预后分析,得到生存曲线和RFS 曲线,结果显示:胃癌患者中PRR11、CLDN7、TPX2、GNL3L、SKA3、MTDH、KIF11、KIF20B高表达组与低表达组生存分析(见图11~20A)和RFS 分析(见图11~20B)无统计学差异。胃癌患者中ADHFE1高表达组生存时间(HR=1.4,LogrankP=0.047,见图16A)以及RFS(HR=1.7,LogrankP=0.0082,见图16B)均低于低表达组,两者具有统计学差异。胃癌患者中CEP55高表达组生存时间高于低表达组,两者具有统计学差异(HR=0.69,LogrankP=0.021,见图18A),CEP55高表达组与低表达组RFS 分析无统计学差异(见图18B)。

图11 基于PRR11 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 11 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with PRR11 gene expression
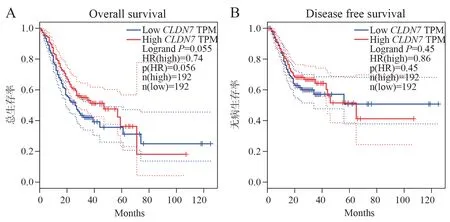
图12 基于CLDN7 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 12 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with CLDN7 gene expression

图13 基于TPX2 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 13 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with TPX2 gene expression

图14 基于GNL3L 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 14 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with GNL3L gene expression
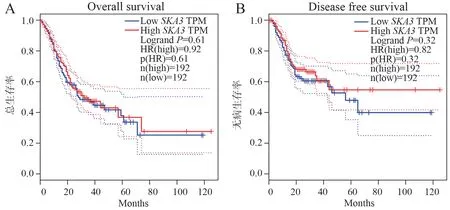
图15 基于SKA3 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 15 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with SKA3 gene expression

图16 基于ADHFE1 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 16 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with ADHFE1 gene expression
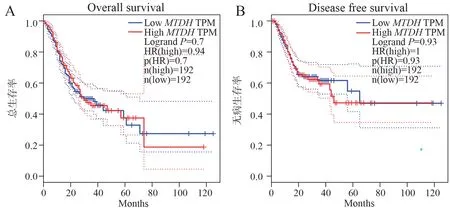
图17 基于MTDH 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 17 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with MTDH gene expression

图18 基于CEP55 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 18 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with CEP55 gene expression

图19 基于KIF11 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 19 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with KIF11 gene expression
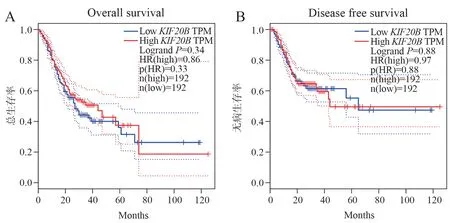
图20 基于KIF20B 基因表达的胃癌患者生存分析(A)和RFS 分析(B)Fig 20 Survival analysis (A) and RFS analysis (B) of gastric cancer patients with KIF20B gene expression
3 讨论
胃癌是世界上最常见的肿瘤之一,发病率和死亡率较高[8]。尽管近些年对该病的诊疗技术有所发展,然而在中国,由于胃癌早期无显著征兆,多数病例病情恶化后才被救治,病死率高[9,10]。寻找能够预测其生存时间的指标具有重要意义[11]。在胃癌疾病发展过程中,某些基因的异常表达会改变肿瘤组织细胞的结构和功能,这些差异表达的基因可能对早期诊断和预后预测具有重要价值。
本研究利用GEPIA 数据库筛选出正常组织和胃癌组织的差异基因,差异排名前10 位的基因为PRR11、CLDN7、TPX2、GNL3L、SKA3、ADHFE1、MTDH、CEP55、KIF11、KIF20B。通过分析这些基因,发现与胃癌患者生存时间密切相关的基因为ADHFE1和CEP55基因。对上述基因进行RFS 曲线分析,发现ADHFE1基因表达与患者的预后存在关联。这些基因有可能成为胃癌诊断和预后分析的重要生物标志。
ADHFE1( Alcohol dehydrogenase 1 containing iron)基因编码的含铁的醇脱氢酶1 是铁活化醇脱氢酶家族的成员,在多种生理过程中起着多种作用[12,13]。研究表明,ADHFE1在结直肠癌组织中表达下调和高甲基化,ADHFE1高表达水平与肿瘤患者的良好预后呈正相关,表明其在结直肠癌中具有抑瘤作用[14-16]。本研究结果显示,胃癌组织中ADHFE1基因的表达显著低于正常组织,ADHFE1基因高表达组患者生存时间明显低于低表达组。RFS分析结果显示ADHFE1基因与胃癌患者的无病生存期呈现负相关。
CEP55(Centrosome protein 55)基因编码的中心体蛋白55 是中心体相关蛋白家族的成员[17]。此基因位于第10 号染色体长臂2 区3 带[18]。已有研究证实,在许多人类肿瘤组织中发现了CEP55的高表达,例如乳腺癌和子宫内膜癌, 并且CEP55基因与肿瘤的恶性程度、侵袭以及不良预后密切相关[19,20]。在本研究中,CEP55基因高表达组和低表达组的生存时间存在显著差异,CEP55基因高表达组患者生存时间显著高于低表达组。
综上所述,差异基因的不同表达水平在胃癌的发生、发展中可能起到重要的作用,并与胃癌的生存预后密切相关,其中ADHFE1的低表达和CEP55的高表达是发生胃癌的危险因素。这些发现为胃癌早期诊断和预后预测研究提供了新的重要线索和基础,探明这些基因在胃癌发生、发展中的作用及分子机制是值得未来研究关注的方向。
作者贡献度说明:
张紫涵:数据处理、作图并撰写论文;陈明明:文献检索并处理数据;刘君:论文的审阅与修改;桑圣刚、张荣光:全程参与、指导数据分析和作图并修改论文。
所有作者声明不存在利益冲突关系。
