2019年云南省昆明市柯萨奇病毒A组16型毒株的分离鉴定及其VP1基因遗传特征分析
2024-04-19刘煜菡张名郭伟冯昌增许丹菡马绍辉
刘煜菡,张名,郭伟,冯昌增,许丹菡,马绍辉
中国医学科学院北京协和医学院医学生物学研究所云南省重大传染病疫苗研发重点实验室,云南 昆明 650118
柯萨奇病毒A组16型(Coxsackievirus A16,CVA-16)属于小核糖核酸病毒科(Picornaviridae)的肠道病毒属(Enterovirus),为单股正链无包膜的RNA 病毒。CVA16 基因组长约7 400 bp,结构蛋白VP1~VP4 组成病毒衣壳,其中VP1 为最重要的抗原决定簇。CVA16与肠道病毒71型(enterovirus 71,EVA71)常交替或共同流行,是手足口病(hand,foot and mouth disease,HFMD)的主要病原体[1-3]。随着EV71 疫苗上市,EV71 已不是HFMD 的主要病原,而CVA16 仍然是其主要病原[4-6]。CVA16 仍引起的HFMD 通常为自限性,但仍有少数患者可发展为肺水肿、脑干脑炎、无菌性脑膜炎等,甚至死亡[7-9]。该病毒RNA 聚合酶无修复错配机制,极易发生重组和突变,从而导致毒力改变[10-12]。因此,本研究对云南省昆明市引起HFMD 的20 株CVA16 进行分离鉴定、全VP1基因测序及遗传进化分析,为制定有效的CVA16 防控策略提供参考。GenBank 上登录的CVA16 毒株的全基因组序列CVA16/guangdong106/CHN/2018(MW030434)和R34/YN/CHN/2012(KY425534)为参考序列,利用Primer-BLAST 对引物序列进行分析,设计覆盖CVA16 毒株的全VP1序列引物CA16VP1-F(5′-GGCYTTGGCAGCAGCTCARG-3′)和CA16VP1-R(5′-ACCACCCTATAGTTGCCCAC-3′)。引物由昆明擎科生物科技有限公司合成。根据PrimeScriptTMOne Step RT-PCR Kit 说明书,利用引物进行RT-PCR 反应。反应条件:50 ℃逆转录35 min,94 ℃逆转录2 min;94 ℃变性0.5 min,52 ℃退火0.5 min,72 ℃延伸1 min,共35 个循环;最后72 ℃再延伸10 min。取PCR 产物2 μL,进行1%琼脂糖凝胶电泳鉴定。
1.6 测序及序列分析 将阳性扩增产物送昆明擎科生物科技有限公司测序,使用NCBI BLAST 工具对序列进行比对。从NCBI GenBank 下载其他CVA16 参考株的VP1基因序列,应用Geneious 9.0.2 和Mega 7.0软件对序列进行分析处理,并构建系统进化树。
1 材料与方法
1.1 毒株及细胞 CVA16 分离株为从2019 年昆明市某医院根据《手足口病诊疗指南(2018)》诊断并收集的HFMD 患者158份粪便中分离获得,共26株;人胚肺二倍体KMB-17 细胞和Vero 细胞由中国医学科学院医学生物学研究所课题组保存并提供。
1.2 主要试剂 病毒RNA 提取试剂盒Axygen Body Fluid DNA/RNA Miniprep Kit购自美国Axygen公司;PrimeScriptTMOne Step RT-PCR Kit 购自日本TaKaRa公司。
1.3 病毒培养 将CVA16 分离株分别接种24 孔板中生长状态良好的Vero 和KMB17 细胞,37 ℃,5%CO2条件下培养5 ~7 d。倒置显微镜每日观察细胞状态,无论细胞是否出现细胞病变效应(cytopathic effect,CPE)均连续盲传2代。
1.4 病毒RNA 提取 按照病毒RNA 提取试剂盒Axygen Body Fluid DNA/RNA Miniprep Kit 说明书提取CPE分离物。
1.5 引物设计及全CVA16VP1基因扩增 以NCBI
2 结果
2.1 CVA16VP1基因扩增产物鉴定 从2019年HFMD患者158份粪便中共分离获得8株KMB-17分离株和18株Vero分离株中,随机挑选20株CVA16云南分离株进行全VP1基因扩增。经1%琼脂糖凝胶电泳分析,可见约1 100 bp的特异性条带。测序结果经NCBI BLAST 比对,并提交至GenBank。将20 株云南分离株分别命名为67V3/YN/CHN/2019、69V3/YN/CHN/2019、82V3/YN/CHN/2019、84K3/YN/CHN/2019、84V3/YN/CHN/2019、86V3/YN/CHN/2019、87V3/YN/CHN/2019、104V3/YN/CHN/2019、111V3/YN/CHN/2019、116V3/YN/CHN/2019、120V4/YN/CHN/2019、122K4/YN/CHN/2019、122V5/YN/CHN/2019、124K3/YN/CHN/2019、124V3/YN/CHN/2019、K31/YN/CHN/2019、K33/YN/CHN/2019、K82/YN/CHN/2019、K127/YN/CHN/2019、K132/YN/CHN/2019;登录号为MI532476-MI532496。
2.2VP1序列遗传进化分析 将获得的20 株完整CVA16VP1序列与GenBank 中登录的70 株CVA16代表株进行序列比对,并以邻近法构建系统进化树,显示Bootstrap 值≥75%的节点。结果表明,昆明市2019 年CVA16 存在2 种基因亚型共同流行:B1a 和B1b。其中17株CVA16属于B1b基因型,3株属于B1a基因型。B1a 基因型中,87V3/YN/CHN/2019、124K3/YN/CHN/2019 和124V3/YN/CHN/2019与广东分离株CVA16-77 GD-CHN 2019-02(MW-197387)单独处于1 个分支群,并且亲缘关系密切(核苷酸同源性为97.5% ~98.7%);B1b 基因型中,67V3/YN/CHN/2019、104V3/YN/CHN/2019、69V3/YN/CHN/2019、111V3/YN/CHN/2019、K82/YN/CHN/2019、K31/YN/CHN/2019 和K33/YN/CHN/2019 与2019 年云南文山株J120-WS-YN-CHN-2019(LC533791)和大理株E44-DLYN-CHN-2019(LC555956)分离株亲缘关系较近;84K3/YN/CHN/2019和84V3/YN/CHN/2019 与2015 年北京分离株S3450/BJ/CHN/2015(MT553-173)亲缘关系较近;86V3/YN/CHN/2019、116V3/YN/CHN/2019、120V4/YN/CHN/2019、122K4/YN/CHN/2019、122V5/YN/CHN/2019 和K127/YN/CHN/2019 与2019 年云南曲靖分离株330-QJ-YN-CHN-2019-CV-A16(LC603099)亲缘关系较近;K132/YN/CHN/2019 和82V3/YN/CHN/2019 与2019 年云南曲靖分离株178-QJ-YN-CHN-2019-CV-A16(LC603076)亲缘关系较近。见图1。
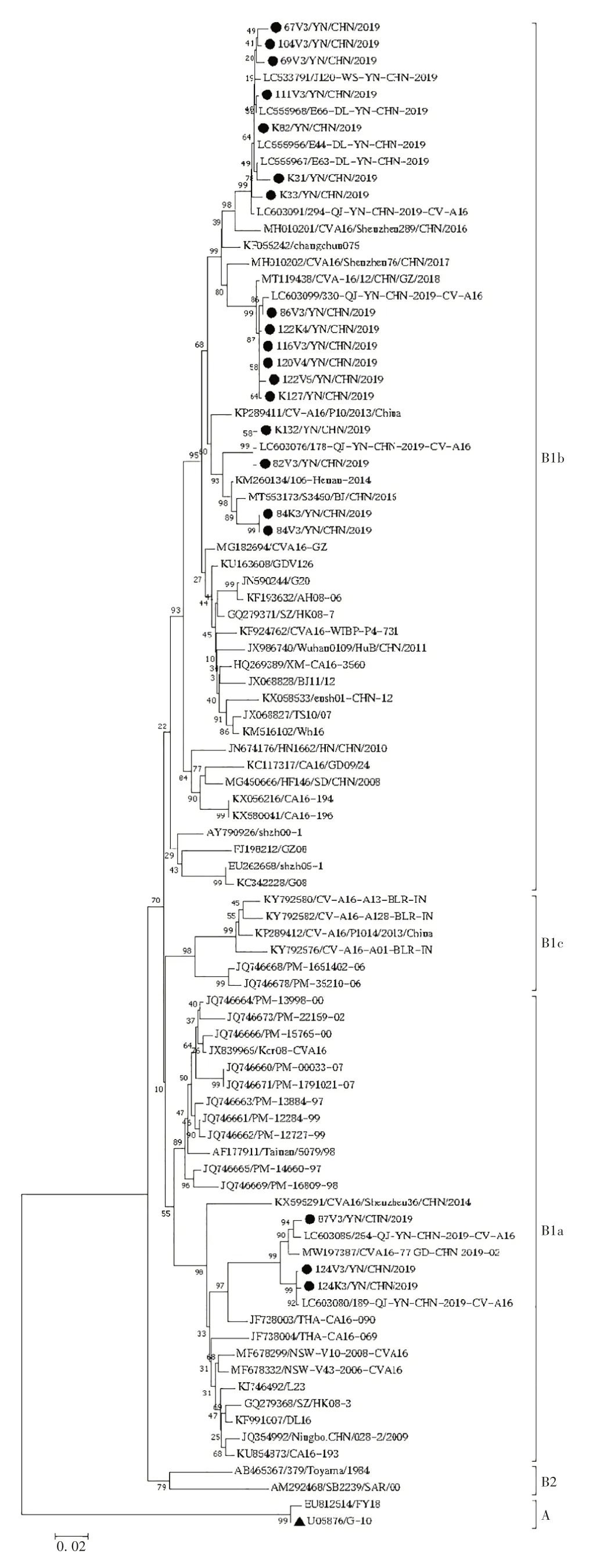
图1 CVA16分离株与参考毒株VP1区系统进化树Fig.1 Phylogenetic tree of VP1 region of CVA16 isolates and reference strains
2.3 全CVA16VP1核苷酸序列分析 对20株CVA16全VP1基因的核苷酸序列两两比对发现,其序列间同源性为87.3% ~100%,其中124V3/YN/CHN/2019 和K31/YN/CHN/2019 同源性最低。17 株CVA16 B1b基因型分离株同源性为93.8%~100%,与国内其他B1b 分离株同源性为91.1% ~99.2%;3株B1a 基因型分离株同源性为98.0% ~98.1%,与国内其他B1a 分离株同源性为88.7% ~98.7%;17株B1b 基因型分离株与其他3 株B1a 基因型分离株同源性为87.4%~88.4%。NCBI BLAST比对发现,84K3/YN/CHN/2019 和84V3/YN/CHN/2019与S3450/BJ/CHN/2015的核苷酸同源性最高,为98.20%;116V3/YN/CHN/2019、120V4/YN/CHN/2019、122K4/YN/CHN/2019、K127/YN/CHN/2019 和122V5/YN/CHN/2019 与2018 年广东分离株CVA16/12/CHN/GZ/2018 同源性最高,为99.33%~100.00%;其余均与2019 年云南不同区域分离株同源性最高,为99.21%~99.89%。见表1。
2.4 全CVA16VP1氨基酸序列分析 20株CVA16全VP1基因的氨基酸序列两两比对发现,其序列间同源性为97.6%~100.0%,其中87V3/YN/CHN/2019与122V5/YN/CHN/2019同源性最低,为96.6%。17株CVA16 B1b基因型分离株同源性为98.3%~100%,与国内其他B1b 分离株同源性为97.3% ~99.0%;3株B1a基因型分离株同源性均为99.3%,与国内其他B1a分离株同源性为98.3%~99.7%;17株B1b基因型分离株与其她3株B1a基因型分离株同源性为97.3%~98.7%。同时也发现多个氨基酸变异。见表2。

表2 20株CVA16分离株与6株云南省分离株CVA16全长VP1氨基酸序列比对Tab.2 Amino acid sequence comparison of full-length VP1 between 20 CVA16 strains and 6 isolated CVA16 strains in Yunnan Province
3 讨论
CVA16VP1基因的平均进化率约为4.49× 10-3替代/(位点·年),其VP1基因是已经广泛用于CVA16的分子流行病学研究的靶基因[13-15]。根据CVA16 全长VP1基因,被分为基因型A、B1和B2,基因型B1又分为亚基因型B1a、B1b和B1c[16-18]。自2008年HFMD大规模暴发以来,中国大陆流行的CVA16 主要为B1基因型,其中又主要为B1a 和B1b[19-20],另外,个别地区偶尔也会分离获得B1c 毒株[21]。本研究分离获得的CVA16 毒株主要为B1b 基因亚型,其次为B1a 基因亚型。2019 年山东青岛流行的基因型同为B1a 和B1b[22];2019 年云南怒江州流行的基因型除分离获得1 株B1c,其余均为B1b[21];2019 年云南文山州流行的基因型同为B1a 和B1b[23];2010—2019 年北京流行的基因型为B1a、B1b 和B1c,但主要为B1b[13];2009 — 2015 年云南昆明流行的基因型为B1a 和B1b[8]。提示昆明地区长期同时存在B1a 和B1b 流行,而以B1b为主。
本研究通过对CVA16全VP1基因的核苷酸和氨基酸同源性比对,发现无论是B1a还是B1b分离株均与GenBank 上最新参考株(2018 和2019 年)的核苷酸及氨基酸同源性最高,且这些分离株也存在一定差异。提示不同或相同地域分离获得的CVA16毒株可能随环境影响发生一定进化。
肠道病毒的VP1 蛋白是病毒的主要中和抗原位点[24],主要受宿主免疫系统的选择压力影响。本研究通过CVA16分离株的全长VP1基因氨基酸序列比对发现,与其他中国株比较以及相互之间也存在多个位点变异,但其氨基酸序列同源性均高于97.3%。另外有研究表明,CVA16 VP1 800-810为其中和表位区域[25],而本研究中,分离株及参考株均未发生变异,这有利于CVA16 疫苗的相关研究。但这些突变位点是否能增加病毒的免疫逃逸机会,并继续在病毒群体中积累,仍需进一步研究。
此外,本研究利用KMB-17 和Vero 细胞对病毒进行分离,经过3 次盲传,发现Vero 细胞分离毒株获得的CVA16要多于KMB-17细胞,提示CVA16对Vero细胞的敏感性高于KMB-17 细胞。这可能与不同细胞表面受体存在差异,从而导致其敏感性不同有关,但尚需进一步研究。
本研究对2019 年云南省昆明市流行的CVA16毒株进行了分离鉴定以及全VP1基因序列分析,有利于了解该地区CVA16 毒株的进化和变异情况,扩展了CVA16 基因库中数据,对加强CVA16 的监测,以及进一步了解我国流行的HFMD 的遗传进化趋势提供了实验室数据。
