线粒体COI 基因序列的DNA 条形码在南海北部多鳞鱚鉴定中的应用
2024-04-14蔡承志
蔡承志
(茂名市农业科技推广中心,广东 茂名 525000)
多鳞鱚在中国近海都有产出,属于比较重要的经济鱼类,又因其肉质细腻鲜美,深受人们的喜爱。作为一种小型底栖鱼类,多鳞鱚也是许多大型鱼类的食物组成成分中不可缺少的部分。目前国内外对多鳞鱚鱼类的研究主要在种类区别、形态学、耳石、生长参数、养殖和仔稚鱼发育等方面,有关其分子遗传学研究相对较少。线粒体COI基因由于其较高的变异速率和易于扩增的特点,被广泛应用于物种鉴定和群体遗传学研究。通过对多鳞鱚线粒体COI基因序列的分析,我们发现该基因在物种鉴定和系统进化关系研究中具有很好的应用潜力。此外,本研究还揭示了多鳞鱚遗传多样性水平较低,环境适应能力较弱,进一步强调了保护措施的紧迫性。本文将详细介绍研究方法、结果和讨论,以期为相关领域的研究提供参考。
1 本研究的目的及意义
多鳞鱚外形特征与鱚属其他鱼类非常相似,特别是与少鳞鱚、中国鱚,主要区别在于三者的侧线上鳞的数量,然而鱚属鱼类的鳞片极易脱落,尤其是在鱼体不新鲜的情况下,单靠形态学的鉴定往往导致多鳞鱚的错误鉴定。因此,有必要对不同地理来源的多鳞鱚样品进行DNA条形码分析,从分子水平来鉴定和区分多鳞鱚,并估算该物种的遗传多样性水平。本研究通过检测采集于南海北部13个不同地点的多鳞鱚线粒体COI基因序列,探究COI条形码在多鳞鱚物种中鉴定能力和评估该物种的遗传多样性,以了解多鳞鱚的系统进化和群体遗传多样性。
2 材料与方法
2.1 试验材料
本实验所用的77尾多鳞鱚样品采集于海南东方渔港、海南文昌和雷州流沙湾等13个南海北部地区(表1)。参考《中国鱼类检索系统》《台湾海峡常见鱼类图谱》《北部湾鱼类图鉴》,对采集的样本进行种类鉴定,剪取适量的鱼体样本的背部肌肉于95%乙醇中的2 mL离心管中,放入冰箱急冻层冷冻保存。

表1 本试验中的多鳞鱚样品
2.2 试验仪器
本研究所用实验仪器:冷冻高速离心机、电热恒温水槽、基因扩增仪、电子天平、可见光凝胶透射仪、电泳仪。
2.3 试验试剂
实验试剂:苯酚、酚-氯仿、灭菌超纯水(DDW)、DNA提取液、蛋白酶K(PK)、异戊醇、氯仿、无水冰乙醇、70%乙醇、EDTA、Tris—base、6×Loading Buffer、10×Taq Buffer(2+Mg)、琼脂糖、SDS、Marker、硼酸、正向引物F1/F2、反向引物F2/R2、10 mM dNTP、EasyTaq DNA polymerase。
2.4 鱼类肌肉DNA的提取
本实验提取鱼肉DNA采用酚/氯仿抽提法,提取DNA加入80 μL的DDW,放置4℃冰箱存放24 h后可使用。
2.5 PCR扩增
2.5.1 扩增线粒体DNA的细胞色素C氧化酶I序列
使用通用引物F1/R1(F2/R2)来扩增多鳞鱚的COI基因片段序列,引物为:
F1:5’-TCA ACC AAC CAC AAA GAC ATT GGC AC-3’;R1:5’-TAG ACT TCT GGG TGG CCA AAG AAT CA-3’。F2:5’-TCG ACT AAT CAT AAA GAT ATC GGC AC-3’;R2:5’-ACT TCA GGG TGA CCG AAG AAT CAG AA-3’。
2.5.2 PCR体系
本试验所用的缓冲体系:39.5 μL灭菌超纯水(DDW)、5 μL 10×Taq Buffer、1 μL10 mol dNTP、1 μL 正向引物(F1/F2)、1 μL反向引物(R1/R2)、0.5 μL Taq DNA Polymerase、2 μL Template DNA。
2.5.3 PCR程序
预变性:94℃ 4 min;循环94℃ 45 s、50℃ 45 s、72℃ 45 s,共35个循环;72℃ 8 min,4℃保存。
2.6 电泳检测与序列分析
2.6.1 电泳检测
本试验主要将5 μL PCR扩增产物或DNA进行1.0%琼脂糖凝胶电泳检测。纯化后的PCR扩增产物记录包装好送到广州生工生物工程股份有限公司进行测序,采用单向测序。
2.6.2 序列分析方法
将得到的多鳞鱚线粒体COI基因序列用SeqManⅡ软件人工校正,剪除COI基因序列前后引物序列。在GenBank中将多鳞鱚COI单倍型基因序列进行BLAST比对与分析;借助MEGA 6.0软件统计多鳞鱚线粒体COI基因序列的单碱基变异位点、保守位点、多态位点、单倍型多样度(Hd)、核苷酸多样性指数(Pi)、转换/颠换比率(R=si/sv)和碱基组成等数据;根据Kimura双参数模型计算出种间和种内的遗传距离;以NJ邻接法(Neighbor joining)构建多鳞鱚单倍型分子系统进化树。
3 结果
3.1 COI基因序列特征
经过PCR扩增与测序后,本试验获得77个多鳞鱚线粒体COI基因片段序列(剪除前后引物序列),序列长度约是593 bp。统计得知,COI基因序列碱基的平均含量分别为T:29.0%、G:19.4%、C:29.2%和A:22.4%,可看出序列反G偏倚,对C和T偏倚。A+T含量(51.4%)明显高于C+G含量(48.6%)。3个密码子的G+C含量差异较大,第2密码子GC含量(56.6%)最大,第1密码子(45.5%)次之,第3密码子(43.6%)最小(表2)。
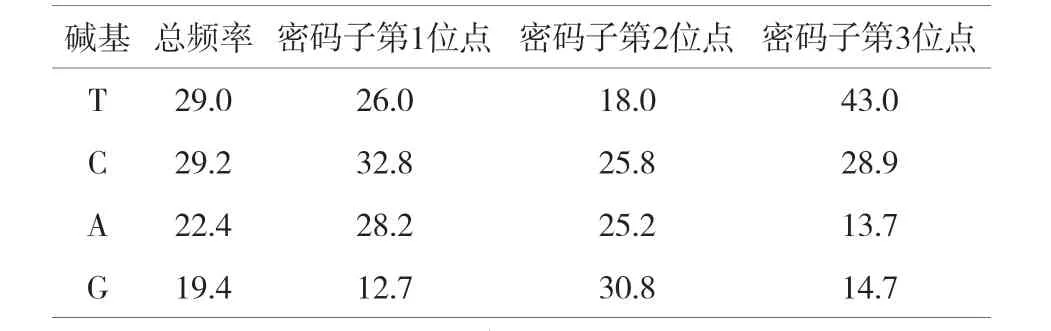
表2 多鳞鱚COI基因序列各碱基平均分布频率(%)
77个多鳞鱚COI基因序列未出现缺失、插入与终止现象。分析得知,保守位点584个,变异位点9个,约占1.5%。在变异位点中,单碱基变异位点有7个,多态信息位点有2个。转换与颠换发生次数比值(R= si/sv)为6.29,3个密码子都没有发生转换和颠换(表3)。
3.2 遗传距离与多样性
依据Kimura双参数模型对鱚属鱼类种间和种内的净遗传距离分析。结果显示,6种鱚属鱼类两两之间的遗传距离为0.1810~0.2493(均值为0.2110),种内遗传距离为0.0000~0.0092(均值为0.0028),种间平均遗传距离为种内的75倍。其中多鳞鱚和斑鱚之间的净遗传距离最小,为0.1810;银鱚与砂鱚之间的净遗传距离最大,为0.2493(表4)。多鳞鱚种内遗传距离为0.0007,种内遗传基因变异程度较低。

表4 6种鱚属鱼类的线粒体COI基因序列的种间遗传距离
对6种鱚属鱼类种内遗传多样性分析,获得个体数(n)、单倍型基因数(Hap)、单倍型基因多样性(Hd)和核苷酸多样性指数(Pi)的信息(表5)。结果显示,多鳞鱚和斑鱚的遗传多样性最低,其核苷酸多样性指数仅0.00067;银鱚、少鳞鱚和中国鱚的核苷酸多样性指数较高,分别为0.0091、0.00616、0.00616。

表5 6种鱚属鱼类的线粒体COⅠ基因序列的种内遗传多样性
3.3 分子系统进化树
基于NJ法所构建的6种鱚属鱼类104个COI基因序列系统树显示(图1),多鳞鱚、少鳞鱚、中国鱚、银鱚、砂鱚、斑鱚等每种不同来源的个体均各自形成一个单系,其置信度均为99%。而6种鱚属鱼类的种间系统进化关系并不明确,未能获得高置信度支持(支持率<70%)。

图1 根据COI基因序列构建的NJ系统数
4 讨论
4.1 多鳞鱚COI基因序列特征
本研究分析77尾多鳞鱚的COI基因序列,获得序列长度约为593bp,4种碱基平均含量分别为T:29.0%、G:19.4%、C:29.2%和A:22.4%;A+T含量(51.4%)明显高于C+G含量(48.6%)。宫亚运等[1]研究中国南海6种绯鲤属鱼类的COI基因,发现其碱基A+T含量(50.6%)稍高于C+G含量(49.4%)。薛泰强等[2]对多鳞鱚、少鳞鱚、斑鱚和银鱚四种鱚属鱼类COI基因片段进行研究,也发现其A+T平均含量(52.9%)高于G+C含量(47.1%)的现象。本研究得出平均碱基含量关系与他们的研究结果类似。
从COI基因片段的碱基C+G含量的贡献来说,第2密码子GC含量(56.6%)最大,第1密码子CG含量(45.5%)次之,第3密码子CG(43.6%)最小,3个密码子CG含量有显著差异。李献儒等[3]在中国近海鲱形目线粒体COI基因研究分析,结果表明第1密码子CG含量(57.5%)最高,第2密码子CG含量(42.7%)次之,第3密码子CG(41.6%)最小。彭居俐等[4]研究4种鲌属鱼类32个COI基因序列得知,第1密码子位点的CG含量(51.6%)最高,第2密码子位点(42.8%)次之,第3密码子位点(42.3%)最小。综合来看,3个密码子CG含量在不同的鱼类中分布有差异,这种差异是否可成为判断鱼类的分类地位的依据还待进一步研究。
4.2 COI条形码在多鳞鱚物种鉴定中的适用性
Hebert等[5]对动物界大量物种研究得知,种内遗传距离大部分要小于0.010,种间遗传距离要比种内遗传距离大10倍以上是COI基因有效鉴定物种条件之一。而本研究对多鳞鱚COI基因序列分析,并与少鳞鱚、中国鱚、银鱚、砂鱚、斑鱚作参照,发现这6种鱚属鱼类两两之间的遗传距离为0.1810~0.2493(均值为0.2110),种内遗传距离为0.0000~0.0092(均值为0.0028),种间平均遗传距离为种内的75倍,种间遗传距离远大于种内遗传距离。这结果符合COI基因有效鉴定物种的条件。
Hebert等[6]经研究又提出了COI基因有效鉴定物种的判断依据,最小种间遗传距离为0.02,种内遗传距离要小于0.02。本研究的77尾多鳞鱚种内遗传距离为0.0007,远小于0.02;多鳞鱚与斑鱚、银鱚、砂鱚、少鳞鱚、中国鱚的种间遗传距离分别为0.1810、0.2136、0.2129、0.2037和0.2156,均大于0.02,与前人研究结论相吻合。在NJ树上,6种鱚属鱼类均可聚成独立分支(置信度99%),不存在交叉情况,与多鳞鱚的形态学判定结果一致,说明线粒体COI基因能有效地对多鳞鱚进行物种鉴定。
4.3 多鳞鱚的遗传多样性水平
研究来自13个地区的77尾多鳞鱚的线粒体COI基因序列,统计得到其保守位点584个,变异位点9个(单碱基变异位点7个和多态信息位点2个),说明不同地理来源的多鳞鱚COI基因序列变异较小,也可能是供研究的各个地区鱼体数量较少造成的。多鳞鱚77个COI基因序列的遗传多样性分析表明,77个个体的COI单倍型基因有10个,单倍型多样性指数为0.329(<0.5),核苷酸多样性指数为0.00067(<0.005),表现出低单倍型多样性和低核苷酸多样性的模式,总体遗传多样性水平较低。本研究中来自南海北部多个地理群体的多鳞鱚遗传多样性较低,提示该物种的南海北部种群的进化潜力较小,这可能与近二十年来南海北部持续的过度捕捞导致种群资源量急剧下降,种群低龄化、小型化等种群衰退有关。本研究表明,南海北部的多鳞鱚遗传多样性较低,其种群的种质资源状况不容乐观,需要引起相当的重视和采取必要的保护措施。
