谷子SWI3基因鉴定及其在盐和干旱胁迫下的表达分析
2024-03-28祁东梅史慎奎王玉芳赵红桃崔素娟王春芳
祁东梅 史慎奎 王玉芳 赵红桃 崔素娟 王春芳


摘 要 SWI/SNF染色质重塑因子在植物的生长发育及逆境应答过程中起重要作用。本研究首先通过序列比对,从谷子基因组中鉴定出6个候选SiSWI3基因,分别命名为 SiSWI3A、 SiSWI3B、 SiSWI3C1、 SiSWI3C2、 SiSWI3D1和 SiSWI3D2。然后对上述基因的结构、编码蛋白、启动子元件、亚细胞定位等进行生物信息学分析和预测,结果表明:SiSWI3蛋白都含有SANT Motif,且亚细胞定位预测显示SiSWI3成员主要被定位在细胞核中; SiSWI3基因启动子区域含有大量与光响应、激素类应答、逆境应答、代谢调控等相关的顺式作用元件。最后采用实时荧光定量PCR(qRT-PCR)检测这些基因在谷子苗期盐胁迫和干旱胁迫下的表达量变化,检测结果表明:SiSWI3基因受盐和干旱不同程度的诱导,说明SiSWI3基因参与谷子苗期盐胁迫和干旱胁迫应答。本研究所得结论为进一步探究SiSWI3染色质重塑因子在抗逆作物谷子的逆境响应中的功能和机制奠定了基础。
关键词 谷子;染色质重塑;SiSWI3基因;盐胁迫;干旱胁迫
谷子(Setaria italica L.)是起源于中国的重要粮食作物,具有生育周期短、植株矮小、单穗结实率高、基因组小等特点,被认为是禾本科黍亚科C4模式植物,适合实验室内功能基因的研究[1]。谷子抗盐抗干旱能力较强,能够在边际土地上种植,同时籽粒营养丰富,富含多种氨基酸,长期以来,谷子作为北方重要的制米作物,是养育华夏民族的哺育作物[2-4]。
染色质重塑是指在基因组DNA序列不改变的前提下,基因表达发生可遗传的现象[5]。根据传统的分类方法,将染色质重塑复体划分为SWI/SNF(SWItching/Sucrose non-Fermenting)、ISWI(imitation SWI)、CHD(Chromotin organization modifier, Helicase, and DNA-binding Domains)和INO80(INOsitol requiring 80)4種类型[6-7]。SWI/SNF家族染色质重塑复体由核心酶、SWI3亚基及其他附属亚基组成[8-9]。SWI3染色质重塑组分在植物生长发育过程中起重要作用:SWI3A和SWI3B参与拟南芥叶片和胚胎的发育[8,10];SWI3B在ABA信号通路中作为正调节子,抑制种子的萌发和生长[11];SWI3C和SWI3D参与叶片和花器官的形态建成及雌蕊发育[8,12];SWI3D同源蛋白ZmCHB101参与玉米叶片和穗轴发育[13]。目前,染色质重塑组分SWI3成员在谷子生长发育和逆境胁迫响应中的具体功能尚不清楚。
近年来,关于谷子抗逆的研究相继被报道。在盐胁迫条件下,转录因子 SiANT1的表达受高盐胁迫诱导[14];在拟南芥中,异源表达 SibZIP42和 SiREM6也表现出抗盐表型[15-16];在谷子中,转录因子 SiNF-YA1和 SiNF-YB8均与抗盐和抗旱相关[17]。在干旱胁迫条件下,谷子自噬基因 SiATG8a和LIM基因家族成员 SiWLIM2b异源超表达使得干旱处理条件下的转基因植株存活率高于野生型[18-19],水稻中超表达谷子转录因子 SiMYB56,转基因植株抗旱能力明显增强[20]。此外,利用遗传转化验证基因功能的相关研究也陆续被报道。Argonaute(AGO1)属于AGO蛋白家族,通过招募小RNAs调节植物生长发育,其在谷子中的缺失突变体 siago1b表现为叶片变窄变卷、花序变小、种子结实率降低且突变体经干旱处理后,比野生型更容易萎蔫,这表明 SiAGO1b在抗旱中发挥功能[21]。在谷子抗旱机制上阐述比较清楚的是ABA依赖的信号通路,其中ABA响应元件结合型转录因子SiAREB可以结合到SiARDP(ABA-responsive DRE-binding protein)基因启动子区域,SiARDP超表达提高谷子的抗旱性[22];SiARDP又可以结合到SiASR4 (ASR:abscisic acid-,stress-,and ripening-induced)和脂质转移蛋白SiLTP基因启动子区域,这两个靶基因超表达后谷子抗旱能力均提高[23-24]。目前,谷子基因组测序已经完成[25-26],而谷子中ATP依赖的染色质重塑复合体成员在盐和干旱胁迫下的响应变化情况还没有详细的阐述。因此,探究SWI3染色质重塑组分的分子机制及其在谷子对盐和干旱逆境胁迫响应中的作用,不仅具有重要理论意义,还对农业的生产实践有重要指导意义。
本研究首先利用生物信息学分析方法,从谷子基因组中鉴定出与拟南芥同源的染色质重塑组分SWI3家族成员,明确该家族基因的结构特征,然后通过实时荧光定量PCR技术分析该家族成员在盐和干旱胁迫下的表达变化情况。本研究结果为探究染色质重塑因子SWI3家族在谷子盐和干旱应答过程中的机制奠定基础,并为谷子种质资源的改良提供参考。
1 材料与方法
1.1 试验材料
选用‘豫谷1号为试验材料,种子由河北民族师范学院生物与食品科学学院分子实验室提供。在玻璃平皿中放一张滤纸,浸湿,把‘豫谷1号种子洒在滤纸上,然后将玻璃平皿放在光培养箱内进行种子萌发,光照周期16 h光照/8 h黑暗,萌发温度(25±2) ℃,湿度60%。
1.2 试验设计
待谷子根的长度达到1~2 cm后移到水培盒中,培养箱光周期设置16 h光照/8 h黑暗,温度25 ℃±2 ℃,湿度60%,用Hogland培养液继续培养至三叶一心期。分别采用150 mmol/L、200 mmol/L、250 mmol/L NaCl溶液进行盐胁迫处理,10%、15%、20% PEG-6000进行模拟干旱处理,在处理的0、2、6和12 h节点取样,设置3次生物学重复。取样后立即将全苗置于液氮中,冷却后置于-80 ℃保存,准备提取RNA。
1.3 试验试剂
试验所用RNA提取试剂盒RNA-easy Isolation Reagent、去基因组反转录试剂盒Hiscript ⅢRT SuperMix for qPCR (+gDNA wiper)和Real-time PCR所用到的ChamQ Universal SYBR qPCR Master Mix均购自南京诺唯赞生物科技有限公司,引物由上海生工生物有限公司合成,其他常规试剂购自生工生物有限公司。
1.4 基因的鉴定及生物信息学分析
通过将本研究获得谷子基因序列与已知的拟南芥和水稻SWI3家族成员基因序列在Phytozome(https://phytozome-next.jgi.doe.gov/)网站上进行BlastP比对,获得具有完整阅读框的谷子同源序列,从中得到谷子中SWI3家族成员信息,并在Phytozome网站获得各基因检索号、基因组序列长度、CDS长度、内含子个数和氨基酸数目等信息。
1.5 RNA提取
RNA提取按照试剂盒说明书进行操作。所用引物由OMIGA 2.0设计,引物序列信息见表1。
1.6 反转录和Real-time PCR分析
反转录过程按试剂盒说明书进行,每个样品先去除基因组DNA,反应体系16 μL:4×g DNA wiper Mix 4 μL,模板RNA 500 ng,RNase-Free H2O补齐至16 μL,42 ℃ 反应2 min后,再加入5×HiScript III qRT SuperMix 4 μL组成反转录体系的20 μL,反应条件37 ℃,15 min,85 ℃ 5 s。反转录合成的cDNA均一化后作为Real-time PCR的模板。以谷子GADPH为内参基因。定量反应体系为10 μL 2×SYBR qPCR Master Mix,0.4 μL上游引物(10 μmol/L),0.4 μL下游引物(10 μmol/L),cDNA模板2 μL,无菌水7.2 μL。反应程序:95 ℃ 30 s;95 ℃ 10 s,60 ℃ 30 s,40个循环。每个处理3个重复,采用2-△△Ct计算目的基因的相对表达量,采用Prism 5.0进行数据的统计分析。
2 结果与分析
2.1SiSWI3基因基本信息及结构分析
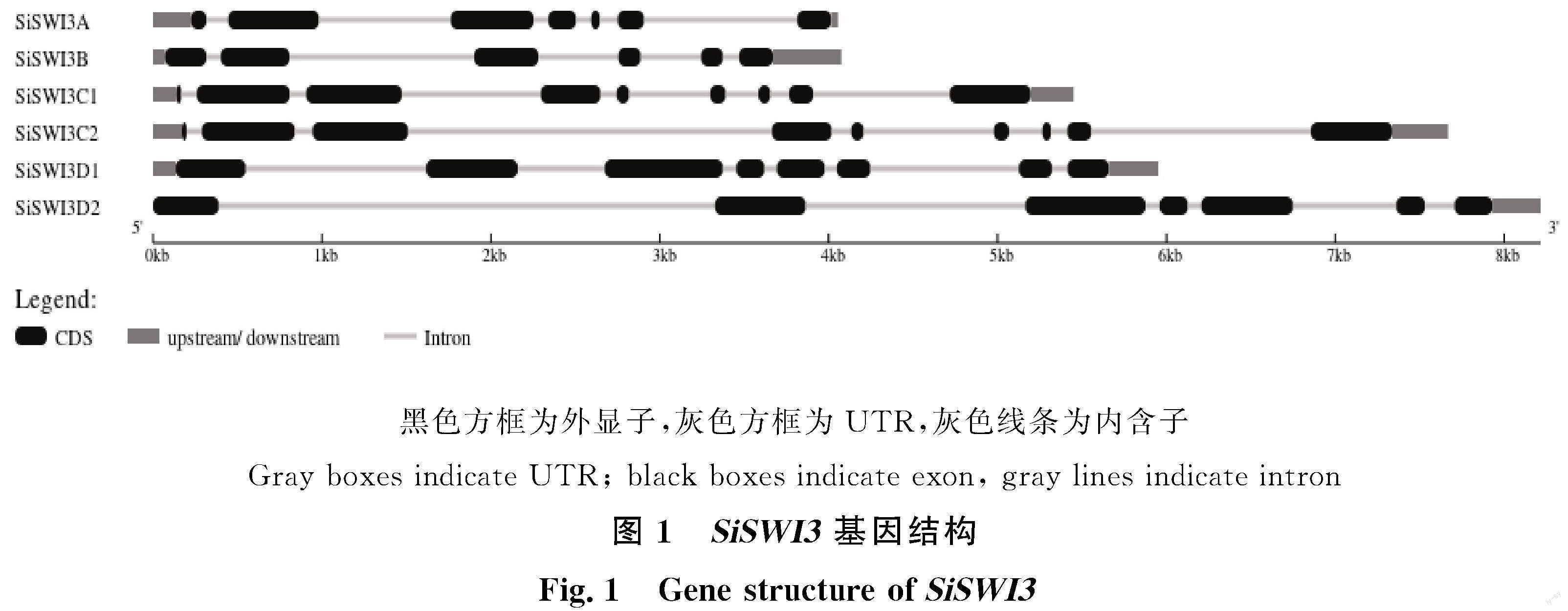
通过序列分析和比对,在谷子中鉴定得到SWI3染色重塑组分,包括6个成员,分别命名为 SiSWI3A、 SiSWI3B、 SiSWI3C1、 SiSWI3C2、 SiSWI3D1和 SiSWI3D2。各成员的理化性质及基因的基本信息如表2所示,基因长度为4 056~8 241 bp,CDS长度为1 494~2 748 bp,氨基酸数目497~915,成员的分子质量为54 169.96~ 99 805.16 u,蛋白质等电点为4.94~ 6.50。此外, SiSWI3A、 SiSWI3B、 SiSWI3C1、 SiSWI3C2、 SiSWI3D1、 SiSWI3D2分别含有6、5、8、8、7、6个内含子(图1)。
2.2 SiSWI3蛋白结构和亚细胞定位分析
使用SMART(http://smart.embl-heidelberg.de/smart/set_mode.cgi?NORMAL=1)在线分析工具对谷子SiSWI3家族成员蛋白结构进行预测,结果表明,6个成员都存在比较保守的SANT结构域,该结构为DNA结合域。此外, SiSWI3C1、 SiSWI3D1、SiSWI3D2还存在ZnF_ZZ结构域(图2)。利用Softberry(http://www.softberry.com/)网页在线工具预测SiSWI3家族成员蛋白亚细胞定位发现,谷子SWI3在细胞核中的定位结果积分值远高于在其他组织中定位积分值,在细胞质、内质网膜、高尔基体积分值为零,因此谷子SWI3蛋白可能主要在细胞核中表达(表3)。
2.3SiSWI3基因系统进化树分析
为进一步了解谷子SWI3基因家族谱系及其功能特点、进化过程,对高粱、水稻、玉米、拟南芥、谷子SWI3基因共同构建系统进化树(图3)。根据构建的系统进化树将其分为3大类,谷子SWI3蛋白不均匀地分布在3大类当中。其中谷子SiSWI3C1与SiSWI3C2亲缘关系最近,与其他4个物种的SWI3聚为一大类,并且在这一大类中谷子SiSWI3A与玉米LOC100285161的亲缘关系最近;SiSWI3D2与SiSWI3D1同属一大类,且谷子SiSWI3D2与水稻LOCOs04g01970的亲缘关系最近;谷子SiSWI3B与高粱LOC8082888和玉米LOC100273414聚合在一起,说明它们亲缘关系较近(同属禾本科C4植物),另一方面也暗示它们的功能具有某些相似性。
2.4 SiSWI3基因启动子调控元件功能分析
利用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在線分析软件对上述SWI3染色质重塑成员的基因启动子进行顺式作用元件预测,每个基因启动子选取长度定为2.0 kb。结果如表4所示,各基因的启动子区存在多个顺式作用原件,包括大量的光响应元件、激素类响应元件(脱落酸、赤霉素、水杨酸、茉莉酸甲酯、生长素)、逆境应答元件(干旱、低温、逆境防御)、对基因转录起始起重要作用的CAAT box和TATA-box以及其他调控生长的相关顺式作用元件,包括分生组织特异性表达、胚乳特异性调控、生物钟调控、玉米蛋白代谢调控、缺氧和厌氧诱导元件等。
2.5SiSWI3基因在苗期盐胁迫下表达分析
为探究SiSWI3基因成员是否响应盐胁迫,对‘豫谷1号的幼苗进行盐胁迫处理,然后通过实时荧光定量PCR对SiSWI3基因的表达量变化情况进行分析,结果如图4所示。在150 mmol/L NaCl胁迫条件下, SiSWI3A在处理 12 h后表达量明显增加, SiSWI3C1、 SiSWI3D1在处理6 h后表达量明显增加, SiSWI3B、 SiSWI3C2表达量没有明显变化。在200 mmol/L NaCl处理胁迫条件下,基因表达出现受抑制的情况,主要表现为: SiSWI3A在处理2 h后表达量明显降低,在12 h后又增加; SiSWI3B在处理2 h和6 h后表达量都降低,在12 h后又明显增加; SiSWI3C1、 SiSWI3C2在处理2 h和 6 h后表达量都明显降低; SiSWI3D1表达量没有明显变化。在250 mmol/L NaCl处理胁迫条件下, SiSWI3A、 SiSWI3C1、 SiSWI3C2、表达量没有明显变化, SiSWI3B在处理6 h后表达量明显增加, SiSWI3D1在处理2 h和12 h表达量明显增加。上述结果表明,SiSWI3家族基因的表达受盐胁迫的诱导,该家族成员参与了盐胁迫的响应。
2.6 SiSWI3基因在苗期干旱胁迫下表达分析
为探究SiSWI3基因成员是否响应干旱胁迫,对‘豫谷1号的幼苗进行模拟干旱处理后通过实时荧光定量PCR对SiSWI3基因的表达情况进行分析,结果如图5所示。由图5可知,在10% PEG-6000处理条件下, SiSWI3A和 SiSWI3B[QX)〗经胁迫处理6 h后基因表达量升高,但 SiSWI3C1和 SiSWI3C2表达量降低。在15% PEG-6000处理条件下, SiSWI3A、 SiSWI3B与 SiSWI3C2表达量没有明显变化, SiSWI3C1的表达量随着时间增加呈现先增加后降低的趋势,但差异并不显著; SiSWI3D1的表达量在处理后2 h升高、6 h降低,表明基因受干旱诱导后其表达又受到抑制。在20% PEG-6000处理条件下, SiSWI3A在干旱处理6 h后基因表达量升高, SiSWI3B与 SiSWI3C1表达量没有明显变化, SiSWI3C2的表达量有上升的趋势,但是差异并不显著, SiSWI3D1的表达量在干旱处理2 h后降低。上述结果表明,SiSWI3家族成员在不同程度上受到干旱胁迫的诱导,暗示其参与了谷子苗期干旱胁迫响应并在谷子苗期干旱响应通路中发挥重要 作用。
3 结论与讨论
本研究通过比对分析在谷子基因组中得到6个SWI3基因,并对6个基因生物信息学特征进行了系统的预测和分析。预测结果显示,SiSWI3家族成员的亚细胞定位主要位于细胞核,因为该家族属于染色质重塑组分,其功能的发挥离不开染色质,而染色质作为遗传物质的主要载体就存在于细胞核中,因此SiSWI3成员的性状和参数非常接近。系统进化树表明,谷子成员与玉米、水稻、高粱亲缘关系较近,与拟南芥相对较远。顺式元件分析表明,在SiSWI3基因启动子区域含有很多顺式作用元件,包括光周期响应元件、激素响应元件、生长代谢调控元件、逆境胁迫响应元件等,这些元件的存在暗示着这些基因很可能参与到相应的信号通路调控过程中[27],例如ABA在非生物胁迫(干旱、盐、高温、低温等)响应中发挥重要作用,在逆境胁迫条件下,植物体内ABA水平升高[28-30],因此推测SiSWI3基因很可能参与植物对非生物逆境胁迫的响应过程。
Real-time PCR检测结果表明,5个SiSWI3基因在两种处理下呈现不同程度的差异性表达,这暗示着谷子SiSWI3基因参与了植物苗期盐胁迫和干旱胁迫的响应过程。结果显示,SiSWI3家族基因在每个盐浓度中均有基因呈现差异性表达变化,在150 mmol/L盐胁迫下, SiSWI3A、 SiSWI3C1、 SiSWI3D1均表现为受盐诱导表达量在不同时间点上调;在200 mmol/L盐胁迫下, SiSWI3A、 SiSWI3B、 SiSWI3C1、 SiSWI3C2的表达量呈现先降低又增加的趋势;在250 mmol/L盐胁迫下, SiSWI3B和 SiSWI3D1表现为受盐诱导表达量增加。可见随着盐浓度增加,基因的响应程度发生变化,响应的基因在各梯度也有所不同,推测原因一是各基因家族成员的表达模式可能不尽相同;二是基因的表达调控非常复杂,植物体内外两方面因素都能对基因表达产生影响,使不同浓度下基因调控通路有差异而导致各基因的响应模式不同。
与盐胁迫下SiSWI3基因的表达模式相比,干旱胁迫下该家族基因的表达情况相对简单。在 SiSWI3A、 SiSWI3B干旱胁迫诱导试验中,10% PEG-6000的浓度下处理6 h,二者表达量均增加,并且 SiSWI3A在20% PEG-6000处理6 h时表达量也增加,并呈现显著性差异; SiSWI3C1、 SiSWI3C2在10% PEG-6000的浓度下处理12 h后表达量下降; SiSWI3D1在10% PEG-6000处理时表达量没有明显变化,在15%和20% PEG-6000处理时分别呈现出先增加后降低以及先降低后增加的趋势。这5个基因在干旱胁迫下的3种表达情况说明SiSWI3家族基因的表达模式不相同。此外, SiSWI3D1在不同浓度PEG-6000下的表达量变化可能是由于外界条件诱导下内源激素或者信号通路调节机制不同。
综上所述,研究初步表明谷子SiSWI3基因受盐和干旱胁迫的诱导,可能参与谷子对逆境胁迫的应答过程,虽然这些结论与分析还需要进一步证实,但为探究染色質重塑因子SiSWI3成员在非生物逆境下的表达特性提供了线索,为进一步阐明 SiSWI3基因在谷子逆境应答中的功能和机制提供了试验依据。
参考文献 Reference:
[1] DEVOS K M,WANG Z M,BEALES J,et al. Comparative genetic maps of foxtail millet (Setaria italica) and rice (Oryza sativa)[J]. Theoretical and Applied Genetics,1998,96(1):63-68.
[2] JAYARAMAN A,PURANIK S,RAI N K,et al. cDNA-AFLP analysis reveals differential gene expression in response to salt stress in foxtail millet (Setaria italica L.)[J]. Molecular Biotechnology,2008,40(3):241-251.
[3] 智 慧,牛振刚,贾冠清,等. 谷子干草饲用品质性状变异及相关性分析[J]. 作物学报,2012,38(5):800-807.
ZHI H,NIU ZH G,JIA G Q,et al. Variation and correlation analysis of hay forage quality traits of foxtail millet [Setaria italica (L.) Beauv.][J]. Acta Agronomica Sinica,2012,38(5):800-807.
[4] DEVOS K M,WANG Z M,BEALES J,et al. Comparative genetic maps of foxtail millet (Setaria italica) and rice (Oryza sativa)[J]. Theoretical and Applied Genetics,1998,96(1):63-68.
[5] BIRD A. Perceptions of epigenetics[J]. Nature,2007,447(7143):396-398.
[6] 郭明欣,何亚莉,许可可,等.植物染色质重塑复合体研究进展[J].安徽农业科学,2019,47(13):16-19.
GUO M X,HE Y L,XU K K,et al. Research advance on chromatin remodeling complex in plants[J]. Journal of Anhui Agricultural Sciences,2019,47(13):16-19.
[7] EISEN J A,SWEDER K S,HANAWALT P C. Evolution of the SNF2 family of proteins:subfamilies with distinct sequences and functions[J]. Nucleic Acids Research,1995(14):2715-2723.
[8] SARNOWSKI T J,RIOS G,JASIK J,et al. SWI3 subunits of putative SWI/SNF chromatin-remodeling complexes play distinct roles during Arabidopsis development[J]. Plant Cell,2005,17(9):2454-2472.
[9] KWON C S,WAGNER D. Unwinding chromatin for development and growth:a few genes at a time[J]. Trends in Genetics,2007,23(8):403-412.
[10] [ZK(#]LIN X W,YUAN C,ZHU B N,et al. LFR physically and genetically interacts with SWI/SNF component SWI3B to regulate leaf blade development in Arabidopsis[J]. Frontiers in Plant Science,2021,12:717649.
[11] SAEZ A,RODRIGUES A,SANTIAGO J,et al. HAB1-SWI3B interaction reveals a link between abscisic acid signaling and putative SWI/SNF chromatin-remodeling complexes in Arabidopsis[J]. Plant Cell,2008,20(11):2972-2988.
[12] HAN S K,SANG Y,RODRIGUES A,et al. The SWI2/SNF2 chromatin remodeling ATPase BRAHMA represses abscisic acid responses in the absence of the stress stimulus in Arabidopsis[J]. Plant Cell,2012,24(12):4892-4906.
[13] YU X M,JIANG L L,WU R,et al. The core subunit of a chromatin-remodeling complex,CHB101,plays essential roles in maize growth and development[J]. Scientific Reports,2016,6(1):38504.
[14] 寧 蕾,王曙光,琚鹏举,等. 过表达谷子SiANT1对水稻耐盐性的影响[J]. 中国农业科学,2018,51(10):1830-1841.
NING L,WANG SH G,JU P J, et al. Rice overexpression of millet SiANT1 gene increases salt tolerance[J].Scientia Agricultura Sinica, 2018,51(10):1830-1841.
[15] 秦玉海,张小红,冯 露,等. 谷子转录因子基因SibZIP42在拟南芥中对高盐和ABA的响应[J]. 中国农业科学,2016,49(17):3276-3286.
QIN Y H,ZHNAG X H,FENG L,et al. Response of millet transcription factor gene SibZIP42 to high salt and ABA treatment in transgenic Arabidopsis[J]. Scientia Agricultura Sinica,2016,49(17):3276-3286.
[16] YUE J ,CONG L ,LIU Y ,et al. A remorin gene SiREM6,the target gene of SiARDP,from foxtail millet (Setaria italica) promotes high salt tolerance in transgenic Arabidopsis[J]. Plos One,2014,9(6):e100772.
[17] FENG Z J,HE G H,ZHENG W J,et al. Foxtail millet NF-Y families:genome-wide survey and evolution analyses identified two functional genes important in abiotic stresses [J]. Frontiers in Plant Science,2015,6:1142.
[18] LI W W,CHEN M,ZHONG L,et al. Overexpression of the autophagy-related gene SiATG8a from foxtail millet (Setaria italica L.) confers tolerance to both nitrogen starvation and drought stress in Arabidopsis[J]. Biochemical and Biophysical Research Communications,2015,468(4):800-806.
[19] YANG R,CHEN M,SUN J C,et al. Genome-wide analysis of LIM family genes in foxtail millet (Setaria italica L.) and characterization of the role of SiWLIM2b in drought tolerance[J]. International Journal of Molecular Sciences,2019,20(6):1303.
[20] XU W Y,TANG W S,WANG C X,et al. SiMYB56 confers drought stress tolerance in transgenic rice by regulating lignin biosynthesis and ABA signaling pathway[J]. Frontiers in Plant Science,2020,11:785.
[21] LIU X T,TANG S,JIA G Q,et al. The C-terminal motif of SiAGO1b is required for the regulation of growth,development and stress responses in foxtail millet [Setaria italica (L.) P. Beauv)][J]. Journal of Experimental Botany,2016,67(11):3237-3249.
[22] LI C,YUE J,WU X W,et al. An ABA-responsive DRE-binding protein gene from Setaria italica, SiARDP,the target gene of SiAREB,plays a critical role under drought stress[J]. Journal of Experimental Botany,2014,65(18):5415-5427.
[23] LI J R,DONG Y,LI C,et al. SiASR4,the target gene of SiARDP from Setaria italica,improves abiotic stress adaption in plants[J]. Frontiers in Plant Science,2016,7:2053.
[24] PAN Y L,LI J R,JIAO L C,et al. A non-specific Setaria italica lipid transfer protein gene plays a critical role under abiotic stress[J]. Frontiers in Plant Science,2016,7:1752.
[25] ZHANG G Y,LIU X,QUAN Z W,et al. Genome sequence of foxtail millet (Setaria italicas) provides insights into grass evolution and biofuel poten-tial[J]. Nature Biotechnology,2012,30(6):549-554.
[26] BENNETZEN J L,SCHMUTZ J,WANG H,et al. Reference genome sequence of the model plant setaria[J]. Nature Biotechnology,2012,30(6):555-561.
[27] NARUSAKA Y,NAKASHIMA K,SHINWARI Z K, et al. Interaction between two cis-acting elements,ABRE and DRE,in ABA-dependent expression of Arabidopsis rd29A gene in response to dehydration and high-salinity stresses[J]. Plant Journal,2003,34(2):137-148.
[28] NAKASHIMA K,YAMAGUCHI-SHINOZAKI K. ABA signaling in stress-response and seed development[J]. Plant Cell Reports,2013,32(7):959-970.
[29] VERMA V,RAVINDRAN P,KUMAR P P. Plant hormone-mediated regulation of stress responses[J]. Bmc Plant Biology,2016,16(1):86.
[30] ZHANG J,JIA W,YANG J,et al. Role of ABA in integrating plant responses to drought and salt stresses[J]. Field Crops Research,2006,97(1):111-119.
Identification of SWI3 Genes from Foxtail Millet (Setaria italica L.) and Its Expression Analysis under Salt and Drought Stresses
Abstract SWI/SNF chromatin remodeling factors play important rolesin plant development and stress response. In this study,six candidate SWI3 genes were identified,namely SiSWI3A,SiSWI3B,SiSWI3C1,SiSWI3C2,SiSWI3D1,SiSWI3D2 from foxtail millet (Setaria italica L.) genome through sequence alignment.Bioinformatics methods were used to analyze the gene structure,protein,promoter sequence,and subcellular localization.The result showed that all six genes contained the conserved characteristic domains of SANT Motif,besides subcellular localization predictions showed that SiSWI3 members were mainly localized in nucleus. cis-elements analysis revealed that light,hormonal,stress,metabolism regulation and other related cis-elements were presented in the promoter region of the SiSWI3 genes. Real-time quantitative PCR (qRT-PCR) was used to detect the expression levels of SiSWI3 genes under different stresses at seedling stage,including salt and drought stress at last. Stress expression analysis showed that the expression levels of SiSWI3 genes were induced under the salt and drought stresses,indicating that five SiSWI3 genes involved in salt and drought stress response at the seedling stage. This study lays a foundation for further research on the function and mechanisms of SiSWI3 chromatin remodeling factors in the stress response of millet.
Key words Foxtail millet; Chromatin remodeling; SiSWI3 gene; Salt stress; Drought stress
